2.2) Production, purification, et obtention des
protéines d'intérêt
Le détail des matériels et méthodes
relatifs à la production à grande échelle, à
la purification, et à l'obtention finale des 4 domaines PRODH figure en
fin de ce chapitre dans
le paragraphe 3.
2.2.1) Obtention du domaine PROcatal
La transposition des meilleures conditions d'expression de
PROcatal à grande échelle
a été entreprise en fernbach contenant 1
litre de culture. Les souches Rosetta ont été
transformées par le vecteur d'expression encodant
la protéine PROcatal fusionnée au partenaire MBP, puis
mises en préculture afin d'ensemencer la culture de 1 litre.
L'expression
de la protéine hétérologue a
été induite à une DO600 de 1.2 par ajout de 1 mM d'IPTG,
puis
maintenue pendant 14 heures à 20°C sous agitation.
Les culots bactériens ont été lysés dans
un tampon Tris-HCl à pH 8.0, et les fractions contenant
les protéines solubles et insolubles
ont été séparées par
centrifugation.
Comme le montre le Tableau 4.2, les résultats de la
transposition à grande échelle en fernbach sont tout à
fait satisfaisants : le taux de protéine hétérologue
PROcatal obtenu dans
la fraction soluble est similaire, voire légèrement
supérieur, à celui obtenu en microplaques.
|
PROcatal
|
Microplaque (250 uL)
|
Fernbach (1 L)
|
|
MBP/Rosetta/20°C
|
S I
++(+) ++
|
S I
+++ +++
|
Tableau 4.2 : Résultats
de la transposition à grande échelle des meilleures
conditions
d'expression PROcatal. Après dépôt
des fractions soluble (S) et insoluble (I) sur gel SDS- PAGE, chaque
bande de surexpression est analysée, et une valeur
semi-quantitative est associée à son intensité, de faible
(+) à très forte (++++). Les fractions se confondant aux
protéines endogènes de E. coli et constituant un doute, sont
associées au sigle +/-. La légende
de ce tableau s'applique également pour les
résultats de la transposition des autres protéines
PRODH (cf. Tableaux 4.3, 4.4, et 4.5).
Sur la base de cette transposition très favorable, la
purification de la protéine de fusion PROcatal a pu être
entreprise. Celle-ci a été menée par chromatographie
d'affinité sur résine d'amylose Amylose Resin (Biolabs)
spécifique au partenaire MBP. La moitié du volume de surnageant
de cassage, provenant de 500 mL de culture, a été
chargée dans la résine d'amylose. Après rinçage,
l'élution a été menée en introduisant un tampon
contenant 10 mM
de maltose. L'ensemble des fractions recueillies ont
été déposées sur gel
d'électrophorèse
SDS-PAGE, et les fractions d'élution ont été
quantifiées par mesure de l'absorbance à 280 nm sur un
spectromètre UV.
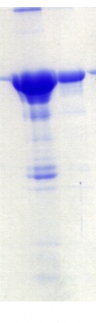
L'analyse SDS-PAGE de la fraction principale
d'élution fait apparaître une bande majoritaire dont la
masse apparente correspond à celle de la protéine de
fusion MBP- PROcatal (Figure 4.3A). La quantité de protéine
recombinante présente dans cette fraction est estimée à
environ 29 mg, ce qui correspond à une production de près de 60
mg par litre de culture. Cependant, nous avons remarqué la
présence de précipités troubles dans les
différentes fractions lors de l'élution de la
protéine. L'analyse SDS-PAGE de la fraction principale,
après centrifugation et reprise du culot insoluble par du SDS,
révèle en effet qu'une partie de la protéine
hétérologue s'est agrégée (Figure 4.3B).

(kDa)
150
100
75
S I
B)
MBP
- PROcatal
50
37
25

A)
Figure 4.3 : Purification de la
protéine de fusion MBP-PROcatal (99 kDa) sur
résine
d'amylose. A) Analyse SDS-PAGE de la fraction principale
d'élution. B) Mise en évidence de l'agrégation partielle
de MBP-PROcatal : analyse SDS-PAGE des fractions soluble (S) et
insoluble (I) après centrifugation de la fraction principale
d'élution.
Nous avons mené une étude de stabilité
à 4°C sur plusieurs jours qui montre que la protéine de
fusion MBP-PROcatal présente une propension lente à
l'agrégation. En effet, alors que la fraction principale de
protéine purifiée contient 29 mg de protéine
soluble en sortie de purification, elle n'en contient plus que 23 mg au bout
de 24 heures, et 18 mg au bout de 48 heures. D'autre part, la conservation de
la protéine à -80°C, après congélation dans
l'azote liquide, n'est pas recommandée car elle provoque une
agrégation drastique à la décongélation. La
stabilité de la protéine de fusion a également
été testée sur plusieurs gammes de concentration. La
dilution des échantillons au 1/2, au 1/5, ou au 1/10 avec du
tampon d'équilibration (NaCl 200 mM, Tris-HCl 100 mM, pH
8.0) n'induit aucune modification du profil de stabilité, ce qui
suggère que la propension à l'agrégation n'est pas
dépendante de la concentration. Dans certains cas, ce type de
comportement peut être observé lorsqu'une protéine est
mise en solution avec un tampon non adapté qui provoque son
agrégation lente. Nous avons donc testé la
solubilité de la protéine MBP-PROcatal avec 9 solutions
tampons différentes : phosphate de sodium, hepès, MES,
acetate de sodium, phosphate de potassium, acétate d'ammonium,
citrate, imidazole, et Tris-HCl (étude non
présentée). Aucun de ces tampons ne permet de ralentir
le phénomène d'agrégation de la protéine de
fusion. D'autre part, l'oxydation de cystéines libres favorise la
formation de ponts disulfures intra ou intermoléculaires non natifs
qui peuvent conduire à l'agrégation. Le domaine PROcatal
possédant 8 cystéines, nous avons ajouté
différents réducteurs dans les surnageants de lyse et dans
les fractions d'élution : DTT, â-mercapto-éthanol, et
TCEP.
L'ajout de ces réducteurs n'a aucune influence sur
le profil de stabilité de la protéine de
fusion. Au vu de l'ensemble des résultats de ces tests, il
semble donc que cette propension à
l'agrégation soit intrinsèque à la
protéine hétérologue MBP-PROcatal.
En parallèle de ces études de stabilité,
nous avons entrepris de cliver le partenaire de fusion MBP par coupure
enzymatique avec la protéase TEV. Cette opération a
été réalisée en solution en ajoutant 640 ug de
protéase à un échantillon contenant 9 mg de
protéine de fusion. Après une incubation à 4°C sous
agitation pendant 14 heures, l'échantillon a été
centrifugé et
la fraction insoluble a été reprise avec du SDS.
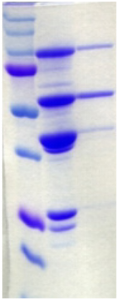
Comme le montre le gel SDS-PAGE de la Figure 4.4, le clivage de la
protéine de fusion par la TEV n'a pas accentué le
phénomène d'agrégation. Trois bandes de forte
intensité dont les masses apparentes correspondent à celles
de MBP-PROcatal, MBP, et PROcatal, apparaissent dans la fraction soluble sur le
gel d'acrylamide. La coupure en solution est donc partielle, et le
rendement peut être estimé à
plus de 60% en comparant les intensités de ces 3 bandes
protéiques.
S I
(kDa)
100
75
50
37
MBP-PROcatal
PROcatal
MBP
25
20
Figure 4.4 : Clivage du
partenaire de fusion de MBP-PROcatal (99 kDa). Analyse de la
solution de clivage par la TEV après centrifugation.
Mise en évidence des bandes protéiques
de MBP-PROcatal, PROcatal (59 kDa), et MBP (43 kDa).
Sur la base de ce bon résultat, nous avons envisagé
de séparer le partenaire de fusion
de la protéine d'intérêt par chromatographie
de pseudo affinité sur résine de nickel HisTrap
HP (Amersham). La solution de clivage, issue de la
réaction de coupure enzymatique par la TEV de 9 mg de protéine de
fusion, a été intégralement chargée sur une colonne
HisTrap HP. Après lavage de la résine avec un
tampon contenant 20 mM d'imidazole, l'élution a été
réalisée avec un gradient linéaire en imidazole de 20 mM
à 300 mM. Les fractions récoltées
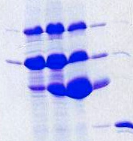
ont été analysées sur gel SDS-PAGE
(Figure 4.5), et la concentration d'imidazole dans les fractions
d'élution a été vérifiée par mesure de
l'absorbance à 300 nm sur un spectromètre
UV.
concentration en imidazole (mM)
20
100
200
chargement lavage élution
SC
MBP- PROcatal
1 2 3 4
5 6 7 8 9
10 11 12
13 14 15

(kDa)
150
100
75

PROcatal
50
MBP
MBP coli 37
TEV
25
Figure 4.5 : Suivi par SDS-PAGE
de la purification de PROcatal sur résine de nickel.
Analyse des fractions de chargement, de lavage,
d'élution, et de la solution de clivage (SC). Les protéines
MBP-PROcatal (99 kDa), MBP clivée (43 kDa), et PROcatal (59
kDa) sont mises en évidence par des flèches rouges, et la
protéase TEV (27 kDa) et la MBP endogène à
E. coli (40 kDa) par des flèches vertes.
L'analyse SDS-PAGE met en évidence une bande
protéique, dont la masse apparente
est très proche de celle du partenaire MBP
clivé, dans les fractions contenant les protéines non retenues
(fractions 1 à 9). Cette bande correspond à la MBP
endogène de E. coli, qui ne possède pas
d'étiquette 6xHis, et qui s'est concentrée sur la
résine d'amylose lors de la première étape de
purification. La faible différence de poids moléculaire
entre les deux formes de MBP est suffisante pour les différencier sur
les gels d'acrylamide (respectivement
43 kDa pour le partenaire clivé, et 40 kDa pour la
protéine endogène).
De manière inattendue, le profil d'élution de
PROcatal, qui ne possède pas d'étiquette
6xHis, est similaire à celui de MBP-PROcatal et MBP
clivée qui en comportent une. Ainsi,
ces trois protéines fixent la résine de nickel
jusqu'à des concentrations d'imidazole de l'ordre
de 100 mM. La chromatographie de pseudo affinité IMAC
est une technique de purification peu spécifique. Ainsi, il est
communément observé que certaines protéines, qui
présentent à leur surface un amas de plusieurs résidus
histidine, sont capables de fixer la résine de nickel
de manière non spécifique à des
concentrations d'imidazole relativement faibles. PROcatal est
un domaine de 486 résidus qui comporte 14
résidus histidine dispersés dans la séquence primaire
; il est donc tout à fait possible qu'il interagisse de manière
non spécifique avec les ions nickel. Cependant, ce type d'interaction ne
suffit pas pour accrocher la résine jusqu'à des
concentrations d'imidazole de l'ordre de 100 mM. Par
conséquent, nous proposons une autre
hypothèse expliquant le profil d'élution de
PROcatal.
En effet, les gels d'acrylamide de la Figure 4.5 montrent que
la présence de la protéine d'intérêt PROcatal dans
les différentes fractions est toujours accompagnée du partenaire
de fusion clivé MBP, ou de la protéine de fusion
résiduelle MBP-PROcatal (fractions 10 à 15). Ceci suggère
que l'élution de PROcatal, à des concentrations d'imidazole de
l'ordre de 100 mM, puisse être due à un effet
d'entraînement de grande envergure, qui s'expliquerait par
l'existence de fortes interactions entre PROcatal et MBP. Ce type
d'interactions de fortes intensités, entre la protéine
d'intérêt et son partenaire de fusion après clivage
par la TEV, suggère un repliement incorrect ou instable du domaine
PROcatal, qui pourrait expliquer la propension à l'agrégation
de la protéine de fusion. De manière intéressante,
ce type de comportement a déjà été
rencontré au LMP avec d'autres protéines dont la
séparation de leur partenaire de fusion, pourtant clivé, n'a
jamais été obtenue.
Dans l'optique de vérifier notre hypothèse,
nous avons envisagé de séparer le partenaire de fusion
MBP de la protéine PROcatal sur résine d'amylose dont
l'affinité est beaucoup plus spécifique. Cependant, lorsque nous
avons préparé cette opération 48 heures après la
purification sur résine de nickel, nous avons constaté que plus
de 80% de la protéine PROcatal s'était agrégée dans
les fractions d'élution.
2.2.2) Obtention du domaine PROinser
Les meilleures conditions d'expression de PROinser obtenues
à l'issu du criblage en microplaques sont identiques à celles
obtenues pour PROcatal (MBP, Rosetta, 20°C). Par conséquent,
nous avons eu recours aux mêmes stratégies et
méthodologies pour produire, purifier, et isoler la protéine
d'intérêt PROinser. Comme le montre le tableau 4.3,
l'expression
de la protéine de fusion MBP-PROinser, en fernbach
contenant 1 L de culture, conduit à un taux de protéine soluble
tout à fait comparable à celui obtenu en microplaques. Sa
purification
est donc envisageable.
|
PROinser
|
Microplaque (250 uL)
|
Fernbach (1 L)
|
|
MBP/Rosetta/20°C
|
S I
++(+) +
|
S I
++ +(+)
|
Tableau 4.3 : Résultats
de la transposition à grande échelle des meilleures
conditions
d'expression PROinser.
La moitié du volume de surnageant de lyse,
provenant de 500 mL de culture, a été
déposé sur résine d'amylose. L'analyse
SDS-PAGE des différentes fractions, après élution
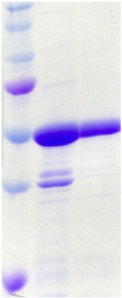
par un tampon maltose, met en évidence une bande
majoritaire dans la fraction principale dont
la masse apparente correspond bien au poids moléculaire de
MBP-PROinser (56 kDa) (Figure
4.6A). Le rendement de l'expression de la protéine de
fusion, obtenu à l'issu de cette première étape de
purification, est d'environ 30 mg de protéine par litre de culture, ce
qui est tout à fait satisfaisant. Cependant, nous avons
également constaté que la protéine MBP-PROinser
présente une tendance à l'agrégation comparable
à celle de MBP-PROcatal. En effet, l'analyse SDS-PAGE de la
fraction principale, après 24 heures d'incubation à
4°C, montre qu'une fraction importante de la protéine s'est
agrégée (Figure 4.6B). Une étude de stabilité,
identique à celle décrite précédemment, a
donc été entreprise dans l'optique d'améliorer la
solubilité de la protéine hétérologue. Les
résultats de cette étude indiquent que MBP- PROinser
présente une propension lente à l'agrégation quels
que soient les paramètres expérimentaux testés
(concentration, solution tampon, réducteur). Ainsi, aucune
condition permettant de ralentir ce phénomène d'agrégation
n'a été déterminée.

(kDa)
150
100
75
50
37
MBP
- PROinser
S I S I

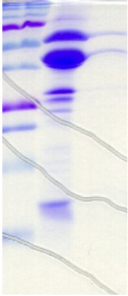
(kDa)
75
50
37
25
20
15
10
MBP-PROinser
MBP
PROinser
25
A) B) C)
Figure 4.6 : Purification et
clivage du partenaire de fusion de MBP-PROinser (56 kDa)
suivis par SDS-PAGE. A) Purification sur résine
d'amylose : analyse de la fraction principale d'élution. B) Mise en
évidence de l'agrégation partielle de MBP-PROinser :
analyse des fractions soluble (S) et insoluble (I) après centrifugation
de la fraction principale 24 heures après l'élution. C) Analyse
de la solution de clivage par la TEV après centrifugation. Mise en
évidence des bandes protéiques de MBP-PROinser, PROinser (13
kDa), et MBP (43 kDa).
Nous avons tout de même entrepris de cliver le
partenaire de fusion par coupure enzymatique avec la TEV. 680 ug de
protéase ont été introduits dans la fraction principale
d'élution de 10 mL contenant environ 8.5 mg de protéine de
fusion. L'analyse SDS-PAGE de
la solution de clivage fait apparaître dans le surnageant
une bande protéique de faible poids
moléculaire à une masse apparente qui
correspond à celle du domaine PROinser (Figure
4.6C). En se basant sur les intensités des bandes
correspondant à MBP-PROinser, MBP, et PROinser, le rendement de coupure
peut être estimé à une valeur proche de 70%. De plus,
l'analyse de la fraction insoluble montre que l'introduction de la
TEV n'a pas accentué le phénomène
d'agrégation.
La quantité de protéine hétérologue
clivée étant suffisante, la séparation du partenaire
de fusion peut être envisagée sur résine
de nickel. La solution de clivage a été intégralement
chargée sur une colonne HisTrap HP. Après
rinçage avec un tampon contenant 20 mM d'imidazole,
l'élution a été menée avec un gradient
linéaire en imidazole de 20 mM à 300 mM. L'analyse des
éluats par SDS-PAGE fait apparaître 2 bandes
protéiques dans les fractions contenant les protéines non
retenues par la résine de nickel (fractions 1 à 6) (Figure
4.7). La première traduit la présence d'une
faible quantité de protéine de fusion MBP-
PROinser, et la seconde correspond à la MBP
endogène de E. coli.
concentration en imidazole (mM)
20
100
200
chargement lavage élution
MBP- PROinser
MBP MBP coli
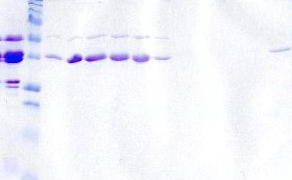
TEV
SC 1 2 3 4
5 6 7
8 9 10 11
12 13 14
15 16 17
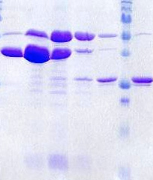

(kDa)
75
50
37
25
20
15
PROinser
10
Figure 4.7 : Suivi par SDS-PAGE
de la purification de PROinser sur résine de nickel.
Analyse des fractions de chargement, de lavage,
d'élution, et de la solution de clivage (SC). Les protéines
MBP-PROinser (56 kDa), MBP clivée (43 kDa), et PROinser (13
kDa) sont mises en évidence par des flèches rouges, et la
protéase TEV (27 kDa) et la MBP endogène à
E. coli (40 kDa) par des flèches vertes.
De manière intéressante, le profil d'élution
du domaine PROinser sur résine de nickel
est tout à fait comparable à celui de
PROcatal. En effet, PROinser, qui ne comporte pas d'étiquette
6xHis, est retenu dans la colonne jusqu'à des concentrations
d'imidazole de
l'ordre de 130 mM ; ce qui est également le cas de la
majeure partie de la protéine de fusion
MBP-PROinser, et de MBP clivée, qui en
possèdent une. Le domaine PROinser ne comportant que 2
résidus histidine dans sa séquence primaire, il est donc
exclu que cette liaison à la résine de nickel soit due à
des interactions non spécifiques. De plus, la présence de
PROinser dans les différentes fractions est toujours accompagnée
du partenaire MBP ou de la protéine de fusion résiduelle
(fractions 13 à 15). A l'instar de PROcatal, il semble donc que le
domaine PROinser interagisse avec son partenaire clivé ou la
protéine de fusion via des liaisons non covalentes de fortes
intensités. L'analyse des profils d'élution sur résine de
nickel suggère donc un repliement incorrect ou instable du
domaine PROinser, qui pourrait expliquer la propension à
l'agrégation de la protéine de fusion, avant et après la
coupure par la protéase TEV.
2.2.3) Obtention de la protéine
PROentier
La production du domaine PROentier a également
été entreprise avec le partenaire de fusion MBP et en souche
Rosetta cultivée à 20°C. Les résultats de la
transposition à grand volume, rassemblés dans le Tableau 4.4,
montrent que le taux de protéine soluble, obtenu en fernbach ou en
erlenmeyer, est très inférieur à celui obtenu en
microplaques. Ainsi, l'expression dans la fraction soluble est à
peine détectable en fernbach contenant 1 L de culture, et elle
est un peu plus prononcée en erlenmeyer de 3L contenant 300 mL de
culture.
|
PROentier
|
Microplaque
(250 uL)
|
Fernbach
(1 L)
|
Erlenmeyer
(300 mL)
|
|
MBP/Rosetta/20°C
|
S
++
|
I
++
|
S
+/-
|
I
++++
|
S
+
|
I
+(+)
|
Tableau 4.4 : Résultats
de la transposition à grande échelle des meilleures
conditions
d'expression PROentier.
Ces quantités de protéine soluble n'étant
pas satisfaisantes, nous avons voulu tester la production à grande
échelle du domaine PROentier en fusion avec le partenaire NusA,
qui conduit au deuxième meilleur taux de protéine soluble
en microplaques. Cependant, un certain nombre de difficultés
inhérentes au vecteur d'expression ont été
rencontrées lors des étapes de transformation et de
précultures, et n'ont pas permis d'exprimer la protéine
hétérologue NusA-PROentier.
La purification de la protéine de fusion MBP-PROentier a
donc été entreprise à partir
de la culture de 300 mL réalisée en
erlenmeyer. La totalité du surnageant de lyse a
été déposée sur résine d'amylose. L'analyse
SDS-PAGE de la fraction principale d'élution fait apparaître une
bande protéique majoritaire, dont la masse apparente correspond bien
à celle de MBP-PROentier (Figure 4.8A), ce qui confirme la surexpression
de la protéine de fusion. La quantité de protéine
recombinante présente dans cette fraction de 11 mL est estimée
à environ
2.3 mg, ce qui correspond à une production relativement
faible d'environ 8 mg par litre de culture. Cependant, contrairement aux
domaines PROcatal et PROinser, aucune tendance à
l'agrégation de la protéine de fusion n'a été
constatée.
Le clivage du partenaire de fusion a donc été
réalisé en ajoutant 180 ug de protéase
TEV dans la fraction d'élution contenant 2.3 mg de
protéine de fusion. Comme le montre le
gel d'acrylamide de la Figure 4.8B, le rendement de
coupure du partenaire MBP est très satisfaisant et atteint
quasiment 90 %. En effet, 2 bandes de moyenne intensité, dont
les masses apparentes correspondent à celles de PROentier et
MBP, apparaissent sur le gel d'acrylamide, alors que la bande
protéique correspondant à MBP-PROentier a quasiment
disparu.

(kDa)
150
100
75
50
37
25

A)
MBP-PROentier
(kDa)
150
100
75
50
37
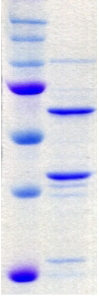
25
B)
MBP-PROentier
PROentier
MBP
Figure 4.8 : Purification et
clivage du partenaire de fusion de MBP-PROentier (107 kDa) suivis par
SDS-PAGE. A) Purification sur résine d'amylose : analyse de la fraction
principale d'élution. B) Analyse de la solution de clivage par
la TEV. Mise en évidence des bandes protéiques de
MBP-PROentier, PROentier (67 kDa), et MBP (43 kDa).
Au vu de ce bon résultat, nous avons
abordé avec confiance la dernière étape de
séparation du partenaire de fusion. La solution de clivage de
11 mL a été déposée sur une colonne
HisTrap HP. La résine a été dans un premier temps
lavée avec un tampon contenant
20 mM d'imidazole, puis l'élution a
été menée avec un gradient linéaire en
imidazole de
20 mM à 200 mM. L'analyse des fractions par SDS-PAGE
montre clairement que le domaine
PROentier n'est pas retenu sur la résine de nickel
(Figure 4.9). En effet, la bande protéique correspondante
n'apparaît que dans les fractions qui contiennent moins
de 40 mM d'imidazole (fractions 1 à 8). On retrouve également
dans ces fractions la MBP endogène de
E. coli issue de la première purification sur
résine d'amylose. Comme attendu, la protéine de fusion
résiduelle MBP-PROentier, et le partenaire clivé MBP,
fixent la résine de nickel jusqu'à des concentrations
d'imidazole de l'ordre de 120 mM. Par conséquent, la
chromatographie de pseudo affinité IMAC sur résine de nickel
permet de séparer le domaine PROentier de son partenaire de fusion, ce
qui n'était pas le cas avec PROcatal, et PROinser.
Au vu de l'analyse SDS-PAGE des fractions contenant
PROentier, un second passage sur résine d'amylose apparaît
cependant nécessaire pour éliminer la MBP endogène de
E. coli, et ainsi atteindre un degré de pureté
satisfaisant. En se basant sur les fractions 6, 7 et 8 contenant
la protéine relativement pure, le rendement obtenu,
à l'issu de cette seconde étape de purification, atteint
à peine 0.9 mg de domaine PROentier purifié par litre
de culture. Il
faudrait donc plus de 10 litres de culture pour espérer
obtenir 10 mg de protéine purifiée.
concentration en imidazole (mM)
20
100
200
chargement lavage élution
MBP- PROentier
PROentier
MBP MBP coli
SC 1 2 3 4
5 6 7 8

9 10
11 12








13 14

(kDa)
150
100
75
50
37
TEV
25
20
Figure 4.9 : Suivi par SDS-PAGE
de la purification de PROentier sur résine de nickel.
Analyse des fractions de chargement, de lavage,
d'élution, et de la solution de clivage (SC). Les protéines
MBP-PROentier (107 kDa), MBP clivée (43 kDa), et PROentier (67 kDa) sont
mises en évidence par des flèches rouges, et la protéase
TEV (27 kDa), et la MBP endogène à
E. coli (40 kDa) par des flèches vertes.
Nous avons enregistré un spectre d'absorbance entre 400 et
600 nm sur l'échantillon
N° 7 contenant 83 ug de protéine PROentier pure
à plus de 90 % (estimation à partir du gel
d'acrylamide). De manière intéressante, ce spectre
fait apparaître un léger pic à 450 nm qui
correspond à la longueur d'onde d'absorbance du
FAD dans le visible (non montré). Ce résultat
suggère donc l'incorporation du cofacteur FAD au sein du domaine
PROentier dont le repliement serait proche de la forme native. Cette
analyse ne fournit cependant qu'une première indication et doit
être confirmée avec un échantillon de protéine plus
concentrée.
2.2.4) Obtention du domaine PROter
A l'issu des tests d'expression en microplaques, nous avons
retenu comme meilleures conditions d'expression de PROter : le partenaire NusA,
la souche Rosetta, et une température
de 20°C. Comme le met en évidence le tableau 4.5,
les résultats de la transposition à grande échelle,
menée en erlenmeyer et en fernbach, ne permettent pas de valider ces
conditions. En effet, l'expression dans la fraction soluble est quasiment nulle
en erlenmeyer de 3L contenant
300 mL de culture, et elle est très faible en fernbach
contenant 1 litre de culture. De plus, la diminution de la vitesse de
croissance bactérienne observée en microplaques, a
également été constatée à grande
échelle ; ce qui confirme l'hypothèse du caractère
toxique du domaine PROter. En parallèle, la production en grand volume
avec le partenaire NusA a été testée en souche C41, qui
permet d'améliorer l'expression des protéines
hétérologues PROter en microplaques. Que ce soit en erlenmeyer
ou en fernbach, aucune expression n'a été
détectée
dans la fraction soluble ou insoluble avec cette souche.
|
PROter
|
Microplaque
(250 uL)
|
Fernbach
(1 L)
|
Erlenmeyer
(300 mL)
|
|
NusA/Rosetta/20°C
|
S
++
|
I
++
|
S
+
|
I
+
|
S
+/-
|
I
+
|
Tableau 4.5 : Résultats
de la transposition à grande échelle des meilleures
conditions
d'expression PROter.
Nous avons tout de même entrepris la purification
de la protéine de fusion NusA- PROter à partir de la
culture menée en fernbach. Aucune résine d'affinité
spécifique à NusA n'étant disponible au laboratoire,
celle-ci a été réalisée sur une colonne de nickel
HisTrap HP.
La moitié du surnageant, provenant de 500 mL de
culture, a été déposée sur la résine.
L'élution a été menée avec un gradient
linéaire d'imidazole de 20 mM à 400 mM. L'analyse SDS-PAGE fait
apparaître une très légère bande à peine
discernable, dont la masse apparente correspond à celle de
NusA-PROter, dans les fractions contenant entre 100 et 200 mM
d'imidazole (fractions 9 à 12) (Figure 4.10A).
concentration en imidazole (mM)
20
100
200
chargement lavage élution
(kDa)
150
100
75
50
37
1 2 3 4
5 6 7 8
9 10 11 12 13

NusA- PROter
10 11 12
(kDa)
150
100
75
50
37
25
25
A) B)
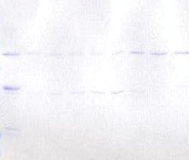
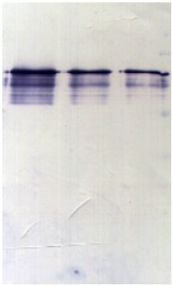
Figure 4.10 : Purification de
la protéine de fusion NusA-PROter (68 kDa) sur résine
de
nickel. A) Analyse des fractions de chargement, de lavage,
et d'élution par SDS-PAGE. B) Analyse des fractions d'élution 10
à 12 par Western-blot avec un anticorps anti-NusA.
Afin de confirmer la présence de la protéine de
fusion, une hybridation Western-blot, beaucoup plus sensible que la
coloration au bleu de Coomassie, a été menée avec
des anticorps anti-NusA (Figure 4.10B). Cette analyse montre que la
bande protéique de faible intensité correspond bien à
la protéine NusA-PROter, mais elle révèle également
un profil de dégradation de la protéine
hétérologue. En effet, la présence de «
traînées » en dessous d'une bande majoritaire sur une
membrane de Western-blot est caractéristique d'une
protéolyse partielle. D'autre part, la quantité de
protéine étant trop faible dans les fractions d'élution,
il
n'a pas été possible de quantifier le rendement de
production par mesure de l'absorbance à
280 nm ; d'autant plus que l'imidazole possède
une absorbance résiduelle à cette longueur d'onde. Au vu de
ces résultats, il n'a pas été envisagé d'aborder
l'étape suivante de clivage du partenaire de fusion.
| 


