2) Caractérisations préliminaires
2.1) Caractérisation de la séquence
primaire et contrôle du marquage
Avant de débuter l'analyse structurale de K2 par RMN, nous
avons voulu nous assurer
de la fiabilité et de l'efficacité de la
méthode employée pour obtenir les échantillons de
protéine. La séquence primaire des protéines K2
simplement et doublement marquée a été
contrôlée par spectrométrie de masse ITD-MS (Ion Trap
Detector-Mass Spectroscopy) sous ionisation par ESI (ElectroSpray
Ionization).
Après concentration sur cellule Amicon, 2 uL d'un
échantillon de K2 ont été prélevés
et introduits dans une solution contenant, 100 uL d'H2O, 100
uL d'acétonitrile, et de l'acide formique (0.25% pour K2 15N,
et 0.75% pour K2 15N / 13C). 10 uL de ce mélange
contenant environ 15 ng/uL de K2 ont ensuite été injectés
dans la source ESI, puis analysés avec un enregistrement de 20
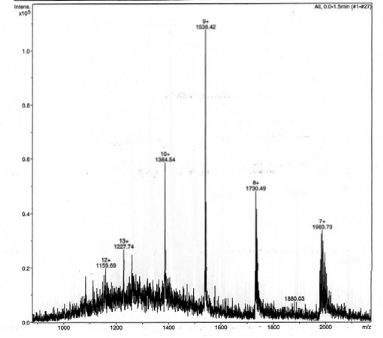
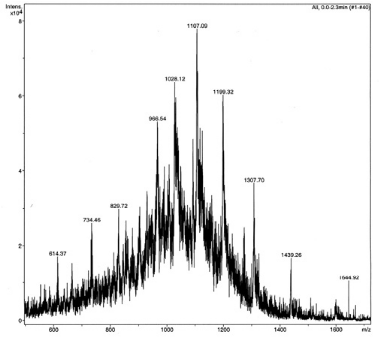
accumulations. Les spectres obtenus sont présentés en Figure 2.7
et les
résultats sont regroupés dans le Tableau 2.3.
15N
9+
1538.42
15N /
13C
13+
1107.09
14+
1028.12
12+
1199.32
10+
1384.54
8+
1730.42
11+
1307.70
7+
1978.73
10+
1439.26
Figure 2.7 : Analyse ESI-ITD-MS des
échantillons de K2 15N et K2 15N /
13C.
Le spectre de masse de K2 simplement marqué
15N fait apparaître une enveloppe d'ions
multichargés centrée autour d'un ion nonachargé. La
déconvolution de cette enveloppe permet de conclure à la
présence d'une forme unique de masse expérimentale 13836 #177; 3
Da. Cette masse correspond tout à fait à celle attendue
et indique que la protéine simplement marquée 15N
a été produite avec un enrichissement isotopique total ou
quasi-total.
Sur le spectre de l'échantillon doublement marqué,
on observe la présence d'un massif
isotopique pour chaque multichargé. L'apparition de ces
amas traduit l'existence de plusieurs formes isotopiques de K2, et
suggère que le double marquage n'est pas total. La
déconvolution automatique d'un tel spectre n'étant pas
possible, nous avons réalisé une déconvolution
manuelle de chaque pic majoritaire correspondant aux 4 ions multichargés
les plus intenses. Les résultats obtenus indiquent que la forme
isotopique prédominante possède une masse expérimentale de
14379 #177; 3 Da pour une masse attendue de 14445 Da. Le taux d'enrichissement
isotopique de l'échantillon 15N / 13C peut donc
être estimé à 92%.
|
Echantillon
|
masse théorique [Da]
|
masse expérimentale
(déconvolution) [Da]
|
taux d'enrichissement
|
|
K2 15N
|
13838,9
|
13836 #177; 3
|
~ 100 %
|
|
K2 15N / 13C
|
14445,3
|
14379 #177; 3
|
~ 92 %
|
Tableau 2.3 :
Caractérisation de la production des échantillons de K2
simplement et
doublement marqués par analyse ESI-ITD-MS. Les
masses théoriques sont indiquées en considérant un
enrichissement isotopique de 100%.
2.2) Caractérisation de l'état
oligomérique
Une fois le poids moléculaire du domaine
protéique vérifié par spectroscopie de masse, nous
avons entrepris une analyse par chromatographie de gel filtration afin
de caractériser l'état oligomérique de K2 en
solution. Cette technique chromatographique d'exclusion permet de
séparer des molécules en fonction de leur poids
moléculaire, et cela en conditions non dénaturantes en
choisissant un tampon d'équilibration adapté.
La colonne de filtration sur gel que nous avons utilisée
est une colonne Superdex75
HR analytique (Pharmacia) de 24 mL dont la gamme de
résolution se situe entre 70 et 3 kDa.
200 uL d'un échantillon concentré de
protéine doublement marquée 15N / 13C (soit
environ
300 ug) ont été introduits dans cette
colonne préalablement équilibrée avec une solution
tampon de PBS (Phosphate Buffer Saline : 150 mM NaCl, 10 mM phosphate, 2.5 mM
KCl,
pH 7.4). L'élution des protéines a
été détectée par suivi de l'absorbance à 280
nm. En amont
de l'analyse de K2, la Superdex75 a été
étalonnée dans les mêmes conditions avec un kit de
calibration contenant un mélange de protéines standard.
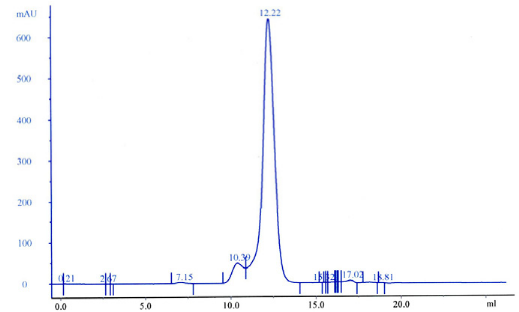
Le chromatogramme de l'analyse de K2 doublement
marquée fait apparaître un pic
ultra majoritaire à un volume d'élution de
12.22 mL (Figure 2.8A). En se basant sur les volumes de
rétention des protéines standard, ce volume correspond
à un poids moléculaire d'environ 18 kDa pour une masse
théorique monomérique attendue de 14.5 kDa. Il semble donc que
la structure quaternaire du domaine K2 soit monodisperse et
sous forme monomérique dans ces conditions d'analyse très
proches des conditions RMN. Ce résultat est cependant nuancé
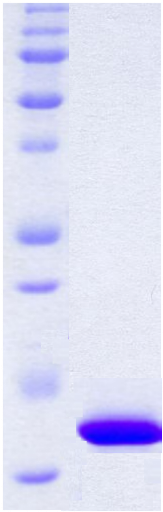
par l'apparition d'un petit pic à 10.39 mL (~38 kDa) qui
pourrait correspondre à une forme dimérique très
minoritaire, ou à une impureté non observable sur le
gel SDS-PAGE (Figure 2.8B). Par ailleurs, la chromatographie
de gel filtration est certes une technique simple et rapide, mais
elle ne fournit qu'une indication de l'organisation quaternaire
d'une protéine dans un tampon donné. Ce résultat doit donc
être considéré avec précaution ou
confirmé par des études plus précises de
diffusion de la lumière ou
d'ultracentrifugation analytique.
A) 12.22 B)
(kDa)
50
37
25
20
15
10

10.39
Figure 2.8 : Caractérisation
de la structure quaternaire de K2 15N / 13C. A)
chromatogramme
de l'analyse par gel filtration en conditions non
dénaturantes. B) analyse SDS-PAGE du même échantillon
(conditions dénaturantes).
2.3) Caractérisation de la structure secondaire et
tertiaire
Le dichroïsme circulaire (DC) est une méthode simple
et sensible qui permet d'évaluer
le niveau de structuration d'un polypeptide. La
technique repose sur les propriétés spectroscopiques des
molécules chirales qui absorbent les composantes polarisées
circulaires droite et gauche de la lumière de manière
inégale. Dans le cas des protéines, le phénomène de
dichroïsme circulaire est essentiellement dû à leur
structure secondaire dans l'UV lointain
(région dominée par l'absorption des liaisons
peptidiques), et à leur structure tertiaire dans
l'UV proche (région dominée par l'absorption des
cycles aromatiques). Aussi, chaque type de conformation présente un
profil d'absorption qui lui est propre (Yang et al., 1986).
Selon ce principe, l'analyse des spectres de DC permet
de distinguer sans aucune ambiguïté une protéine
repliée d'une protéine non structurée. C'est dans
cette optique que nous avons entrepris l'analyse du domaine K2 par
dichroïsme circulaire. Les spectres de DC
ont été enregistrés sur un
échantillon de K2 non marquée afin de vérifier la
structuration de la protéine avant de la produire en grande
quantité en milieu minimum et Algone.
L'analyse a été menée à 25°C en
solvant H2O contenant 50 mM de tampon phosphate
à pH 7.0, et 200 mM de NaCl. Le spectre enregistré
dans l'UV lointain présente un minimum
à 208 nm spécifique de la conformation
hélicoïdale (Figure 2.9). Cependant, aucun autre minimum
n'est clairement discernable dans la région autour de
222 nm. Ce profil d'absorption est caractéristique d'une
protéine structurée en partie en hélice á et en
feuillet â (Venyaminov & Yang, 1996). Par ailleurs, le signal
d'un spectre DC enregistré dans l'UV proche est
généralement très faible en absence de conformation
ordonnée. Dans le cas de la protéine K2, deux minima intenses
apparaissent à 279 et 288 nm et correspondent à
l'absorption de cycles aromatiques de tyrosine et de tryptophane d'une
protéine adoptant une structure tertiaire stable.
30
20
10
0
-10
èx10-3
[degré.cm2.dmole-1]
-20
4


UV lointain UV proche
2
0
-2
-4
-6
-8
-30
200 210 220 230 240
Longueur d'onde ë [nm]
-10
260 280 300 320 340
Figure 2.9 : Spectres de
dichroïsme circulaire enregistrés sur un échantillon de
protéine K2
non marquée solubilisée dans un tampon
phosphate à pH 7.0 et à 25°C.
2.4) Etude préliminaire par Résonance
Magnétique Nucléaire
2.4.1) Enregistrement des premières
expériences
Dans l'optique de confirmer les résultats obtenus par
dichroïsme circulaire, nous avons débuté l'étude
RMN par l'enregistrement des spectres 1D 1H et 2D HSQC
15N-1H sur un échantillon de protéine
simplement marquée 15N. Ces expériences ont
été réalisées sur un spectromètre 600
MHz équipé d'une cryosonde TXI triple
résonance dans les conditions initiales suivantes : une
température de 20°C, un tampon phosphate de pH 7.0, et
une concentration de protéine d'environ 0.7 mM.
Une protéine structurée présente une
signature très différente d'une protéine peu ou
pas structurée sur un spectre HSQC
15N-1H du fait de la variation de l'environnement
chimique induite par les structures secondaire et tertiaire. La dispersion
globale des pics de corrélation sur le spectre HSQC de K2
confirme que la protéine est repliée (Figure 2.10). Ainsi,
les pics situés à gauche de la région centrale, à
un déplacement chimique 1H supérieur à
9 ppm, sont généralement
caractéristiques d'une structuration en feuillet â. D'autre
part, le spectre 1D 1H fait apparaître des raies de
résonance à un déplacement chimique négatif dans
la région des protons aliphatiques. Ces observations
sont typiques d'une protéine repliée
adoptant des éléments de structure secondaire et
tertiaire.












ppm
104
106
108
110
112
114
116
118
120
122
124
126
128
130
10.5
10.0
9.5
9.0
8.5
8.0
7.5
7.0
6.5
ppm
Figure 2.10 : Spectre 2D HSQC
15N-1H de K2 simplement marqué 15N
(tampon phosphate, pH=7.0) enregistré sur un spectromètre 600 MHZ
et à 20°C.
Le nombre de pics de corrélation présents sur ce
spectre est difficile à comptabiliser en
raison d'une superposition importante dans la région
centrale. Il est d'autant plus difficile à déterminer que la
largeur des raies de résonance est importante à cette
température. On peut toutefois estimer ce nombre proche de 100
sachant que le nombre de groupements amides attendu s'élève
à 107 (111 résidus dont 3 prolines, et en considérant que
le groupement amide NH2 du premier résidu est
généralement non observable).
Afin de déterminer si les conditions initiales
choisies étaient adaptées à une étude
complète par RMN, nous avons enregistré une
première série de 4 expériences 3D
hétéronucléaires sur un échantillon
15N / 13C de K2. Il s'agit des expériences
3D HNCO, HNCA, CBCA(CO)NH, et CBCANH, qui permettent l'attribution de la
chaîne principale et
des Câ. Pour chaque expérience, le nombre de pics
qui apparaissent dans les spectres a été comptabilisé et
figure dans le tableau 2.4. Bien que la HNCO soit l'expérience
3D la plus sensible, dans ces conditions d'analyse, 16
déplacements chimiques de carbonyle sont manquants. Les
expériences HNCA et CBCA(CO)NH ne contiennent qu'environ 80% de
données attendues. CBCANH, la moins sensible des 4 expériences,
mais néanmoins majeure dans la stratégie d'attribution, ne
contient qu'un peu plus du tiers de données attendues. Au final, la
quantité d'information recueillie sur les spectres semble insuffisante
pour envisager une attribution quasi-complète du squelette de K2. Par
conséquent, nous avons conclu que les conditions initiales d'analyse
(pH=7.0 et T=20°C) ne sont pas les plus adaptées pour attribuer
l'ensemble des résonances 1H, 15N,
et 13C de la protéine K2.
Expériences
nombre de pics
attendus
nombre de pics
%
observés
HNCO 108 92 85 %
HNCA 216 174 80 %
CBCA(CO)NH 211 162 77 %
CBCANH 420 157 37 %
Tableau 2.4 : Analyse des
spectres HNCO, HNCA, CBCA(CO)NH, et CBCANH de K2
doublement marquée 15N / 13C
(pH=7,0) enregistrés sur un spectromètre 600 MHZ et à
20°C.
2.4.2) Optimisation des conditions de l'analyse par
RMN
A ce stade de l'étude, une optimisation des
conditions de l'analyse par RMN est apparue indispensable avant de
débuter l'analyse d'une longue série d'expériences
pouvant s'échelonner sur plusieurs semaines. Les deux principaux
paramètres qui influent sur la qualité et la quantité
de signal des spectres RMN sont le pH et la température.
L'absence d'une dizaine de corrélations de groupement
amide sur le spectre HSQC de
K2 peut être expliqué par un échange rapide
de protons amides accessibles avec les protons de l'eau. La plupart des
expériences 3D hétéronucléaires utilisées
pour l'attribution reposent sur
la corrélation d'un ou plusieurs atomes avec les
noyaux 1H et 15N amides. Par conséquent,
l'échange rapide des protons amides avec le solvant
affecte également la quantité de signal
sur ces spectres. Une diminution du pH de l'échantillon
permet de réduire la vitesse de cet échange (Figure 2.11).
Cette vitesse est minimum entre pH 3 et 4. Cependant, un pH trop
acide peut déstabiliser le repliement et conduire à une
déstructuration partielle ou totale de la protéine.
L'optimisation de ce paramètre a donc pour objectif de déterminer
la valeur de pH à laquelle l'information enregistrée est maximum
et le risque de dénaturation minimum.
600 ns
60 us
Tk 6 ms
H
0.6 s
1 min
10 min
N
0 2 4 6 8 10
pH
Figure 2.11 : Influence du pH
sur l'échange des protons amides accessibles au solvant. Tk
représente le temps moyen d'échange de proton amide labile de
polypeptide dans un solvant H2O et à 25°C (d'après
Wüthrich, 1986).
Le spectre HSQC de K2 enregistré à 20°C
présente une région centrale encombrée et peu
résolue. La cause de ce manque de résolution est notamment
imputable à la largeur de raie des pics de corrélation. Une
augmentation de la température permet de réduire la
viscosité
de l'eau, et de raccourcir le temps de réorientation
globale ôc de la macromolécule. Ceci se
traduit par un affinement des raies, et donc par une
amélioration de la résolution. De plus,
l'augmentation de l'agitation brownienne peut influer sur la
fréquence de résonance propre de certains noyaux et permettre
l'éclatement d'un massif. Cependant, les échantillons de
protéine sont généralement moins stables à
haute température car la tendance à la
dénaturation, et l'activité des protéases
résiduelles issues de E. coli, sont alors plus
prononcées. Comme pour
le pH, le choix de la température doit donc
résulter d'un compromis entre l'amélioration de la
résolution spectrale et le risque associé de
dénaturation.
L'influence du pH et de la température a
été évaluée sur un échantillon de K2
simplement marqué 15N concentré à 0.8 mM.
Une série d'expériences HSQC 15N-1H a
été entreprise sur un spectromètre 500 MHz dans 6
conditions expérimentales différentes. Chaque spectre a
été enregistré et traité de la même
façon et le nombre de pics (hors groupements NH
ou NH2 de chaîne latérale) a été
comptabilisé pour chaque condition testée (Tableau 2.5).
|
Condition
|
pH=7.0
|
pH=6.5
|
pH=6.0
|
|
T=25°C
|
T=30°C
|
T=25°C
|
T=30°C
|
T=25°C
|
T=30°C
|
|
nb de pics
|
89
|
98
|
101
|
103
|
110
|
110
|
Tableau 2.5 : Analyse des spectres
2D HSQC 15N-1H de K2 simplement marquée
enregistrés
sur un spectromètre 500 MHz dans différentes
conditions de température et de pH.
Tous les spectres HSQC enregistrés présentent le
même profil, similaire à celui obtenu dans les conditions
initiales. Par conséquent, la protéine K2 conserve un repliement
stable aux différents pH et températures testés.
L'effet du pH sur la vitesse d'échange des protons amides
de K2 est clairement visible
sur les spectres HSQC. On observe bien une augmentation du
nombre de résonances avec la diminution du pH. Jusqu'à 110
pics peuvent être comptabilisés à pH 6.0 pour un
nombre attendu de 107. La figure 2.12 illustre l'influence de ce
paramètre sur la région gauche du spectre qui fait
apparaître de nouvelles taches de corrélation à pH plus
acide. D'autre part, nous avons constaté que la diminution du pH permet
d'accroître l'intensité de la majorité des raies de
résonance de K2. Ce gain de signal est particulièrement
intéressant dans l'optique de
recueillir davantage d'information sur les spectres
3D hétéronucléaires. L'effet de la

















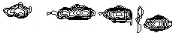






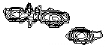












température est également visible sur l'allure
générale des spectres. La résolution de la région
centrale croît avec l'augmentation de la température, ce qui
permet de mieux distinguer les pics de corrélation.
pH=6.0 pH=6.5 pH=7.0
T=25°C
114
116
118
120
122
T=30°C
124
126
9.3
9.0
8.7
Figure 2.12 : Extrait de spectres
2D HSQC 15N-1H de K2 simplement marquée
15N enregistrés
sur un spectromètre 500 MHz dans différentes
conditions de température et de pH.
Finalement, à la vue de l'ensemble de ces
résultats, nous avons retenu comme conditions
expérimentales, une température de
30°C, et un pH de 6.0 pour mener
l'étude structurale de K2 par RMN.
| 


