I.1.1.b. La classification :
Comme tous les cadres taxonomiques, la classification au sein
de ces éléments est actuellement soumise à de
fréquentes révisions en raison de l'identification de nouvelles
familles (Havecker et al., 2004). En fonction de l'ordre des domaines
dans leurs séquences Pol, les rétrotransposons à
LTR sont essentiellement divisés en deux grandes super familles, dont la
mobilité est autonome, la première est ty1/copia
(Pseudoviridae) et la deuxième est ty3/gypsy
(Métaviridae) (in Luchetta et al., 2005). Cependant,
Witte et al. (2001) ont pu découvrir une nouvelle
catégorie appelée TRIM (Terminal Repeat
Retrotransposon In Miniature) (Mhiri et Grandbastien, 2004), aussi un
autre groupe de chercheur a identifié des éléments
regroupé sous le terme de LARDs (pour Large Retrotransposon
Derivatives). (Havecker et al., 2004) la mobilité des
éléments au sein de ces dernières familles est
supposée d'être en trans c'est-à-dire qu'elle
nécessite la présence des protéines d'un autre
élément autonome (ces éléments sont décrits
ultérieurement) (Schulman et Kalendar, 2005).
I.1.1.b1. Les éléments autonomes :
I.1.1.b1.1. La super famille ty1/copia : Pseudoviridae
La taille des éléments de cette super famille
est comprise entre 3925pb12088pb, les LTRs sont entre 156pb-1929pb, et
présentent un TSD (pour Target Site Duplication) de 5pb. L'ordre des
domaines des gènes Pol est : PR-INT-RT-RH (Figure1.1) dont les
protéines sont caractérisées par des motifs hautement
conservés (Pelsy et merdinoglu , 2002). Cette super famille est
rangée en trois familles : les Hémivirus, les Pseudovirus, et
enfin les Sirevirus (la nomenclature n'indique pas qu'ils sont vraiment des
virus). Les Sirevirus se distinguent par leur abondance dans les génomes
végétaux et aussi par la séquence protéique de leur
RT. Les Hémivirus et les Pseudovirus sont caractérisés par
les amorces utilisées pour la transcription inverse qui sont un ARNt
complet et un demi-ARNt respectivement (Havecker et al., 2004). Dans
cette Super Famille il existe entre autres les rétrotransposons
Ty1, Ty2 et Ty4 de la levure Saccharomyces
cerevisiae, les copia -like (copia, mdg1, mdg3)
et 1731 de la drosophile (In Halaimia Toumi, 2006).
I.1.1.b1.2. La super famille ty3/gypsy :
Métaviridae
L'ordre des domaines des gènes Pol est :
PR-RT-RH-INT, qui est semblable à celui des rétrovirus (Figure
1.1). Les membres de la super famille ty3/gypsy sont
d'intérêt, parce que, contrairement à leurs voisins les
rétrovirus, ils possèdent parfois une spécificité
remarquable pour leur site d'insertion, par exemple, l'élément
ty3 de la levure S. cerevisiae s'insère exactement aux
environs 5pb du site de début du gène codant pour l'ARN
polymérase Ø, cela s'avère un excellent choix car ce
gène possède des promoteurs internes, de sorte que l'insertion
ait un effet minime sur l'expression génétique (Malik et Eikbush,
1999). Certains éléments de cette super famille peuvent
acquérir un ORF additionnel codant le gène env des
rétrovirus, c'est le cas des éléments gypsy et
ZAM de la drosophile. Il a été démontré
que l'élément gypsy posséde une capacité
infectieuse. Les analyses phylogénétiques des séquences de
la Reverse Transcriptase montrent que la super famille ty3/gypsy est
généralement divisée en trois familles : les
Métavirus dont l'élément Boudicca de l'homme et
Athlia d'Arabidopsis, les Errantivirus dont
l'élément gypsy et les Sémotivirus qui sont
présents aussi chez les nématodes (Havecker et al.,
2004).
I.1.1.b2. Les éléments non autonomes :
I.1.1.b2.1. La famille des TRIMs :
Les TRIMs (pour Terminal Repeat Retrotransposon
In Miniature) ont été décrits pour la première
fois dans les génomes du pomme de terre (Havecker et al.,
2004), ils présentent la plupart des caractéristiques
structurales des rétrotransposons à LTR, mais sont beaucoup plus
petits (300 à 800 pb) composés de petites LTR encadrant un court
domaine central non codant (Figure1.1). L'origine des TRIM est encore
peu claire et il est probable que leur amplification nécessite une
trans-activation par un autre rétrotransposon (Mhiri et
Grandbastien, 2004), un des éléments connu de ce groupe est
appelé Cassandra, il est une séquence de 565-860 pb,
avec des LTR qui varient selon les espèces, leur taille est comprise
entre 240-350pb. Les LTR d'un élément Cassandra
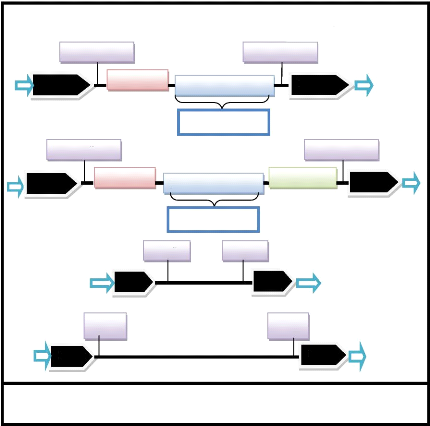
Figure 1.1: L'organisation génomique
des différentes familles des rétrotransposons
à LTR.
(D'après Havecker et al., 2004).
LTR
LTR
LTR
PBS
PBS PPT
PBS PPT LARDs
gag
LTR
gag PR-INT-RT-RH
PBS
PR-RT-RH-INT
Le gène pol
Le gène pol
PPT
LTR
env
TRIM
Ty1/copia
LTR
LTR
PPT
LTR
Ty3/gypsy
totalement séquencé présentent un motif
très conservé : 5'-TG.....CA3'et des ITRs de 6- 12 pb (Kalendar
et al., 2008).
| 


