UNIVERSITE DE RENNES I (U.F.R. Sciences de la vie et de
l'Environnement)
ECOLE NATIONALE D'INGENIEURS DES TECHNIQUES DES INDUSTRIES
AGRICOLES ET ALIMENTAIRES DE NANTES (ENITIAA)
MASTER SCIENCES, TECHNOLOGIES, SANTE : mention
Recherche en Biologie
Spécialité : Microbiologie
Fondamentale et Appliquée
2005/2006
Caractérisation moléculaire des
bactéries lactiques des levains de panification
Mémoire présenté le 27 juin 2006
Par
Ziad RIZK
Stage effectué au Laboratoire de Microbiologie
Alimentaire et Industrielle de l'ENITIAA de Nantes
MASTER SCIENCES, TECHNOLOGIES, SANTE : mention
Recherche en Biologie
Spécialité : Microbiologie
Fondamentale et Appliquée
2005/2006
Caractérisation moléculaire des
bactéries lactiques des levains de panification
Mémoire présenté le 27 juin 2006
Par
Ziad RIZK
Maître du stage :
Xavier DOUSSET -Professeur
Bernard ONNO- Maître de conférence
Stage réalisé au Laboratoire de Microbiologie
Alimentaire et Industrielle de l'ENITIAA de Nantes
Début du stage : 5/Janvier/ 2006
Fin du stage : 25/Juin/2006
Je tiens tout d'abord à remercier Monsieur le
Professeur Hervé PREVOST, responsable du laboratoire de Microbiologie
Alimentaire et Industrielle (LMAI) de l'ENITIAA, de m'avoir accueilli au sein
de son unité.
Je tiens à remercier Monsieur le Professeur
Xavier DOUSSET pour son encadrement scientifique et les conseils
prodigués tout au long de mon stage.
Je remercie également Monsieur Bernard ONNO pour
son encadrement scientifique, sa persévérance et sa contribution
à la réalisation de ce travail.
J'adresse ma vive reconnaissance à
Angélique FOURRIER « Technicienne » du laboratoire
pour m'avoir, initié aux techniques de biologie moléculaire et
pour ses précieuses valeurs humaines. Je la remercie également
pour sa patience, sa bonne humeur et sa joie de vivre.
J'exprime ma gratitude à Maria IGNATOVA et
à Zouheir BEN BELGACHEM « Thésards » à
l'ENITIAA pour leur collaboration et leur disponibilité au cours de ce
travail.
Je remercie conjointement tous les stagiaires et
thésards du laboratoire en particulier : Morgan,
Ségolène, Emmanuel, Sébastien et Stella pour leur conseils
et surtout pour leur accueil qui a rendu mon séjour très
agréable à l'ENITIAA.
Enfin, je n'oublie pas de remercier les enseignants
chercheurs ainsi que le personnel du laboratoire pour leur soutien, leur
disponibilité et leur gentillesse.
BET : Bromure d'éthidium
BSA : Bovine Sérum Albumine
DGGE : Denaturing Gradient Gel
Electrophoresis
EDTA : Ethyl Diamine Tétracide
Acétique
ISR : Intergenic Spacer Region
ITS : Internal Transcribed Spacer
Lb : Lactobacillus
Ln : Leuconostoc
MRS: Man Rogosa Sharpe
RAPD: Randomly Amplified Polymorphic DNA
RFLP: Restriction Fragment Length
Polymorphism
S: Svedberg
SDB: SourDough Bacteria
SDS: Sodium Dodécyl Sulfate
TAE: Tris Acétate EDTA
TBE : Tris base EDTA
TGGE : Temperature Gradient Gel
Electrophoresis
TTGE : Temporal Temperature Gradient Gel
Electrophoresis
UFC : unité formant colonie
Wt/v: Weight per volume
V/V : volume par volume
Caractérisation moléculaire
des bactéries lactiques de levains de panification
Introduction bibliographique
Chapitre I- Etude bibliographique
I. Généralités
1
II. Définition d'un levain de panification
1
III. Les microorganismes des levains de panification
1
III.1. Les levures
1
III.2. Les bactéries lactiques
3
III.3. Les principaux rôles des bactéries
lactiques en panification 4
III.3.1. Le rôle protéolytique des
bactéries
4
III.3.2. La production du gaz carbonique
4
III.3.3. Les aspects aromatiques
5
III.3.4. L'activité anti-microbienne
5
IV. Les méthodes de caractérisation
moléculaire des bactéries lactiques
5
Chapitre II- Matériels et
Méthodes
I. Souches de référence
11
II. Echantillons des levains étudiés
11
III. Détermination du pH et du TTA
11
IV. Milieux de culture
11
IV.1. Milieux de dénombrement des levures
11
IV.2. Milieux de dénombrement, d'isolement et
d'identification des bactéries 12
lactiques
IV.3. Conservation des souches et des isolats
12
V. Dénombrement et isolement des
bactéries lactiques
12
VI. Caractérisation des bactéries
lactiques
12
VI.1.Caractérisation phénotypique
12
1- La morphologie des colonies et la coloration de Gram
12
2- Le test de catalase
12
3- Le test homo/hétérofermentaire
12
VI.2.Caractérisation moléculaire
12
1- Extraction de l'ADN selon le Kit Qiagen
12
2- Quantification de l'ADN
13
3- Les amorces
13
4- La PCR
13
5- L'électrophorèse sur le gel d'agarose
14
6- La restriction enzymatique (RFLP)
15
Chapitre III- Résultats et Discussions
I. Détermination du pH et du TTA
16
II. Identification des bactéries lactiques du
levain Français 16
II.1. Dénombrement des
bactéries lactiques et des levures
16
II.2. Identification phénotypique des
bactéries lactiques
17
II.3. Identification moléculaire des
bactéries lactiques
17
II.3.1. Identification des isolats du
BF-70 17
II.3.2. Identification des isolats du
BF-FC 21
II.3.3.PCR sur colonies
22
Conclusions et perspectives
24
Références bibliographiques
26
Tableaux et figures
29
Le pain est un aliment de base dans de nombreuses
sociétés humaines. Il est fabriqué à partir de
farine, de levure ou levain et d'eau. Ce qui caractérise le pain, c'est
le fait que la pâte est soumise à un gonflement dû à
la fermentation. La farine provient principalement de céréales
panifiables : blé et seigle. En effet, celle-ci se
caractérise par la présence d'une protéine, le gluten, qui
emprisonne les bulles de gaz carbonique dégagées par la
fermentation et permet la levée de la pâte.
Le premier pain produit par l'humanité était
vraisemblablement un pain au levain résultat d'une fermentation mixte
levures-bactéries lactiques. Ce procédé traditionnel de
panification s'est perpétué jusqu'à ce jour, malgré
la place prépondérante occupée par la levure de
boulangerie.
Les industries des produits céréaliers
fermentés (panification, viennoiserie, crackers, boissons aux
céréales) ont actuellement un regain d'intérêt pour
les fermentations mixtes-bactéries lactiques, sur le modèle
levain naturel dans le but d'améliorer les qualités aromatiques,
organoleptiques, nutritionnelles et bioconservatrices des aliments.
La maîtrise de ce procédé de fermentation
mixte s'appuie d'une part sur l'étude de la biodiversité
microbienne des levains et d'autre part sur la caractérisation des
flores, notamment, lactiques à l'aide de méthodes
d'identification biomoléculaires.
La région intergénique ITS qui sépare les
gènes ribosomiques codant pour l'ARNr 16S et 23S de l'opéron
ribosomique met en évidence un polymorphisme de taille et de
séquence suffisant pour permettre l'identification et la
différenciation des genres, des espèces bactériennes et
éventuellement des souches d'une même espèce.
Ce projet a d'abord pour objectif : d'une part de montrer
l'influence de la matière première mise en oeuvre sur la flore
lactique d'un levain et notamment sur la diversité des espèces de
Lactobacillus et d'autre part de valider les méthodes
moléculaires de caractérisation des flores lactiques par
amplification et restriction enzymatique des régions
intergéniques 16S-23S sur des échantillons de levains.
Ces études concernant la caractérisation
moléculaire des levains de panification se réalisent au sein du
laboratoire de Microbiologie alimentaire et industrielle de l'Ecole Nationale
d'Ingénieurs des Techniques des Industries Agricoles et Alimentaires
(ENIT IAA) à Nantes.
I.
Généralités
Quatrième producteur de blé au monde, la France
est un pays de tradition boulangère. Pourtant la consommation de pain en
France n'a cessé de chuter depuis la moitié du 20ème
siècle. Les modifications du mode de vie et le
développement des techniques de réfrigération favorisant
la consommation d'autres aliments peuvent expliquer cette baisse de
popularité.
Dans ce contexte, des efforts sont entrepris, notamment par le
secteur artisanal, pour améliorer la qualité des produits et pour
diversifier l'offre. Les fermentations longues sont un des moyens pour
atteindre cet objectif. La fermentation au levain pourrait, dans ce cadre,
connaître un nouvel essor du fait des caractéristiques
organoleptiques des produits obtenus par ce procédé.
II. La définition
d'un « levain » de panification
Il s'agit à la base, d'un mélange de
farine de blé ou de seigle, du sel et de l'eau potable soumis à
une fermentation lente (24 à 48 h) initiée par des levures et des
bactéries lactiques contenues dans la farine (Vogel et al., 1999). C'est
le levain chef. Il est utilisé comme inoculum pour la fabrication du
pain. Le développement et l'activité des levains sont maintenus
par une incorporation périodique et répétée de
farine et d'eau à la température ambiante entre 20 et 35°C.
Cette opération s'appelle le rafraîchi. Ainsi, les
microorganismes continuent à produire des acides. Le levain suffisamment
actif, prêt à être incorporé dans le
pétrissée est appelé « levain tout
point ». (Onno et Roussel 1994).
Pratiquement, une classification en trois types de
fermentations a été proposée par des chercheurs
allemands:
Le type I : avec des levains maintenus par une
inoculation répétée à la température
ambiante (20-30°C) selon les procédés traditionnels
(rafraîchi) où les microorganismes montrent une activité
métabolique accrue, le type II : levains avec une période
longue et des températures élevées de fermentation (plus
de 5 jours) et une teneur en eau plus élevée, principalement,
utilisée dans les procédés industriels pour
l'acidification des pâtes et le type III: levains qui sont
initiées par des cultures starters définies
généralement utilisées comme suppléments pour
augmenter l'acidité et améliorer les propriétés
aromatiques. Dans ce type les levains sont généralement
déshydratés et sont obtenus par séchage des levains
naturels. Ces levains ont une activité bactérienne assez faible
(Vogel et al., 1999 ).
Un levain peut être préparé à
partir des bactéries lactiques sélectionnées, seules ou en
mélange avec des levures, permettant d'ensemencer une pâte en vue
de l'élaboration rapide d'un levain Dans ce cadre, on distingue deux
types de ferments: les levains liquides et les starters lyophilisés.
(Brochoire et al., 1996)
III. Les microorganismes des
levains de panification
Un levain naturel de panification est constitué d'un
équilibre entre les bactéries lactiques et les levures avec un
ratio moyen de 109 / 107 UFC/g respectivement. Ce sont
les bactéries lactiques qui dominent dans les pains aux levains
(Gobbetti et al., 2005). Ces germes préexistants dans
la farine sont également apportés par l'air ambiant et le milieu
du travail (Brochoire et al ., 1996).
III.1: Les
levures
L'espèce la plus connue en boulangerie est
Saccharomyces cerevisiae. Au cours de la fermentation, la levure
produit du gaz carbonique, de l'alcool et d'autres composés. Le gaz
carbonique va permettre la levée de la pâte et modifier ses
propriétés physiques. Dans le cas du levain, la microflore
levurienne n'est pas uniquement composée de Saccharomyces
Tableau n°1: La diversité des
flores des levains naturels
|
Levains étudiés
|
Bactéries lactiques
|
Levures
|
Auteurs
|
|
Homofermentaires
|
Hétérofermentaires
|
|
Levains français et marocains
|
Lb plantarum
Lb delbrueckii
Pc pentosaceus
Lc lactis subsp. lactis
Lb sakei
Lb curvatus
|
Lb brevis
Lb brevis subsp. lindneri
Ln mesenteroïdes
Ln mesenteroïdes subsp. dextranicum
Ln citreum
|
Non identifiées
|
Bervas, 1991
|
|
Levains français
|
Lb plantarum
Lb casei
Lb delbrueckii sp.
Pediococcus
|
Lb brevis
Lb sanfranciscensis
Lb fermentum
Lb buchneri
Lb fructivorans
|
Saccharomyces
Candida
Torulopsis
|
Larpent, 1992
|
|
Levains italiens
|
Lb plantarum
Lb farciminis
|
Lb brevis subp. lindneri
|
S.cerevisiae
C.krusei
S.exiguus
H.anomala
|
Gobetti et al., 1996
|
|
Levains allemands
|
Lb crispatus
Lb delbrueckii sp.
|
Lb brevis
Lb sanfranciscensis
Lb fermentum
Lb fructivorans
Lb pontis
|
Non identifiées
|
Vogel et al., 1999
|
|
Levains allemands
|
Lb amylovorus
Lb delbrueckii sp.
Lb acidophilus
Lc lactis
|
Lb brevis
Lb sanfranciscensis
Lb fermentum
Lb fructivorans
Lb pontis
Lb panis
|
Non identifiées
|
Vogel et al., 1999
|
|
Levains italiens
|
Lb plantarum
Lb alimentarius
Lb acidophilus
Lb delbrueckii subsp. delbrueckii
Lc lactis
|
Lb brevis
Lb sanfranciscensis
Lb fermentum
Ln citreum
Weissella confusa
|
S.cerevisiae
C.krusei
S.exiguus
|
Corsetti et al., 2001
|
|
Levains français
|
Lb plantarum
Pc pentosaceus
|
Lb brevis
Lb sanfranciscensis
Lb buchneri
|
S.cerevisiae
S.exiguus
C.tropicalis
C.holmii
C.krusei
|
Roussel et Chiron, 2002
|
|
Levains grecs
|
Lb paralimentarius
|
Lb brevis
Lb sanfranciscensis
Weissella cibaria
|
Non identifiées
|
De Vuyst et al., 2002
|
|
Levains allemands
|
Lb crispatus
Lb johnsonii
|
Lb mindensis
Lb sanfranciscensis
Lb fermentum
Lb frumenti
Lb reuteri
Lb pontis
Lb panis
|
Non identifiées
|
Meroth et al., 2003
|
Lb: Lactobacillus, Lc: Lactococcus, Ln: Leuconostoc,
Pc: Pedioc occus, S:Saccharomyces, C:Candida, H: Hansenula, Sp:
species
cerevisiae mais par une combinaison entre des
espèces sauvages comme Candida milleri, Candida krusei et
Saccharomyces exiguus (Corsetti et al., 2001).
Meroth et al., 2003 ont inventorié 23 espèces
de levures présentes dans les levains étudiés.
La caractérisation des levures des levains a
été précisée par les techniques
biomoléculaires. La RFLP de l'ADN mitochondrial a montré son
intérêt pour la détection des populations appartenant au
genre Saccharomyces ou à d'autres espèces. Par ailleurs,
le polymorphisme de la séquence intergénique Ä
(de l'ADN mitochondrial) amplifiée permet l'identification des
souches de S. cerevisiae.
D'autres méthodes d'identification et de suivi
dynamique de la population des levures par PCR-DGGE ont été
décrites par Meroth et al., en 2003 .
III. 2: Les
bactéries lactiques
La caractéristique qui fait l'unité de ce groupe
bactérien est la production de l'acide lactique à partir de
différents substrats carbonés. En dehors de ce point commun, les
nombreux genres et espèces qui constituent ce groupe présentent
une grande diversité de caractéristiques morphologiques et
physiologiques. Cela se traduit par l'existence au sein des espèces de
nombreuses souches possédant des propriétés technologiques
différentes. (Desmazeaud 1998).
Ce sont des cellules vivantes, procaryotes et
hétérotrophes dont les caractéristiques sont les
suivantes: bacilles ou coques gram positif, généralement
immobiles, asporulées, aérotolérants, chimiotrophes et ne
possèdent ni catalase, ni nitrate-réductase et ni cytochrome
oxydase (Stiles et al., 1997 ). Le genre le plus
fréquemment isolé des levains est le genre
Lactobacillus.
Le genre Lactobacillus
Les lactobacilles sont présent naturellement dans la
nature et sont rarement pathogènes. Ce sont des cellules
allongées, régulières en forme de bâtonnets ou
coccobacilles isolés ou en chaînettes de taille variable,
asporogènes, immobiles ou mobiles grâce à des flagelles
péritriches, anaérobies facultatifs. Leurs exigences
nutritionnelles complexes et leurs températures de croissance (2
à 53°C) sont très variables d'une espèce à
l'autre mais elles sont toutes acidophiles avec un pH optimal de croissance de
5,5 a 6,2. Leurs GC% sont de 36 à 47. Leur mode de fermentation est
à la base de la subdivision en trois groupes.
-Groupe I: forme de lactobacilles
homofermentaires stricts qui ne fermentent que les hexoses par la voie
d'Embden-Meyerhof en produisant presque exclusivement du lactate. Ce groupe
comprend notamment Lb. acidophilus, Lb. farciminis, Lb. johnsonii, Lb.
amylovorus, Lb. paralimentarius (Cai et al., 1999), Lb.
delbrueckii subsp. delbrueckii, Lb. delbrueckii subsp. lactis, Lb. delbrueckii
subsp. bulgaricus (Torriani et al., 1999), Lb. nantensis
(Valcheva et al., 2006).
-Groupe II: renferme les lactobacilles
homo-hétérofermentaires facultatifs qui fermentent les hexoses
mais aussi les pentoses en lactate et acétate par la voie
d'Embden-Meyerhof. Il s'agit en particulier de Lb. alimentarius, Lb. casei,
Lb. curvatus, Lb. plantarum, Lb. graminis, Lb. paracasei, Lb. paraplantarum,
Lb. pentosus, Lb. sakei (Stiles et al., 1997). Ces
espèces bactériennes sont présentes dans les
végétaux fermentés comme l'ensilage et dans les produits
carnés et laitiers fermentés.
-Groupe III: forme de lactobacilles
hétérofermentaires stricts qui fermentent les hexoses en lactate,
acétate (ou éthanol) et CO2. Les pentoses
aussi sont fermentés en lactate et en acétate. Ces
Lactobacillus ont un faible pouvoir acidifiant et produisent des
substances aromatiques. Il s'agit notamment de Lb. brevis, Lb.buchneri, Lb.
fermentum, Lb.hilgardii, Lb.fructivorans, Lb.panis (Wiese et al.,
1996) , Lb.pontis, Lb. reuteri, Lb.hammesii (Valcheva et al.,
2005), Lb. sanfranciscensis, Lb. spicheri, Lb. kimchii et Lb.
frumenti (Müller et al., 2000). Ces espèces
se retrouvent dans les levains de panification et les produits laitiers
fermentés (Oheix 2003).
La flore bactérienne peut se classer en deux types
selon l'âge des levains: la flore des levains jeunes est
hétérogène, surtout homofermentaire et d'une
minorité hétérofermentaire et la flore des levains plus
âgés devient majoritairement hétérofermentaire. Elle
comprend en majorité Lb. brevis et Lb.
sanfranciscensis (86 à 95 % de la flore totale) (Spicher 1987).
III.3 : Les principaux rôles des
bactéries lactiques en panification
Parmi les effets bénéfiques des levains de
panification sur la qualité du pain citons: le retard du rassissement,
l'augmentation de l'acidité, la protection contre les moisissures,
l'abaissement de l'index glycémique et l'amélioration du flaveur
et de la texture (De Vuyst et al., 2002).
III.3.1. Le rôle protéolytique des
bactéries lactiques :
La dégradation des protéines est due, soit
à l'activité protéolytique bactérienne, soit
à l'activation des protéases de la farine en milieu acide. Les
bactéries lactiques provoquent une augmentation des protéines
solubles dans la pâte et l'apparition de peptides et d'acides
aminés qui outre le fait qu'ils stimulent la croissance des
microorganismes, interviennent dans la formation de certains composés
aromatiques. Par ailleurs, le pH optimal des protéases de la farine
étant voisin de 4, l'acidification de la pâte favorise leur
activation et la production des acides aminés à partir du gluten
(Onno et Roussel 1994). La dégradation du gluten affecte les
caractéristiques rhéologiques des pâtes et la texture du
pain. Les études ont montré que l'acidification par les acides
lactique et acétique change complètement les
caractéristiques rhéologiques durant la fermentation (Thiele
et al., 2004) et est à l'origine d'une diminution de
viscosité et d'extensibilité. Ce changement est prononcé
pour un pH allant de 3,8 a 4,1.
L'activité protéolytique dépend des
souches bactériennes. Les travaux de Gobbetti et al.,
en 1996 ont montré que Lb. sanfranciscensis CB1 isolé du
levain naturel a une capacité particulière à
dégrader les protéines ou les peptides durant la fermentation.
Les espèces suivantes: Lb. sanfranciscensis, Lb.
brevis 14G et les souches CRL 759 et CRL 778 du Lb.
alimentarius sont potentiellement capables d'hydrolyser la gliadine, la
fraction protéique du gluten responsable de la maladie coeliaque
(Gobbetti et al ., 2005; Rollan et
al., 2005; Di Cagno et al., 2002).
III.3.2. La production du gaz carbonique:
En panification directe, les levures de l'espèce
Saccharomyces cerevisiae sont les seuls agents de fermentation. Les
levures jouent un rôle important dans la production du gaz carbonique
provenant de la dégradation des glucides qui contribue à la
levée de la pâte (Onno et Roussel 1994). Les bactéries
lactiques homofermentaires produisent seulement de l'acide lactique. Seules les
bactéries lactiques hétérofermentaires produisent du gaz
carbonique mais comparativement aux quantités produites par les levures,
elles contribuent faiblement à la levée de la pâte (Onno et
Roussel 1994). Il a été noté que l'association du Lb.
sanfranciscensis avec S. cerevisiae et S. exiguus M14
accroissait la production du gaz carbonique en comparaison avec S.
cerevisiae seule (Gobbetti et al., 1997).
III.3.3. Les aspects aromatiques:
Les caractéristiques aromatiques du pain font le sujet
de plusieurs investigations et plus de 500 composés volatils ont
été signalés dans plusieurs types de pains (Gzerny et
Schieberle 2002). L'arôme du pain doit être attribué non pas
à un seul composé, mais à un mélange complexe de
substances aromatiques, d'origine microbienne et enzymatique.
La qualité aromatique du pain au levain dépend
du pH de la pâte et du rapport acide lactique/ acide acétique
appelé quotient fermentaire. (Czerny et Schieberle
2002 ; Rohrlich 1953).
*Le quotient fermentaire
La fermentation lactique confère aux produits finis un
goût typique du pain au levain, aigrelet par l'acide lactique et plus
piquant par l'acide acétique. La quantité de chaque acide
produite est variable d'un levain à l'autre. On appelle le quotient
fermentaire (QF) le rapport molaire des concentrations en acide lactique et
acide acétique. Le QF dépend des conditions de fermentation et
des espèces présentent dans le levain ou le starter. Pour le pain
de levain de froment (farine de blé tendre) caractéristique du
pain Français, les valeurs optimales du quotient fermentaire pour
définir un bon arôme n'ont pas été
déterminées, mais il est préconisé, à titre
indicatif, un rapport compris entre 4 et 10. Le pain allemand, plus riche en
acide acétique, a un goût acide plus prononcé que celui du
pain français. (Onno et Roussel 1994 ; Roussel et Chiron 2002)
III.3.4. L'activité
anti-microbienne:
Les bactéries lactiques ont un rôle fondamental
dans l'inhibition des flores non lactiques, dont certaines sont
préjudiciables à la qualité du pain. Cette action est due
à l'abaissement du pH (qui inhibe la croissance de la plupart des germes
non lactiques), à la toxicité propre de l'acide lactique, mais
aussi à la sécrétion de bactériocines (facteurs
bactéricides) et des substances antifongiques (Desmazeaud 1998).
-L'activité anti-bactérienne:
A côté des composés comme les acides
organiques, le peroxyde d'hydrogène et le diacétyle, les
bactéries lactiques des levains inhibent les germes indésirables
en produisant des bactériocines comme la plantaricine
libérée par Lb. plantarum ST31, des substances dites
bactériocines-like (BLIS) comme le BLISC57 de Lb.
sanfranciscensis C57 et des antibiotiques comme la reutericycline produite
par Lb. reuteri LTH2584 (Gobbetti et al., 2005
Gobbetti et al., 1997).
-L'activité antifongique:
Des substances comme l'acide phényllactique et l'acide
4-hydroxy-phényllactique isolées des souches de Lb. plantarum
21B et 20B ont montré une activité inhibitrice contre
Aspergillus, Penicillium et Monilia. Par ailleurs,
une gamme d'acides organiques tels que les acides:
acétique, formique, caproïque, propionique,
butyrique et valérique libérées par Lb.
sanfranciscensis CB1, possèdent le même effet contre
Aspergillus, Fusarium, Penicillium et
Monilia. Ces acides agissent d'une manière synergique (Gobbetti
et al., 2005 ; Lavermicocca et al.,
2003 ).
IV. Les méthodes de caractérisation
moléculaire des bactéries lactiques
Pour l'identification de la population microbienne des levains
de panification, des méthodes d'analyses basées sur les
critères phénotypiques (physiologique et biochimique) et
moléculaires (PCR-RFLP, PCR-RAPD, PCR multiplex, PCR avec primers
spécifiques des espèces) ont été utilisées
(Settani et al., 2005 ; Randazzo et al., 2005 ;
Ehrmann et al., 2003 ; Corsetti et al., 2001, Zavaleta
et al., 2001, Müller et al., 2000 ; Zapparoli
et al., 1997). Ces méthodes ont permis l'identification de la
quasi-totalité des bactéries lactiques dans les pains aux levains
(De Vuyst et al., 2002 ; Valcheva et al., 2005). Ces
méthodes sont basées sur le principe de la PCR
(polymerase chain reaction) ou technique d'amplification utilisant
l'ADN-polymérase qui permet d'amplifier in vitro une molécule
d'ADN en plusieurs millions de copies.
De même, la variabilité dans la longueur et la
séquence de la région intergénique 16S/23S ARNr ou ITS
(Internal Transcribed Spacer) rend l'étude de cette région
intéressante pour identifier la microflore lactique (Tannock et
al., 1999). Les ITS sont constitués d'une part de régions
très conservées qui sont les séquences codant pour les
gènes ARNt d'isoleucine et ARNt d'alanine mais aussi des régions
variables utilisables pour l'identification des espèces (Gürtler et
Stanisich 1996).
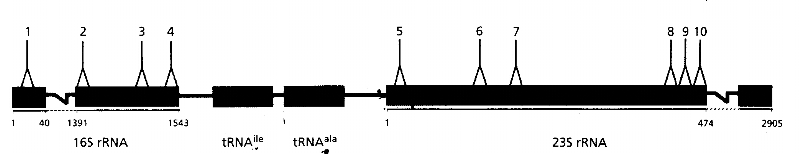
ARNr 16S
ARNtIle
ARNtAla
ARNr 23S
Figure n°1: Positions des
régions conservées de l'opéron ribosomique 16S-23S chez
Escherichia coli. Les lignes noires représentent la
région ISR qui sépare les ARNr 16S et 23S. Cette région
contient les deux gènes d'ARNt (ARNtIle et
ARNtAla) (Gürtler et Stanisich 1996).
Parmi les bactéries, il a été
démontré que la structure de l'ITS reste stable dans la limite
du genre (Kabadjova et al., 2002 ). Les ITS de
Lactobacilles se présentent sous la forme de deux copies
différentes, l'un qui comprend les gènes tARNala et
tARNile, et l'autre sans ces séquences codantes ce qui
explique qu'on observe deux fragments après une amplification de la
région 16S/23S (Nour et al., 1998) . Selon Kabadjova
et al., 2002, le genre Carnobacterium possède trois
types d'opérons ribosomiques. Les bactéries des genres
Lactococcus, Leuconostoc, Weissella, Oenococcus et
Streptococcus possèdent un seul opéron rrn avec une
structure dans l'ordre 5'-16S-seq-tAla-seq-3' qui est visualisée par un
seul produit de la réaction. (Le Jeune et Lonvaud-Funel 1997).
L'organisation de l'opéron rrn
5'-16S-23S-5S-3' est montrée par la figure suivante :
|
A. Lactococcus lactis (Nour
et al ., 1998 )
Leuconostoc oenos (Oenococcus
oeni) (Le Jeune et al., 1997 )
|
16S
23S
ARNtAla
ISR
|
|
B. Lactobacillus (Nour et
al., 1998)
|
16S
23S
ARNtIle
ARNtAla
L-ISR
16S
23S
S-ISR
|
|
C. Carnobacterium (Kabadjova
et al., 2002 ; Rachman et al.,
2004)
|
16S
23S
16S
23S
16S
23S
ARNtIle
ARNtAla
16S
23S
ARNtIle
ARNtAla
16S
23S
ARNtAla
16S
23S
ARNtAla
S-ISR A
S-ISR B
M-ISR A
M-ISR B
L-ISR A
L-ISR B
Figure n°2 : Organisation de
la région intergénique 16S-23S de l'ARNr chez les
bactéries lactiques. La région intergénique est en vert.
S-ISR : petit ISR, M-ISR : moyen ISR, L-ISR : grand ISR. A : groupe
de C. divergens, C. mobile, C. funditum, C.
alterfunditum, C. inhibens, et C. viridans ;
B : groupe de C. piscicola et C. gallinarum
La PCR- RFLP (Restriction Fragment Length Polymorphism) est
une méthode appropriée pour la différenciation des
espèces voir les souches des Procaryotes. Dans cette méthode, on
compare les profils électrophorétiques de différentes
molécules d'ADN digérées par les mêmes enzymes de
restriction. On obtient les fragments de restriction. Si les molécules
d'ADN que l'on compare sont différentes, leurs sites de restriction le
sont aussi; par conséquent, les fragments de restriction obtenus en
électrophorèse seront aussi différents (Caillez 2004). La
méthode PCR-RFLP de la région ISR a permis l'identification de
quatre espèces de Carnobacterium, de certaines souches de
Leuconostoc (Rachman et al., 2004 ; Zavaleta
et al., 1996) et des bactéries acétiques (Ruiz
et al., 2000).
Les endonucléases de restriction souvent
utilisées sont signalées dans le tableau suivant:
Tableau n°2: Exemples de quelques
enzymes de restriction et leurs séquences de reconnaissance
|
Enzymes
|
Source microbienne
|
Séquence
|
|
Hind III
|
Haemophilus influenzae b
|
5'- A - A - G - C - T - T - 3'
3'- T - T - C - G - A - A -5'
|
|
Hinf I
|
Haemophilus influenzae Rf
|
5' - G - A - N - T - C - 3'
5' - C - T - N - A - G - 3'
|
|
Taq I
|
|
5' - T - C - G - A - 3'
3' - A - G - C - T - 5'
|
Enfin, la TTGE (Temporal Temperature Gradient
Gel Electrophoresis) est une méthode qui utilise une
électrophorése en conditions dénaturantes pour
détecter la diversité, le suivi dynamique et l'activité
des communautés bactériennes dans des différents aliments
(Randazzo et al., 2005 ; Meroth et al., 2003 ;
Duthoit et al., 2003 ; Ogier et al., 2002,
Cocolin et al., 2000, Zoentendal et al.,
1998). La TTGE est une forme parallèle de la DGGE
(Denaturing Gradient Gel Electrophoresis), où un gradient de
température est appliqué au lieu du gradient chimique. La
température est la même quelque soit la position sur le gel mais
elle change en fonction du temps. Plusieurs fragments de Tm (melting
temperature) différentes peuvent donc être séparés
sur le même gel de TTGE. Au cours de l'électrophorèse,
l'ADN migre d'abord à l'état double brin, puis rencontre des
conditions qui dénaturent le domaine de fusion moins stable de la
molécule, formant une structure partiellement simple brin, dite
ramifiée. Cette structure est formée grâce
à l'utilisation de GC clamp, une séquence de
guanines (G) et cytosines (C) de 30 à 50 nucléotides,
ajoutée et attachée à l'extrémité 5' d'une
de deux amorces utilisées. Cette séquence co-amplifiée et
introduite dans les fragments amplifiés empêche la
dénaturation complète ; l'ADN double brin ne se transforme
donc pas en simple brin. Cette dénaturation partielle entraîne une
réduction de la mobilité électrophorétique de sorte
que sa position finale dans le gel dépend exclusivement de la
température de fusion (Tm) du domaine le moins stable. La
révélation des fragments sous UV est réalisée en
général en utilisant le SYBR Green I comme colorant (agent
intercalant). (Rachman 2004 ; Müyzer et Smalla 1998 ; Vasquez
et al., 2001).

Produit PCR région V6-V8 de l'ARNr 16S issu d'un
même échantillon
Figure n°3: La technique TTGE
5'
3'
Amorce 1
Amorce 2
GC clamp
30-50
nucléotides
Domaine de haute Tm
ADN ramifié
Figure n°4: Formation d'un brin
d'ADN ramifié grâce à la présence de GC clamp.
(Rachman et al., 2004)
Cette technique est utilisée dans le but
d'étudier l'écologie microbienne. L'extraction de l'ADN
bactérien est effectuée directement à partir de la matrice
à analyser. L'ADN bactérien d'une région variable V6-V8
est amplifié par PCR et est ensuite soumis à une
électrophorése dénaturante par la température. Les
fragments obtenus correspondant aux bactéries recherchées peuvent
être récupérés et séquencés afin de
confirmer leur identité. La technique TTGE permet donc d'obtenir des
empreintes électrophorétiques, donc génétiques d'un
écosystème microbien. Cette technique a été
appliquée par Meroth et al., 2003 à l'étude de
la dynamique des flores lactiques de levain de panification.
Figure n°5 : Schéma
générale de l'étude
Coloration de Gram
Test au catalase
Test homo/hétérofermentaire
Extraction d'ADN (souche Gram+, Cat-)
PCR avec les primers 16S/2 et 23S/7
2 fragments
Autres espèces de
Lactobacillus
(Kabadjova et al,2002)
PCR avec les primers tala et
23/10
à partir de l'ADN total extrait
RFLP (Hind III, Hinf I, Taq
I)
Différenciation des espèces de
Lactobacillus
Visualisation des gels sous UV
Coloration par le BET
Analyse par le logiciel
Bioprofile 1D++
Quantification de l'ADN (20 à 100
ng/ul)
3 fragments
Lb.sanfranciscensis
(Valcheva et al,2006)
10g du levain+90 ml TS
Dénombrement
(CFU/g)
Bactéries lactiques (BL) sur SDB et MRS m agar
Levures sur Sabouraud glucose agar
Echantillon
pour la détermination du pH
et du TTA
Echantillon à congéler
pour la TTGE
Isolement de 20 colonies BL
sur MRS m en bouillon
Caractérisation
phénotypique
Mise en souchier à -20°C
Caractérisation
moléculaire
Repiquage des colonies sur SDB et MRS m en
bouillon
levain BF
pour la TTGE
pour la TTGE
PCR avec primers-spécifiques
des espèces
thrthhh
I. Souches de référence
Tableau n°3: Liste des souches de
référence
|
Souches
|
Références
|
Souches
|
Références
|
|
Lactobacillus acidophilus
|
DSM 20079
|
Lactobacillus hilgardii
|
DSM 20176
|
|
Lactobacillus alimentarius
|
CIP 102986T
|
Lactobacillus panis
|
DSM 6036
|
|
Lactobacillus amylovorus
|
DSM 20531
|
Lactobacillus paralimentarius
|
DSM 13238
|
|
Lactobacillus brevis
|
DSM 20556
|
Lactobacillus plantarum
|
ATCC 14917
|
|
Lactobacillus brevis
|
DSM 1268
|
Lactobacillus pontis
|
DSM 8475
|
|
Lactobacillus curvatus
|
DSM 20019
|
Lactobacillus reuteri
|
DSM 20016
|
|
Lactobacillus delbrueckii subsp bulgaricus
|
CIP 55.1
|
Lactobacillus sanfranciscensis
|
ATCC 43322
|
|
Lactobacillus delbrueckii subsp delbrueckii
|
DSM 20074
|
Lactobacillus sanfranciscensis
|
CIP 103252T
|
|
Lactobacillus farciminis
|
DSM 20180
|
Lactobacillus frumenti
|
DSM 13145
|
|
Lactobacillus fermentum
|
DSM 20052
|
Lactobacillus casei
|
DSM 20011
|
|
Lactobacillus mindensis
|
|
Lactobacillus kimchii
|
DSM 13961
|
|
Lactobacillus spicheri
|
DSM 5753
|
|
|
DSMZ:Deutsche Sammlung Von Microorganismen
and Zelkulturen GmbH, BRAUNSCHWEIG, GERMANY, CIP: Collection
de l'Institut Pasteur, PARIS - FRANCE, ATCC: American Type
Culture Collection, ROCKVILLE, MD, USA, T: Type
strain.
II. Echantillons des levains
étudiés
Dans l'objectif d'observer l'influence de la farine
utilisée lors du renouvellement du milieu (rafraîchi), deux
levains naturels français de panification provenant de la région
du pays de la Loire ont été analysés : le levain BF
(BioFournil), utilisé par l'entreprise du même nom, avec
rafraîchi régulier sur farine témoin type 70 et ce
même levain BF après rafraîchi sur la farine du blé
concassé. Les analyses ont été effectuées à
partir de 10 g de levain de chaque échantillon dilués avec 90 ml
d'une solution de Tryptone-sel stérile. La solution sera ensuite
homogénéisée à l'aide d'un stomacher (Lab-Blender
400) et utilisée pour toutes les analyses physico-chimiques et
microbiologiques.
III. Détermination du pH et du TTA
Après mesure du pH, l'acidité titrable totale
(TTA-Total Titrable Acidity) est dosée à l'aide d'une solution de
soude N/10 pour 10 ml de la solution initiale du levain. Le volume de soude
nécessaire pour atteindre la valeur de pH de 8,5 est mesurée et
le résultat est exprimé en ml/NaOH N/10/10 g de levain.
IV. Milieux de culture
IV.1. Milieux de dénombrement des
levures
Le dénombrement des levures est effectué sur
milieu Sabouraud glucose agar avec du chloramphénicol (Biokar, France).
Toutes les boîtes sont ensemencées en surface avec 0,1ml et
incubées à 25°C pendant 48 heures en aérobiose.
IV.2. Milieux de dénombrement, d'isolement
et d'identification des bactéries lactiques
Le milieu MRS agar modifié (1% de maltose, 0,5% de
fructose, 0,5g/l de cystéine et 5 ml d'extrait de levure fraîche)
à pH final de 6,2 et le milieu SDB (SourDough Bacteria) sont
utilisés pour l'analyse des échantillons et la culture des
souches de référence. (Kline et Sugihara 1971).
IV.3. Conservation des souches et des
isolats:
Les souches ainsi que les isolats sont conservés
à -20 °C dans un mélange de bouillon MRS modifié et
glycérol à 40% (V/V) pour les bactéries lactiques (Biokar,
Beauvais, France).
V. Dénombrement et isolement des
bactéries lactiques
Après dilutions décimales de la solution
mère dans une solution de Tryptone-sel stérile jusqu'à
10-6. 0,1 ml des dilutions 10-5 et
10-6 seront déposés sur les milieux cités
précédemment. Les dénombrements se feront sur les milieux
MRS modifié agar et sur SDB agar après 72 heures d'incubation et
à 30°C dans des conditions d'anaérobiose (anaerocult). 20
colonies sont isolées du milieu MRS modifié en bouillon pour
l'identification des bactéries lactiques.
VI. Caractérisation des bactéries
lactiques
VI.1.Caractérisation
phénotypique
1- La morphologie des colonies et la coloration de Gram
L'observation microscopique est nécessaire pour
définir la forme des cellules bactériennes (coques ou bacilles).
La coloration de Gram des isolats après 72 h de culture sur MRS
modifié permet de s'assurer que nous sommes effectivement en
présence des bactéries gram positive (coloration violette).
2- Le test de catalase
La catalase est une enzyme contenant du fer, elle catalyse la
décomposition du peroxyde d'hydrogène en eau et en
oxygène. Le test de détection de cette enzyme dans une souche
bactérienne, consiste à ajouter du peroxyde d'hydrogène
aux cellules bactériennes, la présence de la catalase se marque
par la formation des bulles de gaz (oxygène).
3- Le test homo/hétérofermentaire
La souche est cultivée dans un bouillon MRS
modifié en bouillon. Le dégagement du gaz carbonique retenu par
la cloche de Durham met en évidence le caractère
hétérofermentaire.
(Sriranganathan et al., 1973).
VI.2.Caractérisation
moléculaire
1- Extraction de l'ADN avec le Kit Qiagen
1 ml de la culture bactérienne en phase exponentielle
de croissance est centrifugé à 7500 rpm pendant 10 minutes.
Après avoir éliminé le surnageant, le culot
bactérien est resuspendu avec 180ul de tampon de lyse (20mM Tris-HCl pH
8 ; 2mM EDTA ; 1,2% Triton X-100 ; 20 mg/ml lysozyme) suivi
d'une incubation à 37°C pendant une heure. 25ul de
protéinase K et 200ul de tampon AL sont ajoutés pour
dégrader toutes les protéines après une incubation
à 70°C pendant 30 minutes. Cette étape est suivie par
l'addition de 200 ul d'éthanol absolu. Le mélange est ensuite
transvasé sur une colonne, puis centrifugé à 8000 rpm
pendant une minute. L'éluant est éliminé et l'ADN est
récupéré sur une colonne dans un tube de 2 ml à
lequel on ajoute 500 ul de tampon AW1. On centrifuge à 800rpm pendant
une minute et on jette l'éluant et le tube. De nouveau, l'ADN est
récupéré dans un tube de 2 ml à lequel on ajoute
500 ul de tampon AW2. On centrifuge à 15 000 rpm pendant 3 minutes
et on jette l'éluant et le tube. On place la colonne sur un tube de
1,5ml et on ajoute cette fois-ci 100 ul de tampon AE directement sur la
membrane. On incube à température ambiante pendant une minute et
on centrifuge à 8 000 rpm pendant une minute. Cette démarche
est renouvelée puis, on jette la colonne et on conserve l'ADN pur
extrait à -20°C.
2- Quantification de l'ADN
Le but est d'obtenir une concentration d'ADN entre 20 et 100
ng/ul pour chaque échantillon avant de réaliser le PCR. La
quantification se fera grâce au spectrophotomètre UVIKON BIOTEK.
La concentration d'ADN sera calculée selon la formule suivante:
D.O à 260 nm x F x A
[ADN] ug/ ul =
---------------------------
1000
Où:
F= facteur de
dilution =10
A= Absorption
ADN= 50; Absorption ARN= 40
3- Les amorces
Les amorces oligonucléotidiques utilisées dans
cette étude sont obtenues d'Invitrogen
Tableau n°4 : Les amorces de
l'étude
|
Code
|
Localisation
|
Séquences oligonucléotidiques
Primers (5'?3')
|
T (°C)
Hybridation
|
Références
|
|
16S/2
|
16S
1389-1407 de E.coli
|
CTT GTA CAC ACC GCC CGT C
|
60°C
|
Gürttler et Stanisich 1996
|
|
23S/10
|
23S ARNr
456-474 de E.coli
|
CCT TTC CCT CAC GT ACT G
|
60°C
|
Idem
|
|
23S/7
|
23S ARNr
188-208 de E.coli
|
GGT ACT TAG ATG TTT CAG TTC
|
60°C
|
Idem
|
|
ARNtala
|
ITS
|
TAG CTC AGC TGG GAG AGC
|
60°C
|
Idem
|
|
Ls1
|
|
AAGTCGCCCAATTGATTCTTAGT
|
65°C
|
Zapparoli et al., 1997
|
|
Ls2
|
|
TTCACCCTAATCATCTGTCCCA
|
65°C
|
Zapparoli et al., 1997
|
|
TGGE (L1401)
|
V6-V8
|
GCG TGT GTA CAA GAC CC
|
56°C
|
Zoetendal et al., 1998
|
|
TGGE
(U968-GC)
|
V6-V8
|
CGC CCG GGG CGC GCC GGG GAA CGC GAA GAA CCT TAC
|
56°C
|
Vasquez et al., 2001
Zoetendal et al., 1998
|
4- La PCR
Ø Avec les primers 16S/2 et
23S/7
Le mélange final pour les réactions de PCR a un
volume de 25 ul et contient 1x PCR Mix du tampon Taq polymérase
sans MgCl2, 2,5 mM MgCl2 (Biolabs, New England),
1uM pour chaque amorce (Invitrogen), 0,25 mM pour chaque dNTP
(Eurogentec), 1U de Taq polymérase (Biolabs,
New England) et 20-50ng/ml d'ADN. La réaction du PCR s'effectue dans le
thermocycleur PTC-100 (MJ Research Inc., Watertown MA, USA). L'amplification
des ISRs comprend 35 cycles : 1 minute de dénaturation à
94°C, 1 minute d'annealing ou d'hybridation à 60°C et 1
minute d'extension à 72°C.
Ø Avec les primers tRNAala et
23S/10
La composition du mélange PCR et les conditions
d'amplifications sont les mêmes que celles qui sont décrites
ci-dessus. Cette deuxième réaction d'amplification est faite
après l'obtention des résultats de la première
amplification (présence de 2 ou 3 fragments).
Ø Avec des primers spécifiques des
espèces
La composition du mix PCR est la même que
précédemment. L'amplification des ISRs comprend 35 cycles avec
un changement au niveau de la température d'hybridation des amorces qui
est de 65°C.
Ø PCR sur colonies
C'est une méthode rapide et efficace pour la
détection et l'identification de la population des levains de
panification. Des colonies de diamètre compris entre 1-1,5 mm ont
été choisies et mélangées avec 5 ul de l'eau
stérile. Cette suspension sera incubée 5 minutes à
95°C. On centrifuge à l'aide d'une micro-centrifugeuse et 1-3,5 ul
sont utilisés pour la réaction de PCR. Les conditions
d'amplifications sont les mêmes que celles qui sont décrites plus
loin et la composition du mélange PCR reste le même à
laquelle on ajoute 0,1 x BSA.
5- L'électrophorèse sur le gel d'agarose
Les molécules placées dans un champ
électrique migrent vers l'anode. Dans le gel, la mobilité
électrophorétique dépend de la taille des molécules
et de leur conformation. Les molécules de forme linéaire migrent
d'autant plus vite qu'elles sont de petites tailles. Les produits
d'amplifications seront séparés sur un gel
d'électrophorèse à 1,5% d'agarose dans une solution tampon
TAE colorée au BET. La migration se fait sous un voltage de 100V.
L'observation de chaque gel se fera sur le transilluminateur à UV
grâce à la fluorescence sous UV du complexe ADN-UV. Afin de
déterminer la taille de chaque fragment, le marqueur de poids
moléculaire 100 pb DNA ladder sera utilisé.
1517 pb
1200 pb
1000 pb
900 pb
800 pb
700 pb
600 pb
500 pb
400 pb
300 pb
200 pb
100 pb

Figure
n°6 : Le marqueur 100 pb DNA Ladder-Biolabs
6- La restriction enzymatique (RFLP)
Les produits du PCR avec les primers tRNAala -
23S/10 sont digérés par des enzymes de restriction. Les enzymes
utilisées sont : HindIII, HinfI et
áTaqI (Biolabs). La réaction de enzymatique se
réalise avec un volume final de 20 ul contenant 1x du tampon 0,1 x BSA,
10U de l'enzyme et 16 ul du produit du PCR. Les produits de digestion sont
séparés sur un gel d'électrophorèse dans 2%
(wt/vol) agarose-résophore dans le TBE comme tampon (1x). Les gels sont
colorés par 0,5 ug/ml de BET et visualisés sous UV. Les
photographies sont prises par une vidéo caméra (Sony, Clichy,
France) et sauvegardés. Ils seront analysés par le logiciel Bio
Profile 1D++ (Vilbert Lourmat, Marne La Vallée, France).
TTGE
Ø Extraction et purification de l'ADN total
bactérien
L'extraction de l'ADN total bactérien est décrit
par Meroth et al., (2002) avec des petites modifications. 10g de
levain sont homogénéisés pour 5 minutes par un stomacher
dans un sac contenant 90 ml d'une solution de tryptone-sel. Un aliquote (50 ml)
est centrifugé à 4°C pour 5 minutes à 100 rpm. Le
surnageant est centrifugé pour 15 minutes à 4000 rpm et le culot
cellulaire est stocké à -20°C. Le culot est ensuite
déposé sur glace et lavé 3 fois par 1 ml d'une solution
tampon de phosphate saline et une fois par 1 ml de l'eau. Puis on le resuspend
dans 130 ul d'un tapon lyse ( 6 ,5% sucrose, 50mM Tris HCl pH 8, 0,1 mM
EDTA, 20 mg/ml lysozyme et 100ug/ml RNase). Après une incubation d'une
heure à 37°C, on ajoute 30 ul du NaCl (5M) et le tout est
incubé pour 10 minutes à 65°C. Ensuite, on ajoute
successivement 20 ul de protéinase K (15mg/ml) et 10 ul de sodium
dodécyle sulfate (20%). Le nouveau mélange est incubé
pendant 30 minutes à 60°C. Enfin, 200 ul de phénol
(65°C, pH =7) ont été ajoutés à la mixture et
incubée pour 6 minutes à 60°C. Après le
refroidissement sur glace, on procède 2 fois à l'extraction et
avec des volumes égales de phénol-chloroforme-isoamyl alcool
(25 :24 :1) et 2 fois par du chloroforme. Après la
précipitation par de l'éthanol, l'ADN est gardé dans 100ul
de Tris-HCl (10 mM, pH =8).
La PCR-TTGE est obtenue en utilisant les deux primers
amplifiant les régions variables V6-V8 du gène ARNr. La PCR est
effectuée dans un volume total de 50 ul contenant 1x tampon PCR sans le
chlorure de magnésium, 2,5 mM MgCl2, 1 ug/ml ADN, 0,3 uM pou
chaque primer, 0,25 mM dNTP et 1U de Taq polymérase. On a les
mêmes conditions des PCR précédentes. Les produits du PCR
sont séparés sur 1,5 % (wt/vol) du gel d'agarose (Eurobio,
France) et consécutivement visualisés sous la lumière UV
après une coloration par le BET.
Ø Les analyses du TTGE
10 ul du produit amplifié par PCR sont analysés
par la TTGE sur le gel du polyacrylamide (composition et préparation
dans l'Annexe 1). L'analyse du TTGE est effectuée en utilisant DCode
Universal Mutation Detection System (Bio Rad, Marnes la coquette, France). Le
gel du polyacrylamide 16 cm/16cm/1 mm est utilisé avec le tampon 1x TAE
(2M tris base, 1M acide acétique glacial, 50mM EDTA). Les gels ont
été préparés avec 11 % (wt/vol) de la solution
d'acrylamide (acrylamide-bisacrylamide ; 37,5 :1) et une
concentration d'urée finale de 9,7M. Pour séparer les
bactéries étudiées, les conditions suivantes ont
été admises : voltage -55V, température initiale
-65,5°C, température finale - 70,5°C, gradient de
température 0,2°C/h et le temps de migration est de 18 heures. 10
ul du produit de PCR sont introduites dans les pores. En outre, le gel est
coloré pour 15 minutes par le colorant SYBR Green I
(Molecular Probes, Eugene OR, USA) et photographié sur
la table disposant d'un transilluminateur à UV.
L'objectif de cette étude est de montrer l'influence de
la matière première mise en oeuvre sur la flore lactique d'un
levain et notamment sur la diversité des espèces du genre
Lactobacillus. Pour cela deux échantillons de levains
ont été analysés : BF -70 entretenu sur
farine de type 70, et BF-FC rafraîchi sur farine de
blé concassé.
I. Détermination du pH et du TTA
Les résultats sont consignés dans le tableau
n°5 :
Tableau
n°5 : pH et TTA des échantillons des levains
BF
|
Echantillon
|
pH
|
TTA ml/N/10/10g
|
|
BF-70
|
4.03
|
8.04
|
|
BF-FC
|
4.08
|
9.30
|
L'acidité plus élevée du levain BF-FC
peut-être mise en relation avec le taux de cendres supérieur et la
quantité plus importante de parties périphériques du grain
dans le cas du blé concassé.
II. Identification des bactéries lactiques des
levains Français
II.1. Dénombrement des
bactéries lactiques et des levures :
Les dénombrements sont effectués sur les milieux
MRS modifié agar et SDB agar pour les bactéries lactiques et sur
le milieu Sabouraud glucose agar avec chloramphénicol pour la flore
levurienne (Tableau n°6).
Tableau n°6:
Dénombrement des levures et des bactéries lactiques dans les
levains BF
|
Echantillons
|
Bactéries lactiques
|
Levures
|
|
MRS m
UFC/g
|
SDB UFC/g
|
Sabouraud glucose agar
UFC/g
|
|
BF-70
|
1,4.109
|
5,1.109
|
1,5 .108
|
|
BF-FC
|
1,9.109
|
3,1.109
|
1,5.108
|
La population bactérienne observée sur SDB agar
est plus importante que sur MRS modifié agar. Les colonies sont plus
petites sur SDB agar et elles présentent pratiquement la même
morphologie. Les populations obtenues ne présentent pas de
différences significatives entre les deux levains analysés.
Selon les analyses antérieures sur ce levain, Lb.
sanfranciscensis est la flore lactique dominante du levain BF. Le milieu
SDB, de part sa composition (pH qui vaut 5,6 et présence d'extrait frais
de levure) est favorable à la croissance et au dénombrement cette
espèce bactérienne (Kline et Sugihara, 1971). A l'inverse, le
milieu MRS m est moins favorable et ne permet pas le dénombrement de la
totalité des flores présentes dans le levain.
II.2. Identification phénotypique des
bactéries lactiques :
Les étapes d'identification consistent à
réaliser les différents tests phénotypiques comme la
coloration de Gram, le test catalase et le test
homo-hétérofermentaire.
A partir du milieu MRS m, 20 isolats de chaque
échantillon ont été purifiés et soumis à la
coloration de Gram et au test catalase. Les 20 isolats provenant de la farine
BF-70 sont des bacilles Gram positifs, catalases négatives, 18 sont
hétérofermentaires et 2 homofermentaires. Sur la farine
concassée, le même travail a été entrepris. Les 20
isolats obtenus sont des bacilles Gram positif, catalase négative et
tous sont hétérofermentaires.
Tableau n°7 : Ratio
des isolats homo/hétérofermentaire dans les levains
|
Echantillons de levains
|
Bacilles hétérofermentaires
|
Bacilles homofermentaires
|
|
BF-70
|
98 %
|
2%
|
|
BF-FC
|
100 %
|
0 %
|
II.3. Identification moléculaire des
bactéries lactiques :
II.3.1. Identification des isolats du
BF-70 :
L'amplification par PCR avec les amorces 16S/2-23S/7 des 16
isolats hétérofermentaires du BF-70 montre un profil de 3 bandes
sur le gel d'agarose. Ce profil correspond à l'espèce Lb.
sanfranciscensis type de notre étude. En effet selon Valcheva
et al., 2006 la présence de ces 3 fragments est uniquement
observée chez Lb. sanfranciscensis alors que toutes les autres
espèces de Lactobacillus présentent seulement 2 types
d'opérons ribosomiques.
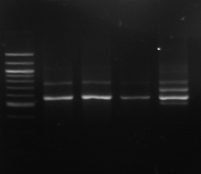
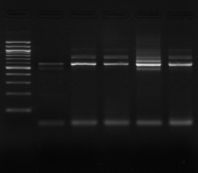
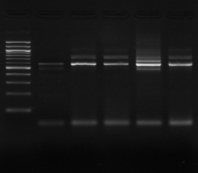
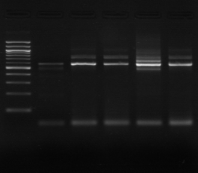
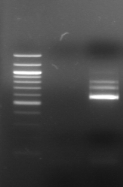
M 1 2 3 4 M
M1 M2 M3 M4 M5 M6 M7
M M8 M9 M10 M11 M M12 M13
M M14 M15 M16
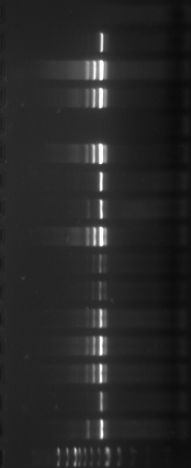
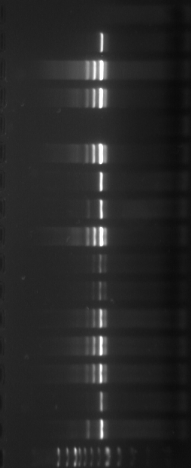
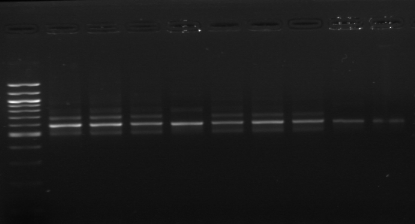
Figure n°7: PCR avec les amorces
16S/2-23S/7 des 16 isolats de BF-70.
M-marqueur 100 pb DNA ladder (biolabs) et
les numéros 1 à 4 correspondent à
l'amplification des souches de
référence utilisées dans cette étude :
1-Lb.
paralimentarius, 2-
Lb.spicheri,3-Lb. kimchii, 4-Lb. sanfranciscensis
ATCC
43322.
M1 à M16 : isolats BF-70.
Après avoir déterminé de manière
probable l'espèce qui domine dans l'échantillon BF-70, nous avons
confirmé ce résultat en procédant à
l'amplification de la région tRNAala -23S/10 par les amorces
correspondantes à cette séquence nucléique dans le but de
faire ensuite la restriction enzymatique .On observe donc la présence
d'un seul fragment d'amplification sur la figure n°8 :
M R M1
M8 M16
740 pb
Figure n°8: Amplification par
les amorces tRNAala -23S/10 (souches : 1,8 et 16)
M-marqueur 100 pb DNA
(Biolabs).
R : souche de
référence Lb. sanfranciscensis ATCC 43322.
On observe ainsi que toutes les souches présentent un
seul fragment de taille identique de 740 pb environ. L'étape suivante va
consister à générer des profils de restriction en
utilisant des enzymes permettant de différencier Lb.
sanfranciscensis. Les 3 enzymes choisis dans notre étude
sont : Hind III, Hinf I et á Taq I.
M M
M M
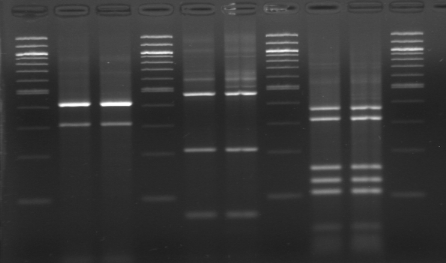
Figure
n°9: RFLP par Hind III, Hinf I et á Taq I
Pour tous les isolats il génère le même
profil connu et caractéristique pour Lb. sanfranciscensis ATCC
44322.
Seul l'enzyme á Taq I donne un profil unique pour cette
souche de Lb. sanfranciscensis ATCC 43322. Les tailles des fragments
digérés par l'enzyme á Taq I sont les suivantes :
110,140, 160, 310 et 380 pb.
Les autres enzymes génèrent des profils communs
à Lb. paralimentarius DSM13238, Lb. alimentarius CIP
102986T et Lb. delbrueckii subsp. delbrueckii DSM 20074.
Les tailles des fragments des fragments digérés par Hinf I
sont : 60, 210 et 470 pb et par Hind III sont : 320 et 420 pb.
La taille des fragments a été obtenue à
l'aide du logiciel Bio Profile 1D++ (Vilbert Lourmat). Lors de cette
étude, nous avons pris les valeurs moyennes arrondies de tous les essais
par souche et par enzyme de restriction. L'intervalle de confiance
estimé à environ 7% pour chaque fragment à l'aide du
logiciel Bio 1D++.
Pour la confirmation finale des résultats on a choisi
de faire la PCR avec des primers spécifiques de Lb.
sanfranciscensis (Zapparoli et al., 1997) sur les isolats. Elle
permet l'identification définitive de Lb. sanfranciscensis
ATCC 43322 pour les isolats étudiés comme le montre la figure
n°10 où on oberve un seul fragment de 1460 pb spécifique
de cette espèce (Zapparoli et al., 1997) .
M R
M1 M8 M16
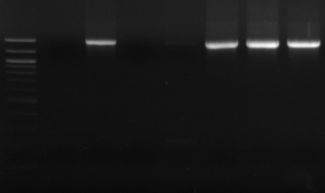
Figure n°10: PCR avec
primers spécifiques pour Lb. sanfranciscensis
M- marqueur 100 pb DNA ladder
(Biolabs)
R : souche de
référence
M1, M8 et M10 : isolats BF-70.
II.3.2. Identification des isolats du
BF-FC :
On a procédé tout d'abord à
l'amplification de la région ITS par les amorces 16S/2-23S/7. Sur 20
isolats, seules 12 souches ont été amplifiées. Les isolats
proviennent du milieu MRS modifié.
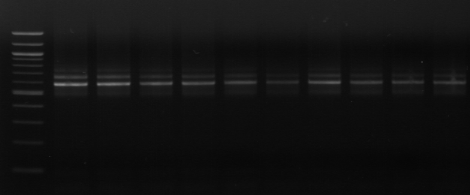
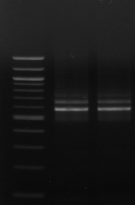
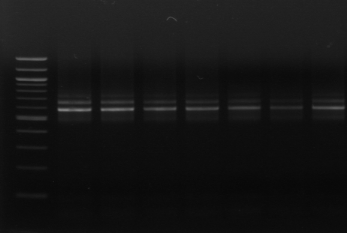
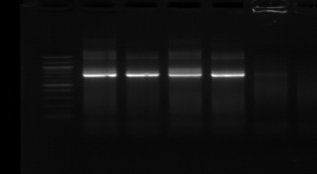
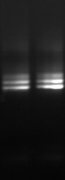
M M1 M2 M3 M4 M7 M9 M10 M11
M12 M15 M M18 M20
Figure n°11: PCR avec
les amorces 16S/2-23S/7 de 12 isolats de BF-FC
On recueille un profil de 3 bandes pour toutes les souches
étudiées (figure n°11). Lb. sanfranciscensis
représenterait encore une fois 100% de la flore lactique de la farine
concassée. On procède aux mêmes démarches que celles
utilisées pour l'étude de la flore lactique de BF-70.
Après une amplification de la région
tRNAala -23S/10 et une restriction enzymatique par les enzymes de
restriction utilisés pour les isolats du BF-70, on observe sur la figure
n°12 pour l'isolat M9 choisi comme exemple que celui-ci comme les autres
isolats de ce levain présente un profil de restriction identique
à celui de Lb. sanfranciscensis ATCC 44322. .
PCR Hinf I
Hind III á Taq I




Figure n°12: PCR
tala/10 et RFLP par Hinf I, Hind III et á Taq I de l'isolat
M9
En outre, pour confirmer que les isolats appartiennent
à l'espèce Lb. sanfranciscensis, une PCR avec les
amorces spécifiques a été faite. La figure n°13
ci-dessous montre le résultat d'amplification des 3 isolats avec les
amorces spécifiques.
R
M1 M9 M20
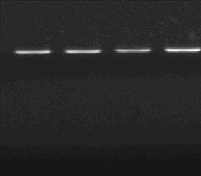
1460 pb
Figure n°13 : PCR avec
primers spécifiques de l'espèce Lb. sanfranciscensis
ATCC 43322
R : souche de référence Lb.
sanfranciscensis ATCC 43322 et les isolats M1,
M9 et M20.
II.3.3.PCR sur colonies:
Les approches classiques pour la caractérisation et
l'identification des bactéries lactiques des levains prennent beaucoup
de travail et du temps.
Dans cette étude, nous avons essayé de
développer la PCR sur colonie comme méthode pour la
détection rapide de la population des bactéries lactiques.
Différents protocoles avec différentes modifications ont
été employés. Les petites colonies ont été
sélectionnées et inoculées dans 5 ul de l'eau
stérile. Ensuite elles seront incubées pendant 5 minutes à
95°C et 2 à 3 ul de la suspension est utilisée pour y
être amplifiée. Des modifications au niveau du mix PCR ont
été faites.
Cette méthode permet de détecter rapidement la
population et le pourcentage de Lb. sanfranciscensis dans
l`échantillon de levains. Dans notre cas, la PCR sur colonies a
confirmé que ces isolats provenant des deux échantillons
appartenaient à l'espèce Lb. sanfranciscensis.
M 1 2 M
3 M 4 5 6
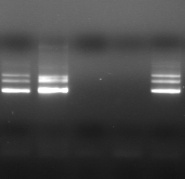


M 7 8 M 9
M 10
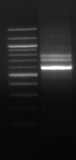
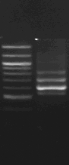
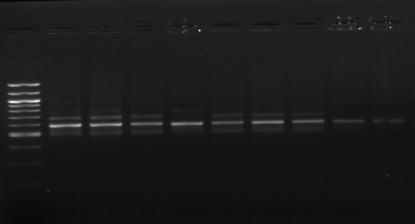
Figure n°14:
Amplification ITS avec la PCR sur colonies
M-Marqueur DNA ladder
(Biolab)
1 à 5 colonies de
BF-70 et 6 à 10 colonies de BF-FC
III. La TTGE
Cette technique est choisie pour étudier le suivi
dynamique des communautés bactériennes. Ainsi, chaque
communauté est représentée par un profil où chaque
bande électrophorétique correspond à une espèce
définie. L'identification des espèces se fait grâce
à un référentiel préalablement établi
à partir de fragments d'ADN de souches pures.
L'extraction d'ADN bactérien est effectuée
directement de la matrice à analyser. Dans notre cas on a choisi
à amplifier la région V6-V8 du 16S rDNA des deux
échantillons.
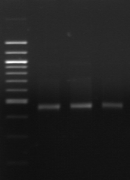
M R
1 2
Figure
n°15 : Amplification de la région V6-V8 du 16S
rDNA.
M: Marqueur 100 pb
DNA ladder (Biolab)
R: souche de
référence Lb. sanfranciscensis ATCC 44322
1- BF-70
2-BF-FC
1 2
3 4

Figure n°16 : TTGE.
1- 3 :Lb. sanfranciscensis ATCC 43322
2- BF-70 4-BF-FC
Le gel ci-dessus montre le profil des échantillons BF. Les
fragments correspondent 100 % à Lb. sanfranciscensis ATCC
44322.
Il faut noter que la TTGE ne permet pas d'identifier les
populations sous-dominantes, qui peuvent avoir un intérêt.
Ce projet avait pour objectifs : d'une part de montrer
l'influence de la matière première mise en oeuvre sur la flore
lactique d'un levain et notamment sur la diversité des espèces de
Lactobacillus et d'autre part de valider les méthodes
moléculaires de caractérisation des flores lactiques. Les
principaux résultats obtenus sont les suivants :
- L'acidité plus élevée obtenue à
partir du levain BF-FC est intéressante.
- Les populations obtenues ne présentent pas de
différences significatives entre les deux levains analysés.
- Les 20 isolats provenant de la farine BF-70
sont des Lactobacillus, 18 d'entre eux sont
hétérofermentaires et 2 homofermentaires. Sur la farine
concassée, les 20 isolats obtenus sont des Lactobacillus
hétérofermentaires.
- L'amplification par PCR avec les amorces
16S/2-23S/7 a mis en évidence la présence de 3 fragments
d'amplification et a conduit à l'identfication présumée de
Lb. sanfranciscensis pour les 16 isolats
hétérofermentaires du BF-70 et les 12 isolats BF-FC .
- L'étape suivante (RFLP) consistant
à générer des profils de restriction en utilisant des
enzymes Hind III, Hinf I et á Taq I a permis d'identifier
spécifiquement Lb. sanfranciscensis et l'enzyme
Taq I s'est révélée être la plus
discriminante.
- Enfin l'utilisation des des primers
spécifiques de Lb. sanfranciscensis (Zapparoli et al.,
1997) a permis de conclure que tous les isolats
hétérofermentaires appartenaient à l'espèce
Lb. sanfranciscensis .
En conclusion, dans les levains de panification
réalisées à partir de la farine concassée, la flore
lactique semble être constituée uniquement de l'espèce
Lb. sanfranciscensis alors que la biodiversité
bactérienne semble être plus importante dans les levains
fabriqués à partir de la farine BF-70 (une faible pourcentage de
lactobacillus homofermentaires).
Les travaux futurs viseront à :
? Compléter l'étude de la biodiversité
des flores lactiques des levains français de panification en
identifiant les espèces de bactéries non encore
identifiées par d'autres méthodes moléculaires telles
que la PCR multiplex et la RAPD.
? Valider les méthodes moléculaires de
caractérisation et de détection des flores
céréaliers par PCR-RFLP et PCR spécifiques sur les
régions intergéniques 16S-23S sur un échantillon plus
large de levains et constituer une banque de profils avec les souches de
référence et les souches sauvages des levains.
? Contribuer à une meilleure connaissance de la
dynamique des écosystèmes par des méthodes
moléculaires cultures indépendantes comme la TTGE.
? Utiliser des souches pour reconstituer des levains pour
obtenir des propriétés technologiques et nutritionnelles
intéressantes.
? Analyser quantitativement les populations
bactériennes par des méthodes moléculaires comme la PCR
quantitative (Real Time PCR).
REFERENCES BIBLIOGRAPHIQUES
|
1- Brevas E., 1991. Mise au point des levains
bactériens pour la panification. Thèse de doctorat.
Université Blaise Pascal (Clermont II). 179 pages.
2- Brochoire G., Netter A., Onno B., Rio I., Roussel
Ph., Stephan C., 1996. Les nouvelles de la boulangerie
pâtisserie, supplément technique.
3- Cai Y., Okada H., Mori H., Benno Y., Nakase T.,
1999. Lactobacillus paralimentarius sp. nov.,
isolated from sourdough. International Journal of Systematic
Bacteriology, 49: 1451-1455.
4- Caillez J-C 2004 La biologie
moléculaire, 1ère édition, Ellipses-238p.
5- Cocolin L., Manzano M., Cantoni C., Comi
G., 2000. Development of a rapid method for the identification of
Lactobacillus ssp. isolated from fermented Italian sausages using a
polymerase chain reaction-temperature gradient gel electrophoresis. Letters in
Applied Microbiology, 30:126-129.
6- Corsetti A., Lavermicocca P., Morea M., Tosti N.,
Gobbeti M., 2001. Phenoypic and molecular identification and
clustering of lactic acid bacteria and yeasts from wheat (species Triticum
durum & Triticum aestivum) sourdoughs of Southern Italy.
International Journal of Food Microbiology, 64: 95-104.
7- Czerny M., Schieberle P., 2002. Important
aroma compounds in freshly ground wholemeal and white wheat
flour-identification and quantitive changes during sourdoughs fermentation.
Journal of Agricultural and Food Chemistry, 50: 6835-6840.
8- Desmazeaud M., 1998. Bactéries
lactiques et qualité des fromages. Laboratoire de recherches
laitières, INRA.
9- DeVuyst L., Schrijvers V., Paramithiotis S., Hoste
B., Vancanneyt M., Swings J., Kalantzopoulos G., Tsakalidou E., Messens W.,
2002. The biodiversity of lactic acid bacteria in Greek traitionnal
wheat sourdoughs is reflected in boh composition and metabolite formation.
Applied & Environnemental Microbiology, 68, N°12:
6059-6069.
10- Di Cagno R., De Angelis M., Lavermicocca P., De
Vincenzi M., Giovannini C. Faccia M., Gobbetti M., 2002. Proteolysis
by sourdough lactic acid bacteria: Effects on wheat flour protein fractions and
gliadin peptides involved in human cereal intolerance. Applied and
Environnemental Microbiology, 68, N°2: 623-633.
11- Duthoit F., Godon J-J., Montel M-C.,
2003. Bacterial community dynamics during production of registered
designation of origin salers cheese as evaluated by 16S rRNA gene single-strand
conformation polymorphism analysis. Applied & Environmental Microbiology,
69, N°7: 3840-3848.
12- Ehrmann M., Müller M., Vogel R.,
2003. Molecular analyses of sourdough reveals Lactobacillus
mindensis sp. nov., .International Journal of Systematic and
Evolutionary Microbiology, 53:7-13.
13- Gobbetti M., Corsetti A., 1997.
Lactobacillus sanfrancisco a key sourdough lactic acid bacterium. Food
Microbiology, 14: 175-187.
14- Gobbetti M., De Angelis M., Corsetti A., Di Cagno
R., 2005 Biochemistry and physiology of sourdough lactic acid
bacteria. Trends in Food Sciences and Technology, 16:57-69.
15- Gobbetti M., Smacchi E., Corsetti A.,
1996. The proteolytic system of Lactobacillus sanfrancisco
CB1: purification and characterization of a proteinase, a dipeptidase, and an
aminopeptidase. Applied and Environmental Microbiology, 62,
N°9: 3220-3226.
16- Gürtler V., Stanisich VA., 1996. New
approaches to typing and identification of bacteria using the 16S-23S rDNA
spacer region. Microbiology, 142: 3-16.
17- Ignatova M. 2005.
Identification moléculaire des levains de panification. Mémoire
de stage. ENITIAA. 41p.
18- Kabadjova P., Dousset X., Le Cam V., Prevost H.,
2002. Differentiation of closely related Carnobacterium food
isolates based on 16S-23S ribosomal DNA Intergenic Spacer Region polymorphism.
Applied and Environmental Microbiology, 68, N°11:
5358-5366.
19- Kitahara M., Sakata S., Benno Y., 2005.
Biodivesity of Lactobacillus sanfranciscensis strains isolated from
five sourdoughs. Letters in Applied Microbiology,
40:353-357.
20- Kline L., Sugihara T.F., 1971.
Microorganisms of the San Francisco sourdough bread process. Applied
Microbiology, 21, N°3: 439-465.
21- Larpent J-P., 1992. La microbiologie de
la fermentation panaire. Agro-alimentaire Information CDIUPA. Université
Blaise Pascal-Clermont II.
22- Lavermicocca P. Valerio F., Visconti A.,
2003. Antifungal activity of phenyllactic acid against molds isolated from
bakery products. Applied and Environmental Microbiology, 69,
N°1: 634-640.
23- Le Jeune C., Lonvaud-Funel A.,
1997. Sequence of DNA 16S/23S spacer region of Leuconostoc
oenos (Oenococcus oeni): application to strain differentiation.
Res. Microbiol., 148:79-86.
24- Meroth C., Walter J., Hertel C., Brandt M., Hammes
W., 2003. Monitoring the bacterial population dynamics in sourdough
fermentation process by using PCR-denaturing gradient gel
electrophoresis. Applied and Environmental Microbiology, 69,
N°1: 475-482.
25- Müller M., Ehrmann M., Vogel R.,
2000. Lactobacillus frumenti sp. nov., a new lactic acid
bacterium isolated from rye-bran fermentations with a long fermentation period.
International journal of systematic and Evolutionary Microbiology,
50: 2127-2133.
26- Müller M., Ehrmann M., Vogel R.,
2000. Multiplex PCR fort he detection of Lactobacillus pontis and two
related species in a sourdough fermentation. Applied and Environmental
Microbiology, 66, N°5: 2113-2116.
27- Müyzer G., Smalla K., 1998.
Application of denaturing gel electrophoresis (DGGE) and temperature gradient
gel electrophoresis (TGGE) in microbial ecology. Antoine Van
Leuwenhoek, 73: 127-141.
28- Nour M., 1998. 16S-23S and 23S-5S
Intergenic spacer regions of lactobacilli: nucleotide sequence, secondary
structure and comparative analysis. Res. Microbiol. 149:
433-448.
29- Oheix N., 2003. Caractérisation
moléculaire des bactéries lactiques des levains de panification.
Mémoire d'ingénieur. ENITIAA. 81p.
30- Onno B., Roussel P., 1994. Technologie et
microbiologie de la panification au levain. Bactéries lactiques Volume
II. Edition Lorica. Pages 293-321.
31- Ogier J-C., Son O., Gruss A., Tailliez P.,
Delacroix-Buchet A., 2002. Identification of the Bacterial Microflora
in Dairy Products by Temporal Temperature Gradient Gel Electrophoresis. Applied
and Environmental Microbiology, 68, N°8: 3691-3701.
32- Rachman C., 2004. Développement
d'outils moléculaires d'identification et d'analyse de la
biodiversité de l'écosystème bactérien du saumon
fumé. Thèse de Doctorat. ENITIAA. 230 p.
33- Rachman C., Kabadjova P., Valcheva R., Prevost H.,
Dousset X., 2004. Identification of Carnobacterium species by
restriction fragment length polymorphism of the 16S-23S rRNA gene intergenic
spacer region and species-specific PCR. Applied and Environmental Microbiology,
70, N°8: 4468-4477.
34- Randazzo C.L., Heilig H., Giudici P., Caggia C.,
2005. Bacterial population in traditionnal sourdough evaluated by
molecular methods. Journal of Applied Microbiology, 99,
251-258.
35- Rohrlich M., 1953. Beiträge zur
kenntnu der biologie und biochemider sauerteiggärung II Mit: Untersuchum
gen uber denbiochemischen Mechanismus der Barkteriellen sauerteiggärung,
z. lebens. Unkers. Forsch. 96, p.24-40.
36- Rollan G., De Angelis M., Gobbetti M., Valdez
G.F, 2005. Proteolytic activity and reduction of gliadin-like
fractions by sourdough lactobacilli. Journal of Applied Microbiology,
99: 1495-1502.
37- Roussel Ph., Chiron H., 2002. Les pains
Français, 1 ère édition,
MAÉ-ERTI-433p.
38- Ruiz A., Poblet M., Mas A., Guillamon J.,
2000. Identification of acetic acid bacteria by RFLP of PCR-amplified
16S and 16S-23S rDNA Intergenic Spacer. International Journal of Systematic and
Evolutionary Microbiology, 50: 1981-1987.
39- Settanni L., Van Sinderen D., Rossi J., Corsetti
A., 2005. Rapid differentiation and in situ detection of 16 sourdough
Lactobacillus species by multiplex PCR. Applied and Environmental
Microbiology, 71, N°6: 3049-3059.
40- Spicher G., 1987. Die Mikroflora des
sauerteiges XXII Mitt: Die in Weizensauerteign Vorkommenden lactobacillen;
z.lebens.Unters.Forsch. 184, p.300-303.
41-Sriranganathan N., Seidler R., Sandine W., Elliker
PR., 1973. Cytological and deoxyribonucleic acid-deoxyribonucleic acid
hybridization studies on Lactobacillus isolates from San Francisco
sourdough. Applied Microbiology, 25, N°3: 461-470.
42- Stiles M., Holzapfel W., 1997. Lactic
acid bacteria of foods and their current taxonomy. Areview J. Food Microbiol,
36: 1-29.
43- Tannock GW., Timisjarvi A., Rodtong S., NG J.,
Munro K., Alatossava T., 1999. Identification of Lactobacillus
isolates from the gastrointestinal tract, silage, and yoghurt by 16S-23S rRNA
gene Intergenic spacer region sequence comparisons. Applied and Environmental
Microbiology, 65, N°9: 4264-4267.
44- Thiele C., Grassl S., Gänzle M.,
2004. Gluten hydrolysis and depolymerization during sourdough
fermentation. Journal of agricultural and food chemistry, 52:
1307-1314.
45- Torriani S., Zapparoli G., Dellaglio F.,
1999. Use of PCR-based methods for rapid differentiation of
Lactobacillus delbrueckii subsp. bulgaricus and L. delbrueckii
subsp.lactis.
Applied and Environmental Microbiology,
Vol.65, N°10: 4351-4356.
46- Valcheva R., Korakli M., Onno B., Prevost H.,
Ivanova I., Ehrmann M., Dousset X., Gänzle M., Vogel R., 2005.
Lactobacillus hammesii sp. nov., isolated from french sourdough.
International Journal of Systematic and Evolutionary
Microbiology, 55: 763-767.
47- Valcheva R., Ferchichi M., Korakli M., Ivanova I.,
Gänzle M., Vogel R., Prévost H., Onno B., Dousset X. 2006.
Lactobacillus nantensis sp. nov., isolated from French wheat
sourdough. International Journal of Systematic and Evolutionary Microbiology,
56: 587-591.
48- Vasquez A., Ahrné S., Pettersson B., Molin
G., 2001. Temporal temperature gradient gel electrophoresis (TTGE) as
a tool for identification of Lactobacillus casei, Lactobacillus
paracasei, Lactobacillus zeae and Lactobacillus
rhamnosus. Letters in Applied Microbiology, 32:
215-219.
49- Vogel R., Knorr R., Muler M., 1999.
Non-dairy lactic fermentations: the cereal world.
50- Wiese B., Strohmar W., Rainey F., Diekmann
H., 1996. Lactobacillus panis sp. nov., from sourdough with a
long fermentation period. International Journal of Systematic
Bacteriology, 46, N°2: 449-453.
51- Zapparoli G., Torriani S., 1997. Rapid
identification and detection of Lactobacillus sanfrancisco in
sourdough by species-specific PCR with 16S rRNA-targeted primers. System. Appl.
Microbiol. 20: 640-644.
52- Zavaleta A., Martinez A., Rodriguez F.,
1996. 16S-23S rDNA intergenic sequences indicate that Leuconostoc
oenos is phylogenetically homogeneous. Microbiology, 142:
2105-2114.
53- Zoetendal E., Akkermans A., De Vos W.,
1998. Temperature gradient gel electrophoresis analysis of 16S rRNA from human
faecal samples reveals stable and host-specific communities of active bacteria.
Applied and Environmental Microbiology, 64, N°10:
3854-3859.
Tableau n°1: La diversité des
flores des levains naturels
2
Tableau n°2: Exemples de quelques
enzymes de restriction et leurs sites de reconnaissances
8
Tableau n°3: Liste des souches de
référence
11
Tableau n°4: Les amorces de
l'étude
13
Tableau n°5: pH et TTA des
échantillons des levains BF
16
Tableau n°6: Dénombrement des
levures et des bactéries lactiques dans les levains BF 16
Tableau n°7: Ratio des isolats
homo/hétérofermentaires dans les levains
17
Figure n°1 : Positions des
régions conservées de l'opéron ribosomique 16S-23S chez
Escherichia coli
6
Figure n°2 : Organisation de la
région intergénique 16S-23S de l'ARNr chez les bactéries
lactiques
7
Figure n°3 : La technique TTGE
9
Figure n°4 : Formation d'un brin
d'ADN ramifié grâce à la présence de GC clamp
9
Figure n°5 : Schéma
générale de l'étude
10
Figure n°6 : Le marqueur 100pb DNA
Ladder-Biolabs
15
Figure n°7 : PCR avec les amorces
16S/2-23S/7 de 16 isolats de BF-70 18
Figure n°8 : Amplification par les
amorces tRNAala -23S/10 (souches : 1,8 et 16)
19
Figure n°9: RFLP par Hind III, Hinf I et
á Taq I
19
Figure n°10 : PCR avec primers
spécifiques pour Lb. sanfranciscensis
20
Figure n°11 : PCR avec les amorces
16S/2-23S/7 de 12 isolats de BF-FC 21
Figure n°12 : PCR
tala/10 et RFLP par Hinf I, Hind III et á Taq I de la souche
M9 21
Figure n°13 : PCR avec primers
spécifiques de l'espèce Lb. sanfranciscensis
ATCC 43322
22
Figure n°14 : Amplification ITS
avec la PCR sur colonies
23
ANNEXE I
Les milieux de culture
1. MRS (Man Sharpe Rogosa) modifié:
Biokar diagnostics
La composition en gramme par litre est la suivante :
|
Composés
|
Quantité (g)
|
|
polypetone
|
10
|
|
Extrait de viande
|
10
|
|
Extrait autolytique de levure
|
5
|
|
Glucose
|
20
|
|
Tween 80
|
1,08
|
|
Phosphate dipotassique
|
2
|
|
Acétate de sodium
|
5
|
|
Citrate d'ammonium
|
2
|
|
Sulfate de magnésium
|
0,2
|
|
Sulfate de manganèse
|
0,05
|
|
Agar
|
15
|
|
pH à 25°C= 6,5+/-0,2
|
55,3 g du milieu est mélangé avec : 10 g/l
de maltose, 5 g/l de fructose, 0,5 g/l de cystéine et 5 ml de FYE et mis
en suspension dans un litre d'eau distillée, ensuite chauffé sous
agitation magnétique jusqu'à homogénéisation
complète. Stériliser à l'autoclave à 121°C
pendant 15 minutes. Le milieu est coulé dans des boîtes de
pétri à 45°C en milieu stérile.
Le bouillon MRS modifié (55g/l) a la même
composition sauf qu'il ne contient pas de l'agar biologique.
2. SDB (SourDough Bacteria) : Kline et
Sugihara, 1971
La composition en gramme par litre est la suivante :
|
Composés
|
Quantité (g)
|
|
Maltose
|
20
|
|
Extrait de levures
|
3
|
|
FYE
|
0,5% (v/v)
|
|
Tween 80
|
3ml de 10% solution par litre
|
|
Peptone de soja
|
12
|
|
Agar
|
15
|
|
pH à 25°C= 5,6
|
Le bouillon SDB a la même composition sans l'agar
biologique.
3. Tryptone sel
Ce milieu contient 2 g de peptone et 17 g de chlorure de
sodium. La solution est répartie dans des tubes de 9 ml avant
d'être stérilisée à 121°C pendant 15 minutes et
stockée à 4°C. Cette solution sert pour les dilutions.
4. Préparation du FYE :
20% de suspension commerciale de levure boulangère est
mélangée dans de l'eau distillée. Le mélange est
stérilisé pendant 30 minutes à 121°C. Après
une nuit de repos et de décantation entre 2 et 8°C, le
mélange est ensuite centrifugé. Le surnageant est
récupéré et stocké à -20°C.
5. Sabouraud glucose agar +
Chloramphénicol :
La composition en gramme par litre est la suivante :
|
Composés
|
Quantité (g)
|
|
Peptone pepsique de viande
|
10
|
|
Glucose
|
35
|
|
Chloramphénicol
|
0,5
|
|
Agar agar biologique
|
15
|
60g/l sont mis en suspension dans un litre d'eau
distillée. Le mélange st chauffé lentement sous agitation
magnétique jusqu'à la dissolution complète.
Stériliser à l'autoclave à 121°C pendant 15
minutes.
6. La préparation du gel
d'Agarose :
On mélange 50% d'agarose-réosphore
(référence 018649 Eurobio) et 50% d'agar standard
(référence 018054 Eurobio). On ajoute 100 ml du tampon TBE sur la
mixture ci-dessus.
On vérifie que l'agarose est complètement
dissous et on ajoute 15 ul de BET pour la coloration.
7. La préparation du gel
d'acrylamide :
La composition du gel est la suivante :
12,6 g d'urée dans 14,5 ml d'eau (mélange
à 45°C), 1ml d'ammonium persulfate, 6ml d'acrylamide, 750ul du
tampon TAE 50x, 27,5ul d'une solution TEMEO et 275 ul d'APS à 10%. Le
gel demande 2 heures pour y être polymérisé.
8. La préparation du colorant SYBR Green
I :
On mélange 30 ul du colorant SYBR Green avec 300 ul d'une
solution tampon TBE 1X.
9. La préparation du PCR
mix :
|
Composition
|
Concentration initiale
|
Concentration finale
|
Volume par réaction (ul)
|
|
H2 O
|
|
|
19 ,283
|
|
Solution tampon
|
10x
|
1x
|
2,5
|
|
dNTP
|
20mM
|
0,2mM
|
0,2
|
|
Amorce 1
|
100umole
|
1uM
|
0,225
|
|
Amorce 2
|
100umole
|
1uM
|
0,225
|
|
MgCl2
|
25mM
|
1,5mM
|
1,5
|
|
Taq polymérase
|
15U/ul
|
1U
|
0,067
|
|
ADN
|
|
|
1
|
- Taq ADN polymérase :
Marque : Q-Biogene ;
Concentration : 2000U (15 U/ul)
- Solution Tampon : Incubation mix sans
MgCl2
Marque: Q-Biogene; Concentration: 10X
- MgCl2 :
Marque: Q-Biogene; Concentration: 25mM
- dNTP Master Mix :
Marque: Eurogentec; Concentration: 20mM
- Oligonucléotides :
* 23S/10 : Concentration :
100uM
Marque :
Eurobio
* tRNAala :
Concentration : 100uM
Marque :
Eurobio
? Programme du PCR
Dénaturation de l'ADN : 94°C pour
5minutes
Dénaturation 94°C
pour 1minute
Hybridation 60°C
1minute
Elongation 72°C
1minute
Elongation terminale 72°C
5minutes
Refroidissement 4°C
10. RFLP mix :
|
Composition
|
Volume/réaction (ul)
|
|
á Taq I
|
Hinf I
|
Hind III
|
|
H2 O
|
1,85
|
0,75
|
1,75
|
|
Spesific (10X)
|
2,5
|
2,5
|
2,5
|
|
BSA (0,1X)
|
0,25
|
0,25
|
0,25
|
|
Produit du PCR
|
20
|
20
|
20
|
|
Enzyme (10 U/réaction)
|
0,4
|
1
|
0,5
|
Les enzymes :
? á Taq I
Concentration : 20000U/ml
Contient : 0,2ml (5000U)
Marque : Biolabs
Tampon : Tampon Ne pour Taq I 10X Ref B0149S
Température d'incubation : 65°C pour 4 heures
minimum
? Hinf I
Concentration: 10000U/ml
Contient: 0,5ml (5000U)
Marque: Biolabs Ref R0155S
Tampon: Tampon Ne 2 Biolabs 10X
Température d'incubation : 37°C pour 4 heures
minimum
? Hind III
Concentration: 20000U/ml
Contient: 0,5ml (10000U)
Marque: Biolabs Ref R0104S
Tampon: Tampon Ne 2 Biolabs 10X
BF-70
|
Milieux et
dilutions
Echantillon
|
Sabouraud glucose agar
|
MRS m agar
|
SDB agar
|
|
10-5
|
10-6
|
10-5
|
10-6
|
10-5
|
10-6
|
|
BF-1
|
13,5
|
15
|
31
|
14
|
90
|
51
|
|
BF-2
|
61,5
|
9
|
119
|
13,5
|
173,5
|
45,5
|
|
BF-3
|
46,5
|
3,5
|
57,5
|
7,5
|
108,5
|
36
|
BF-FC
|
Milieux et
dilutions
Echantillon
|
Sabouraud glucose agar
|
MRS m agar
|
SDB agar
|
|
10-5
|
10-6
|
10-5
|
10-6
|
10-5
|
10-6
|
|
BF-1
|
1,5
|
0
|
24
|
3
|
30
|
4,5
|
|
BF-2
|
5,5
|
0
|
59,5
|
18
|
90
|
28,5
|
|
BF-3
|
0
|
0
|
42
|
4
|
32,5
|
3,5
|
|
TESTS PHENOTYPIQUES
|
BF-70
|
Tests
N° tubes
|
Morphologie et aspect des colonies
|
La coloration de Gram
|
Test au catalase
|
Test homo-hétérofermentaire
|
|
1
|
Colonies de petite taille (1-1,5 mm), blanches
crémeuses, bombées, lisses et rondes.
|
+
|
-
|
Hétéro
|
|
2
|
idem
|
+
|
-
|
Hétéro
|
|
3
|
idem
|
+
|
-
|
Hétéro
|
|
4
|
idem
|
+
|
-
|
Hétéro
|
|
5
|
idem
|
+
|
-
|
Hétéro
|
|
6
|
idem
|
+
|
-
|
Hétéro
|
|
7
|
idem
|
+
|
-
|
Hétéro
|
|
8
|
idem
|
+
|
-
|
Hétéro
|
|
9
|
idem
|
+
|
-
|
Hétéro
|
|
10
|
idem
|
+
|
-
|
Hétéro
|
|
11
|
idem
|
+
|
-
|
Hétéro
|
|
12
|
idem
|
+
|
-
|
Hétéro
|
|
13
|
idem
|
+
|
-
|
Hétéro
|
|
14
|
idem
|
+
|
-
|
Hétéro
|
|
15
|
idem
|
+
|
-
|
Homo
|
|
16
|
idem
|
+
|
-
|
Hétéro
|
|
17
|
idem
|
+
|
-
|
Hétéro
|
|
18
|
idem
|
+
|
-
|
Hétéro
|
|
19
|
idem
|
+
|
-
|
Hétéro
|
BF-FC
|
Tests
N° tubes
|
Morphologie et aspect des colonies
|
La coloration de Gram
|
Test au catalase
|
Test homo-hétérofermentaire
|
|
1
|
Colonies de petite taille (1-1,5 mm), blanches
crémeuses, bombées, lisses et rondes.
|
+
|
-
|
Hétéro
|
|
2
|
idem
|
+
|
-
|
Hétéro
|
|
3
|
idem
|
+
|
-
|
Hétéro
|
|
4
|
idem
|
+
|
-
|
Hétéro
|
|
5
|
idem
|
+
|
-
|
Hétéro
|
|
6
|
idem
|
+
|
-
|
Hétéro
|
|
7
|
idem
|
+
|
-
|
Hétéro
|
|
8
|
idem
|
+
|
-
|
Hétéro
|
|
9
|
idem
|
+
|
-
|
Hétéro
|
|
10
|
idem
|
+
|
-
|
Hétéro
|
|
11
|
idem
|
+
|
-
|
Hétéro
|
|
12
|
idem
|
+
|
-
|
Hétéro
|
|
13
|
Grosses colonies
|
+
|
-
|
Hétéro
|
|
14
|
Colonies de petite taille (1-1,5 mm), blanches
crémeuses, bombées, lisses et rondes.
|
+
|
-
|
Hétéro
|
|
15
|
idem
|
+
|
-
|
Hétéro
|
|
16
|
idem
|
+
|
-
|
Hétéro
|
|
17
|
idem
|
+
|
-
|
Hétéro
|
|
18
|
idem
|
+
|
-
|
Hétéro
|
|
19
|
Grosses colonies
|
+
|
-
|
Hétéro
|
|
20
|
idem
|
+
|
-
|
Hétéro
|
|
ENZYMES
|
SOURCE MICROBIENNE
|
SEQUENCE
|
|
Alu I
|
Arthrobacter luteus
|
?
5'-A-G-C-T-3'
3'-T-C-G-A-5'
|
|
Bam HI
|
Bacillus amyloliquefaciens H
|
?
5'-G-C-A-T-C-C-3'
3'-C-C-T-A-G-G-5'
?
|
|
Eco RI
|
Escherichia coli
|
?
5'-G-A-A-T-T-C-3'
3'-C-T-T-A-A-G-5'
?
|
|
Eco RII
|
Escherichia coli
|
?
5'-C-C-T-G-G-3'
3'-G-G-A-C-C-5'
?
|
|
Hae III
|
Hæmophilus ægyptius
|
?
5'-G-G-C-C-3'
3'-C-C-G-G-5'
?
|
|
Hind III
|
Hæmophilus influenzæ b
|
?
5'-A-A-G-C-T-T-3'
3'-T-T-C-G-A-A-5'
?
|
|
Hinf I
|
Hæmophilus influenzæ Rf
|
?
5'-G-A-N-T-C-3'
3'-C-T-N-A-G-5'
?
|
|
á TaqI
|
|
?
5'-T-C-G-A-3'
3'-A-G-C-T-5'
?
|
|
Code
|
Localisation
|
Séquences oligonucléotidiques
Primers (5'?3')
|
T (°C)
Hybridation
|
Références
|
|
16S/2
|
16S
1389-1407 de E.coli
|
CTT GTA CAC ACC GCC CGT C
|
60°C
|
Gürttler et Stanisich 1996
Kabadjova et al, 2002
|
|
23S/10
|
23S ARNr
456-474 de E.coli
|
CCT TTC CCT CAC GT ACT G
|
60°C
|
idem
|
|
23S/7
|
23S ARNr
188-208 de E.coli
|
GGT ACT TAG ATG TTT CAG TTC
|
60°C
|
idem
|
|
ARNtala
|
ITS
|
TAG CTC AGC TGG GAG AGC
|
60°C
|
idem
|
|
TTGE
|
|
GCG TGT GTA CAA GAC CC
|
|
|
|
TTGE
|
|
CGC CCG GGG CGC GCC GGG GAA CGC GAA GAA CCT TAC
|
|
|
|
Psfpar1
|
ITS
|
5' CTC TGT TAT ATT TGA ATA AGTACT 3'
|
|
|
|
psfspich
|
ITS
|
5' GAC CGC AAG GTC ATC ATT AT3'
|
|
|
L-ITS : Lb. paralimentarius
>437 pb
CTAAGGATATGGTACTTTGGTACCAACGGAAATACACAATTGTTAAAACTTTGTTTAGTTTTGAGGGGTCTACCTGGCGCTTTCGGGCCTATAGCTCAGCTGGTTTAGAGCGCACGCCTGATAAGCGTGAGGTCGATGGTTCAAGTCCATTTAGGCCCATTTTCTCTGTTATAATTGAATAAGTACTATGGGGGTTTAGCTCAGCTGGGAGAGCACCTGCTTTGCAAGCAGGAGGTCATCGGTTCGATCCCGTTAACCTCCATTGACAGAGATGTCATACTCGTACCTTGTAAACTGGATATTAAGGATTAATGAATTAAAGAAACACCGAAAACTGCGCGGCAATCTTTTTGATTGCCAGCTGTGACGAAAGTCATGGCAAATTGTTTTAAATTACTTTTTGTAATTTAATTTAGGTTAAGTTATAAAGGGCGCAC
Sequence tRNA Bounds tRNA Anti Intron Bounds
Cove
Name tRNA # Begin End Type Codon Begin End
Score
-------- ------ ---- ------ ---- ----- ----- ----
------
, 1 85 159 Ile GAT 0 0 78.68
, 2 190 262 Ala TGC 0 0 65.92
S-ITS: Lb. paralimentarius
>238 pb
CTAAGGATATGGTACTTTGGTACCAACGGAAATACACAATTGTTAAAACTTTGTTTAGTTTTGAGGGGTCTACCCTCAAACTCGTACCTTGAAAACTGGATATTAAGAATTAATGAATTAAAGAAACACCGAAAACTGCGCGGCAATCTTTTTGATTGCCAGCTGTGACGAAAGTCATGGCAAATTGTTTTAAATTACTTTTTGTAATTTAATTTAGGTTAAGTTATAAAGGGCGCAC
L-ISR : Lb. mindensis
>418 pb
CTAAGGAATATACGGAGGCTACACATACTTGTTGAAACAATGTTCAGTTT
TGAGGGGCTTACCTCTCAACTAAATGATTTTGGGCGTATAGCTCAGCTGG
TTTAGAGCGCACGCCTGATAAGCGTGAGGTCGATGGTTCGAGTCCATTTA
TGCCCATTGACCGCAAGGTCATCATTATGGGGAATTAGCTCAGATGGGAG
AGCACCTGCTTTGCAAGCAGGGGGTCAGCGGTTCGATCCCGCTATTCTCC
ATTGGCACGAGAGTGCCAAGGAATTTGTTCTTTGAAAACTAGATATTATC
AATTATTTTCCTTTAATTATTAAGATAATTAAACCGAGAAACAACTGCGT
ATTTTTGAGTTTTTTAATTAGTTTATCGCTAATACTCAATTAATTTGACG
ATCACGAAGTGACCGTTA
Sequence tRNA Bounds tRNA Anti Intron Bounds Cove
Name tRNA # Begin End Type Codon Begin End Score
-------- ------ ---- ------ ---- ----- ----- ---- ------
, 1 82 156 Ile GAT 0 0 78.13
, 2 179 251 Ala TGC 0 0 65.35
S-ISR: Lb. mindensis
>196 pb
>readseq.input(1), 370 bases, CB80CAB5 checksum.
CTAAGGATATGGTGCTTTAGCACCAACGGAAATACACAACGTTAAAACTTTGTTTAGTTTTGAGAGGTCTACTCTCAAACTCGTACCTTGAAAACTGGATATTAAGAATTAATGATTTAAAAGAAACACCGAAAACTGCGCGATAAAAGTGGATGTATTTCCATATTCTGTCAAGATTAAGTTATAAAGGGCGCAC
L-ISR : Lb. spicheri
>427 pb
CTAAGGAATAATACGGAGGCTACACATACTTTGTCGAAACAATGGTCAGT
TTTGAGGGGTCTACCCTCAAAGCCTTATGTGCTGTTTTGGGCGTATAGCT
CAGCTGGTTTAGAGCGCACGCCTGATAAGCGTGAGGTCGATGGTTCGAGT
CCATTTATGCCCATTGACCGTAAGGTCGATATG GGGAATTAGCTCAGATG
GGAGAGCACCTGCCTTGCAAGCAGGGGGTCAGCGGTTCGATCCCGCTATT
CTCCATTGGTGCGAGAGCATCAAGATAACTTGTTCTTTGAAAACTAGATA
TTATCAATTATTTTCCTTTAATTATTAAAGATAATTAAACCGAGAACACC
GCGTTTATTTTGAGTTTTTTAATTAGTTTTAAAATTCGCTAATACTCAAT
TAATCGTTCATCACGACGTGATGAATA
Sequence tRNA Bounds tRNA Anti Intron Bounds Cove
Name tRNA # Begin End Type Codon Begin End Score
-------- ------ ---- ------ ---- ----- ----- ---- ------
, 1 89 163 Ile GAT 0 0 78.13
, 2 183 255 Ala TGC 0 0 67.17
S-ISR : Lb. spicheri
>220 pb
CTAAGGAATAATACGGAGGCTACACATACTTTGTCGAAACAATGGTCAGTTTTGAGGGGTCTACCCTCAAACTTGTTCTTTGAAAACTAGATATTATCAATTATTTTCCTTTAATTATTAAAGATAATTAAACCGAGAACACCGCGTTTATTTTGAGTTTTTTAATTAGTTTTAAAATTCGCTAATACTCGATTAATCGTTCATCACGACGTGATGAATA
>434 pb
L-ISR: Lb. hammesii
CTAAGGAATATACGGAGGCTACACATACTTTGTCGAAACAATGGTCAGTT
TTGAGGGGTCTACCCTCATAATTATATGATTTTACTAAATGATTTTGGGC
GTATAGCTCAGCTGGTTTAGAGCGCACGCCTGATAAGCGTGAGGTCGATG
GTTCGAGTCCATTTATGCCCATTGACCGCAAGGTCATCATTTTGGGGAAT
TAGCTCAGATGGGAGAGCACCTGCTTTGCAAGCAGGGGGTCAGCGGTTCG
ATCCCGCTATTCTCCATTGGTGCGAAAGCATCAAGGAATTTGTTCTTTGA
AAACTAGATATTATCAATTATTTTCCTTTAATTATTAAGATAATTAAACC
GAGAAACAACTGCGTATTTTTGAGCTTTTTAATTAGTTTATATCGCTAAT
ACTCAATTAATCAGCGATTACGAAGTAATCGTTA
Sequence tRNA Bounds tRNA Anti Intron Bounds
Cove
Name tRNA # Begin End Type Codon Begin End
Score
-------- ------ ---- ------ ---- ----- ----- ----
------
, 1 97 171 Ile GAT 0 0 81.21
, 2 194 266 Ala TGC 0 0 72.85
S-ISR: Lb. hammesii
>216 pb
CTAAGGAATATACGGAGGCTACACATACTTTGTCGAAACAATGGTCAGTTTTGAGAGGTCTACTCTCAAACTTGTTCTTTGAAAACTAGATATTATCAATTATTTTCCTTTAATTATTAAGATAATTAAACCGAGAAACAACTGCGTACTTTTGAGTTTTTTAATTAGTTTATATCGCTAATACTCAATTAATCAGCGATTACGAAGTAATCGTTA
S-ITS: Lb. Frumenti
>204 pb
CTAAGGAATAAAACGGAAACCTACACATAGTTGAAACTTTGTTTAGTTTTGAGGGGTTTACCCTCAGAGCTTGTACTTTGAAAACTAAATAATATCATTTTTCTTTATTTAACAAAACAAACCGAGAACACCGCGTTATTTTGAGTTGTTTAATGAAGATAATCGCTAACTCAATTAATCCGATGATCACTTAGTGATCATCAA
L-ITS: Lb. Frumenti
>397 pb
CTAAGGAATAAAACGGAAACCTACACATAGTTGAAACTTTGTTTAGTTTTGAGAGGTTTACTTCTCAAAACATCAGACGCTTATTTGGGCCTATAGCTCAGCTGGTTTAGAGCGCACGCCTGATAAGCGTGAGGTCGATGGTTCAAGTCCATTTAGGCCCATTATGGGGAATTAGCTCAGCTGGGAGAGCACCTGCTTTGCAAGCAGGAGGTCATCGGTTCGATTCCGTTATTCTCCATTGTCAACCGTGAGGTTGACAGAGCTTGTACTTTGAAAACTAAATAATATCATTTTTCTTTATTTAACAAAACAAACCGAGAACACCGCGTTATTTTGAGTTGTTTAATGAAAATAATCGCTAACTCAATTAATCCGATGATCACTTAGTGATCATCAA
Sequence tRNA Bounds tRNA Anti Intron Bounds
Cove
Name tRNA # Begin End Type Codon Begin End
Score
-------- ------ ---- ------ ---- ----- ----- ----
------
, 1 87 161 Ile GAT 0 0 78.68
, 2 166 238 Ala TGC 0 0 66.64
L-ITS : Lb. panis
>405 pb
CTAAGGAATAAAACGGAACCTACACATCAGTTGAAACTTTGTTTAGTTTTGAGAGGTTTACCTTTCAAAACGTATGACGCTTATTTGGGCCTATAGCTCAGTTGGTTTAGAGCGCACGCCTGATAAGCGTGAGGCCGATGGTTCAAGTCCATTTAGGCCCATCATATTTTGGGGAATTAGCTCAGCTGGGAGAGCACCTGCTTTGCAAGCAGGAGGTCATCGGTTCGATTCCGTTATTCTCCATTGCTAGTCGTAAGATTAGCAGAGCTTGTACTTTGAAAACTAAATAATATCTAATTTCTTTATTTATTAAAACAATAAACCGAGAACACCGCGTTATTTGAGTTTTATTAACGAAATAATCGCTAACTCAATTAATCAAGGTGATCACTTCGTGATCATCAA
Sequence tRNA Bounds tRNA Anti Intron Bounds
Cove
Name tRNA # Begin End Type Codon Begin End
Score
-------- ------ ---- ------ ---- ----- ----- ----
------
, 1 87 161 Ile GAT 0 0 77.04
, 2 171 243 Ala TGC 0 0 66.64
S-ITS: Lb. panis
>222 pb
ACTATAGGGAAAGCTCGGTACCACGCATGCTGCAGACGCGTTACGTATCGGATCCAGAATTCGTGATTCTTGTACACACCGCCCGTCACACCATGGAAGTTTGTAACGCCCAAAGTCGGTGGCCTAACCATTTGGAGGGAGCCGCCTAAGGCGGGACAGATGACTGGGGTGAAGTCGTAACAAGGTAGCCGTAGGAGAACCTGCGGCTGGATCACCTCCTTT
L-ITS: Lb. pontis
>403 pb
CTAAGGAATAAAACGGAGCCTACACATTGTCGAAACTTTGTTTAGTTTTGAGGGGTTTACCTCAAAACTTACTGAGACGCTTATTTGGGCCTATAGCTCAGCTGGTTTAGAGCGCACGCCTGATAAGCGTGAGGTCGATGGTTCAAGTCCATTTAGGCCCATATATGGGGAATTAGCTCAGCTGGGAGAGCACCTGCTTTGCAAGCAGGAGGTCATCGGTTCGATTCCGTTATTCTCCATTGCCAATCGTAAGGTTGGCACGAGCTTGTACTTTGAAAACTAAATAATATCTAATTTCTTTATTTATTAAAAACAATAAACCGAGAACACCGCGTTATTTGAGTTTTATTAACGAAATAATCGCTAACTCAATTAATTGAGATGACCACTAAGTGGTCATCAA
Sequence tRNA Bounds tRNA Anti Intron Bounds
Cove
Name tRNA # Begin End Type Codon Begin End
Score
-------- ------ ---- ------ ---- ----- ----- ----
------
, 1 87 161 Ile GAT 0 0 78.68
, 2 167 239 Ala TGC 0 0 66.64
S-ISR: Lb. pontis
>207 pb
CTAAGGAATAAAACGGAACCTGCACATTGTCGAAACTTTGTATAGTTTTGAGGGGTTTACCCTCAGAGCTTGTACTTTGAAAACTAAATAATATCTAATTTCTTTATTTATTAAAAACAATAAACCGAGAACACCGCGTTATTTGAGTTTTATTAACGAAATAATCGCTAACTCAATTAATTGAGATGACCACTAAGTGGTCATCAA
L-ITS : Lb. nantensis
AY690836
>389 pb
CTAAGGATATGATGCGTAAGCATCAACGGAAATACACAATTGTTAAAACTTTGTTTAGTTTTGAGAGGCCTACCGGGCGTTTTTCGGGCCTATAGCTCAGCTGGTTTAGAGCGCACGCCTGATAAGCGTGAGGTCGATGGTTCAAGTCCATTTAGGCCCATTCTCATATTAATAAGTACCATATGGGGGCTTAGCTCAGCTGGGAGAGCACCTGCTTTGCAAGCAGGAGGTCATCGGTTCGATCCCGTTAGCCTCCATTGACAGAAATGCCAATGTTCGTACCTTGAAAACTGGATATTAAGAAATAATGAATTAAAGAAACACCGAAAACTGCGCGATAAAAATGATTAATTTCATATTTTGTCAAGATTAAGTTATAAAGGGCGCAC
tRNA 86..160
tRNA 185..257
S-ITS : Lb. nantensis
AY690835
>196 pb
CTAAGGATATGATGCGTAAGCATCAACGGAAATACACAATTGTTAAAACTTTGTTTAGTTTTGAGGGGTTTACCCTCAGTAACTCGTACCTTGAAAACTGGATATTAAGAAATAATGAATTAAAGAAACACCGAAAACTGCGCGATAAAAATGATTAATTTCATATTTTGTCAAGATTAAGTTATAAAGGGCGCAC
Pour alignement
>nantensis
CTAAGGATATGATGCGTAAGCATCAACGGAAATACACAATTGTTAAAACTTTGTTTAGTTTTGAGAGGCCTACCGGGCGTTTTTCGGGCCTATAGCTCAGCTGGTTTAGAGCGCACGCCTGATAAGCGTGAGGTCGATGGTTCAAGTCCATTTAGGCCCATTCTCATATTAATAAGTACCATATGGGGGCTTAGCTCAGCTGGGAGAGCACCTGCTTTGCAAGCAGGAGGTCATCGGTTCGATCCCGTTAGCCTCCATTGACAGAAATGCCAATGTTCGTACCTTGAAAACTGGATATTAAGAAATAATGAATTAAAGAAACACCGAAAACTGCGCGATAAAAATGATTAATTTCATATTTTGTCAAGATTAAGTTATAAAGGGCGCAC
>hammesii
CTAAGGAATATACGGAGGCTACACATACTTTGTCGAAACAATGGTCAGTT
TTGAGGGGTCTACCCTCATAATTATATGATTTTACTAAATGATTTTGGGC
GTATAGCTCAGCTGGTTTAGAGCGCACGCCTGATAAGCGTGAGGTCGATG
GTTCGAGTCCATTTATGCCCATTGACCGCAAGGTCATCATTTTGGGGAAT
TAGCTCAGATGGGAGAGCACCTGCTTTGCAAGCAGGGGGTCAGCGGTTCG
ATCCCGCTATTCTCCATTGGTGCGAAAGCATCAAGGAATTTGTTCTTTGA
AAACTAGATATTATCAATTATTTTCCTTTAATTATTAAGATAATTAAACC
GAGAAACAACTGCGTATTTTTGAGCTTTTTAATTAGTTTATATCGCTAAT
ACTCAATTAATCAGCGATTACGAAGTAATCGTTA
>spicheri
CTAAGGAATAATACGGAGGCTACACATACTTTGTCGAAACAATGGTCAGT
TTTGAGGGGTCTACCCTCAAAGCCTTATGTGCTGTTTTGGGCGTATAGCT
CAGCTGGTTTAGAGCGCACGCCTGATAAGCGTGAGGTCGATGGTTCGAGT
CCATTTATGCCCATTGACCGTAAGGTCGATATGGGGAATTAGCTCAGATG
GGAGAGCACCTGCCTTGCAAGCAGGGGGTCAGCGGTTCGATCCCGCTATT
CTCCATTGGTGCGAGAGCATCAAGATAACTTGTTCTTTGAAAACTAGATA
TTATCAATTATTTTCCTTTAATTATTAAAGATAATTAAACCGAGAACACC
GCGTTTATTTTGAGTTTTTTAATTAGTTTTAAAATTCGCTAATACTCAAT
TAATCGTTCATCACGACGTGATGAATA
Les industries des produits céréaliers
fermentés (panification, viennoiserie, crackers...) ont actuellement un
regain d'intérêt pour les fermentations mixtes La maîtrise
de ce procédé de fermentation mixte s'appuie d'une part sur
l'étude de la biodiversité microbienne des levains et d'autre
part sur la caractérisation des flores, notamment, lactiques à
l'aide des outils biomoléculaires .L'objectif était
d'étudier la biodiversité des flores lactiques des levains
français de panification et l'influence du paramètre type de
farine sur la flore lactique, ainsi que, la validation des méthodes
moléculaires (PCR, PCR-RFLP) de caractérisation de ces flores.
Deux échantillons de levains français ont
été analysés : un levain BF rafraîchi avec une
farine de type 70 et un levain BF rafraîchi avec la farine du blé
concassé. La flore lactique du levain de panification est
constituée principalement de bactéries du genre
Lactobacillus. La région intergénique (ITS) 16S-23S
présente un polymorphisme de taille et de séquence suffisant
entre les espèces pour permettre une caractérisation
moléculaire de ces bactéries. L'amplification de cette
région intergénique par les amorces 16S/2 et 23S/10 a permis de
confirmer les genres des bactéries de référence et ceux
des isolats de levains. Celle-ci a généré un profil
électrophorétique de 3 bandes qui est particulier à
l'espèce Lb. sanfranciscensis.
L'amplification de la séquence tARNala -
23S/10 suivie d'une digestion enzymatique (PCR-RFLP) par trois enzymes de
restriction a généré différents profils de
restriction ont montré que, dans les deux échantillons,
Lb. sanfranciscensis était l'espèce prédominante.
La banque de données informatisée contenant ces profils de
restriction a permis une confirmation des résultats d'identification
des isolats de levains de panification. L'amplification d'une séquence
spécifique de la région rDNA 16S de cette espèce
bactérienne a contribué à la validation des
résultats obtenus par PCR-RFLP.
Cette étude semble montrer que la farine
utilisée pour le rafraîchi des levains n'a pas d'influence sur la
flore majoritaire dans les conditions de l'étude. Toutefois, il est
nécessaire d'identifier un nombre plus important d'isolats, de tester
d'autres types de farine et sur un nombre plus important de
rafraîchis.
Mots-clés : Lactobacilles,
bactéries lactiques, Lb. sanfranciscencis, levains,
panification, PCR-RFLP, ITS, flore lactique.
Industries of the fermented cereal products (panification,
viennoisery, crackers...) currently have a renewed interest for mixed
fermentations the control of this process of mixed fermentation is based on the
one hand on the study of the microbial biodiversity of the leavens and on the
other hand on the characterization of the flora, in particular, lactic using
the biomolecular tools. The objective was to study the biodiversity of the
lactic flora of the French leavens of panification and the influence of the
standard parameter of flour on the lactic flora, like, the validation of the
molecular methods (PCR, PCR-RFLP) of characterization of this flora.
Two French leaven samples were analyzed: a leaven BF refreshed
with a flour of the type 70 and one leaven BF refreshed with the flour of
bruised grain. The lactic flora of the leaven of panification is made up
mainly of bacteria of the Lactobacillus kind. The intergenic area (ITS) 16S-23S
has a sufficient polymorphism of size and sequence between the species to allow
a molecular characterization of these bacteria. The amplification of this
intergenic area by the primers 16S/2 and 23S/10 made it possible to confirm the
kinds of the bacteria of reference and those of the leaven isolates. This one
generated an electrophoretic profile of 3 bands which is particular with the
species Lb sanfranciscensis.
The amplification of the sequence tARNala - 23S/10 followed by
an enzymatic digestion (PCR-RFLP) by three enzymes of restriction generated
various profiles of restriction showed that, in the two samples; Lb.
sanfranciscensis was the prevalent species. The computerized bank of data
containing these profiles of restriction allowed a confirmation of the results
of identification of the leaven isolates of panification. The amplification of
a specific sequence of the area rDNA 16S of this bacterial species contributed
to the validation of the results obtained by PCR-RFLP.
This study seems to show that the flour used for refreshed
leavens does not have an influence on the majority flora under the conditions
of the study. However, it is necessary to identify a more significant number of
isolates, to test other types of flour and on a more significant number of
refreshed.
Key words: Lactobacilles, lactic acid
bacteria, Lb. sanfranciscencis, leavens, panification, PCR-RFLP, ITS,
TTGE, lactic flora.



