|

Ministère de l'Enseignement Supérieur
Université du 7 Novembre à Carthage
Institut National des Sciences Appliquées et de
Technologie
Projet de Fin d'Etudes
Pour l'obtention du
Diplôme National d'Ingénieur en Sciences
Appliquées et en Technologie
Filière : Biologie Industrielle
Sujet :
Clonage et expression de la protéine CFP10
de
Mycobactérium tuberculosis dans
Pichia pastoris
Réalisé par : Bilel
Masmoudi
Laboratoire d'accueil :
Institut Pasteur de Tunis
Laboratoire d'Immunopathologie,
Vaccinologie et Génétique Moléculaire
Responsable laboratoire : Pr. Mohammed Ridha
Barbouche
Responsable INSAT : Pr. Mohammed Néjib
Marzouki
Année Universitaire : 2005 / 2006
Ce travail a été effectuée au
Laboratoire d'Epidémiologie et d'Ecologie Parasitaire de l'Institut
Pasteur de Tunis sous la direction scientifique de Dr. Ikram
GUIZANI.
Il a bénéficié du soutien
financier de l'OMS-EMRO-COMSTECH (programme Applied Biotechnologiy and Genomics
in health) et du Ministère de la Recherche Scientifique, de la
Technologie et du Développement des Compétences (contrat
programme 2004 - 2008).
DEDICACES
Je dédie ce travail
A mes parents,
En témoignage de mon amour et de ma gratitude pour
tous les sacrifices et le soutien qu'ils ont consenti pour moi.
A toute ma famille et à tous mes amis.
Remerciements
Si ce Projet de Fin d'Etude peut paraître aujourd'hui,
c'est grâce à l'initiative et au soutien de plusieurs personnes
qu'il m'est agréable de remercier.
Professeur Mohammed Ridha BARBOUCHE, chef de service à
l'institut pasteur de tunis je le remercie profondément pour m'avoir
accueilli, permis de réaliser ce travail dans son service et qui a
assumé la responsabilité de m'encadrer et de guider mes pas dans
le domaine de la recherche. Qu'il trouve ici l'expression de ma grande
reconnaissance et mon profond respect.
Professeur Mohammed Néjib MARZOUKI Professeur à
l'Institut National des Sciences Appliquées et de Technologie, je le
remercie pour ces conseils, son soutien permanent et son attention constante.
Je lui témoigne ma profonde gratitude.
Professeur Mokhtar HAMDI, Professeur à l'Institut
National des Sciences Appliquées et de Technologie, je le remercie pour
avoir eu l'amabilité de présider le jury, qu'il veuille accepter
ma profonde gratitude et mon respect.
Professeur Michel ODONOHOU, je le remercie très
respectueusement pour avoir bien voulu juger ce travail. Puissent ces quelques
lignes lui témoigner ma très grande estime.
Mr. Chaouki BENABDESSELEM, étudiant en thèse de
doctorat, à l'Institut Pasteur de Tunis, je le remercie de m'avoir
initié à la pratique des techniques de Biologie
Moléculaire, de sa compétence de ces précieux conseils et
de son assistance pour la réalisation de ce travail. Qu'il veuille
accepter mon profond respect.
Melle Meherzia BEN FADHEL, ingénieur que je
la remercie pour son aide précieuse dans le séquençage.
Les membres du Laboratoire d'immunopathologie vaccinologie et
génétique moléculaire à l'Institut Pasteur de Tunis
pour leur grand soutien moral et pour leurs encouragements.
Sommaire
Introduction :
A] Etudes bibliographiques :
I) Les protéines
recombinantes.....................................................................10
II) Les critères de choix d'un système
d'expression............................................10
II.1) Choix des hôtes
cellulaires.....................................................11
II.2) Choix du vecteur
d'expression................................................11
III) Les systèmes d'expressions
utilisés.............................................................12
III.1) Les hôtes utilisées pour la
production des protéines recombinantes.....12
III.1.1)
E.Coli..................................................................12
III.1.2) Saccharomyces
cerevisiae..........................................12
III.1.3) Les cellules CHO (Chinese
Hamster Ovary).....................13
III.1.4) Autres
bactéries.......................................................13
III.1.5) Autres levures et
champignons filamenteux......................13
III.1.6) Les plantes
transgéniques...........................................14
III.1.7) Les animaux
transgéniques..........................................14
III.2) Les vecteurs et cassettes
d'expression........................................14
IV) Le système d'expression Pichia
pastoris.......................................................15
IV.1) Principaux avantages du
système.............................................15
IV.2) Le métabolisme du
méthanol..................................................17
V) Stratégie d'expression chez Pichia
pastoris......................................................17
V.1) Les vecteurs
d'expression.......................................................17
V.2) Mode d'intégration du gène
d'intérêt..........................................18
V.3) Le nombre de copie de la cassette
d'expression..............................19
V.4) Les Modifications post traductionnelles
chez Pichia pastoris................19
V.5) Les facteurs agissant sur la
cinétique de croissance.........................19
V.6) Les facteurs agissant sur la
cinétique de production........................20
VI) La
tuberculose...................................................................................20
VI.1) Les bacilles de la
tuberculose..................................................21
VI.2) Pouvoir pathogène de
Mycobactérium tuberculosis.........................21
VI.3) Diagnostic
bactériologique.....................................................22
VI.4)Séquençage de génome
de la souche H37Rv.................................22
VI.5) Le gène Rv3874 et
l'antigène CFP10.........................................23
B] Matériel et méthodes :
I) Matériels :
I.1) Souches bactériennes et
levures utilisées.......................................25
I.2) Vecteur de clonage et
d'expression.............................................25
I.2.1)
pcDNA3/Rv3874...........................................................25
I.2.2)
pPICZáA......................................................................................25
I.3) Les
enzymes.......................................................................26
I.3.1) Les enzymes de
restrictions...............................................26
I.3.2) Les autres
enzymes.........................................................26
I.4) Les marqueurs de poids
moléculaires..........................................27
I.4.1) Les marqueurs de poids
moléculaires d'ADN.........................27
I.4.2) Les marqueurs de poids
moléculaires de protéines....................28
I.5) Les
oligonucléotides..............................................................28
I.6) Autres
matériels...................................................................28
II) Méthodes
II.1)
PCR.................................................................................29
II.2) Electrophorèse sur gel
d'agarose...............................................29
II.3) Purification d'ADN sur gel
d'agarose.........................................30
II.4) La
digestion.......................................................................30
II.5) La
ligation........................................................................31
II.6) Transformation des
bactéries...................................................31
II.7) Colonie
PCR.....................................................................32
II.8) Préparation des
plasmides......................................................32
II.8.1) Préculture
bactérienne....................................................32
II.8.2) Extraction de l'ADN
plasmidique.......................................32
II.9) Eléctroporation de la levure
Pichia pastoris..................................33
II.10) Production de la protéine
recombinante dans Pichia pastoris............33
II.11) Electrophorèse des
protéines sur gel polyacrylamide en présence de
SDS..............................................................................34
II.12) Immunoblot et
révélation par
ECL................................................35
C] Résultats et interprétation
I. Clonage du gène Rv3874 dans le vecteur
pPICZáA........................................37
I.1) Stratégie de
clonage...............................................................37
I.2)
PCR..................................................................................38
I.3) Digestion et purification du
produit PCR et du plasmide pPICZáA........39
I.4) Ligation et transformation des
bactéries compétentes E-coli top 10 f'....40
I.5) Colonie
PCR........................................................................41
I.6) Carte de restriction de plasmide
recombinant.................................41
I.7) Séquençage
automatique.........................................................42
II) Transformation et expression du gène Rv3874 dans la
levure Pichia pastoris
KM71H.............................................................................................43
II.1) Linéarisation de
pPICZáA / Rv3874..........................................43
II.2) Transformation par électroporation de
la souche KM71H de Pichia
pastoris..............................................................................................44
II.3) Production de la protéine
recombinante CFP10-myc epitope-6his.........44
II.4) Identification de la protéine
recombinante par western blot................45
D] Conclusions et perspectives
Références bibliographiques
Avant propos
Grâce aux avancées
scientifiques spectaculaires des vingt dernières années en
matière de génie génétique et de biologie
moléculaire, il est aujourd'hui envisageable de modifier le patrimoine
génétique d'un organisme vivant en lui incorporant un fragment
d'ADN provenant d'une espèce différente et de lui faire exprimer
cette information génétique étrangère. Ces
savoir-faire nouveaux de la génétique ont permis d'ouvrir des
voies de recherche et de développement nouvelles aux perspectives
considérables. Il devient possible aujourd'hui, en s'appuyant sur ces
méthodologies, de reprogrammer le matériel
génétique d'un micro-organisme en lui insérant un
gène précis, qui lui permettra de produire une protéine
recombinante précieuse (à usage médical :
interféron, insuline, hormone de croissance... ou industriel :
enzymes...).
Cependant, le processus de conversion de l'information
génétique en protéine biologiquement active se heurte
à plusieurs problèmes aussi bien d'ordre qualitatif que
quantitatif tels que le problème de repliement intracellulaire des
protéines. En effet de nombreuses protéines recombinantes ne sont
pas produites dans leur état natif, et s'agrègent dans un
état biologiquement inactif. Pour cette raison, il est souvent
souhaitable d'intervenir en amont, sur le système d'expression pour
minimiser ces problèmes et donc obtenir une protéine recombinante
la plus proche possible de la protéine native.
La tuberculose est une maladie infectieuse chronique,
c'est l'un des plus grands fléaux de l'humanité avec plus de dix
millions de nouveaux cas et trois millions de décès chaque
année.
Cette situation s'aggrave avec l'émergence de souches
multirésistantes et le SIDA. Ainsi, la génomique, la
protéomique et la transcriptomique ont été rapidement
adaptés pour la recherche des gènes à haut potentiel
diagnostic ou à intérêt vaccinal.
Mycobactérium. tuberculosis l'agent
pathogène de la tuberculose sécrète toute une famille des
protéines dites CFP (culture filtrat protéine) dont la
protéine CFP10.
Dans ce travail, nous nous sommes donc intéressé
au gène Rv3874 qui code pour la protéine CFP10 précocement
sécrétée par M.tuberculosis dans le milieu de
culture ainsi que dans la première phase de l'infection par ce
pathogène. CFP10 est un antigène immunodominant et candidat
potentiel pour le diagnostic de la tuberculose.
Dans le but d'obtenir une protéine sous forme
recombinante nous avons donc procédé au clonage du gène
Rv3874 de M.tuberculosis dans le vecteur pPICZáA destiné
à la transformation de la levure méthylotrophique Pichia
pastoris que nous avons ensuite transformée par
éléctroporation et à la production de cette
protéine sous forme recombinante soluble.
I) les protéines recombinantes :
Une protéine recombinante est le résultat
d'un transfert du gène d'intérêt qui code pour cette
protéine dans une cellule hôte en vue de la produire. L'insuline
fut la première protéine recombinante humaine produite au
début des années 80 par la technologie de l'ADN recombinant.
Cette technologie a bénéficié des avancées
scientifiques de ces deux dernières décennies, en matière
de génie génétique, de biologie moléculaire ainsi
que du développement des techniques de culture des microorganismes
à haute densité cellulaire. Au sens large, un système de
production adapté à la fabrication d'une protéine
recombinante donnée, est un processus biotechnologique qui s'appuie
principalement sur :
· L'emploi d'un vecteur d'expression (en
général un plasmide ou un virus), jouant le rôle de
transporteur génétique du gène d'intérêt
codant pour la protéine recherchée.
· L'utilisation d'une cellule hôte, chargée
d'exécuter les instructions fournies par le gène
d'intérêt qui lui est inséré, dans l'objectif de
synthétiser la protéine recherchée.
· Une phase de production proprement dite permettant de
fabriquer la protéine souhaitée.
· Enfin, sa purification à partir du milieu de
culture.
Au sens restreint, un système de production de
protéines recombinantes est caractérisé par un couple
constitué d'un vecteur d'expression et d'un hôte (cellules ou
organismes entiers tels que des animaux ou des plantes, au quel on a
transféré le ou les gènes codant pour ces
protéines.).
II) Les critères de choix d'un système
d'expression :
Le choix du système d'expression est
principalement guidé par les modifications que la protéine doit
subir pour être biologiquement active. Bien qu'il existe des techniques
permettant le transfert rapide des gènes clonés entre
différents systèmes d'expression, le vecteur
génétique, utilisé pour exprimer le gène
recombinant, doit être adapté à l'hôte choisi. De
même, il est important de connaître les compartiments cellulaires
dans lesquels la protéine d'intérêt se replie ou exerce son
activité biologique naturelle. Selon que cette protéine est
cytoplasmique, membranaire ou extracellulaire, le vecteur devra contenir des
séquences spécifiques permettant l'adressage correct de la
protéine. Pour cela il est nécessaire d'intervenir en amont en
optimisant les conditions d'expression de la protéine. Le choix du
système d'expression constitue l'une des étapes critiques dans le
processus de production des protéines hétérologues.
(2).
II.1) Choix des hôtes cellulaires :
L'hôte idéal doit
exécuter correctement les instructions fournies par le gène
étranger et réaliser les modifications post-traductionnelles
permettant d'obtenir une protéine identique à la protéine
naturelle. Les cellules hôtes doivent se prêter à la culture
en masse en fermenteur. Elles doivent être résistantes, capables
de se multiplier à densité élevée, et peu
exigeantes en éléments nutritifs.
Le choix de l'hôte dépend également de
l'utilisation ultérieure de la protéine
hétérologue :
· S'il s'agit d'une protéine d'eucaryote
supérieur, dont on veut tester l'activité biologique, on n'en a
pas besoin de produire une grande quantité. On va préférer
l'expression transitoire du gène en question dans les cellules de
mammifère.
· Si on veut produire des grandes quantités de
protéine recombinante pure, on évalue les différents
systèmes d'expression cellulaire possibles et on sélectionne le
plus performant. Ces systèmes sont choisis en fonction de la
protéine en question, les trois systèmes opérationnels
actuellement sont le colibacille, la levure, et la cellule animale ou
végétale.
· Si les protéines doivent subir des modifications
post traductionnelles pour assurer leurs activités biologique, on ne
peut pas utiliser E-Coli qui, en tant que procaryote, en est
incapable. Les levures ou les cellules animales ou végétales
offrent la meilleure alternative. (9) ; (20).
II.2) Choix du vecteur d'expression :
Un vecteur doit posséder un
promoteur, des régions régulatrices de transcription, des signaux
d'initiation et de fin de traduction, éventuellement des signaux de
sécrétion s'il s'agit de protéines
sécrétées dans le compartiment extracellulaire. Il doit
également assurer la stabilité du gène dans la cellule
hôte. La présence d'un promoteur inductible en amont du
gène cloné est une condition importante pour l'obtention d'une
grande quantité de protéine recombinante. Ainsi un vecteur
d'expression doit posséder une origine de réplication bien
caractérisée et un marqueur de sélection, permettant la
propagation et la maintenance de la construction.
III) Les systèmes d'expression
utilisés :
Les hôtes les plus utilisés
à l'heure actuelle sont incontestablement la bactérie
Escherichia coli, la levure Saccharomyces cerevisiae et les
cellules CHO extraites des ovaires de hamster chacun d'entre eux
présentent des avantages et des inconvénients.
III.1) Les hôtes utilisés pour la production
des protéines recombinantes :
III.1.1) E. coli :
Avantages : sa génétique est très
bien connue. De nombreux vecteurs plasmidiques ont été construits
et sont donc disponibles afin d'insérer et d'exprimer un gène
étranger au sein de la bactérie. Elle est par ailleurs facile
à utiliser, se prête très bien à la culture de masse
en fermenteur. Enfin, les taux d'expression obtenus sont élevés,
c'est-à-dire qu'elle permet de produire des quantités
appréciables de protéines (jusqu'à plusieurs grammes par
litre).
Inconvénients : sécrétant mal les
protéines, il est souvent nécessaire de "casser" la
bactérie afin de récupérer la protéine (ce qui pose
des problèmes de purification et nécessite parfois des
étapes de solubilisation et de renaturation..., quelquefois au
détriment des rendements). Autre inconvénient majeur : E.
coli n'effectue pas les modifications post-traductionnelles des
protéines (en particulier la glycosylation, la carboxylation, etc.), qui
constituent souvent une condition nécessaire pour l'activité de
la protéine. Enfin, E. coli étant une
entérobactérie, il est donc nécessaire de s'assurer de
l'absence d'endotoxines dans les protéines purifiées. (17).
III.1.2) Saccharomyces cerevisiae :
Il s'agit de la levure de boulanger, utilisée
depuis des millénaires dans l'alimentation humaine.
Avantages : son matériel génétique
est simple et elle ne présente aucune toxicité. Bons vecteurs
d'expression disponibles aujourd'hui. Les taux d'expression des
protéines sont relativement intéressants (de l'ordre de centaines
de milligrammes par litre). La levure est capable de fabriquer des
protéines complexes et de réaliser des modifications
post-traductionnelles (glycosylation, phosphorylation, acylations...).
Inconvénients : le problème majeur pour
les protéines synthétisées dans
Saccharomyces cerevisiae est l'hyperglycosylation des
protéines produites. Les protéines sont souvent obtenues à
l'intérieur du cytoplasme nécessitant ainsi de casser la cellule
afin de les récupérer. La sécrétion est possible,
mais en général au détriment du rendement. Elle fonctionne
bien pour des petits polypeptides tels que l'insuline, mais beaucoup moins bien
pour de grandes protéines. (17).
III.1.3) Les cellules CHO (Chinese Hamster Ovary)
Avantages : méthode déjà
employée dans la production d'un vaccin contre l'hépatite B, les
cellules CHO se prêtent très bien à la culture en masse
dans le bioréacteur. Un avantage discriminant de ces cellules
réside dans leur capacité à synthétiser des
protéines complexes de poids moléculaire élevé.
Certains vecteurs d'expression (BPV -Bovine Papilloma Virus-, SV40...)
permettent d'introduire et de faire exprimer des gènes humains dans ces
cellules
Inconvénients : rendement faible (de l'ordre de
10 milligrammes par litre au maximum). Par ailleurs, les cellules CHO
s'avèrent fragiles et leur culture est plus onéreuse. (18).
III.1.4) D'autres bactéries :
D'autres systèmes de
production, c'est-à-dire d'autres couples hôtes-vecteurs, se
développent également aujourd'hui. Les hôtes sont
principalement : Bacillus subtilis, Streptomyces..., ceux-ci
possèdent des capacités de sécrétion
supérieur à E. coli. Cependant leur
génétique est moins connue, et le niveau de production de
protéines est inférieur à celui obtenu avec E.
coli. (17).
III.1.5) D'autres levures et champignons filamenteux
:
Quatre autres levures pourraient bien
supplanter Saccharomyces cerevisiae il s'agit surtout de Pichia
pastoris, Kluyveromyces lactis, Hanseluna polymorpha,
Yarrowia lipolytica. Elles présentent l'avantage de
sécréter beaucoup plus facilement les protéines
hétérologues que Saccharomyces cerevisiae. Ces souches
de levures son déjà utilisées dans
l'alimentation : Kluyveromyces lactis et présente dans
toutes les fromageries, Yarrowia lipolytica, Pichia pastoris sont
autorisées pour l'alimentation humaine et animale. De plus, elles se
développent sur des milieux simples. (4).
Une autre voie consiste aujourd'hui à utiliser des
bioréacteurs vivants en tant que système de production de
protéines recombinantes, c'est-à-dire des plantes ou des animaux
vivants, transgéniques.
III.1.6) Les plantes transgéniques :
Différentes techniques sont employées afin
de transférer un gène d'intérêt dans le patrimoine
génétique d'une plante : vecteurs bactériens, injection de
cellules embryonnaires totipotentes, éléctroporation du
protoplaste. La mise au point de plantes transgéniques à partir
de végétaux comme le tabac, le colza ou encore la pomme de terre,
permet de produire une variété de protéines recombinantes
précieuses : interféron, interleukine, facteur VII de la
coagulation... Dans ce domaine, restent principalement à résoudre
les problèmes de niveau d'expression des protéines (rendements
faibles), ainsi que l'extraction et la purification. Les plantes
transgéniques pourraient représenter un moyen de production peu
coûteux. (16).
III.1.7) Les animaux transgéniques :
Les principales techniques de transgénèse
utilisées sont la micro-injection dans le cytoplasme de l'embryon, les
vecteurs rétroviraux et les cellules ES (embryonnaires souches de
mammifères). Les animaux transgéniques peuvent être
utilisés afin de produire des protéines
hétérologues : production de facteur IX de la coagulation dans le
lait de brebis transgéniques, lactoferine humaine obtenue dans le lait
de vache transgénique, hormone de croissance humaine obtenue dans le
lait de souris, hémoglobine humaine produite dans le sang du porc...
L'intérêt d'une production de protéines recombinantes dans
le lait ou le sang d'animaux transgéniques se heurte cependant à
des niveaux d'investissements très lourds pour des marchés a
priori très restreints. (15).
III.2) Les vecteurs et cassettes
d'expression :
Pour la production des protéines recombinantes,
les vecteurs ainsi que les cassettes d'expression jouent un rôle
primordial dans l'amélioration de la qualité et la
quantité de la protéine d'intérêt.
En ce qui concerne les cassettes d'expression, elles
consistent en une organisation de différentes séquences
intervenant non seulement dans la régulation et le contrôle de
l'expression des gènes, mais aussi dans le maintien et la
stabilité du vecteur.
Dans le cas de la levure on distingue essentiellement 4
types de vecteurs qui différent par le mécanisme de leur maintien
:
YIP (Yeast Integrating Plasmid) : ce sont des vecteurs
auxquels il manque l'origine de réplication dans la levure, donc qui
doivent se propager comme un élément intégratif dans le
chromosome de la levure et le plus souvent dans une seule copie par
génome.
YRP (Yeast Replicatif Plasmid) : ce sont des vecteurs
qui présentent une séquence de réplication autonome
(ARS), ce sont des plasmides instables et à réplication
autonome.
YCP (Yeast Centromer Plasmid) : ces vecteurs
présentent une origine de réplication et une séquence
centromérique, ce sont des plasmides stables et qui se propagent en un
faible nombre de copies.
YEP (Yeast Episomal Plasmid) : ces vecteurs
présentent un fragment de plasmide de levure de 2um, ils se propagent en
un grand nombre de copies et ils ont une réplication autonome et une
ségrégation irrégulière.
Il est possible de diriger l'intégration du vecteur
ou du moins une partie dans un site spécifique dans le génome de
la levure, où il peut se répliquer d'une façon stable et
se maintient comme une partie du chromosome de l'hôte.
Vu que ces plasmides peuvent s'amplifier et s'isoler
facilement à partir d' E.coli, chacun de ces vecteurs est
désigné comme un vecteur navette qui inclut des séquences
permettant la sélection et le maintien du plasmide dans les deux
systèmes hôtes, des séquences qui permettent la
transcription et la traduction efficace du gène étranger, des
séquences qui permettent une bonne terminaison de la transcription et la
présence d'un promoteur constitutif et régulable. (12).
IV) Le système d'expression Pichia
pastoris :
IV.1) Principaux avantages du système :
Depuis 1980, la société
américaine Phillips Petroleum a créé une filiale, la
société SIBIA (Salk Institut Biotechnology Industrial
Associates), qui se consacre au développement et l'exploitation de
Pichia pastoris.
SIBIA a breveté cette levure, et a produit comme essai,
l'antigène HBs : son expression intracellulaire est
impressionnante. Les taux obtenus sont 5 à 100 fois plus
élevés que S.cerevisiae.
La société a également annoncé
quelle avait obtenue 6 à 10 g de TNF purifié par litre de
culture.
Lorsque les protéines hétérologues sont
secrétées, les taux obtenues sont plus faible mais ils restent
extrêmement intéressants : l'antigène HBs a
été produit à raison de 2 g/l, l'invertase 2,3g/l et un
taux qui dépasse 1g/l pour la sérum albumine humaine.
Plusieurs avantages sont offerts suite à l'utilisation
de pichia pastoris parmi ces avantages on peut citer :
· La facilité de manipulation.
· Taux d'expression important (10 à 100 fois plus
élevé que S.cerevisiea ).
· Pichia pastoris offre un choix d'expression de
la protéine recombinante sous forme intracellulaire ou
secrétée. (5) ; (7).
· Capacité d'effectuer plusieurs modifications
post-traductionnelles : formation de ponts disulfure, phosphorylation,
acylation, avec un repliement correct des protéines. (19).
· Etant méthylotrophiques, Pichia
pastoris possède la faculté d'assimiler le méthanol
comme une seule source de carbone et d'énergie, en effet cette levure
possède une voie d'utilisation de méthanol hautement inductible,
elle est capable de convertir 30 à 40% de son poids en protéines,
ces protéines qui sont synthétisées lorsque la levure
pousse sur le méthanol sont indétectable lorsque la levure pousse
sur le glucose ou le glycérol. (22).
· Les problèmes d'hyperglycosylation
observés chez S.cerevisiea sont beaucoup moins importants pour
Pichia pastoris.
· La stabilité de la cassette d'expression dans le
génome de la levure, sa capacité de sécrétion, les
faibles taux de protéines contaminants dans le milieu de culture et la
rapidité de croissance de la levure sur des milieux non coûteux et
dans les fermenteurs font de Pichia patoris un système de choix
pour l'expression de protéines hétérologues. (8).
Vu ces caractéristiques intéressantes :
certaines protéines (exemple la protéine G) qui n'ont pas
étaient sécrétées d'une manière efficace
chez les bactéries ou chez saccharomyces cerevisiae ou dans les
cellules d'insectes ont était obtenues avec succès dans
Pichia pastoris. (9) ; (19).
IV.2) Le métabolisme du
méthanol :
Etant méthylotrophiques,
Pichia pastoris possède la faculté d'assimiler le
méthanol comme une seule source de carbone, en effet, la première
étape du métabolisme consiste en une oxydation du méthanol
en formaldéhyde en présence de l'alcool oxydase.
Cette enzyme étant faiblement affine à
l'oxygène, se trouve produite en grande quantité.
Deux gènes distincts(AOX1 et AOX2) codent pour l'alcool
oxydase, le gène AOX1 est plus fortement induit par le méthanol
que le gène AOX2 et se trouve donc majoritairement responsable de
l'activité de l'enzyme dans la cellule.
L'expression de l'AOX1 se fait selon le mécanisme de
répression induction. En effet, la présence de n'importe quelle
source de carbone autre que le méthanol empêche l'expression du
gène, cependant, l'absence de cette source n'implique pas son
expression. Ainsi le gène AOX1 a été isolé, son
promoteur a été introduit dans des vecteurs pour diriger
l'expression du gène codant pour la protéine
désirée. (7).
V) Stratégie d'expression chez Pichia
pastoris :
V.1) Les vecteurs d'expression :
Les vecteurs d'expression construits
sont des YIP capables de se maintenir et de se propager dans les deux
systèmes hôtes E.coli et Pichia pastoris. Par
exemple des vecteurs utilisés pour l'expression des protéines
recombinantes dans P.pastoris on peut citer les vecteurs de la famille
pPICZ (figure1) qui comprennent :
- une séquence promotrice et terminatrice du
gène AOX1, séparées par plusieurs sites de restriction qui
servent à insérer la séquence à exprimer et par
deux séquences codant pour le myc epitope et un queue polyhistidine
(his6).
- un gène de résistance à la
zéocine et un gène HIS4 pour la sélection des souches
recombinantes.
- une origine de réplication chez E.coli.
Le vecteur peut inclure également une séquence
signal de sécrétion en amont de la séquence
d'intérêt pour obtenir une protéine
sécrétée.
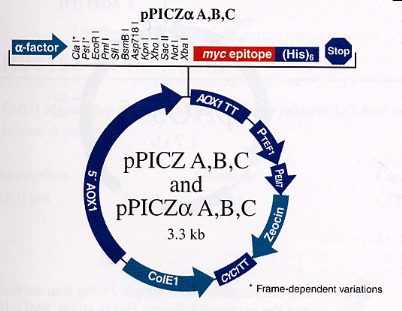
Figure1 : La famille des vecteurs pPICZ
V.2) Mode d'intégration du gène
d'intérêt :
Les vecteurs utilisés pour
l'expression des protéines recombinantes sont des YIP
c'est-à-dire qu'ils ne possèdent pas d'origine de
réplication chez la levure et donc ne peuvent se propager que par leur
intégration dans le génome de l'hôte.
L'insertion de la cassette d'expression se fait par
recombinaison homologue entre l'ADN plasmidique et les régions qui lui
sont homologues dans le génome de Pichia pastoris, cette
recombinaison dirigée par le gène AOX1 montre une grande
stabilité surtout lorsque le gène d'intérêt est
présent en plusieurs exemplaires.(9).
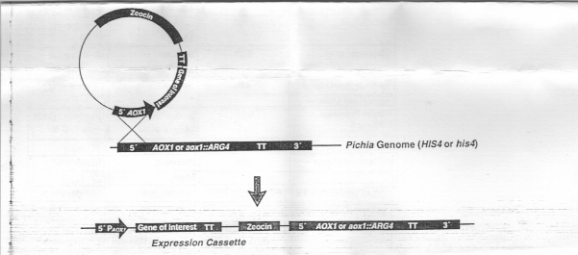
Figure2 : Schéma explicatif de
l'insertion de la cassette d'expression dans le génome de pichia
pastoris
V.3) Le nombre de copies de la cassette
d'expression :
Généralement on montre que le nombre
élevé de cassettes d'expression (>10) dans le génome de
la levure se traduit par une augmentation du taux d'expression de la
protéine d'intérêt. Plusieurs exemples montrent que
l'insertion d'une simple cassette d'expression est largement suffisante pour un
optimum de production et l'augmentation du nombre de copies n'à pas
toujours un effet significatif sur la production des protéines
d'intérêt. (9).
V.4) Modifications post traductionnelles chez Pichia
pastoris :
Parce que chaque protéine
présente un cas particulier et parce que les propriétés et
la conformation de la protéine produite doivent être proches de
l'état natif, certains d'entre elles doivent subir des modifications
post traductionnelles pour remplir leur fonction.
Chez Pichia pastoris, la glycosylation des
protéines recombinantes est semblable à celle de
saccharomyces puisqu'elle met en jeu des résidus mannoses
N-liés et O-liés. Cependant, Pichia pastoris
présente un avantage par rapport à
saccharomyces : c'est que l'hyperglycosylation est beaucoup moins
importante. En effet, le nombre de résidus mannose ajouté aux
protéines est de 50 à 150 par chaîne pour
saccharomyces alors qu'il n'est que de 8 à 14 chez
Pichia. (20).
V.5) Les facteurs agissant sur la cinétique de
croissance de Pichia pastoris :
La cinétique de croissance des
organismes microbiens dépend de plusieurs paramètres : la
composition du milieu de culture, le mode d'alimentation (Batch, fed batch,
continu), Les conditions physico-chimiques (pH, Température,
agitation...).
Les conditions optimales pour Pichia pastoris sont
les suivantes :
Température : 30°C
PH: 5
Pression d'oxygène : 50% en fermenteur et un
rapport volume/volume=0,2 en Erlenmeyer.
Il faut signaler aussi que chez Pichia pastoris, la
nature et la concentration de la source de carbone influent directement sur sa
cinétique de croissance, en effet, une haute densité cellulaire
est obtenue sur glycérol, par contre, lorsque le méthanol est
utilisé comme source de carbone, le rendement en biomasse est plus
faible. (9).
V.6) Les facteurs agissant sur la cinétique de
production :
Chez Pichia pastoris, la phase
de production est découplée à la phase de croissance, ce
qui représente un avantage considérable, du fait qu'on peut
suivre la production de la protéine recombinante par le suivi de la
consommation du substrat inducteur : le méthanol.
En effet, pendant la phase de croissance le substrat
utilisé est le glycérol. Ce substrat exerce une répression
sur le promoteur AOX1 qui régule l'expression du gène
d'intérêt. Une fois le glycérol est épuisé du
milieu, la répression est levée et la phase de production peut
être initiée par l'utilisation du méthanol, substrat qui
induit l'expression des gènes sous le contrôle du promoteur AOX1
et donc la production de la protéine d'intérêt. Ainsi, la
concentration du méthanol représente un des facteurs les plus
influents sur le taux de production. (11).
VI) La tuberculose :
La tuberculose est une infection
bactérienne, qui reste l'une des plus grands fléaux qui touche
l'homme avec plus de dix millions de nouveaux cas et trois millions de
décès chaque année. (13). Elle est commune à
l'homme et à de nombreuses autres espèces animales. Elle est due
essentiellement aux bactéries appartenant au complexe
Mycobactérium tuberculosis et à diverses espèces
bactériennes appartenant au genre mycobactérium.
Le tiers de la population humaine est infecté par
M. tuberculosis, pathogène intracellulaire facultatif, mais
seulement 5 à 10% de cette population risquent de développer la
maladie. (21). Cette situation devient de plus en plus alarmante avec
l'émergence de souches multirésistantes et le SIDA. (14).
De 1908 à 1920 Calmette et Guérin
élaborent un vaccin qui porte leur nom : Bacille Calmette
Guérin (BCG). Il est employé pour la première fois
en 1921 et marque une étape importante dans la prophylaxie de la
maladie. La vaccination avec le BCG reste le moyen le plus efficace
jusqu'à nos jours mais cette efficacité et de plus en plus mise
en cause (1). Le taux de protection de ce vaccin varie, en fait de 0 à
80%. (3).
Cette situation de plus en plus alarmante, rend urgent
l'élaboration d'outils plus efficaces pour la lutte antituberculeuse,
à savoir, le développement des tests de diagnostic à haut
potentiel prédictif de la maladie, des nouveaux produits
thérapeutiques antituberculeux et de préférence, un vaccin
plus efficace que le BCG.
VI.1) Les bacilles de la tuberculose :
Les bactéries du genre Mycobacterium
sont des bacilles qui ne se colorent pas facilement mais qui, une fois
colorées, résistent à la décoloration par l'acide
et l'alcool et sont de ce fait dits bacilles «
acido-alcoolo-résistants ». Le genre comprend de nombreuses
espèces saprophytes ou commensales et des espèces
pathogènes dont les deux principales sont : Mycobacterium
tuberculosis, agent de la tuberculose, et Mycobacterium leprae,
agent de la lèpre.
M.tuberculosis est un bacille aérobie strict
immobile sans capsule et sans spore. Après coloration de ZIEHLNEELSEN
(fuchine phéniquée à chaud, décoloration par
acide-alcool, recoloration par le bleu de méthylène), il
apparaît comme un bacille rouge de 0,2 à 0,3 micron de large sur 3
à 5 microns de long, légèrement incurvé, à
extrêmités arrondies. (3).
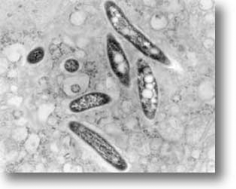
Figure3 :
Mycobactérium tuberculosis
VI.2) Pouvoir pathogène de M.
tuberculosis :
Contrairement aux autres bactéries
pathogènes, M. tuberculosis ne possède pas de facteur de
virulence classique, de type toxine. Chez l'homme, M. tuberculosis
utilise l'appareil respiratoire comme principale voie d'entrée. Il
résiste à la dégradation par les macrophages
alvéolaires, dans lesquels il est capable de se multiplier. Un des
éléments cruciaux pour la survie de M. tuberculosis dans
les phagosomes des macrophages est sa capacité à en diminuer
l'acidification, ce qui a pour conséquence de bloquer ou de retarder la
fusion du phagosome avec le lysosome. Au niveau moléculaire, ce
phénomène n'est pas clairement élucidé, la
détermination des mécanismes impliqués constitue un des
enjeux majeurs de la recherche dans ce domaine. La réponse immunitaire
de l'hôte contrôle la sévérité de l'infection
par M. tuberculosis. Cette réponse immunitaire met en jeu la
voie Th1, qui recrute des cellules dendritiques, des macrophages et des
lymphocytes de type CD4 et CD8. Les bases moléculaires de la
pathogénicité sont donc multifactorielles, impliquant à la
fois la bactérie et l'hôte. (3).
VI.3) Diagnostic bactériologique :
Le diagnostic de certitude de la tuberculose repose sur
la mise en évidence de Mycobacterium tuberculosis dans les
prélèvements pathologiques : crachats et tubages gastriques pour
la tuberculose pulmonaire ; urines, liquides de ponction des séreuses,
etc... pour les autres localisations tuberculeuses. (10).
L'examen microscopique des frottis colorés par la
méthode de ZIEHL-NEELSEN permet de mettre en évidence des
bacilles acido-alcoolo-résistants. Lorsqu'il est positif il permet un
diagnostic de forte présomption de tuberculose. (10).
Seul la culture permet un diagnostic de certitude de la
tuberculose. M.tuberculosis ne pousse pas sur les milieux usuels. Il
nécessite des milieux très enrichis. Le plus employé est
un milieu à l'oeuf, le milieu de LOEWENSTEIN-JENSEN. Sur ce milieu il
donne des colonies de teinte crème beige, sèches, à
surface rugueuse, en chou-fleur, tout à fait caractéristiques.
Fait important, les colonies n'apparaissent qu'en 21 jours en moyenne (temps de
division de M.tuberculosis = 20 heures).
Or le délai long entre le début de l'exploration
et le traitement augmente la morbidité et la mortalité par la
maladie pour cela et depuis très longtemps, les chercheurs tentent
d'utiliser la sérologie pour un diagnostic rapide. (3).
VI.4) Séquençage de génome de la
souche H37Rv :
M. tuberculosis H37Rv a
été la première souche pathogène isolée en
1905, c'est la souche de référence et la plus largement
utilisée dans les recherches dans le domaine de la tuberculose.
En 1998, le séquençage total du
génome de la souche H37Rv a constitué un tournant
dans l'histoire des bacilles tuberculeux ouvrant ainsi
plusieurs voies de recherches. Ainsi, l'identification de nouveaux gènes
à potentiel vaccinal, diagnostic et thérapeutique est rendue, de
loin, plus facile par la génomique, la transcriptomique et la
protéomique.
Le génome de M. tuberculosis H37Rv comprend
4.411.529 paires de bases et 3924 cadres de lecture ouverts ont
été identifiés. L'analyse des banques de données a
permis d'attribuer à 40% de ces séquences codantes une fonction
prédite. (6).
VI.5) Le gène Rv3874 et l'antigène
CFP10 :
L'analyse du génome a aussi indiqué la
présence de gènes codant pour des protéines
présentes dans le filtrat de culture de M. tuberculosis et donc
sécrétées. Un de ces protéines est CFP10 (culture
filtrat protein), une petite protéine codée par le gène
Rv3874, c'est un antigène qui est fortement reconnu par les lymphocytes
T humains et provoque une production massive d'IFNã cytokine
nécessaire à l'activité bactéricide du macrophage.
Des études récentes ont montré que cette protéine
est impliquée dans le pouvoir pathogène de M.
tuberculosis. Lors des études de génomique
comparative employant différentes techniques d'hybridation,
plusieurs régions de différence (RD1-RD14), codant pour
environ 140 protéines, ont été trouvées
absentes chez M. bovis BCG, la souche vaccinale, par rapport
à la souche virulente M. tuberculosis H37Rv. Ces
résultats permettent d'étudier de façon
approfondie les différences génétiques potentiellement
impliquées dans la virulence de certains membres du
complexe M. tuberculosis. Par exemple, la région RD1,
qui contient le gène Rv3874, est la seule région absente dans les
souches atténuées M. bovis BCG, et M. microti,
mais elle est présente chez tous les autres membres du complexe M.
tuberculosis. Comme le BCG, la plupart des souches de M. microti
sont inoffensives pour l'homme. Ainsi, M. microti a été
utilisée comme vaccin dans les années 1960 en
Tchécoslovaquie et a fait l'objet de larges essais cliniques au
Royaume-Uni. Lors de la réintroduction de la région RD1 par
complémentation dans le BCG et dans M. microti, la virulence de
ces deux souches vaccinales est partiellement restaurée chez la souris
immunodéprimée. En revanche, la réintroduction de cinq
autres régions de différence (RD3, RD4, RD5, RD7 et RD9),
suspectées d'être impliquées dans la virulence, ne semble
pas avoir d'effet sur le pouvoir pathogène de ces deux souches. Comme la
région RD1 comprend aussi le gène codant pour l'antigène
protéique CFP10, il est maintenant évident que toutes les souches
vaccinales employées à grande échelle dans l'histoire de
la vaccination contre la tuberculose étaient dépourvues de cet
antigène, qui est immunodominant et semble diriger la réponse
immunitaire de l'hôte vers la voie Th1, entraînant la production
d'IFNã. En effet, l'expression de cet antigène dans le contexte
d'un BCG recombinant, ainsi que celle d'autres protéines de la
région RD1 impliquées dans la sécrétion duCFP10,
augmente l'efficacité du BCG vis-à-vis d'une inoculation avec
M. tuberculosis dans différents modèles animaux.
Dès lors, les protéines de la région RD1 sont
considérées comme des cibles potentiellement très
intéressantes dans la prévention (antigène protecteur), le
diagnostic (remplaçant le PPD - épreuve à la tuberculine
-) et la thérapie (cible de médicament). (3).
I) MATERIEL :
I.1) Souches Bactériennes et levures
utilisées :
La souche bactérienne d'Escherichia coli
utilisé est :
- Top10F' : F' [
lacIq Tn10 (TetR) ]
mcrA Ä(mrr-hsdRMS-mcrBC)
80lacZÄM15 ÄlacX74deoR recA1
araD139 Ä(ara-leu) 7697 galU galK
rspL (StrR) endA1 nupG.
- La souche de la levure Pichia pastoris
utilisée est KM71H de phénotype :
arg4 aoxl : ARG4 et de génotype Muts,
Arg+. Elle est fournie par la société Invitrogen.
Cette levure présente plusieurs avantages pour
l'expression de protéines recombinantes par rapport aux systèmes
classiques :
- Un taux d'expression élevé de protéine
de fusion : pouvant aller jusqu'à 12 g/l.
- Un « Scale up » plus facile : le
passage à l'expression en fermenteur est plus facile avec
Pichia qu'avec les bactéries.
- Pichia pastoris s'adapte plus facilement aux
conditions de fermentation que ce soit à des petits volumes (< 1
litre) qu'à des grands volumes (>10 000 litres).
- Un système de sélection simple, varié
et efficace.
Pichia est un système d'expression eucaryote
d'où l'avantage de réaliser des modifications
post-traductionnelles des protéines eucaryotes.
I.2) Vecteur de clonage et d'expression :
I.2.1) pcDNA3/Rv3874 :
Le gène Rv3874 a
été cloné entre les sites Bam H I et Xho I dans le vecteur
pcDNA 3 cette construction qui a servi comme matrice pour amplifier l'ADNc
du gène Rv3874 nous a été aimablement fournie par le Dr.
Helmi MERDASSI.
I.2.2) pPICZA (Invitrogen):
C'est un vecteur navette de clonage et d'expression
dans Pichia pastoris de 3593 pb, qui appartient à la famille
pPICz(fig.1).
Les sites de clonage au niveau de ce vecteur se trouvent entre
la séquence du côté COOH terminal codant pour -factor, en
amont, qui est un peptide signal de saccharomyces cereviseae
permettant la sécrétion de la protéine recombinante dans
le milieu extracellulaire ; et en aval du côté NH2
terminal les séquence codant pour :
- L'épitope myc qui permet une détection facile
des protéines recombinantes soit par ELISA ou par Western Blot.
- Une queue poly-Histidine (6 His) permettant la purification
et la détection rapide des protéines recombinantes
exprimées dans Pichia pastoris
Ce vecteur de clonage présente également :
- Le promoteur AOX1 inductible par le méthanol, ce
promoteur permet l'obtention de taux élevés d'expression de
protéines recombinantes.
- Le gène de résistance à la
zéocine sous le contrôle du promoteur EM7 pour une
sélection dans E.coli et le promoteur TEF1 pour une
sélection dans Pichia pastoris
- une origine de réplication ColE1 dans
E.coli
Le vecteur pPICZA est utilisé pour le clonage du
gène Rv3874 et la transformation de la souche Top10F'. Il a
été également utilisé pour la transformation de
Pichia pastoris (voir figure1).
I.3) Les enzymes :
I.3.1) Les enzymes de restriction :
Ces sont des endonucléases qui coupent de
manière définie et reproductible l'ADN double brin.
Les enzymes utilisées au laboratoire sont des enzymes
de type II qui coupent donc au niveau d'un site de reconnaissance
spécifique.
Au cours de ce travail nous avons utilisé les enzymes
de restriction suivantes :
Tableau 1 : les enzymes de restriction utilisées au
cours de ce travail et leurs propriétés
|
Enzymes
|
Unité/ul
|
Fournisseur
|
T° d'incubation
|
|
BstX I
|
10
|
Amersham
|
45°C
|
|
Eco RI
|
15
|
Amersham
|
37°C
|
|
Xba I
|
15
|
Amersham
|
37°C
|
I.3.2) Les autres enzymes :
Ampli Taq DNA polymérase (Amersham) :
Utilisée lors des réactions de PCR et de
séquençage automatique extraite de Thermus aquaticus.
Cette enzyme possède une mutation dans le domaine
amino-terminal qui élimine l'activité nucléase 5'-3' de
l'Ampli Taq DNA polymrase.
L'enzyme est aussi équipée d'une pyrophosphatase
inorganique thermostable qui élimine les problèmes
associés à la pyrophosphorolyse.
T4 DNA ligase (Amersham) :
Cette enzyme extraite de bactéries infectées par
le phage T4 assure la ligation des brins d'ADN aussi bien à bouts francs
que cohésifs.
I.4.) Les marqueurs de poids
moléculaire :
I.4.1) Marqueurs de poids moléculaire
d'ADN :
2
1

Figure 4 : Marqueurs de poids moléculaire d'ADN
utilisés
1: PhiX-DNA-HaeIII Markers
2: Lambda DNA-HindIII Digest
I.4.2) Marqueur de poids moléculaire de
protéines :

Figure
5 : Marqueurs utilises de poids moléculaire de protéines
RPN 800 (Amersham)
I.5) Les oligonucléotides:
Le couple d'amorces CH1/CH2 (Progligo) : a servi pour l'amplification par
PCR et le clonage du gène Rv3874 dans le plasmide pPICZA entre les sites
de restriction EcoRI et XbaI.
CH1 :
CCGGAATTCGCAGAGATGAAGACC
CH2 :
GC TCTAGACGGAAGCCCATTTGCGA
Le couple d'amorces AOX1F/AOX1R (invitrogen) a servi au
séquençage automatique du gène Rv3874 cloné dans
pPICZA.
I.6) Autres matériels :
-
Les kits :
Kit de purification d'ADN à partir de gel d'agarose : Quiaquick Gel
Extraction Kit (QIAGEN).
Kit de purification d'ADN plasmidique (mini préparation des
plasmides) :QIAprep Spin Miniprep Kit (Qiagen).
Kit de purification d'ADN plasmidique (midi préparation des
plasmides) :QIAprep Spin Midiprep Kit (Qiagen).
-
Appareil PCR: Perkin Elmer ,Gene Amp9700.
-
Incubateur agissant thermostaté :INFORS AG , Rittergasse27,
CH-4103 Bottmingen.
-
Séquenceur automatique : ABI PRISMTM 377 sequencer
(PERKIN ELMER Applied Systems).
II)
METHODES:
II.1) PCR :
L'extraction de l'ADNc de Rv3874, à partir du plasmide p CDNA 3, a
été réalisée par PCR. En fait le couple d'amorces
CH1/ CH2 a été utilisé pour amplifier le gène
Rv3874, en introduisant les sites de clonage EcoRI en amont et XbaI en
aval.
Les
conditions de la réaction PCR sont les suivantes :
-
1à10 ng d'ADN/tube.
-
Tampon 10X :5l
-
dNTP(25Mm) :0,5l
-
MgCl2 (25mM) : 2,5 ul
-
Primer forward (20 uM) : 0.5 ul
-
Primer reverse (20 uM) : 0.5 ul
-
Taq poly (5 U/ul) : 0.5 ul.
-
H2O qsp 50 ul.
Les
cycles d'amplification sont comme suit :
-
2 min 94°C
-
30 cycles: - 30 s 94°C
- 30 s 55°C
- 30 s 72°C
-
7 min 72°C.
II.2) Electrophorèse sur gel
d'agarose :
Les profils éléctrophorétiques des produits de PCR, de
digestion, de mini ou midi préparation ont été
analysés après électrophorèse sur gel d'agarose de
0.8 % à 1%, TAE 1 X. Les échantillons ont été
contrôlés sur le gel après avoir été
mélangés avec un tampon d'échantillon (50%
glycérol, 10 mM EDTA et du bleu de bromophénol à 1%)
à une dilution finale de 1/6.
II.3) Purification d'ADN à partir du gel :
Après migration sur gel d'agarose « low
melting » les bandes spécifiques de l'ADN à purifier
sont coupées sous U.V et pesées.
Le
Kit Qiagen permet la purification des fragments d'ADN qui se fait sur une
colonne Tip-20 (colonne de silice chargée positivement) en utilisant les
réactifs du kit.
L'élution
se fait avec 50ul de tris-HCI pH 8, la purification est contrôlée
par électrophorèse sur gel d'agarose.
II.4) La digestion :
Afin de cloner le gène Rv3874, dans le bon sens, dans le vecteur pPICZA
entre les sites choisis EcoR I et Xba I, l'insert et le plasmide sont
digérés par ces enzymes de restriction.
Les
conditions de digestion sont les suivantes :
Tableau
2 : Mélange réactionnel de la digestion par Xba I et EcoR
I
Le tampon 10 X M est choisi parce qu'il permet une activité de 100% pour
les enzymes EcoR I et Xba I.
Le
mélange réactionnel est incubé à une
température de 37°C dans un bain marie pendant six heures.
Les
produits de digestions sont contrôlés sur gel d'agarose, si la
digestion est totale nous procédons à la ligation, après
avoir purifié le produit de digestion.
II.5) La ligation :
La T4 DNA Ligase est une enzyme qui catalyse la formation d'une liaison
phopho-diester entre deux bouts d'ADN, 3'-OH libre et 5'-phosphate. Cette
réaction qui nécessite de l'ATP permet l'insertion du gène
désiré au sein du plasmide digéré et parfois la
recircularisation du plasmide lui-même.
La
réaction de ligation est réalisée à 16°C avec
une incubation de 16 heures du mélange réactionnel suivant :
-L'
ADN plasmidique digéré par les enzymes de restriction choisies et
purifiées.
-
l'insert à cloner est digéré par les mêmes enzymes
de restriction et purifié.
-
Le tampon de la T4 DNA ligase est dilué au 1/10 (final).
-
de la Ligase (10 U/ul) (sachant qu'1UI de T4 DNA ligase permet de liguer 1 ug
d'ADN).
II.6) Transformation des bactéries :
La souche Top 10 F' a été transformée par le produit
de ligation pPICZa/Rv3874 pour l'amplification du plasmide recombinant.
Pour
assurer la transformation des souches bactériennes on doit passer en
premier lieu par la préparation de cellules compétentes.
Plusieurs
méthodes sont disponibles mais nous avons utilisé au cours de ce
travail la méthode de TSS(Transforming and Storage Solution).
Méthode
du TSS.
Une colonie ( Top 10f') isolée est suspendue stérilement
dans respectivement 5 ml de milieu LBLS, et mise en culture pendant une nuit
à 37°C sous agitation à 220 rpm. Un ml de cette culture est
transféré dans un erlen de 500 ml stérile contenant 50 ml
de milieu LBLS.
Les
cellules sont incubées à 37°C sous agitation à 220
rpm jusqu'à ce que DO600 nm atteigne 0,4 à 0.6
unité. La culture est alors mise dans de la glace pendant 30 min pour
stopper la prolifération bactérienne. Les cellules sont par la
suite centrifugées à 1200 g pendant 15 min à
4°C.
Le
culot cellulaire est resuspendu dans 4 ml de solution de TSS froide, contenant
le DMSO, agent fragilisant la membrane cellulaire, puis le mélange est
réparti dans des tubes à raison de 150 ul par tube eppendorf.
A
ce stade on a des cellules compétentes qu'on incube par la suite en
présence de 7 à 15 ng d'ADN plasmidique tout en gardant tout dans
la glace pendant 30 min, puis tout est transféré dans un bain
marie à 42°C pendant 1min afin d'effectuer un choc thermique
permettant l'entrée des plasmides dans les bactéries.
On
ramène de nouveau les tubes dans la glace. On ajoute 350 ul de milieu
LBLS et on incube pendant 45 à 60 min à 37°C.
100
ul du produit de la transformation sont étalés sur milieu LBLS
contenant 1,5 % d'agar + 25 ug/ml zéocine. Les boites de culture sont
incubées pendant une nuit à 37°C.
Les colonies qui poussent sont criblées par colonie PCR
II.7) Colonie PCR :
C'est une PCR qui s'effectue directement sur une colonie
bactérienne permettant ainsi un criblage rapide et fiable des clones
transformés.
Une
colonie est resuspendue directement dans le mélange réactionnel
de la PCR, chauffée pendant 4 min à 95°C (hot start), on
effectue par la suite une PCR comme déjà décrit.
II.8) Préparation des plasmides :
II.8.1) Préculture
bactérienne :
A partir d'un stock de bactéries, des colonies sont
isolées par ensemencement sur boîte de LBLS(Luria Bertani Low Solt
Medium) contenant 20% d'agar et de zéocine à 25
ug/ml.
Une
colonie isolée est incubée dans 5 ml de milieu LBLS à 25
ug par ml de zéocine pendant une nuit à 37°C sous agitation
de 220 rpm.
Cette
préculture sert à ensemencer 50 ml de milieu LBLS liquide qui
seront incubés dans les mêmes conditions.
II.8.2) Extraction de l'ADN plasmidique :
Nous avons utilisé le kit Qiagen pour l'extraction de l'ADN
plasmidique. La technique d'extraction se base sur la lyse alcaline des
cellules bactériennes. Le kit miniprep permet d'extraire l'ADN
plasmidique à partir de 5 ml de culture.
Après
extraction, on contrôle les formes et les tailles de l'ADN plasmidique
par électrophorèse sur gel d'agarose ;
On
peut également doser l'ADN par spectrophotométrie à 260
nm, en utilisant la relation : une unité de DO correspond à
une solution d'ADN de concentration égale à 50 ug/ml.
La
pureté de l'ADN est estimée par une lecture de la densité
optique à 280 nm, le rapport DO260/280 doit être compris entre 1,6
et 2.
II.9) Electroporation de la levure Pichia
pastoris :
Une colonie de KM71H (sensible à la zéocine), isolée
sur boîte YPD(Yeast extract Peptone Dextrose Medium), est mise en culture
dans 100 ml de BMGY(Buffered complex medium containing glycerol):
jusqu'à ce que la DO600 atteigne une valeur entre 1.3 et 1.5.
Le
culot cellulaire est lavé avec 100ml d'eau stérile froide
après avoir stoppé la culture dans la glace.
Le
culot est ensuite lavé avec 4 ml de sorbitol 1M, très froid
(0°C) puis resuspendue dans 250 ul de cette solution.
A ce stade on a des cellules compétentes.
Dans
une cuve d'éléctroporation on mélange 80 ul de cellules
compétentes (D.O = 1 ,5) avec 5 à 10 ug du plasmide
recombinant pPICZA/Rv3874 qui a été linéarisé par
Bst X I et purifié.
Après
avoir fixé le voltage à 1500 V et la capacitance sur 25 uF, la
cuve est mise dans un éléctroporateur « BIORAD gene
pulser ». On procède de telle manière à ce que
la durée du choc électrique soit entre 5 et 10 ms.
Après
l'addition du sorbitol 1M et l'incubation des cellules dans l'étuve
à 30°C pendant 2 heures, on étale les cellules sur des
boites YPDS zéocine (150ug/ml) à raison de 250 ul par boite.
Les
boites sont incubées à 30°C pendant 2 à 3 jours, les
colonies qui poussent sont normalement des transformants.
L'éléctroporation
permet d'internaliser le vecteur de clonage propre à Pichia
pastoris, qui va s'intégrer par recombinaison homologue dans le
génome de la levure. Cette recombinaison est dirigée par les
séquences homologues du gène AOX1.
II.10) Production de la protéine recombinante
dans Pichia pastoris :
Le clone choisi pour la production est mis en culture dans 50 ml de BMGY
sous agitation de 250 rmp à 30°C, jusqu'à ce que la DO600
atteigne une valeur comprise entre 2 et 6.
Le
culot cellulaire est ensuite resuspendu dans 5 ml (1/10ème de
volume initiale) de BMMY (Buffred Methanol-complex Medium) à 1% final de
méthanol.
Chaque
24 heures, on ajoute du méthanol à 1% final et on
récupère 100 ul de culture pour suivre la cinétique de la
production à 24, 48 et 72 heures.
II.11) Electrophorèse des protéines sur gel de
polyacrylamide en présence de SDS
(SDS-PAGE :SDS-Polyacrylamid Gel Electroporesis) :
Pour un gel de migration prévoir 15 ml de gel de séparation
et 5 ml de gel de concentration :
a)
Le gel de séparation : 15 %
b/
Le gel de concentration :
-
Monter les plaques sur le support de polymérisation.
-
Couler d'abord 5ml de gel de séparation (jusqu'au premier écran
du support).
Compléter
avec de l'eau pour aplatir la surface du gel. Après
polymérisation du premier gel, éliminer l'eau avec du papier
filtre et couler en haut le gel de concentration. Laisser polymériser
après avoir mis le peigne.
-
Après polymérisation, placer le montage sur le support de
migration et poser l'ensemble dans la cuve vide (sans tampon). Couler ensuite
le tampon de migration.
-
Déposer les échantillons (préalablement
additionnées de 1/3 de volume de tampon d'échantillon 3X et
dénaturer 5' à 100°C). Déposer aussi le marqueur de
taille RainBow (3ul). Faire migrer pendant environ 45' sous un voltage de 120
V.
A
la fin de la migration, les protéines séparées selon leur
poids moléculaire sont soit colorées au bleu de Coomassie soit
transférées sur membrane de nitrocellulose (technique
d'immunoblot).
II.12) Immunoblot et révélation par
ECL : « Enhanced ChemiLuminescence » :
Après migration et révélation de protéines par
le bleu de Coomassie on veut vérifier si la protéine
récupéré dans le surnageant est bien la protéine
recombinante CFP10-myc-6 his.
Pour
cela on procède à la réalisation d'un immunoblot.
Le
système western Blot ECL (AMERSHAM) est une méthode non
radioactive d'émission de lumière pour la détection
d'antigène spécifique immobilisé, conjugués
directement ou indirectement à des anticorps couplés à la
HRP (Horse Radish Peroxydase).
Après
migration sur gel de polyacrylamide les protéines sont
transférées sur une membrane de nitrocellulose.
La
membrane est ensuite mise dans une solution de saturation (PBS 1X, tween 0.2%,
lait écrémé 3%) puis lavée avec une solution de
lavage (PBS 1 X, tween 1%).
Après
incubation avec les anticorps marqués, on lave de nouveau pour
éliminer les fixations non spécifiques des anticorps et on
révèle par autoradiographie en utilisant le kit ECL d'Amersham.
I)
Clonage de gène Rv3874 dans le vecteur pPICZA :
I.1) Stratégie de Clonage :
L'ADNc
du gène Rv3874 dont la séquence en acides nucléiques est
ci-dessous a une taille de 303 pb et a été extrait à
partir du plasmide pcDNA3:
>Mtub_H37RV:|CFP10|Rv3874
atggcagagatgaagaccgatgccgctaccctcgcgcaggaggcaggtaatttcgagcgg
atctccggcgacctgaaaacccagatcgaccaggtggagtcgacggcaggttcgttgcag
ggccagtggcgcggcgcggcggggacggccgcccaggccgcggtggtgcgcttccaagaa
gcagccaataagcagaagcaggaactcgacgagatctcgacgaatattcgtcaggccggc
gtccaatactcgagggccgacgaggagcagcagcaggcgctgtcctcgcaaatgggcttc
tga.
Cette séquence a été analysée par le logiciel
BioEdit® qui nous a fourni la carte de restriction totale du
gène Rv3874 :
BalI
AvaI
EcoRII
Bal
I
HincII
EcoRII
255
250
153
121
101
90
1
pb
300
pb
-Carte
de restriction simplifiée du gène Rv3874-
Pour le clonage nous avons choisi les deux enzymes EcoR I et Xba I, qui
ne coupent pas au niveau de gène Rv3874 et qui coupent au niveau de site
de clonage multiple de plasmide pPICZA qui est un vecteur navette
destiné à la transformation de la levure Pichia
pastoris.
A
fin d'amplifier spécifiquement le gène d'intérêt
nous avons conçu les amorces de telle façon à obtenir les
jonctions suivantes tout en respectant les cadres de lectures de la
protéine d'intérêt et ceux des peptides en aval.
a
/ Jonction Foward avec EcoR I :
5'...GCT
CAA GCT GAA TTC GCA GAG ATG AAG ACC...3'
3'...CGA
GTT CGA CTT AAG CGT CTC TAC TTC TGG...5'
á Factor EcoRI CFP10
b / Jonction Reverse avec Xba I:
5'...CTG
CAA ATG GGC TTC CGT CTA GAA CAA AAA...3'
*
*
3'...GAC
GTT TAC CCG AAG GCA GAT CTT GTT TTT...5'
CFP 10 XbaI myc
épitope
* A fin de respecter le cadre de lecture de l'épitope myc deux bases C
et G ont été ajoutées en s'assurant que l'acide
aminé ajouté soit neutre qui est l'alanine.
Après
cette étude nous avons choisi les amorces de clonage
suivantes :
CH1 :
CCGGAATTCGCAGAGATGAAGACC
Tm=
46°C.
CH2 :
GC TCTAGACGGAAGCCCATTTGCGA
Tm=
54°C.
Les
parties soulignées correspondent aux séquences Rv3874.
I.2) PCR :
Les
amorces CH1 et CH2 ont été utilisés pour amplifier le
gène Rv3874 en introduisant les sites de restrictions EcoRI et XbaI pour
le clonage dirigé dans pPICZáA.
Après
optimisation on a amplifié par PCR le gène Rv3874 pour avoir une
quantité d'ADN suffisante pour le clonage.
Le
produit de PCR est contrôlé sur un gel d'agarose 1%.
M
1
2
300 pb

Figure1 :
Produits de PCR de gène Rv3874
M :
marqueur de poids moléculaire PhiX.
1 :
2ul de produit d'amplification de gène Rv3874 par PCR.
2 :
Contrôle négatif de PCR.
La
taille du gène Rv3874 est de 303pb, ce qui correspond bien à la
taille observée au niveau de piste 2.
I.3) Digestion et purification du produit PCR et du plasmide
pPICZáA :
Afin de cloner le gène Rv3874 dans le vecteur
pPICZáA le plasmide ainsi que l'insert ont été
digéré par les deux enzymes de restrictions
EcoRI et XbaI puis purifiés à partir de gel d'agarose
à faible température de fusion (low melting).
M
![]()
2
1
Figure 2 : Digestion totale de
pPICZáA par EcoRI et XbaI.
Gel
d'agarose 0,8%
M :
marqueur de poids moléculaire lambda DNA-HindIII Digest
1 : pPICZáA digéré totalement avec
EcoRI et XbaI.
2 : pPICZáA non digéré.
La
digestion totale du plasmide pPICZáA (piste1) montre une
linéarisation du plasmide d'où la disparition des
différentes formes de plasmide observées au niveau de la piste
2.
Après
digestion, le vecteur pPICZáA et le produit de PCR sont purifiés
en utilisant la technique d'extraction sur gel.
Les
différentes élutions d'ADN purifiés sont
contrôlés sur un gel d'agarose 0,8
3
2
1
M
![]()
300 pb
Figure 3 : Purification de produits de PCR et pPICZáA
digérés par EcoRI et XbaI.
Gel d'agarose 0,8%.
M :
marqueur de poids moléculaire ÖX.
1 :
1,5 ul de produit d'amplification par PCR de gène Rv3874.
2 :
1,5 ul première élution de pPICZáA
digéré.
3 :
1,5 première élution de produits de PCR
digéré.
Les
deux premières élutions contrôlées sont
utilisées pour la ligation.
I.4) Ligation et transformation des bactéries
compétentes E.coli top 10F'.
Le produit de la ligation a été utilisé pour
transformer des bactéries E.coli top 10 F' compétentes
initialement sensibles à la zéocine.
1.5)
Colonie PCR :
Les
colonies qui poussent sur des boites LBLS agar/zéocine sont des
transformants ayant intégré le plasmide recombinant, la
présence de l'insert est en premier lieu vérifiée par
colonie PCR.
Le
produit de colonie PCR est contrôlé sur gel d'agarose 1%.
5
4
3
2
1
M
C
300 pb
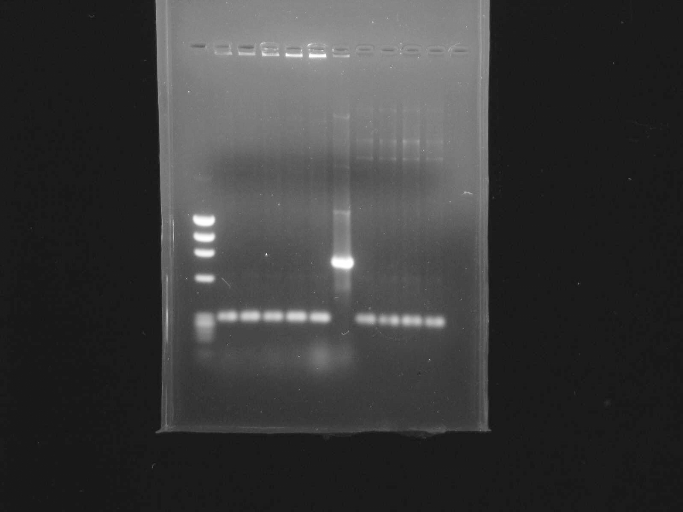
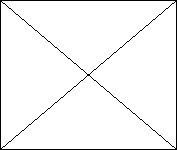
Figure 4 : Contrôle de la colonie PCR,
Gel d'agarose 1%. 5ul de dépôt par piste.
C : Contrôle négatif de PCR.
M : marqueur de poids moléculaire ÖX
1 : contrôle positif de PCR, la matrice étant
l'ADN plasmidique pcDNA3/Rv3874.
2, 3, 4, 5 : colonie positives.
Les quatre clones testés montrent une amplification
identique au témoin positif (C+) correspondant à l'amplification
spécifique par PCR du gène Rv3874.
I.5) Carte de restriction du plasmide
recombinant :
Pour vérifier l'intégrité de notre
construction, le plasmide recombinant a été digéré
par les mêmes enzymes de clonage EcoRI et XbaI, afin de libérer
l'insert du plasmide.
Le produit de digestion est contrôlé sur gel
d'agarose 0,8%.
2
1
300 pb
![]()
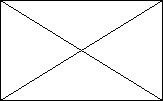
Figure 5: Digestion du plasmide recombinant
pPICZáA/Rv3874 par EcoRI et XbaI
1 : pPICZáA/Rv3874 digéré par EcoRI et
XbaI
2 : Marqueur de poids moléculaires PhiX
La digestion libère un fragment d'ADN de 303 pb qui
correspond au gène Rv3874, donc on peut conclure de la réussite
du clonage. La construction obtenue est représentée par la figure
suivante :

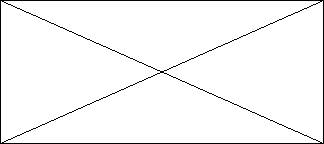
Figure 6 : plasmide recombinant
pPICZáA/Rv3874
I.6) Séquençage automatique :
Vu que la Taq polymérase peut introduire des
mutations au niveau des séquences amplifiées,
l'intégrité de la séquence nucléotidique de notre
construction a été vérifiée par
séquençage automatique du plasmide recombinant en utilisant
l'amorce AOX (F) et CH1 et de séquençage à l'aide d'un
séquenceur automatique : ABI PRISMTM 377 sequencer
(PERKIN ELMER Applied Systems).
1
6His
Xba I
myc epitope
Rv3874
EcoR I
Rv3874
á Factor
1
2
**
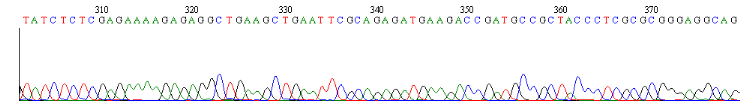
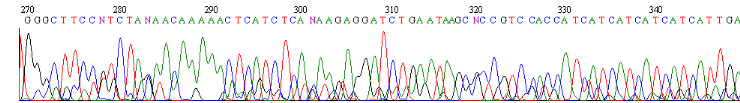
Figure7 : Résultat de
séquençage automatique du plasmide recombinant
pPICZáA/Rv3874
Schéma 1 : séquençage par l'amorce AOX
(F).
Schéma 2 : séquençage par l'amorce
CH1.
Le résultat de séquençage montre bien
l'intégrité de notre cassette d'expression, par la
présence des séquences du myc épitope, du 6histidine, des
deux sites de restriction EcoR I et Xba I, le á factor et aussi notre
gène d'intérêt Rv3874 ainsi que les deux bases qui ont
été ajouté pour garder le cadre de lecture de l'epitope
myc et le 6histidine.
II) Transformation et expression du gène Rv3874
dans la levure Pichia pastoris KM71H :
II.1) Linéarisation de
pPICZáA/Rv3874 :
A partir d'une extraction du plasmide recombinant nous
avons linéarisé 10ug d'ADN plasmidique par l'enzyme de
restriction BstX1
Le produit de digestion a été utilisé
après purification pour transformer des levures
Pichia pastoris par éléctroporation.
Le produit de purification est contrôlé sur un
gel d'agarose 0,8%
1
2
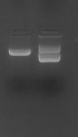
Figure8 : Contrôle du plasmide recombinant
pPICZáA/Rv3874 digéré par BstXI et
purifié.
Piste 1 : 1ul de plasmide recombinant
pPICZáA/Rv3874 digéré par BstXI et
purifié.
Piste 2 : 1ul de plasmide recombinant
pPICZáA/Rv3874 non digéré.
II.2) Transformation par éléctroporation
de la souche KM71H de Pichia pastoris :
Les levures KM71H ont été
sensibilisées par le sorbitol pour subir une
éléctroporation en présence du plasmide recombinant
linéarisé.
L'insertion du gène d'intérêt dans le
génome de Pichia
pastoris se fait par recombinaison homologue.
Après étalement sur le milieu de culture
YPDS+Zéocine les boites sont incubées dans l'étuve
à 30°C pendant trois à quatre jours.
Les clones qui poussent sur zéocine sont
utilisés pour la production de la protéine recombinante.
II.3) Production de la protéine recombinante
CFP10 :
Le gène Rv3874 est cloné en aval d'une
séquence dite á factor qui code pour un peptide signal servant
à la sécrétion de la protéine recombinante dans le
milieu extracellulaire.
Les surnageants de culture de la production, après une
induction avec le méthanol 1% pendant 24, 48 et 72 heures, sont
analysés sur gel de polyacrylamide SDS-PAGE 15% dans des conditions
dénaturantes en présence de â2
mercaptoéthanol. Ce mélange subit également une
dénaturation à la chaleur, 100°C pendant 5 minutes.
L'analyse des deux premières inductions ne montre rien
par contre on observe une bande concernant l'analyse de la troisième
induction (72 heures).
1
15kDa
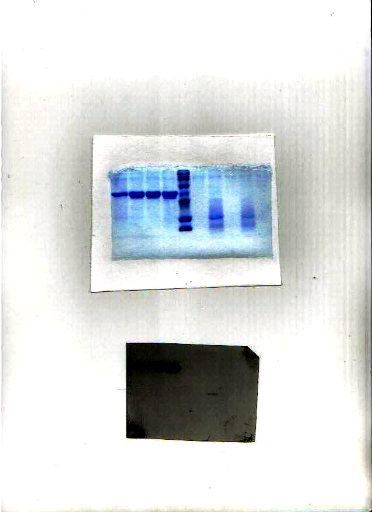
Figure9 : Analyse sur SDS-PAGE 15% du surnageant
de culture après 72 heurs de la
production de CFP10.
1 : Marqueur de poids moléculaire RPN 800.
2 : Protéine recombinante CFP10.
Le poids moléculaire théorique de la
protéine CFP10 est de 10 KDa. En ajoutant le poids moléculaire de
myc épitope et de la queue histidine plus les modifications post
traductionnelles réalisées par Pichia pastoris on
s'attend à une taille plus grande ce qui explique que la bande au niveau
de la figure 9 est à peu prés de 15 KDa.
II.4) Identification de la protéine recombinante
par immunoblot blot :
A fin de confirmer l'identité de la
protéine révélée au niveau du gel (fig 9) nous
avons réalisé la méthode de western blot en utilisant
l'anti myc HRP, un anticorps couplé à la peroxydase qui
reconnaît spécifiquement l'épitope myc
CFP 10
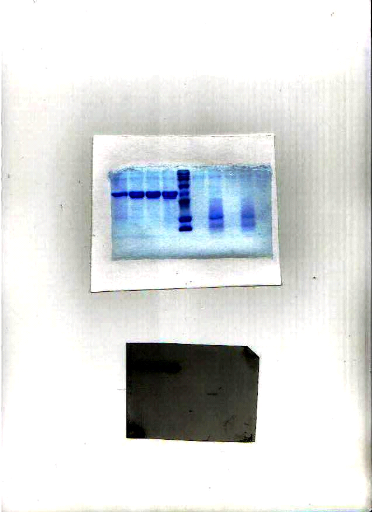
Figure 10 : Révélation par ECL du
wester blot réalisé sur la protéine CFP10 en utilisant
l'anti myc HRP.
La durée d'exposition de film en autoradiographie = 5
minutes.
L'anticorps Anti-myc reconnaît de façon
spécifique l'épitope myc. Cette reconnaissance montre que la
protéine produite contient bien l'epitope myc.
La tuberculose constitue un réel problème
de santé publique à l'échelle mondiale.
Les recherches ne cessent de progresser par l'approche
moléculaire de la tuberculose ce qui peut aider à
l'amélioration du traitement et de la prévention de cette
maladie.
Le séquençage total, du génome de M.
tuberculosis H37Rv en 1998 a constitué un tournant qui ouvre
plusieurs voies de recherche et permis d'identifier de nombreux gènes
dont les fonctions reste mal connues.
Pour découvrir le rôle de ces gènes les
chercheurs doivent passer par les étapes de clonage et d'expression
afin de convertir l'information génétique en une protéine
biologiquement active
à partir de laquelle on peut appliquer plusieurs tests
afin de dégager les caractéristiques de la protéine et
donc savoir l'intérêt du gène en question.
Au cours de ce travail nous avons réussi à
cloner le gène Rv3874, qui code pour la protéine CFP10, dans le
vecteur pPICZáA destiné à la transformation de la levure
Pichia pastoris qui est un excellent système de production des
protéines hétérologues vu la croissance rapide de la
levure qui se maintient en fermenteur et qui pousse sur des milieux simples.
Par ailleurs, il s'agit d'un système eucaryote qui réalise,
normalement, les modifications post- traductionnelles. Pour ce qui est du
clonage de gène Rv3874 les résultats on étés
confirmés par colonie PCR, par carte de restriction et par
séquençage automatique.
Nous avons choisi de produire la protéine recombinante
sous forme sécrétée soluble dans le surnageant de culture
ce qui facilite sa purification puisque, la levure Pichia pastoris
sécrète de très faibles quantités de
protéines autres que la protéine d'intérêt.
La protéine CFP 10 a été produite dans un
premier temps dans E-Coli. Ce systéme de production
présente des limitations puisque la bactérie est incapable de
réaliser les modifications post traductionnelles qui peuvent être
importantes pour l'activité biologique de la protéine.
De plus E-Coli, sécrète très mal
les protéines et si la protéine recombinante est exprimée
dans le cytoplasme il faut casser la membrane plasmique pour y accéder,
ce qui nécessite une purification très poussée.
On peut signaler aussi q'une autre protéine appartenant
au complexe de M. tuberculosis qui est la CFP32 et qui était le
sujet d'un DEA d'un étudiant au laboratoire a été produite
avec succès dans la levure Pichia pastoris avec un rendement
appréciable, de l'ordre de 0,5g/l. (5).
Donc, au cours de ce travail nous avons montré que
Pichia pastoris peut être utilisée comme système de
production des protéines de Mycobactérium
tuberculosis.
Nous avons réussi à produire sous forme
recombinante la protéine CFP10-myc-6his et nous avons identifié
la protéine recombinante par western-blot en utilisant l'anticorps
marqué anti-myc HRP.
Finalement on peut enrichir ce travail en réalisant la
purification de cette protéine et également développer un
test ELISA qui sera utilisé pour la recherche d'anticorps anti-CFP10
dans le sérum des patients tuberculeux.
Références bibliographiques
B
1. Behr, M. A., and Small, P.M. (1999). A historical and
molecular phylogeny of BCG strains. Vaccine, 17, pp: 915-922.
2. Betton J. M . (2005). Repliement et production de
protéines recombinantes. Médecine sciences Vol 21 : 6-7.
3. Brosch, R ; Durieux, P ; Gauselus, J ;
Guérin, N ; Infuso, A ; Jarlier,V ; Marchal, G ;
Zylberman, P. (2004). La place de la vaccination dans le contrôle de la
tuberculose en France ; expertise collective. Vol 2 : 25-42.
4. Benabdesselem, C. (2003) Clonage et expression dans
Pichia pastoris, et caractérisation d'une forme recombinante de
la protéine CFP32 spécifique du complexe mycobactérium
mtuberculosis ; Mastère de biotechnologie et immunologie
appliquées aux maladies trasmissibles : Faculté de
pharmacie-Monastir ; Tunisie.
C
5. CereghinoJ.L., Cregg J.M. (2000) Heterologous protein
expression in the methylotrophic yeast Pichia pastoris. FEMS MICROBIOLOGY
REVIEWS 24: 45-66.
6. Cole, S. T., R. Brosch, J. Parkhill, T. Garnier, C.
Churcher, D. Harris and B. G. Barrel. (1998). Deciphering the biology of
mycobactérium mtuberculosis from the complete genome sequence.
Nature, 393, pp: 537-544.
7. Cregg, J, M., Barringer, K. J., Hessler, A. Y. and Madden,
K, R (1985). Pichia pastoris as a hot system for transformation. Mol.
Cell. Biol; 5:3376-3385.
8. Cregg, J.M., Vedvick, T.S., and Raschke, W.C. (1993) Recent
advances in the expression of foreing genes in Pichia pastoris. Biotechnology
11: 905-910.
D
9. Daly, R. and Hearn, T.W.M (2005) Expression of heterologous
proteins in Pichia pastoris: a useful experimental tool in protein
engineering and production. J. Mol. Recognit. 18: 119-138.
G
10. Grosset J. place des examens microbiologiques et
anatomopathologiques dans la décision diagnostique et
thérapeutique. Med Mal Infect 1995 ; 25 : 327-333.
H
11. Hu, Y., Meng, X.L., Xu, J.P., Lu, W., Wang,J. (2005)
Cloning and expression of earthworm fibrinolytic enzyme PM246 in
Pichia pastoris. Prot. Exp. Purif. 43 pp 18-25.
I
12. Issa, I. (2004). Production par un nouveaux vecteur
navette de protéine de fusion avec la GST dans la levure Pichia
pastoris: cas de l'interféron á. -Tunis :
Faculté des sciences, -DEA en microbiologie.
13. Issar, S. (2003). Mycobactrium tuberculosis pathogenesis
and molecular determinants of virulence. Clinical Microbiology Reviews vol.16,
No3, pp: 464.
L
14. Lipsitch, M. (2001). The rise and fall of antimicrobial
resistance. Microbiology, 148, pp:2967-2973.
M
15. Montgomery, S. A. (2004). Transgenic Animals: Walking
Bioreactors. BioProcess INTERNATIONAL Chap 4: 40-50.
R
16. Rosin, L. J. (2004). Transgenic plants: biofarming of the
future. BioProcess INTERNATIONAL Chap5: 52-61.
S
17. Scott, C. (2004)a. Microbial fermentation: the oldest form
of Biotechnology. BioProcess INTERNATIONAL Chap1: 8-20.
18. Scott, C. (2004)b. Animal cell culture: High maintenance
but Worth the trouble. BioProcess INTERNATIONAL Chap2: 22-32.
19. Sue Marcauley-Patrick., Mariana L. Fazenda., Brian McNeil
& Linda M. Harvey. (2005). Heterologous protein production using the Pichia
pastoris expression system.Wiley Inter Science Review. Yeast. 22: 249-270.
T
20. Tschopp, J. F., Sver low, G., Kosson, R., Croig, W and
Grinna, L. (1997). High level secretion of glycosylated invertase in the
methylotrophic yeast, Pichia pastoris Bio/Technology; 5: 1305-1308.
V
21. Van Crevel., R., Ottenhoff, T.M and Van Der Meer, J.M.
(2002). Innate Immunity to Mycobacterium tuberculosis. Clinical Microbiology
Reviews, 15 N° 2, pp: 294-309.
W
22. Wegner, G.H; Harder, W. 1986. Methylotrophic
yeast-microbial growth on C1 compounds. In: Van Versveld, H.W.and
Duine, J.A.,(Eds), Proceedings of the 5th International symposium.
Martinus Nijhoff, Derdecht, pp: 139-149.
ANNEXES
Tampons et milieux de culture
?Les tampons
- PBS (Phosphate buffered saline): 1.4M NaCL, 27Mm KCL, 101Mm
Na2HPO4 (pH7.3).
- TE: Tris EDTA: 10Mm Tris HCL (pH8), 1Mm EDTA (pH8).
- TBE 5X (Tris Borate EDTA) : 0.045Mm Tris Borate, 0.001M
EDTA.
- TAE 1X (Tris Acetate EDTA) : 0.04M Tris Acetate, 0.001M
EDTA.
- Un tampon d'échantillon : 50%glycérol,
10Mm EDTA et du bleu de bromophénol à 1%.
?Les milieux de culture
- LBLS (Luria Bertani Low Solt Medium) : 1%
Bactotryptone, 0.5% Extrait de levure, 0.5% Nacl, le milieu est
stérilisé par autoclavage 20 min à 121°C a une
pression de 1.5 Bar.
- LBLS Agar : LBLS + 20g/l Agar.
- BMGY (Buffered complex medium containing glycerol): 1%
extrait de levure, 2% peptone, 100Mm phosphate de potassium pH6, 1.34% Yeast
Nitrogen Base (YNB) et 1% glycerol, 4×10-5% biotin.
- BMMY (Buffred Methanol-complex Medium (de même
composition que le BMGY mais on remplace le glycérol par par 1% ou 2% de
méthanol).
- YPD (Yeast extract Peptone Dextrose Medium) : 1%
extrait de levure, 2% peptone, 2% Dextrose.
- YPDS Agar : YPD + 18.2% Sorbitol 1M, 0.2% Agar
| 


