|
Diplôme d'Etudes Approfondies
« Biologie des Cellules
Sanguines »
TLS, une protéine du spliceosome, est impliquée
dans le mécanisme d'action de l'acide rétinoïque
à travers les
régulations post-transcriptionnelle
et transcriptionnelle
Eric Le Corvec
Université Paris 7 Denis Diderot
Juillet 2002
Laboratoire de Biologie Cellulaire
Hématopoïétique
EMI 00-03, Univ. Paris 7 EA 316, Saint-Louis
UF474
Institut Universitaire d'Hématologie,
Hôpital Saint-Louis
75475 Paris Cedex 10
Directeur du laboratoire et maître de
stage
Christine Chomienne
Laurent Delva
Je remercie chaleureusement Christine Chomienne de
m'avoir permis de réaliser ce DEA.
Je remercie tout spécialement Laurent Delva
qui m'a fourni aide et conseils, et sans qui ce travail n'aurait pu être
possible.
Je remercie Gilles Despouy pour son soutien moral et
scientifique.
Je remercie Nicole Balitrand pour ses conseils
avisés.
Je remercie Sylvie Deshaie pour tout ce qu'elle m'a
apporté pendant l'année.
Merci amical à Alexandra Mazharian pour sa
bonne humeur.
Enfin, je voudrais remercier toute l'équipe du
LBCH pour leur accueil et leur sympathie.
RESUME
Les régulations transcriptionnelle et
post-transcriptionnelle sont impliquées fortement dans la
diversification de l'expression protéique.
Le fait que TLS soit surexprimée dans 60% des LAM et
qu'il interagisse avec RXR, l'un des principaux partenaires du RANC, nous
permet d'émettre l'hypothèse que TLS serait liée à
la voie de signalisation de l'AR dans les cellules
hématopoïétiques. TLS, une protéine
ubiquitairement exprimée, intervient dans de nombreux mécanismes
moléculaires. De nombreuses études montrent qu'elle est
liée à la machinerie transciptionnelle aussi bien qu'à
celle de l'épissage.
Nous avons initié l'étude de l'action de TLS et
de l'AR sur l'activité transcriptionnelle dans les cellules
myéloblastiques HL-60. L'appartenance de TLS au complexe
transcriptionnel des récepteurs à l'acide rétinoïque
est évaluée par la technique de retard sur gel avec les deux
récepteurs nucléaires aux rétinoïdes (RAR et RXR). De
plus, le rôle de TLS et de l'AR sur l'épissage a été
étudié par une technique d'analyse in vivo, sur
l'épissage alternatif en 5' du minigène E1A dans les cellules
hématopoïétiques K562 en absence ou en présence
d'acide rétinoïque.
Nos travaux ont permis de caractériser un rôle de
co-activateur de la transcription par les récepteurs aux
rétinoïdes en présence d'acide rétinoïque dans
les cellules HL-60 et Cos-6, son appartenance au RANC et un rôle de
facteur d'épissage favorisant la sélection du site 5' distal
d'épissage du minigène E1A dans les cellules
myéloïdes K562. Nous avons montré que TLS agit aussi avec la
voie de signalisation des rétinoïdes qui accentuent les effets
observés de TLS sur l'épissage.
L'acide rétinoïque est ainsi impliqué
à la fois dans le transcriptome et le spliceosome à travers TLS.
Ainsi, c'est la première fois que la voie de signalisation des hormones
est impliqué dans l'épissage et la transcription à travers
un facteur d'épissage.
LISTE DES ABREVIATIONS
ABL : ABeLson
ADN : Acide DésoxyriboNucléique
ADNc : ADN complémentaire
AF : Activation function
AR : Acide Rétinoïque
ARN : Acide RiboNucléique
ARN polII : ARN ploymérase II
ARNpré-m : ARN pré-messager
ATRA : All-Trans Retinoic acid
AR 9-cis : Acide
Rétinoïque 9-cis
BCR : Breakpoint Cluster Region
CHOP : C/EBP HOmologous Protein
DBD : DNA Binding Domain
DMSO : DiMethyl SulfOxide
DR : Direct Repeat
E1A : Enhancer 1 Adenovirus
ER : Estrogen Receptor
ERG : ETS-Related Gene
EWS : EWing Sarcomas
GR : Glucocorticoid Receptor
hnRNP : heterogeneous nuclear
RiboNucleoProtein
LAM : Leucémie Aiguë
Myéloïde
LBD : Ligand Binding Domain
NPM : NucleoPhosMin
NuMA : Nuclar Matrix Apparatus
PCR : Polymerisation in Chain Reaction
PLZF : Promyelocytic Leukemia Zinc Finger
PML : ProMyelocytic Leukemia
RANC : Retinoic Acid-dependent Nuclear
Complex
RAR : Retinoic Acid Receptor
RARE : Retinoic Acid Response Element
RGG : Arginine-Glycine-Glycine
RNP-CS : RNP-Consensus Sequence
RRM : RNA Recognition Motif
RT : Reverse Transcriptase
RT-PCR : RT suivie d'une PCR
RXR : Retinoid X Receptor
snRNP : small nuclear RiboNucleoProtein
TAF : TBP Associated Factor
TBP : TATA Binding Protein
TLS : Translocated in LipoSarcomas
TR : Thyroid Receptor
VDR : Vitamin D Receptor
SOMMAIRE
RESUME
1
SOMMAIRE
3
I - INTRODUCTION
4
BUT DU PROJET
9
II - MATERIELS ET METHODES
10
A) MATERIELS :
10
a) Lignées cellulaires :
10
c) Agents inducteurs :
10
d) Plasmides :
10
e) Oligonucléotides :
11
B) METHODES
11
a) Cultures cellulaires
11
b) Préparation de l'ADN plasmidique
11
c) Transfections cellulaires
11
d) Epissage in vivo
12
e) Test de transactivation
13
f) Technique de Western Blot
13
g) Technique du retard sur gel
14
III- RESULTATS
15
1. TLS est un co-activateur du RANC
15
2. TLS fait parti du RANC
17
3. Analyse in vivo de
l'activité de TLS et de l'acide rétinoïque sur
l'épissage alternatif en 5' de E1A
19
IV - DISCUSSION
23
V - REFERENCES
28
I - INTRODUCTION
Un des principaux thèmes d'études de notre
laboratoire concerne l'étude de l'hématopoïèse et des
leucémies, dans un but de diagnostic, de pronostic et de ciblage
thérapeutique. Ainsi, la compréhension des mécanismes
moléculaires qui contrôlent la différenciation des cellules
myéloïdes est un élément essentiel.
L'hématopoïèse est un processus actif,
hautement régulé, de prolifération et de
différenciation des précurseurs et des progéniteurs
hématopoïétiques. Au sein du compartiment actif des cellules
souches, un programme de différenciation cellulaire se met en place.
Dans un premier temps, la cellule souche perd sa capacité
d'auto-renouvellement, puis progressivement sa capacité de
prolifération. La cellule engagée dans ce processus va s'orienter
vers l'une des voies de différenciation
hématopoïétique (granulo-monocytaire,
mégacaryocytaire, érythroïde ou lymphocytaire).
Les signaux, intra- ou extra-cellulaires, qui mettent en
cycle la cellule souche et qui gouvernent secondairement les premières
étapes de son déterminisme sont peu connus. Deux modèles
sont actuellement proposés pour rendre compte de ce
phénomène. Le premier est dit « modèle
stochastique » dans lequel ces étapes se font en absence de
tout signal extérieur, de façon intrinsèque au programme
génétique. Le deuxième modèle est dit
« modèle déterministe », où la cellule
reçoit de l'extérieur, de façon extrinsèque, un
signal qui va déclencher le programme génétique de
différenciation. Quel que soit le modèle considéré,
ces mécanismes conduisent de façon ultime à l'activation
de facteurs de transcription qui activent les gènes responsables de la
différenciation cellulaire (Felsenfeld et coll., 1996). C'est
vraisemblablement la combinatoire des facteurs de transcription exprimés
par la cellule qui va aboutir à l'expression
préférentielle des gènes spécifiques de
lignée. En d'autres termes, c'est l'expression génique qui influe
sur la différenciation.
Il a été démontré que les
rétinoïdes stimulaient de façon préférentielle
la granulopoïèse (Douer et coll., 1982 ; Gratas et coll.,
1993). Les rétinoïdes agissent en se fixant sur des
récepteurs spécifiques qui sont les récépteurs
nucléaires aux rétinoïdes. Les récepteurs
nucléaires (Figure 1) appartiennent à une famille de facteurs de
transcription qui régulent l'expression des gènes en fonction de
la fixation d'un ligand. Les membres de la superfamille des récepteurs
nucléaires incluent les récepteurs aux hormones
stéroïdiennes, tels que les récepteurs aux
glucocorticoïdes (GR) et aux oestrogènes (ER), des
récepteurs pour des hormones non stéroïdiennes comme le
récepteur aux hormones thyroïdiennes (TR), le récepteur
à la Vitamine D3 (VDR), les récepteurs aux
rétinoïdes, et également des récepteurs pour divers
métabolites lipidique comme les acides gras et les prostaglandines
(Green et Chambon, 1986).
La superfamille des récepteurs nucléaires
comprend également une grande famille de récepteurs dits
orphelins lorsque le ligand n'a pas encore été identifié
ou n'existe pas.
A
B
C
E/F
AF-1
AF-2
Activation
indépendante du ligand
DBD
Dimérisation
LBD
Activation dépendante du ligand
Dimérisation
D
Figure 1. Structure générale des
récepteurs nucléaires
Deux classes de récepteurs aux rétinoïdes
ont été identifiées : les récepteurs à
l'acide rétinoïque (RAR) et les récepteurs au
rétinoïde X (RXR). Trois types de RARs (, et ) codés par
des gènes distincts localisés sur des chromosomes
différents sont présents chez l'Homme, chez la Souris, puis dans
d'autres espèces (Giguère et coll., 1987 ; Petkovich et
coll., 1987 ; Krust et coll., 1989 ; Zelent et coll., 1989). Les RARs
sont capables de fixer deux métabolites actifs de la Vitamine A, l'acide
rétinoïque tout-trans (ATRA) et l'acide
rétinoïque 9-cis (Allegretto et coll., 1993 ; Allenby
et coll., 1993). La comparaison des séquences protéiques des
différents RARs et des autres membres de la superfamille des
récepteurs nucléaires a permis de les subdiviser en
différents domaines possédant des fonctions distinctes. Ces
domaines sont plus ou moins conservés d'un récepteur à
l'autre et d'une espèce à l'autre (Kastner et coll., 1994). La
deuxième classe de récepteurs aux rétinoïdes, les
RXRs, lie spécifiquement et uniquement l'acide rétinoïque
9-cis. Les gènes codant pour les trois types de RXRs (, et )
ont été clonés chez la Souris et chez l'Homme. Dans le
domaine d'activation des récepteurs nucléaires, on trouve deux
séquences portant des fonctions activatrices : AF-1 (domaine A/B),
indépendant du ligand et AF-2 (domaine E/F), dépendant du ligand.
Ces deux domaines (mais principalement AF-2) peuvent servir au recrutement des
co-activateur et des co-répresseurs (Kastner et coll, 1994).
La structure des RARs est divisée en six régions
fonctionnelles de A à F alors que les RXRs ne possédent pas de
région F. Les différents types de RXRs présentent une
forte identité de séquence dans leurs régions C et E, mais
diffèrent fortement au niveau de leurs régions A/B et D. Cette
différence est ainsi spécifique d'un type donné de RXR
(Chambon, 1996).
Les récepteurs nucléaires exercent leur action
par différents mécanismes. Ils peuvent activer ou réprimer
des gènes cibles en se fixant directement sur des séquences
spécifiques d'ADN appelées éléments de
réponse sous la forme d'homo-dimère (exemple des
récepteurs stéroïdiens et des RXRs) ou
d'hétéro-dimères (exemple des RARs, du VDR et des TRs)
avec les RXRs comme partenaires. Ou en fixant d'autres classes de facteurs de
transcription (Chambon, 1996). Certains récepteurs nucléaires
tels que le TR et le RAR, peuvent réprimer des gènes cibles en
absence ou en présence du ligand. Ces effets sont liés à
l'interaction des récepteurs nucléaires avec des classes de
protéines intermédiaires dont la fonction est soit d'activer (les
co-activateurs), soit de réprimer (les co-répresseurs) la
transcription. Le complexe transcriptionnel des récepteurs
nucléaires à l'AR est dénommé RANC. analyse des
régions promotrices des gènes cibles de l'acide
rétinoïque a permis de décrire des éléments de
réponse, les RARE (Retinoic Acid Response Element). Ces
éléments de réponse sont constitués de deux
séquences hexa-nucléotidiques conservées PuG(C/T)TCA
disposées en répétitions directes (DR), en palindromes ou
inversées et séparées par un nombre de nucléotides
variant de 1 à 5 (Giguère, 1994). Ainsi, un espacement de 1
nucléotides (DR1) entre les deux motifs consensus AGGTCA définit
un élément de réponse de l'homo-dimère RXR-RXR, un
espacement de 5 nucléotides (DR5) entre les deux motifs aboutit à
un RARE.
L'altération de RAR provoque des perturbations au
niveau des mécanismes de remodelage de la chromatine. Ces
altérations sont dues aux gènes de fusion impliquant le
gène RAR lors de translocations chromosomiques responsables de la
leucémie aiguë promyélocytaire (LAM3 selon la classification
FAB). Il s'agit de la première pathologie maligne humaine
répondant à une thérapeutique différenciatrice
(pour revue voir Chomienne et coll., 1996 ; Fenaux et coll., 1994). Elle
présente ainsi une caractéristique unique par rapport aux autres
sous-types de leucémies aiguës (LAM, anomalie de la
myélopoïèse).
De façon intéressante, il a été
révélée que dans 60% des LAM, TLS, une protéine
ubiquitaire, était très fortement exprimée (Aman et coll.,
1996). D'autre part, des interactions directes entre TLS et le récepteur
aux rétinoïdes RXR ont été démontrées
(Power et coll., 1998). Ces deux observations nous incitées à
étudier les liens éventuels entre TLS et les voies de
signalisation aux rétinoïdes dans un contexte
hématopoïétique normal et altéré.
Le gène TLS fut, en effet, d'abord identifié
dans une tumeur maligne à partir de la translocation t(12;16) comme
gène codant pour la partie N-terminale de TLS/CHOP, une
oncoprotéine de fusion qui est exprimée invariablement
associée au liposarcome myxoïde humain (Crozat et coll.,
1993 ; Rabbits et coll., 1993). D'autres translocations chromosomiques
(à l'origine de sarcomes humains et de leucémies), fusionnent
soit TLS soit un gène similaire, EWS à un grand nombre de
facteurs de transcription (Zinszner et coll., 1994). Le point commun de ces
diverses oncoprotéines de fusion est la présence du domaine
N-terminal de TLS ou de EWS. Ce domaine joue un rôle essentiel dans la
transformation confirmé par des expériences de transformation
utilisant des lignées de souris (Ichikawa et coll., 1999) ou des
cellules normales hématopoïétiques (Pereira et coll., 1998).
De nombreuses expériences ont permis de mettre en
évidence certains rôles de TLS, notamment la réalisation de
deux lignées de souris nullizigotes pour TLS. Les souris homozygotes
portant une mutation induite de TLS sont stériles avec une importante
augmentation d'axes chromosomiques non appariés ou
mésappariés dans les spermatocytes
préméïotiques. Ces résultats montrent un rôle
de TLS dans l'appariement de l'ADN homologue et dans la recombinaison.
L'analyse de ces Souris indique que TLS est essentielle pour la survie du
nouveau-né, influence le développement lymphocytaire, a un
rôle dans la réponse proliférative des lymphocytes B
à des stimuli mitogènes, et est nécessaire pour le
maintien de la stabilité génomique (Hicks et coll.,
2000). La capacité de TLS à se lier à
l'ADN est entre autre induite par sa phosphorylation par la PKCII qui est
permise par l'activité tyrosine kinase de BCR/ABL. Ces résultats
suggèrent que TLS joue un rôle de régulateur dans la
leucémogénèse causée par BCR/ABL, favorisant
l'indépendance envers les facteurs de croissance et empêchant la
différenciation, en modulant l'expression de récepteurs de
cytokines (Perroti et coll., 1998) confirmant que sa surexpression dans les LAM
puissent être une des causes des désordres observés.
TLS interagit aussi avec l'ARN polII par son domaine
N-terminal et est impliquée dans la formation du complexe TFIID. Il
pourrait ainsi agir comme un régulateur de la transcription basale
participant à la reconnaissance de promoteur transcriptionnel et
à l'initiation de la transcription (Yang et coll., 2000). De plus, TLS
interagit avec RXR et TR par leur domaine N-terminal (Powers et coll., 1998).
Des facteurs d'épissage interagissent aussi avec TLS, l'impliquant dans
ce mécanisme. TLS interagit avec Spi-1/PU.1 une protéine ETS
capable de réguler la transcription et la maturation ARN dans les
cellules myéloïdes (Hallier et coll., 1998). Trois protéines
SR, SC35, TASR-1 et TASR-2 interagissent avec TLS par son domaine C-terminal.
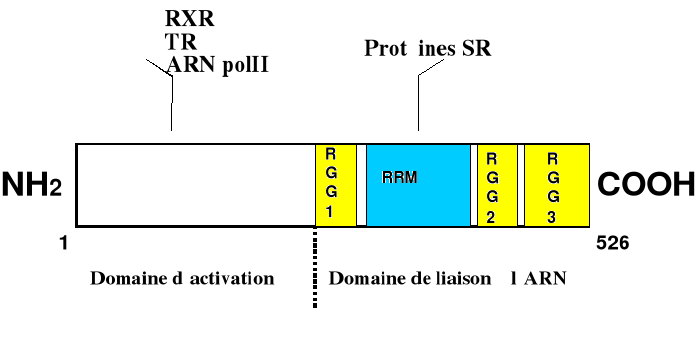
Figure 2 . TLS est une hnRNP et fait partie de la
famille de EWS et hTAFII68.
L'étude de TLS a permis de préciser sa
structure (Figure 2). TLS appartient à une famille de protéines
incluant EWS (Delattre et coll., 1992) et TAFII68 (Bertolotti et coll., 1996).
Le domaine N-terminal des protéines de cette famille est riche en
glutamine, sérine et tyrosine, qui sont les acides aminés
trouvés dans les domaines d'activation de la transcription. Dans des
chimères oncogèniques, l'addition du domaine N-terminal de TLS
aux régulateurs transcriptionnels CHOP, FLI-1 ou ERG-1
génère des protéines dont l'activité
transcriptionnelle diffère de celle de ces constituants respectifs. De
plus, le domaine N-terminal de TLS présent dans ces chimères peut
avoir un effet dominant négatif sur la fonction de TLS (provenant de la
lignée germinale) comme suggère la récente identification
de déterminants oncogéniques dans le domaine N-terminal de TLS.
Le domaine C-terminal contient plusieurs motifs : une séquence
consensus de ribonucléoprotéine (RRM : ou RNP-CS), des
répétitions arginine-glycine-glycine (RGG 2/3), étant la
signature des protéines de liaison à l'ARN (Burd et coll., 1994)
et un doigt de zinc C2C2. TLS lie l'ARN in vivo
ou in vitro (Zinszner et coll., 1997b). In vitro, les ARNs
sélectionnés par TLS partagent un motif GGUG. TLS reconnaît
un ARN contenant ce motif dans un extrait cellulaire. Chacun des domaines de
liaison à l'ARN (les trois boîtes RGG et le motif de
reconnaissance de l'ARN) contribuent à la spécificité de
l'interaction TLS-ARN (Lerga et coll., 2001). TLS est aussi appelée FUS
ou hPOMp75. Cette dernière dénomination a été
donnée lorsqu'il fut montré que TLS se lieny à l'ADN
simple et double brins. TLS favorise l'hybridation de brins
complémentaire d'ADN et l'incorporation d'un oligonucléotide
d'ADN simple brin dans une super hélice d'ADN pour former une
« D-loop », suggérent que TLS soit impliquée
dans la recombinaison homologue (Baetchtold et coll., 1999).
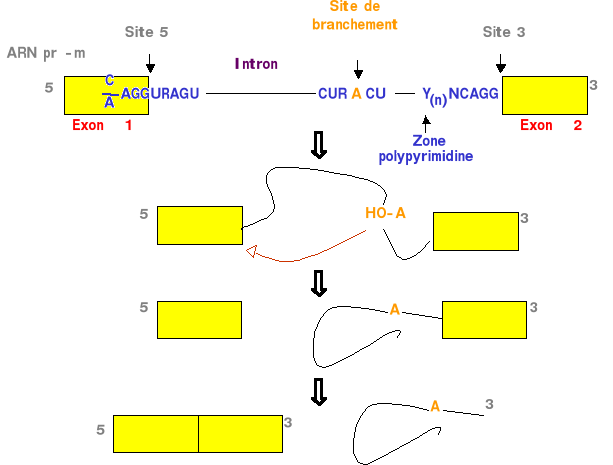
Figure 3. Réaction de
transestérification dans le mécanisme de l'épissage de
l'ARN pré-m.
Chez les Eucaryotes supérieurs, l'expression
génique implique la transcription, le « 5'
capping », l'épissage de l'ARN pré-m, le « 3'
processing », l'exportation de l'ARNm et éventuellement la
traduction dans le cytoplasme (Misteli et coll, 2000). L'épissage
(dérivation d'un terme de marine qui signifie mettre bout à bout
les extrêmités de deux cordages) consiste en une
réaction de transestérification éliminant certaines
séquences d'un ARN pré-m (les introns) et en laissant d'autres
séquences (les exons). Des séquences consensus ont
été identifiées aux jonctions exons/introns, elles sont
définies comme les sites d'épissage donneur en 5' de l'intron
(dinucléotide GU), et accepteur, à l'extrêmité 3' de
l'intron (dinucléotide AG) (Breatnach et Chambon, 1981). Les fonctions
des snRNA U1, U2, U4, U5, et U6 dans l'épissage sont liées
à leur inclusion au sein de complexes composés de snRNA et de
nombreuses autres protéines, appelées les snRNP. L'agencement des
cinq snRNP avec une cinquantaine de protéines sur la matrice de l'ARN
pré-m constitue un complexe ribonucléoprotéique dynamique,
le « spliceosome ». Les protéines SR participent
à ce complexe, ce sont des facteurs d'épissage essentiels
caractérisés par un motif riche en dipeptide
sérine-arginine ayant un rôle dans la définition des exons
et la sélection des sites alternatifs d'épissage (Valcarcel et
coll., 1996). Enfin, la capacité à sélectionner
différentes combinaisons d'exons lors de l'épissage, afin de
générer des protéines de fonctions distinctes. Ce
phénomène d'épissage alternatif est indispensable au
développement de nombreux organismes. Environ 60% des gènes
seraient exprimés selon un tel mécanisme (Croft et coll.,
2000).
TLS fait partie de la famille hnRNP. Et elle a
été identifiée comme la hnRNP p2 dans un complexe
protéique assemblé sur l'ARN pré-m des adénovirus.
TLS est engagée dans un complexe avec les hnRNPs A1 et C1/C2 et
associée avec plusieurs snRNP du « spliceosome ».
TLS est complexée avec des transcrits de l'ARN polII dans des extraits
de cellules de HeLa irradiées (Perroti et coll., 1998). En outre, un
inhibiteur de l'ARN polII induit la relocalisation de TLS du noyau au
cytoplasme, dépéndant de la présence de son domaine
C-terminal. Toutes ces proptiétés sont caractéristiques
des hnRNPs. TLS fut trouvée associée avec le dinucléotide
AG durant l'étape de reconnaisance du site 3' d'épissage et
participe à la sélection du site 5' d'épissage alternatif
dans l'ARN pré-m du minigène E1A. TLS peut recruter par son
domaine C-terminal, deux régulateurs d'épissage de la famille des
protéines SR. Ainsi, TLS est un de facteur d'épissage.Lorsqu'elle
est surexprimée dans des cellules érythroïdes, TLS induit de
préférence l'utilisation du site 5' distal d'épissage,
durant la maturation de l'ARN pré-m de E1A (Hallier et coll., 1998).
Cette préférence est contrebalancée par Spi-1,
suggérant que TLS peut faire partie d'un réseau de
protéines impliquées dans la régulation de la maturation
ARN.
Plusieurs pathologies liées à des
altérations de l'épissage ont été
caractérisées. Certaines affections comme les amyotrophies
spinales héréditaires ou les maladies
neurodégénératives liées à la
protéine tau, représentent de véritables problèmes
de santé publique et donnent lieu à des coopérations
fructueuses entre laboratoires hospitaliers et universitaires (Philips et
Cooper, 2000). Pour exemple de pathologies impliquant l'épissage
alternatif, on retrouve Spi-1/PU.1 dans l'érythroleucémie de
Friend et CD44 dans certaines transformations tumorale (Stickeler et coll.,
1999).
Le rôle de TLS dans la régulation
transcriptionnelle n'est pas encore clairement élucidé. TLS
augmente la transactivation dirigée par NFB induite par des stimuli
physiologiques tels que le TNF, IL-1 et la surexpression d'une kinase induisant
NFB. TLS augmente l'activité du promoteur NFB-dépendant d'un
gène d'adhésion intercellulaire et du gène de l'IFN. Ces
résultats suggèrent que TLS agit comme un co-activateur de NFB
(Uranishi et coll., 2001).
BUT DU PROJET
Le fait que TLS soit surexprimée dans 60% des LAM et
qu'il interagisse avec RXR, l'un des principaux partenaires du RANC, nous
permet d'émettre l'hypothèse que TLS serait liée à
la voie de signalisation de l'AR dans les cellules
hématopoïétiques. Nous nous proposons d'étudier deux
fonctions de TLS qui pourraient être mises en jeu dans les LAM, en tant
que co-activateur de la transcription et son rôle de facteur
d'épissage. L'action de TLS (en présence d'AR) comme
co-activateur de la transcription est testée dans deux modèles
cellulaires, HL-60 et Cos-6, par le dosage de l'expression d'un gène
cible sous la dépendance d'un promoteur fixant le RANC. L'appartenance
de TLS au RANC est observée par la mise en présence du RANC (RAR,
RXR, DR5 avec ou sans AR) et de TLS. L'effet de TLS et de l'AR sur
l'épissage sont analysés qualitativement par l'obtention de
profil d'épissage alternatif du minigène E1A et l'étude
quantitative des isoformes produites.
II - MATERIELS ET METHODES
A) MATERIELS :
a)
Lignées cellulaires :
Trois lignées cellulaires ont été
utilisées :
- La lignée HL-60 fut obtenue à partir de
cellules d'un patient atteint d'une leucémie aiguë
myéloblastique de type 2 selon la classification FAB
(French-American-British). Les cellules HL-60 ont la caractéristique
d'être bi-potentielles, c'est à dire de se différencier
soit vers la voie granulocytaire, soit vers la voie monocytaire, selon que
l'ATRA ou la VD3 sont utilisées (Breitman et coll., 1980).
- La lignée K562 fut obtenue à partir de
cellules d'un patient atteint d'une leucémie chronique
myéloïde. La population cellulaire a été
identifiée comme indifférenciée et de série
granulocytique.
- La lignée Cos-6 provient d'un rein de Singe.
b) Anticorps :
Plusieurs anticorps ont été
utilisés : l'anticorps monoclonal de Souris anti-myc (anti myc-TLS,
IgG1, Kappa, Roche Molecular Biochemicals), l'anticorps polyclonal de Lapin
Anti-RXR (RXR 444, don de Cécile Rochette-Egly et Pierre Chambon),
l'anticorps polyclonal de Lapin Anti-RAR (RAR 115, don de Cécile
Rochette-Egly et Pierre Chambon) et l'anticorps polyclonal de Lapin Anti-TLS
(don de Françoise Moreau-Gachelin).
c)
Agents inducteurs :
L'ATRA (Hoffmann La Roche) est dissout dans le DMSO à
une concentration de 10-2 M, conservé à -80°C
à l'abri de la lumière, dilué dans du RPMI pour obtenir
une concentration de 10-4 M, stocké à -20°C et
utilisé dans les milieux de culture à la concentration finale de
10-6 M. L'AR 9-cis est réalisé de la
même façon et utilisé à la concentration finale de
10-6 M.
d)
Plasmides :
- Le plasmide Tkgal est un plasmide dans lequel est
cloné l'ADNc de la -galactosidase sous le contrôle d'un fragment
délété du promoteur TK du virus de l'Herpès (don de
H. de Thé).
- Le plasmide RARE est un plasmide pXp2, dans lequel l'ADNc de
la luciférase est sous le contrôle du promoteur du gène
RAR2 (inséré en amont du promoteur, don de H. de Thé).
- Le vecteur d'expression pCS3-MT (Myc-Tag) contient le
promoteur/enhancer du cytomégalovirus simien IE94, le promoteur SP6 et
six copies de l'épitope Myc 9E10 (don de Françoise
Moreau-Gachelin).
- pCS3-MT-TLS code pour une protéine contenant
l'épitope Myc et les acides aminés de TLS de 1-526 (don de
Françoise Moreau-Gachelin).
- pCS3-MT-E1A fut généré par insertion du
fragment EcoRI-XbaI de E1A du plasmide Sp4 dans le site EcoRI-XbaI du vecteur
pCS3-MT (don de Françoise Moreau-Gachelin).
- Le pSG5-hRAR et le pSG5-mRXR sont des plasmides
d'expression contenant l'ADNc des récepteurs nucléaires RAR et
RXR (don de Cécile Rochette-Egly et Pierre Chambon).
- Le CMV-luc est un plasmide puC18 de 6 kb, sans "polylinker",
dans lequel est cloné l'ADNc du gène luciférase sous le
contrôle du promoteur du cytomégalovirus, qui est un promoteur
fort (don de H. de Thé).
e)
Oligonucléotides :
Dans la technique de retardement sur gel :
DR5: 5'-GATCAGGGTTCACCGAAAGTTCACTCGCATATATTAG-3'
(Eurogentec).
Dans la technique de RT : amorce RT :
5'-GGAGAGCTTGGGCGACC-3 ' (Genset).
Dans la technique de PCR : amorce PCR-E1A :
5'-ATTATCTGCCACGGAGGTGT-3': (Genset).
B) METHODES
a)
Cultures cellulaires
Les cellules de lignées HL-60 et K562 sont
ensemencées à la concentration de 3x105 cellules par
ml et les cellules Cos-6 à la concentration de 4x105 cellules
par ml.
Les cellules HL-60 et K562 sont cultivées en RPMI 1640,
additionné de sérum de veau foetal (SVF,
décomplémenté par la chaleur (20 min. à 56°C))
respectivement à 15 et 10 %, de glutamine 2 mM et de
Pénicilline/Streptomycine à 100 ug/ml. Les cellules sont
maintenues en phase exponentielle de croissance par une dilution, deux fois par
semaines.
Les cellules adhérentes Cos-6 sont
cultivées en DMEM, additionné à 6 % de SVF, de glutamine 2
mM et de Pénicilline/Streptomycine à 100 ug/ml. Les cellules sont
maintenues en phase exponentielle de croissance par une dilution, deux fois par
semaines, après un lavage avec le milieu sans sérum et un
décollement par deux fois une addition de 1 ml de trypsine pendant 5
min. à 37°C.
b)
Préparation de l'ADN plasmidique
L'ADN plasmidique est obtenu par culture de bactéries
préalablement transformées par les plasmides
d'intérêt, dans du milieu LB plus ampicilline 1X. L'ADN
plasmidique est préparé selon le protocole de purification
plasmidique (Plasmid Purification, Qiagen). Il subit ensuite une
précipitation au chlorure de césium, cette étape augmente
la pureté de l'ADN mais n'est pas obligatoire. La qualité de cet
ADN est analysée par spectrophotométrie pour évaluer la
quantité et par migration sur gel d'agarose 1 % pour évaluer la
qualité.
c)
Transfections cellulaires
1) Transfection transitoire de cellules Cos-6 par
phosphate de calcium
Vingt quatre heures avant la transfection, les cellules sont
mises dans des puits de culture de 6 cm de diamètre à une
concentration de 600000 dans 5 ml de milieu. Le jour suivant, le milieu est
remplacé par 4 ml de milieu frais. Une solution de 1 ml contenant 5
à 30 ug d'ADN plasmidique dans 450 ul de H2O, 50 ul de
CaCl2 (2,5 M) et 500 ul de HBS 2X (NaCl 280 mM, KCl 10 mM,
Na2HPO4 12 mM, Glucose 12 mM, Hépès 50 mM)
préalablement incubée 30 min. à température
ambiante est ajoutée sur les cellules. Elles sont remises dans
l'étuve 12 heures, puis lavées au PBS 1X. Du milieu frais est
ajouté et ainsi que le ligand, éventuellement. Les cellules sont
de nouveau incubées de 24 heures pour un dosage de la luciférase
à 2 jours pour une extraction protéique à 37°C. Avant
l'extraction des protéines, les cellules sont lavées au PBS
1X.
2) Transfection transitoire de cellules HL-60 par
électroporation
Les cellules sont passées la veille à la
concentration habituelle correspondant au type cellulaire et les cuves à
éléctroporation sont préalablement refroidies à
4°C. Chaque cuve est utilisée pour 30x106 cellules.
Trois heures avant la transfection, les cellules sont mises à
1x106 par ml puis incubées à 37°C. Puis elles
sont centrifugées, lavées et regroupées à 4°C
dans un volume d'OPTIMEM-1 équivalent à 10 ml pour
100x106 cellules. Elles sont ensuite reprises dans un volume
contenant 100 ul d'OPTIMEM-1 par cuve. Après en avoir placé 100
ul dans chaque cuve, le mélange est incubé pendant 10 min.
à 4°C. Les cellules sont ajoutées dans les cuves, à
une solution plasmidique contenant 100 ul d'OPTIMEM-1 par cuve.
L'électroporation est effectuée dans un électroporateur
dont les paramètres sont : C= 960 uF et V=250 V, elles sont ensuite
mises à incuber à température ambiante pendant 30min. Les
cellules sont récupérées avec 1 ml de milieu complet
préalablement chauffé à 37°C. Les cellules sont
incubées à 37°C pendant trois heures, puis remises en
culture.
3) Transfection transitoire de cellules K562 par la
lipofectamine
L'ADN plasmidique est préparé dans un volume de
100 ul par puits avec 5 % de lipofectamine plus et complété par
95 ul d'OPTIMEM-1 qui est laissé incubé pendant 15 min. Une
solution de 12,5 ul de lipofectamine et de 87,5 ul d'OPTIMEM-1 par puits est
préparée. Les deux solutions sont mélangées dans
des tubes stériles en polypropylène, en ajoutant la solution
plasmidique goutte à goutte. Ce mélange est incubé une
heure à température ambiante. Les cellules sont
conditionnées au nombre de 2x106 par puits dans 0,8 ml
d'OPTIMEM-1 (la concentration initiale des cellules dans leur milieu
était de 500000 par ml) et incubées une heure à 37°C.
Les 200 ul préparés sont déposés goutte à
goutte dans chaque puits. Puis les cellules sont incubées deux heures
à 37°C et enfin 3,5 ml de milieu sont ajoutés et le ligand
éventuellement.
d)
Epissage in vivo
Les cellules sont récoltées 24 heures
après la transfection. La co-transfection avec le plasmide
pCMV-luciférase permet la standardisation de la RT en fonction des
différences observées dans l'efficacité de transfection.
Une fraction aliquotée de ces cellules est utilisée pour mesurer
l'activité luciférase et le reste est utilisé pour
effectuer une extraction totale des ARNs. Les ARNs totaux sont
préparés à partir des cellules en utilisant le protocole
de purification d'ARN et d'après les instructions du fournisseur
(RNeasy, Qiagen) et traité deux fois par la Dnase I (Qiagen). La
qualité des ARNs obtenus est observée par migration sur un gel
d'agarose dénaturant. Les isoformes de l'ARN E1A sont analysées
par RT-PCR comme décrit précédemment (Hallier et coll.,
1998). Un microgramme d'ARN est rétro-transcrit par la SuperscriptII
transcriptase reverse du virus de la leucémie murine de Moloney
(Gibco-BRL) en présence de 50 uM de dNTP et 2 picomoles du primer 3'RT
E1A dans une solution de 8 ul. Environ un dixième de l'ADNc est
utilisé pour la réaction d'amplification (PCR)
réalisée avec la Taq polymérase ADN (Perkin
Elmer) en présence de l'amorce 5'E1A marquée à son
extrémité 5' par la T4 polynucléotide kinase (Boehringer)
et de l'ATP P32 (Amersham) comme précédemment
décrit (Hallier et coll., 1998). Le minimum de cycles PCR est
utilisé (18 à 22 cycles) afin de détecter le signal dans
une fenêtre de détection adéquate. Les réactions de
contrôle de la RT-PCR contiennent une matrice ARN n'ayant pas subi de
transcription inverse. Les produits de la RT-PCR de E1A sont mis à
migrer sur un gel de polyacrylamide/urée 6 % dénaturant,
autoradiographiés et quantifiés avec un système Biorad..
L'expression des protéines codées par les plasmides est
vérifiée par la technique du Western Blot effectuée sur
des échantillons de cellules transfectées d'où viennent
les ARNs totaux.
e)
Test de transactivation
La veille de la transfection, les cellules Cos-6 sont mises en
puits, dans 5ml de DMEM-6% SVF décomplémenté à
raison de 0,6x106 par puits. Le jour suivant, le milieu est
changé pour 4 ml de milieu frais. Dans chaque puits, sont ajoutés
une solution de 1 ml préalablement incubée à
température ambiante comprenant une solution plasmidique (TKGal,
RARE-Luciférase,...), 450 ug d'eau bi-distillée en plus de 50 ul
de CaCl2 (2,5 M) et 500 ul de HBS 2X (NaCl 280 mM, KCl 10 mM,
Na2HPO4 1,2 mM, Glucose 12 mM, Hépès
50 mM).
Les cellules HL-60 sont électroporées avec une
solution plasmidique équivalente.
Après une incubation de 12 heures à
37oC, les cellules sont lavées 2 fois au PBS 1X. Un tampon de
lyse (200 ul) est ajouté dans chaque puits et les cellules sont
décollées à l'aide d'un grattoir. Les cellules sont
récupérées dans des tubes Eppendorff, vortexées et
centrifugées 2 min. à 12000g et à 4oC.
L'activité de la luciférase (selon le protocole de Luciferase
Assay System, Promega, Madison, EUA) dans le lysat est mesurée dans un
luminomètre et exprimée en unité arbitraire.
L'activité de la -Galactosidase (selon le protocole de BM
Chemiluminescence ELISA Substrate -Gal, Boehringer Mannheim, Mannheim,
République Fédérale d'Allemagne) est également
mesurée afin de normaliser les mesures obtenues de l'activité
luciférase.
f)
Technique de Western Blot
Des extraits protéiques totaux sont
réalisés. Les cellules sont lavées deux fois au PBS 1X, le
culot est repris dans 300ul de tampon d'extraction (Hepès 20 mM pH
8, NaCl 450 mM, EDTA 0,4 mM et glycérol 25%) contenant des
inhibiteurs de protéases (pepstatine, leupeptine, aprotinine). Pour la
lyse des cellules, trois bains successifs dans l'azote et à 37°C
sont effectués. Après centrifugation (15min. à 14000 rpm),
le surnageant correspondant à l'extrait total est prélevé,
constitué en fractions aliquotes et congelé à
-80°C.
Après une dénaturation de 10 min. à
100oC, les échantillons de protéines sont
déposés sur un gel dénaturant d'acrylamide 12 %. La
migration se fait en 2 étapes, tout d'abord à 80 V pendant 15
min. puis à 125 V pendant une heure. Les protéines sont ensuite
transférées sur une membrane de nitrocellulose par
électrotransfert à 600 mA pendant 60 min. au froid. Pour utiliser
les anticorps anti-RAR et anti-RXR, la membrane est lavée deux fois avec
du PBS 1X puis saturée avec du lait 5 % pendant 2 heures à
température ambiante. Durant toute la nuit, la membrane est
incubée à 4oC avec l'anticorps primaire anti-RAR
dilué au 1/100è et anti-RXR dilué au
1/100è. Lavée 5 fois 10 min. avec du PBS 1X, la
membrane est re-saturée avec du lait (2,5 %) pendant 10 min. puis
incubée avec l'anticorps secondaire pendant 30 min. (Protéine A
diluée au 1/10000è). Puis, 5 lavages sont
effectués au PBS 1X contenant 0,01 % de Tween.
Pour l'anticorps anti-myc, la membrane est saturée avec
du PBS Tween 20 à 0,1 % pendant 2 heures à température
ambiante. Plusieurs lavages au PBS Tween sont faits pendant deux min. La
membrane est incubée à 4oC toute la nuit avec
l'anticorps primaire anti-myc dilué au 1/10000è.
Lavée plusieurs fois pendant 5 min. avec 500 ml de PBS Tween, la
membrane est incubée avec l'anticorps secondaire pendant une heure
(anti-kappa de souris dilué au 1/1500è). Puis les
membranes sont lavées avec 1l de PBS Tween pendant au moins trois fois
15min..
Enfin, la membrane est mise en présence d'une solution
de détection (ECL, Amersham, Piscataway, NJ, EUA) durant une minute. La
radiographie est faite en exposant un film à des temps variables
(Hyperfilm ECL, Amersham, Piscataway, NJ, EUA).
g)
Technique du retard sur gel
La sonde DR5, qui correspond à l'élément
de réponse RARE du promoteur de RAR, est marquée à son
extrémité 5' en incubant pendant 60 min. à
37oC: 30 ng de la sonde, du tampon kinase 10X, 3 l
d'ATPP32, 1 l de T4 polynucléotide kinase (5 unités).
Après avoir purifié la sonde marquée sur une colonne
(centri.spin-40, préalablement hydratée avec 0,5 ml de tampon TE
1X pendant 30 min. et centrifugée 2 min. à 700 g à
température ambiante) en la centrifugeant 2 min. à 700 g à
température ambiante, le contrôle de l'activité
spécifique est réalisé grâce à un compteur
à scintillation. La sonde double brin est élaborée en
ajoutant à la sonde marquée 60 ng du brin complémentaire,
du tampon kinase 10X et 150 mM de NaCl. La solution est dénaturée
à 100oC puis progressivement refroidie à
37oC.
Les extraits protéiques et les anticorps sont
incubés 20 min. sur la glace avec une solution contenant 2 ul de tampon
Darnell (KCl 400 mM, Hepès 200 mM, MgCl2 10 mM, EGTA 1
mM, DTT 5 mM, Ficoll 4%, pH 7,5), 1 ul de poly dI-dC (1 ug/ul) et 40000
cpm de la sonde radiomarquée. Les extraits sont ensuite
déposés sur un gel de polyacrylamide 4 % et migrés
à 200 V durant 82 min. Le gel est séché à vide
pendant 120 min à 80°C. Une autoradiographie est
réalisée avec un écran amplificateur en présence
d'un film Kodak X-Omat AR à -800C durant 12 heures.
III- RESULTATS
1. TLS est un co-activateur du RANC
Premièrement, nous avons analysé l'effet d'une
surexpression transitoire de TLS sur l'expression d'un gène rapporteur
(luciférase) sous le contrôle du promoteur RARß2 (Figure
4A). Nous avons utilisé comme modèle cellulaire les cellules
hématopoïétiques HL-60. Ces cellules sont
électroporées en présence de Tkßgal, pour
évaluer l'efficacité de transfection et normaliser les
résultats, de ßRARE-luc, utilisé comme gène
rapporteur pour évaluer l'activité transcriptionnelle et de
différentes quantités de vecteur d'expression de TLS, comme
indiquées. pCS3, un plasmide contrôle est utilisé de telle
façon que toutes les transfections aient la même quantité
totale de plasmide d'expression (Figure 4). Les cellules sont
récoltées 24 h après et l'activité
luciférase est mesurée. Les valeurs représentent les
moyennes des doublets réalisés. Des résultats similaires
furent obtenus dans plusieurs expériences indépendantes.
Les résultats exprimés correspondant à
l'augmentation de l'activité de la luciférase par rapport
à un témoin négatif (cellules transfectées avec
pCS3 seul, en absence de ligand).
Lors de la transfection en présence d'AR seul (1'), une
augmentation de l'activité luciférase est observée. La
co-transfection par pCS3-MT-TLS seul et RARE-luc (2 et 3) n'a pas d'effet sur
l'induction de l'activité luciférase, sauf en présence
d'AR (ATRA 10-6 M) (2' et 3', multiplié par 25, en
présence de 5 ug et 10 uG de TLS (Figure 4B). Ces résultats
suggèrent que TLS augmente l'activité transcriptionnelle induite
par les récepteurs endogènes dans les cellules HL-60.
Afin de déterminer que la co-activation par TLS agit
par et grâce au RANC, nous avons utilisé des cellules Cos-6
possédant peu de récepteurs aux rétinoïdes
endogènes que nous avons transfectées avec les plasmides
d'expression de RAR et de RXR. Les cellules Cos-6 sont donc
co-transfectées au chlorure de calcium avec les plasmides TKgal et RARE,
comme décrit précédemment.

Figure 4 . TLS augmente l'activité
transcriptionnelle des récepteurs nucléaires à l'acide
rétinoïque dans les cellules HL-60. A, schéma
descriptif de la construction utilisée dans le test de transactivation.
B, L'activation transcriptionnelle du RARE a lieu avec l'AR seul ou avec TLS de
façon dépendante de l'AR dans les cellules HL-60.
Dans la Figure 5, les contrôles (1 et 4) indiquent la
transactivation basale du gène rapporteur. Lors de la transfection en
présence d'AR seul (1' et 4', multiplié par 40) ou avec TLS (2',
3', 5', 6' et 7'), une induction est observée. L'induction est optimale
avec 2 ug de TLS (3' et 5', multiplié par 85). TLS est donc un
co-activateur du complexe transcriptionnel des récepteurs RAR et RXR,
dépendant de l'AR. A des quantités plus importantes, 5 ug et 10
ug de TLS (6' et 7'), une diminution de l'induction en présence d'AR,
inférieure au niveau atteint en absence de TLS, est observée.
Cette diminution pourrait être dû à un effet de
« quenching » ou à l'effet de TLS qui, en grandes
quantités, empêcherait RXR de se lier à l'ADN.
L'ensemble de ces résultats indiquent que la
protéine TLS est un co-activateur de la transcription par les
récepteurs à l'AR. TLS semble donc impliquée dans la voie
de signalisation de l'AR.

Figure 5 A et B. TLS agit comme co-activateur du
RANC, de façon dose dépendante. Les deux
expériences sont réalisées indépendamment.
2. TLS fait parti du RANC
L'effet de TLS sur la transcription passant par la voie de
signalisation de l'AR suggère que TLS puisse faire partie du RANC,
comprenant les récepteurs nucléaires. Afin de déterminer
l'appartenance de TLS au RANC, des expériences de retard sur gel ont
été réalisées.
Des extraits protéiques de Cos-6 transfectés par
chlorure de calcium avec les plasmides d'expression de RAR, RXR et TLS sont
utilisés dans les expériences de retard sur gel.
Ces extraits protéiques totaux de Cos-6 sont
préalablements vérifiés par Western Blot après
immunoblotting avec des anticorps spécifiques, dont l'anti-myc pour la
protéine étiquetée TLS (Figure 6).

Figure 6 : Analyse par Western Blot de l'expression
protéique des Cos-6 transfectées
La technique de retard sur gel est réalisée avec
les extraits protéiques totaux testés précédemment
en présence ou en absence de TLS (Figure 7).
RXR RAR
DR5
Figure 7. schéma descriptif de la construction
utilisée dans le retard sur gel et de la fixation du RANC
Le signal en 1 correspond à la fixation de RAR et RXR
sur le DR5. Le complexe observé est ainsi constitué des
récepteurs nucléaires et est appelé RANC. Le signal 2
indique que RXR en fait partie. L'intensité et le retard des bandes 3 et
4 indiquent que TLS fait partie du RANC. A partir de 1 ul (bande 4), plus la
quantité de TLS est augmentée, plus le complexe semble
déstabilisé (bande 5 et 6). La quantité maximale de TLS
pour sa participation au RANC est de 1 ul d'extraits protéiques totaux.
Au delà de cette confirmation et en moyennant des expériences de
contrôles des phénomènes de compétition ou
d'allostérie, TLS pourrait interférer avec le complexe, pouvant
empêcher RXR de se lier à l'ADN.
En conclusion, ces résultats préliminaires
suggèrent fortement que TLS augmenterait la transcription de
gènes cibles de l'AR de manière dépendante du ligand dans
les cellules myéloïdes et que ceci serait lié à une
participation directe de TLS dans le complexe transcriptionnel RANC. Les
expériences dans les cellules Cos-6 montrent que cette régulation
par TLS et l'AR pourrait être un phenomène
général.

Figure 8. Retard sur gel réalisé avec
RAR et RXR, en présence ou absence de TLS.
L'effet de la dose de TLS sur le RANC est
déterminé.
3. Analyse in vivo de l'activité de
TLS et de l'acide rétinoïque sur l'épissage alternatif en 5'
de E1A
A la lumière des résultats obtenus, il est clair
que TLS agit sur la voie de signalisation aux rétinoïdes au moins
par son activité transcriptionnelle.
Etant donné que TLS peut jouer un rôle de facteur
d'épissage en favorisant la sélection du site 5' distal
d'épissage de l'ARN pré-m du minigène E1A dans les
cellules érythroïdes murines IW1-32, nous avons
étudié le rôle de TLS sur l'épissage de l'ARN
pré-m du minigène E1A dans un modèle
hématopoïétique humain, la lignée
myéloïde K562.
L'ARN pré-m E1A contient des sites 5' d'épissage
qui mènent à la formation de plusieurs isoformes principales
d'ARN (dont les 13S, 12S et 9S). Ces isoformes peuvent être mises en
évidence et quantifiées par PCR à l'aide d'amorces
spécifiquements choisies (Figure 9).
 Figure 9. Représentation schématique de
l'épissage alternatif des transcrits de E1A. Les isoformes de
l'ARN pré-m de E1A et la taille correspondante des produits de RT-PCR
sont montrés. Les amorces pour l'analyse en RT-PCR sont indiquées
par des flèches. Figure 9. Représentation schématique de
l'épissage alternatif des transcrits de E1A. Les isoformes de
l'ARN pré-m de E1A et la taille correspondante des produits de RT-PCR
sont montrés. Les amorces pour l'analyse en RT-PCR sont indiquées
par des flèches.
Afin d'étudier le rôle éventuel de TLS ou
de l'AR sur l'épissage en 5' du minigène E1A, nous avons
co-transfecté transitoirement les cellules K562 par la lipofectamine
avec les plamides d'expression de E1A, de pCS3 (pour uniformiser les
quantités de plasmide utilisés), de TLS (les quantités
utilisées sont indiquées) et de CMV-luc (pour mesurer
l'efficacité de la transfection). Lorsque la luciférase
s'exprime, les ARNs sont extraits, puis rétrotranscrits. Puis une PCR
est réalisée. La détection des produits de PCR est faite
après séparation sur un gel PAGE-urée par autoradiographie
(Figure 10). Enfin, l'analyse quantitative des résultats est obtenu par
le système Molecular Analyst de Biorad (Figure 11).
Après s'être assuré de l'efficacité
de la transfection, il convient d'obtenir les conditions expérimentales
adéquates pour s'assurer de la détection de toutes les isoformes.
Il s'agit de faire varier soit la quantité de produits de RT
utilisé pour la PCR, soit le nombre de cycles de la PCR, soit
l'exposition de l'autoradiographie. Chaque expérience est faite en
doublet et reproduite pour s'assurer de la reproductibilité des
résultats obtenus. L'étude est basée sur le choix du site
5' d'épissage du minigène E1A que l'on déduit de
l'expression en pourcentage des isoformes 13S, 12S et 9S. Il est, de plus,
impératif de visualiser une bande correspondant à la forme non
épissée de E1A pour que les résultats soient
interprétables.
En comparant les résultats sans AR (1) aux
résultats avec l'AR (2), il est possible d'affirmer, sous réserve
de contrôles supplémentaires (entre 40 et 60 % de 9S) qu'il ne
semblerait pas que l'AR seul ait un effet significatif sur l'épissage de
E1A dans K562.
Par contre, les résultats en présence de TLS
sont reproductibles et confirment que dans les cellules K562, la
protéine TLS permet un épissage préférentiel de
l'isoforme 9S au détriment des isoformes 13S et 12S. Ce
phénomène serait augmenté en présence d'AR. Les
résultats (3) montrent que TLS seule favorise la formation de l'isoforme
9S (75 % de 9S), alors que lorsque l'AR est utilisé avec TLS, l'isoforme
9S est considérablement augmentée (90 % de 9S).
Ces résultats indiquent que TLS agit sur la
sélection du site 5' distal d'épissage, favorisant la formation
de l'isoforme 9S du minigène E1A, phénomène
accentué par l'AR. Ainsi, ces résultats suggèrent que TLS
agit sur l'épissage dans les cellules
hématopoïétiques humaines et que l'AR intervient sur
l'épissage à travers TLS.
En conclusion, ces résultats montreraient pour la
première fois que l'AR serait impliqué directement dans le
contrôle post-transcriptionnel à travers la protéine
TLS.

Figure 10. L'épissage alternatif in
vivo du minigène E1A dans les cellules K562.
L'intensité des bandes correspondant aux
différentes isoformes de E1A pour chaque profil est quantifiée
par densitométrie. Les résultats sont exprimés
en pourcentage des isoformes 13S, 12S et 9S. Les études sont faites
en doublets. Le RT- représente le contrôle négatif de la
RT, elle a été réalisée sans enryme RT et aucun ADN
n'est révélé. Les ARNs extraits et utilisés sont
ainsi exempts d'ADN.

Figure 11. Quantification des isoformes des ARN
pré-m de E1A par un logiciel Molecular Analyst de Biorad . Le
pourcentage des isoformes 13S, 12S et 9S est représenté.
IV - DISCUSSION
Ces travaux de recherche concernent l'étude du
rôle de TLS dans les voies de signalisation de l'acide
rétinoïque. TLS est une protéine bi-fonctionnelle dans le
mécanisme d'action de l'AR au niveau de la régulation
transcriptionnelle et de la régulation post-transcriptionnelle par l'AR.
En effet, des expériences de transactivation et de retard sur gel
mettent en évidence que TLS participe au complexe transcriptionnel de
l'hétérodimère RXR-RAR, appelé RANC permettant
d'augmenter la transactivation dépendante de l'AR sur les promoteurs de
gènes cibles. Dans un premier temps, des techniques de transfection
transitoire dans des cellules Cos-6 et dans les cellules myéloblastiques
HL-60 démontrent que TLS est capable d'augmenter l'activité
transcriptionnelle du promoteur naturel de RAR sensible à l'action de
l'acide rétinoïque en présence d'acide
rétinoïque. Il avait été précédemment
mis en évidence que TLS possédait une activité
transcriptionnelle per se. En effet, la partie N-terminale de TLS
impliquée dans la protéine de fusion TLS/ERG est
considérée comme un pré-requis pour le facteur de
transcription ERG altéré dans son potentiel
leucémogène en augmentant son activité transcriptionnelle
et/ou en changeant sa spécificité au niveau des gènes
cibles (Zinszner et coll., 1994 ; Ichikawa et coll. , 1999). En outre, il
a été mis en évidence que la partie N-terminale de TLS
fusionnée au domaine de liaison de l'ADN de Gal4 possède une
forte activité transcriptionnelle (Uranishi et coll., 2001).
Afin de déterminer si TLS faisait partie du RANC, des
expériences de retard sur gel ont été effectuées en
utilisant comme sonde oligonucléotidique DR5. A cette fin, des extraits
de cellules Cos-6 transfectées par RAR, RXR ou TLS ont été
utilisées. Les résultats démontrent que TLS fait partie de
RANC. Une autre étude réalisée in vitro montre
que cette interaction est directe et indépendante de la présence
de l'acide rétinoïque et semble spécifique aux
récepteurs à l'acide rétinoïque RXR, aux
glucocorticoïdes, aux oestrogènes, et à l'hormone
thyroïdienne (Powers et coll., 1998). Cette interaction directe est
indépendante de la présence du ligand. Cette observation est due
au fait que le complexe se forme entre le domaine de liaison de l'ADN (domaine
C) et la région D du récepteur nucléaire avec la partie
N-terminale de TLS (Powers et coll., 1998). La région C, composée
de 66 acides aminés, est la plus conservée parmi tous les membres
de la superfamille des récepteurs nucléaires. Elle contient deux
structures de « doigt à zinc »
et correspond au domaine de liaison du récepteur nucléaire
à l'ADN. La région D, appelée également zone
charnière, se situe entre le domaine de liaison à l'ADN et la
région E. Elle est subdivisée en trois sous-régions (D1,
D2 et D3) dont la région D1 N-terminale, la plus conservée,
contient de nombreux acides aminés basiques qui correspondraient
à un signal de localisation nucléaire. La région centrale
D2 est plus variable. Il est intéressant de noter que l'interaction de
TLS avec le domaine de liaison à l'ADN de RXR n'altère pas la
liaison du récepteur nucléaire sur son élément de
réponse spécifique.
La fonction co-activatrice de TLS au niveau du complexe
transcriptionnel des récepteurs à l'acide rétinoïque
pourrait sembler paradoxale dans le fait que l'interaction de TLS avec RXR
implique son domaine de liaison de l'ADN. Cependant, cette interaction n'est
pas déstabilisatrice du complexe protéine/ADN. Bien au contraire,
puisqu'il est établi que cette interaction
protéine/protéine soit stabilisatrice du complexe RXR-ADN (Powers
et coll., 1998). De plus, cette interaction n'empêche pas le changement
de conformation des récepteurs nucléaires. Ainsi, TLS par son
action stabiliserait le complexe co-activateur des récepteurs à
l'acide rétinoïque. Les principaux complexes co-activateurs des
récepteurs nucléaires comprennent le complexe Brg (SWI/SNF), les
co-intégrateurs CBP et p300, la famille des protéines p160, la
protéine p/CAF et les complexes TRAP/DRIP/ARC.
TLS a déjà été impliquée
dans le complexe transcriptionnel d'un autre facteur de transcription, NFB.
Ainsi, TLS augmente la transactivation dépendante de NFB induite par des
stimuli physiologiques tels que TNF et l'IL-1 (Uranishi et coll., 2001). TLS
agit donc en tant que co-activateur de divers facteurs de transcriptions.
Enfin, TLS partage des caractéristiques structurales
avec hTAFII68. Ces protéines ont été retrouvées
associées avec les complexes TFIID (Bertolotti et coll., 1997 et 1998)
et sont impliquées dans l'activation transcriptionnelle (Prasad et
coll., 1994 ; Zinszner et coll., 1994 ; Bertolotti et coll.,
1999 ; Ichikawa et coll., 1999). TLS interagit in vivo avec TFIID
(Uranishi et coll., 2001). De plus, TLS est associée à l'ARN
polII via son domaine N-terminal (Yang et coll., 2000). L'ensemble de
ces interactions pourrait permettre à TLS de faire le lien
moléculaire entre des facteurs de transcription et le complexe
d'initiation de la transcription.
L'ensemble de ces études implique de façon
indéniable TLS dans la régulation transcriptionnelle au niveau
des voies de signalisation des rétinoïdes.
La seconde partie du projet de recherche concerne le lien
éventuel entre les voies de signalisation des rétinoïdes et
la régulation post-transcriptionnelle à travers la modulation de
l'épissage alternatif. En effet, le fait que TLS, facteur
d'épissage, soit impliquée dans les voies de signalisation de
l'AR à travers son rôle direct dans la coactivation
transcriptionnelle dépendante du RANC incite à étudier le
rôle de l'acide rétinoïque et de ses récepteurs dans
l'épissage.
Les résultats expérimentaux montrent que TLS
agit in vivo sur la sélection du sites 5' d'épissage
alternatif de l'ARN pré-m E1A dans les cellules
hématopoïétiques K562. La sélection du site 5' distal
de E1A correspondant à la formation de l'isoforme 9S est
favorisée au détriment de celle des sites proximaux 12S et 13S.
Certaines données nous aiguillent sur le rôle de
TLS dans la régulation de l'épissage alternatif in vivo.
TLS est associée aux RNPs en formant in vivo un complexe avec
la hnRNP A1, un facteur d'épissage capable de favoriser la
sélection du site 5' distal d'épissage pendant un épissage
alternatif de n'importe quel ARN pré-m (Uranishi et coll., 2001). Il a
été précédemment montré que TLS agissait sur
la sélection du site distal de E1A dans les cellules
érythroblastiques de Souris IW1-32 (Hallier et coll., 1998). Les
expériences effectuées sur les cellules K562 transfectées
par E1A permettent de mettre en évidence une augmentation du taux
d'isoforme 9S en présence d'AR et de TLS. L'interférence
fonctionnelle entre TLS et l'acide rétinoïque peut être due
à des interférences moléculaires, c'est à dire des
interactions physiques protéine/protéine. Cette hypothèse
peut s'appuyer sur l'interaction directe montrée par des travaux
précédents. Par conséquent, ces résultats
identifient l'acide rétinoïque en tant qu'acteur impliqué
dans la régulation de l'épissage. C'est la première fois
qu'il est mis en évidence l'action d'une hormone sur l'épissage.
Ces travaux permettent de définir un nouveau niveau de régulation
des voies de signalisation des rétinoïdes.
Le fait que TLS interagisse avec RXR, TR et GR permet de
raisonnablement penser que ce phénomène se retrouve à un
niveau plus général.
L'épissage de l'ARN, étape critique de
l'expression des gènes, est de plus en plus considéré
comme un événement co-transcriptionnel. Des évidences
expérimentales indiquent désormais que les étapes de
transcription, le « capping » et la polyadénylation,
sont intimement liées à l'ARN polII à travers son
association avec les facteurs impliqués dans ces mécanismes
moléculaires (Cho et coll., 1997 ; Hirose et coll., 1998). Les
protéines de régulation de l'épissage, les
protéines SR, sont associées à l'ARN polII. Cependant, les
moyens par lesquels cette association s'effectue n'étaient pas
identifiés. Le co-activateur transcriptionnel p52 est capable
d'interagir avec la protéine SR, ASF/SF2. p52 agit ainsi en tant
qu'adaptateur afin de coordonner la transcription et l'épissage (Ge et
coll., 1998). TLS interagit aussi avec l'ARN polII et les protéines SR
(Yang et coll. , 2000). TLS pourrait donc agir en tant que molécule
recruteuse des facteurs de régulation de l'épissage SR vers l'ARN
polII, couplant ainsi la transcription avec l'épissage. TLS pourrait
aussi agir directement sur l'épissage par son interaction avec l'ARN
(Lerga et coll., 2001).
TLS contient trois domaines potentiellement impliqués
dans la liaison à l'ARN, RGG1, RRM et RGG2-3. Le mécanisme
impliqué dans la reconnaissance de l'ARN par TLS est encore inconnu. La
structure secondaire de l'ARN représente une part importante des
interactions protéine/ARN. Les séquences
sélectionnées se fixent à TLS avec des affinités de
l'ordre de 250 nM à 600 nM, le Kd de 250 nM correspondant au complexe
ggugARN/TLS (Lerga et coll., 2001). Par conséquent, TLS semble
être une protéine possédant une faible affinité pour
sa séquence ARN. Ainsi, il n'est pas exclu que le Kd du complexe
TLS/ggugARN déterminé in vitro soit
éloigné du Kd d'un complexe ARN/protéine au niveau du
spliceosome. Cependant, une faible affinité d'un facteur
d'épissage pour sa séquence ARN cible pourrait représenter
une condition compatible avec l'assemblage du
« spliceosome » qui se déroule à travers
l'échange et le replacement d'un grand nombre de protéines au
niveau du substrat,l'ARN pré-m.
Les fonctions de TLS dans la régulation de la
transcription et dans l'épissage permettent d'envisager les
conséquences d'une altération de TLS dans certaines pathologies.
Dans les cellules leucémiques myéloïdes possédant la
translocation t(16;21) et dans les cellules de liposarcome dans la
translocation t(12;16), un seul allèle est interrompu alors que l'autre
allèle est intact (Crozat et coll. , 1993 ; Rabbitts et coll.,
1993 ; Yamamoto et coll., 1997). Ces observations suggèrent un
rôle de protéine de fusion générée de type
dominante négative. La protéine de fusion TLS/ERG,
observée dans la leucémie myéloïde humaine t(12;16),
pertuberait l'épissage in vivo de E1A observé dans des
cellules HeLa (Yang et coll., 2000). Il est à noter que TLS
n'était pas considérée capable d'agir sur E1A dans ces
cellules, ce qui semble erroné à partir d'autres observations
effectuées (F. Moreau-Gachelin, communication personnelle).
Néanmoins, TLS est capable d'inhiber la fonction des TASR sur
l'épissage de l'ARN pré-m E1A (Yang et coll., 2000). De plus, il
est intéressant de noter que l'épissage de CD44 est
altéré par la présence de la protéine de fusion
TLS/ERG dans des clones stables générés à partir de
cellules de la lignée K562 (Yang et coll., 2000). Le gène
CD44 code pour une molécule d'adhésion constituée
de dix exons constitutifs et dix exons variables. Différentes
combinaisons formées par les exons variables est à l'origine
d'une grande variété d'isofomes d'épissage de CD44 qui
diffèrent au niveau de leur domaine extracellulaire. L'épissage
anormal de l'ARN pré-m CD44 a été retrouvé dans
diverses tumeurs solides et des leucémies (Cooper et coll., 1995). En
outre, Il a été suggéré que les variations en taux
protéique des SR selon les différentes étapes du
développement du cancer du sein puissent être directement
liées aux variations des diverses isoformes de CD44 (Stickeler et coll.
, 1999). Deux mécanismes sont envisagés pour expliquer l'effet
dominant négatif de TLS/ERG sur l'épissage de CD44. Le premier
considère que TLS s'accrochant aux isoformes spécifiques de CD44,
TLS/ERG bloquerait cette voie de régulation, à l'origine d'une
dégradation prématurée de l'ARN pré-m de CD44 non
épissé complètement. Dans le second mécanisme, si
l'une des voies de régulation est bloquée et qu'une autre voie de
régulation de l'épissage alternatif existe, l'inhibition de la
première voie par TLS/ERG pourrait inciter l'épissage de CD44
à travers une autre voie de régulation, augmentant ainsi le
risque de générer un épissage aberrant.
Si TLS est exprimée de façon ubiquitaire, il
est intéressant de noter des différences d'expression dans les
cellules hématopoïétiques normales et pathologiques. Les
cellules souches hématopoïétiques purifiées de sang
de cordon ont des taux inférieurs de TLS comparées aux cellules
myéloïdes. D'autre part, une expression importante de TLS fut mise
en évidence dans des cellules de LAM (Mills et coll., 2000). Il est
intéressant de noter que TLS a été également
identifiée par son taux d'expression diminué dans les cellules
HL-60 traitées par l'acide rétinoïque pendant une heure. De
plus, l'expression de TLS est fortement diminuée dans les cellules HL-60
au cours de la granulopoïèse induite par le DMSO ou l'ATRA. Il
existe une spécificité à la granulopoïèse
puisque la différenciation monocytaire induite par le TPA ou la vitamine
D3 n'agit pas sur l'expression de TLS (Mills et coll., 2000). Cette diminution
d'expression est associée avec la baisse de la prolifération
cellulaire dans les diverses LAM. L'ensemble de ces observations suggère
que l'expression de TLS est un régulateur clef de la
myélopoïèse où un fort taux d'expression de TLS
favorise la prolifération cellulaire au détriment de la
différenciation.
Les perspectives de ces travaux s'articulent sur deux axes.
Le premier concerne les mécanismes moléculaires fondamentaux qui
impliquent TLS dans les voies de signalisation de l'acide
rétinoïque. Nous envisageons d'étudier TLS dans la
régulation de l'épissage constitutif et l'épissage
alternatif en 3' d'épissage. L'effet de l'AR sur l'épissage de
E1A sera aussi étudié à travers les rôles de RAR et
RXR. Il s'agit aussi de déterminer avec précision l'association
de TLS dans le complexe transcriptionnel de l'AR. Ainsi, des expériences
d'identification des interactions directes entre TLS et RAR seront
effectuées sachant qu'elles ont été mises en
évidence avec RXR (GST pull down et co-immunoprécipitation). Nous
voulons étudier la coordination de l'épissage et de la
transcription pour savoir si ces mécanismes sont dépendants ou
indépendants l'un de l'autre (une construction réunissant les
gènes E1A et la luciférase sous la dépendance d'un
promoteur muté sensible à l'AR, ne permettant pas au RANC de se
fixer pour augmenter la transcription). Le rôle biologique de TLS sera
déterminé par son invalidation transitoire (RNAi). Le
deuxième axe consiste à confirmer le lien entre TLS et la
pathologie. Il s'agit de comprendre les conséquences de
l'altération de l'expression de TLS et son ciblage dans le cadre d'une
nouvelle prospective thérapeutique et diagnostique.
V - REFERENCES
Allegretto, E. A., McClurg, M. R., Lazarchik, S. B., Clemm, D.
L., Kerner, S. A., Elgort, M. G., Boehm, M. F., White, S. K., Pike, J. W., and
Heyman, R. A. (1993). Transactivation properties of retinoic acid and retinoid
X receptors in mammalian cells and yeast. Correlation with hormone binding and
effects of metabolism. J. Biol. Chem. 268, 26625-26633.
Allenby, G., Bocquel, M. T., Saunders, M., Kazmer, S., Speck,
J., Rosenberger, M., Lovey, A., Kastner, P., Grippo, J. F., and Chambon, P.
(1993). Retinoic acid receptors and retinoid X receptors: interactions with
endogenous retinoic acids. Proc. Natl. Acad. Sci. U.S.A. 90, 30-34.
Aman, P., Panagopoulos, I., Lassen, C., Fioretos, T.,
Mencinger, M., Toresson, H., Hoglund, M., Forster, A., Rabbitts, T. H., and
Ron, D., (1996). Expression patterns of the human sarcoma-associated genes FUS
and EWS and the genomic structure of FUS. Genomics 37, 1-8.
Beaschtold, H., Kuroda, M., Sock, J., Ron, D., and Lopez, B.L.
(1999). Human 75 Kda DNA-pairing Protein is identical to the pro-oncoprotein
TLS/FUS and is able to promote D-loop formation. J. Biol. Chem. 274,
34337-34342.
Bertolotti, A., Lutz, Y., Heard, D. J., Chambon, P., and Tora,
L. (1996). hTAF(II)68, a novel RNA/ssDNA-binding protein with homology to the
pro-oncoproteins TLS/FUS and EWS is associated with both TFIID and RNA
polymerase II. EMBO J. 15, 5022-5031.
Bertolotti, A., Melot, T., Acker, J., Vigneron, M., Delattre,
O., and Tora, L. (1998). EWS, but not EWS-FLI-1, is associated with both TFIID
and RNA polymerase II: interactions between two members of the TET family, EWS
and hTAFII68, and subunits of TFIID and RNA polymerase II complexes. Mol. Cell.
Biol. 18, 1489-1497.
Bretnach, R., and Chambon, P. (1981). Organization and
expression of eukaryotic split genes coding for proteins. Annu. Rev.
Biochem., 50, 349-384.
Breitman, T. R., Selonick, S. E., and Collins, S. J. (1980).
Induction of differentiation of the human promyelocytic leukemia cell line
(HL-60) by retinoic acid. Proc. Natl. Acad. Sci. U.S.A. 77,
2936-2940.
Burd, C. G., and Dreyfuss, G. (1994). RNA binding specificity
of hnRNP A1: significance of hnRNP A1 high-affinity binding sites in pre-mRNA
splicing. EMBO J. 13, 1197-1204.
Chambon, P. (1996). A decade of molecular biology of retinoic
acid receptors. FASEB J. 10, 940-954.
Chen, Z., Brand, N.J., Chen A., Chen, S.J., Tong, J.H., Wang,
Z.Y., Waxman, S., and Zelent, A. (1993). Fusion between a novel
krüpper-like zinc finger gene and the retinoic acid receptor-alpha locus
due to a variant t(11 ;17) translocation associated with acute
promyelocytic leukemia. EMBO J. 12, 1161-1167.
Cho, E.J., Takagi, T., Moore, C.R., Buratowski, S. (1997).
mRNA capping enzyme is recruited to the transcription complex by
phosphorylation of the RNA polymerase II carboxy terminal domain. Genes Dev
11, 3319-3326.
Chomienne, C., Fenaux, P., and Degos, L. (1996). Retinoid
differentiation therapy in promyelocytic leukemia. FASEB J. 10,
1025-1030.
Cooper, D.L., and Dougherty G.J. (1995). To metastasize or
not? Selection of CD44 splice sites. Nat. Med. 1, 635-637.
Croft, L., Schandorff, S., Clark, F., Burrage, K., Arctander,
P., and Mattick, J.S. (2000). ISIS, the intron information system, reveals the
high frequency of alternative splicing in the human genome. Nat. Genet.
24, 340-341.
Crozat, A., Aman, P., Mandahl, N., and Ron, D. (1993). Fusion
of CHOP to a novel RNA-binding protein in human myxoid liposarcoma. Nature
363, 640-644.
Delattre, O., Zucman, J., Plougastel, B., Desmaze, C., Melot,
T., Peter, M., Kovar, H., Joubert, I., de Jong, P., and Rouleau, G. (1992).
Gene fusion with an ETS DNA-binding domain caused by chromosome translocation
in human tumours. Nature 359, 162-165.
Douer, D., and Koeffler, H. P. (1982). Retinoic acid.
Inhibition of the clonal growth of human myeloid leukemia cells. J. clin.
invest. 69, 277-283.
Felsenfeld, G., Boyes, J., Chung, J., Clark, D., and
Studitsky, V. (1996). Chromatin structure and gene expression. Proc. natl.
acad. sci. u.s.a. 93, 9384-9388.
Fenaux, P., Chastang, C., Chomienne, C. and Degos, L.
(1994).All-trans retinoic acid (ATRA) in combination with chemotherapy improves
survival in newly diagnosed acute promyelocytic leukemia (APL). Lancet,
343, 1033.
Ge, H., Si, Y., and Roeder, R. G. (1998). Isolation of cDNAs
encoding novel transcription coactivators p52 and p75 reveals an alternate
regulatory mechanism of transcriptional activation. EMBO J. 17,
6723-6729.
Giguère, V. (1994). Retinoic acid receptors and
cellular retinoid binding proteins: complex interplay in retinoid signaling.
Endocr. Rev. 15, 61-79.
Giguere, V., Ong, E. S., Segui, P., and Evans, R. M. (1987).
Identification of a receptor for the morphogen retinoic acid. Nature
330, 624-629.
Gratas, C., Menot, M. L., Dresch, C., and Chomienne, C.
(1993). Retinoic acid supports granulocytic but not erythroid differentiation
of myeloid progenitors in normal bone marrow cells. Leukemia 7,
1156-1162.
Green S. and Chambon P. (1986). Carcinogenesis: a superfamily
of potentially oncogenic hormone receptors. Nature 324, 615-617.
Hallier, M., Lerga, A., Barnache, S., Tavitian, A.,
Moreau-Gachelin F. , (1998). The transcription factor Spi-1/PU.1 interacts with
the potential splicing factor TLS. J. Biol. Chem..273(9), 4838-42.
Hicks, G.G., Singh, N., Nashabi, A., Mai, S., Bozeck, G.,
Klewes, L., Arapovic, D., White, E.K., Koury, M.J., Oltz, E.M., Van Kaer, L.,
and Ruley, E.H. (2000). Fus deficiency in mice results in defective
B-lymphocyte development and activation, high levels of chromosomal instability
and perinatal death. Nature Genetics 24, 175-179.
Hirose, Y., and Manley, J.L. (1998). RNA polymerase II is an
essential mRNA polyadenylation factor. Nature 395, 93-96.
Ichikawa, H., Shimizu, K., Katsu, R., and Ohki, M. (1999) Dual
transforming activities of the FUS (TLS)-ERG leukemia fusion protein conferred
by two N-terminal domains of FUS (TLS). Mol. Cell. Biol. 15,
2513-2526.
Kastner, P., Grondona, J. M., Mark, M., Gansmuller, A.,
LeMeur, M., Décimo, D., Vonesch, J. L., Dollé, P., and Chambon,
P. (1994). Genetic analysis of RXR alpha developmental function: convergence of
RXR and RAR signaling pathways in heart and eye morphogenesis. Cell
78, 987-1003.
Krust, A., Kastner, P., Petkovich, M., Zelent, A., and
Chambon, P. (1989). A third human retinoic acid receptor, hRAR-gamma. Proc.
natl. acad. sci. u.s.a. 86, 5310-5314.
Lerga, A., Hallier, M., Delva, L., Orvain, C., Gallais, I.,
Marie, J., Moreau-Gachelin F. (2001), Identification of an RNA binding
specificity for the potential splicing factor TLS.J .Biol .Chem..
276(9), 6807-16.
Mills, K.I., Walsh, V., Gilkes, A.F., Sweeney, M.C., Mirza,
T., Woodgate L.J., Brown G. and Burnett A.K. (2000). High FUS/TLS expression in
acute myeloid leukaemia samples. Brit. J. Hem. 108, 316-321
Misteli, T. (2000). Cell biology of transcription and pre-mRNA
splicing: nuclear architecture meets nuclear function. J Cell Sci 113
11, 1841-1849.
Pereira, D.S., Dorrell, C., Ito, C.Y., Gan, O.I., Murdoch,
B., Rao, V.N., Zou, J.P., Reddy, E.S., and Dick, J.E. (1998). Retroviral
transduction of TLS/ERG initiates a leukemogenic program in normal human
hematopoietic cells. Proc. Natl. Acad. Sci. USA 95, 8239-8244..
Perroti, D., Bonatti, S., Trotta, R., Martinez, R., Skorski,
T., Salomoni, P., Grassili, E., Lozzo, R.V., Cooper,D. R., and Calabretta B.
(1998). TLS/FUS a pro-oncogene involved in multiple chromosomal translocations,
is a novel regulator of BCR/ABL-mediated leukemogenesis. EMBO J. 17,
4442-4455.
Petkovich, M., Brand, N.J., Krust, A., and Chambon, P. (1987).
A human retinoic acid receptor which belongs to the family of nuclear
receptors. Nature 330, 444-450.
Philips, A. V., and Cooper, T. A. (2000). RNA processing and
human disease. Cell. Mol. Life. Sci 57, 235-249.
Rabbits, T.H., Foster, A., Larson, R., Nathan P. (1993).
Fusion of the dominant negative transcription regulator CHOP with a novel gene
FUS by translocation t(12;16) in malignat liposarcoma. Nature Gen. 4,
175-180.
Powers, C.A., Mathur, M., Raaka, B.M., Ron, D., and Samuels,
H.S. (1998). TLS (Translocated in LipoSarcoma) is a high affinity interactor
for steroid, thyroid hormone and retinoid receptors. Molecular Endocrinology
12, 4-18.
Stickeler, E., Kittrell, F., Medina, D., and Berget, S.M.
(1999). Stage-specific changes in SR splicing factors and alternative splicing
in mammary tumorigenesis. Oncogene 18, 3574-3582.
Umesono, K. (1998). Endocrine signaling system mediated by
nuclear receptor. Nippon Rinsho 56, 1693-1698.
Uranishi, H., Tetsuka, T., Yamashita, M., Asamitsu, K.,
Shimizu, M., Itoh, M., and Okamoto, T. (2001). Involvement of the
pro-oncoprotein TLS (translocated in liposarcoma) in nuclear factor-kappa B
p65-mediated transcription as a coactivator. J. Biol Chem 276,
13395-13401.
Valcarel, J., Gaur, R.K., Singh, R., and Green, M.R. (1996).
Interaction of U2AF RS region with pre-nRNA of branch point and promotion base
pairing with U2snRNA. Science 273, 1706-1709.
Yang, L., Embree, L.J., and Hickstein, D.D. (2000). TLS-ERG
leukemia fusion protein inhibits RNA splicing mediated by serine-arginine
proteins. Mol Cell Biol 20(10), 3345-54.
Yamamoto, K., Hamagushi, H., Nagata, K., Kobayashi, M.,
Tanimoto, F., and Taniwaki, M. (1997), Establishment of anovel human acute
myeloblastic leukemia cell line (YNH-1) with t(16;21), t(1;16) and 12q13
translocations. Leukemia 11, 599-608.
Zelenetz, A.D., Wilman, C.L., Chen, Z., Chen, S-J, Zelent, A.,
Macintyre, E., Veil, A., Cortes, J., Kantarjian, H. and Waxman, S. (1995)
Clinical and molecular characterization of a rare syndrome of acute
promyelocytic leukemia associated with translocation (11;17). Blood,
85, 1083-1094.
Zelent, A., Krust, A., Petkovich, M., Kastner, P. and Chambon,
P. (1989) Cloning of murine alpha and beta retinoic acid receptors and a novel
receptor gamma predominantly expressed in skin. Nature 339,
714-717.
Zinszner, H., Albalat, R., and Ron, D. (1994). A novel
effector domain from the RNA-binding protein TLS or EWS is required for
oncogenic transformation by CHOP. Genes and Dev 8, 2513-2526.
Zinszner, H., Immanuel, D., Yin, Y., Liang, F.X., and Ron, D.
(1997). A topogenic role for the oncogenic N-terminus of TLS: nucleolar
localization when transcription is inhibited. Oncogene 14, 451-461.
Zinszner, H., Sok, J., Immanuel, D., Yin, Y., and Ron, D.
(1997). TLS (FUS) binds RNA in vivo and engages in nucleo-cytoplasmic
shuttling. J. Cell. Sci. 110 , 1741-1750.
|
|



