2) Le motif Winged Helix de KIN17 est-il capable
de lier l'ADN
ou l'ARN ?
L'étude préliminaire bio-informatique de la
protéine KIN17, qui a été présentée dans
le premier chapitre, suggère la présence d'un motif
de liaison aux acides nucléiques de type
« doigt de zinc » entre les résidus 28
et 50 en amont du domaine K2. Les chercheurs du Laboratoire de
Génétique de la Radiosensibilité (LGR) du CEA de
Fontenay-aux-Roses ont confirmé l'existence de ce motif en
démontrant sa capacité à lier l'ADN, et notamment
l'ADN courbe, de manière dépendante aux ions Zn2+
(Mazin et al., 1994). Au cours de cette étude, les auteurs ont
également montré qu'il existe un second domaine de liaison
à l'ADN situé dans la région N-terminale de KIN17
entre les résidus 71 et 281. De manière
intéressante, cette région de KIN17 comporte le motif
Winged Helix du domaine K2 (65 à
157). Récemment, de nouvelles investigations
menées par les chercheurs du LGR ont mis en évidence la
capacité de KIN17 à lier l'ARN et notamment l'ARN riche en bases
guanine et uracile (Pinon-Lataillade et al., 2004). Dans l'optique
d'améliorer la connaissance des fonctions précises et des
mécanismes d'action de KIN17, il serait intéressant de
déterminer quelle est l'implication du motif Winged Helix
du domaine K2 dans la liaison à l'ADN et l'ARN. Aussi, d'un
point de vue structural et fonctionnel, ce motif a-t-il la capacité de
lier
l'ADN ou l'ARN ?
2.1) Approche structurale
2.1.1) Reconnaissance de l'ADN par les protéines
à motif Winged Helix
Les principales fonctions des domaines protéiques
à motif Winged Helix sont la reconnaissance et la
fixation de l'ADN. En 1999, près d'une dizaine de structures
de complexe Winged Helix-ADN résolues par
radiocristallographie ou par RMN étaient disponibles dans la
Protein Data Bank. A partir de ces structures, Gajiwala
et Burley ont proposé deux modes de reconnaissance de
l'ADN par les protéines à motif Winged Helix
(Gajiwala & Burley, 2000) :
· Le premier mode dit « classique » est
à ce jour le plus rencontré dans les complexes Winged
Helix-ADN. Il a été découvert pour la
première fois à partir de la structure cristallographique
de la protéine HNF-3 liée à un fragment d'ADN (Clark et
al., 1993). Dans ce modèle, l'hélice de reconnaissance H3 plonge
dans le sillon majeur de l'ADN et établit plusieurs contacts avec
les bases nucléotidiques (contacts spécifiques) et le
squelette phosphate (contacts non spécifiques). La boucle W1
située entre les brins S2 et
S3 participe également à la liaison à
l'ADN en établissant quelques contacts non spécifiques avec
le squelette phosphate et le ribose dans le petit sillon de l'ADN (Figure
5.3). L'hélice H3 constitue donc
l'élément majeur de ce mode de reconnaissance en assurant
la spécificité de l'interaction, alors que la boucle W1 a pour
principale fonction d'augmenter l'affinité de la liaison (Huffman &
Brennan, 2002). Dans certains cas, des résidus de l'hélice H2
ou de la région N-terminale de H1 interagissent également
avec l'ADN et contribuent à améliorer l'affinité de
l'interaction. La plupart des interactions Winged Helix-ADN dans
ce mode d'action impliquent des résidus à chaîne
latérale polaire et notamment chargée positivement. Par
conséquent, les domaines Winged Helix qui adoptent ce mode
de reconnaissance classique présentent une surface
d'interaction
H3-W1 largement positive.
· Le second mode dit « atypique » n'a
été identifié à ce jour qu'à une seule
reprise dans la structure cristalline de la protéine RFX1
liée à un fragment d'ADN (Gajiwala et al.,
2000). Il implique également les éléments H3
et W1 mais leur rôle est inversé. Ainsi, la
spécificité de l'interaction est ici assurée par la boucle
W1, riche en résidus basiques, et
qui établit plusieurs contacts avec les bases du
grand sillon de l'ADN (Figure 5.3). La
surface de l'hélice H3 apparaît comme neutre et
interagit principalement avec le squelette
phosphate du sillon mineur. De manière
intéressante, plusieurs domaines Winged Helix capables de
lier l'ADN comme par exemple ORC2 (Singleton et al., 2004), ou SmtB
(Cook et al., 1998), présentent une surface neutre au niveau de
l'hélice H3 et un amas de résidus basiques dans la boucle W1. Il
est donc fort probable que ces domaines utilisent le mode de reconnaissance
atypique pour lier l'ADN.
« mode classique »
« mode atypique »
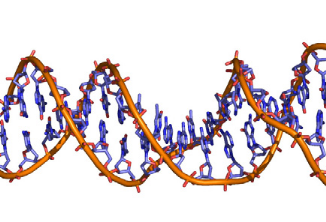
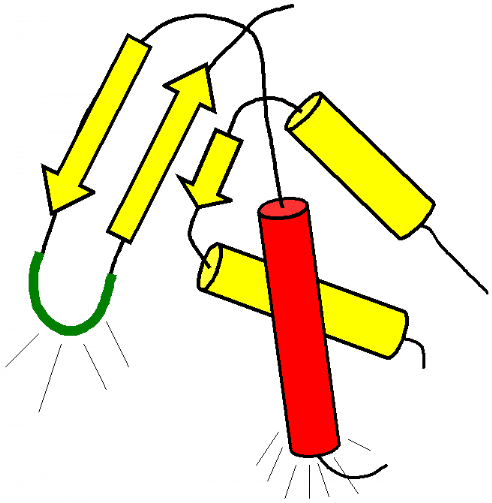
sillon mineur
sillon majeur
sillon mineur
sillon majeur
Figure 5.3 : Illustration des modes
de reconnaissance de l'ADN par les motifs Winged Helix.
L'hélice H3 est représentée en rouge et
la boucle W1 en vert.
2.1.2) Comparaison structurale du domaine K2 avec des
motifs Winged Helix
de liaison à l'ADN
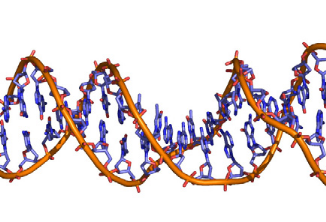
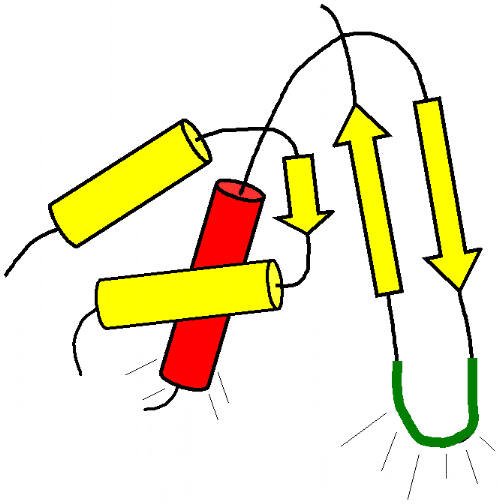
L'analyse de la structure du domaine K2 par l'algorithme
DALI fait apparaître une homologie de structure importante entre
K2 et les protéines ORC2 (Singleton et al., 2004),
PA-Fur (Pohl et al., 2003), TFIIEâ (Okuda et
al., 2000), BlaI (Safo et al., 2005), MecI (Garcia-Castellanos et al.,
2004), et DP2 (Zheng et al., 1999). Ces protéines sont des
régulateurs de la transcription des gènes impliqués
dans des mécanismes variés comme le contrôle de
l'incorporation du fer (PA-Fur), la résistance bactérienne aux
antibiotiques (BlaI et MecI), la régulation du cycle cellulaire
(ORC2 et DP2), et l'initiation de la transcription (TFIIEâ). Pour
toutes ces protéines, le domaine Winged Helix homologue
à K2 est un domaine
de liaison à l'ADN. Parmi ces domaines structuralement
proches, figure le motif Winged Helix
des protéines BlaI, MecI, et DP2 dont la structure en
complexe avec un fragment d'ADN a été
résolue par cristallographie des rayons X. Aussi, il est
à noter que la structure de la protéine
BlaI a été initialement
déterminée par RMN en solution par les chercheurs de
l'IBS Jean- Pierre Ebel de Grenoble (Van Melckebeke et al., 2003). Ces trois
motifs adoptent un mode de reconnaissance de l'ADN de type classique. Nous
avons superposé la structure du domaine K2 avec celle de ces 3
protéines en complexe afin d'évaluer les potentialités du
Winged Helix de
K2 à fixer l'ADN selon ce mode de reconnaissance.
La topologie du domaine Winged Helix de la
protéine MecI liée à un double brin d'ADN de 25
paires de bases est du type H1-H2-T-H3-S1-S2. Le programme DALI a
été utilisé pour identifier les régions
structuralement proches de K2 et MecI. Malgré une absence d'homologie
de séquence entre ces 2 protéines (< 10 %), la
déviation moyenne « rmsd » atteint 2.7 Å sur 66
carbones á. Comme le montre la Figure 5.4, les 2 structures
se superposent de manière quasi parfaite au niveau du feuillet â,
de la boucle W1, et de l'hélice
H2 (en arrière plan). L'hélice H1 du
domaine K2 comporte 2 tours de plus que son homologue de MecI et
son orientation est un peu moins ouverte par rapport au reste de la
structure. La plus grande différence structurale entre ces 2
protéines se situe au niveau de l'hélice de reconnaissance
H3 et de la région du coude entre H2 et H3. En effet, l'hélice
H3
du domaine K2 est d'une part, plus courte que son homologue de
MecI d'un peu moins de 2 tours et d'autre part, son orientation sensiblement
plus ouverte ne lui permet pas de pénétrer le sillon majeur de
la même manière que H3 de MecI. Il apparaît ainsi
en superposant ces 2 structures que la large boucle entre H2 et H3, et
notamment l'hélice 310 H2.5, sont dans une
position beaucoup plus favorable pour interagir avec les bases du
grand sillon de l'ADN.
H1
H1
W1
W1
H3 H3
H2 K2 de KIN17
H2 MecI
H2.5
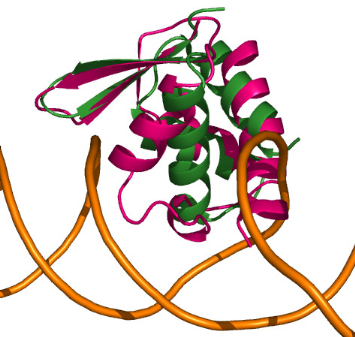
Figure 5.4 : Vue d'ensemble de la
superposition de structure du domaine K2 avec la protéine
MecI en complexe avec un double brin d'ADN de 25 paires de
bases (PDB : 1SAX). Le squelette phosphate de l'ADN est
représenté en orange.
Dans la structure cristallographique du complexe MecI-ADN, 7
résidus de l'hélice de
reconnaissance H3 sont impliqués dans la liaison avec
l'ADN et parmi ceux-ci, les 3 résidus K43, T47, et R51
établissent des contacts majeurs et spécifiques avec 4 bases
nucléotidiques. Ces trois résidus, mis en évidence dans la
Figure 5.5, appartiennent à la région N-terminale de
H3 qui ne se superpose pas avec l'hélice H3 de K2. A la
place de l'arginine R51 de MecI qui forme 2 liaisons hydrogène avec
une guanidine, on trouve dans la même orientation le tryptophane
W72 de K2, d'une part plus volumineux et hydrophobe, et d'autre part qui n'a
pas la capacité à former 2 liaisons hydrogène. La lysine
K43 de MecI qui forme une liaison hydrogène avec une thymine se
superpose quasi parfaitement à l'asparagine N59 de l'hélice
310 de K2. Cette asparagine pourrait également
former cette liaison hydrogène mais ne possède pas la
basicité d'une lysine. Il est d'ailleurs intéressant de noter que
le domaine K2 ne présente quasiment aucun résidu basique dans la
région contenant la large boucle et l'hélice H3. Aussi,
l'orientation des chaînes latérales négatives des
résidus E64 et D68 vers le double
brin d'ADN n'est pas très favorable à l'approche du
squelette phosphate chargé négativement.
H3 W72
D68 H3
T47
R51
K2 de KIN17
MecI
N59
E64
K43
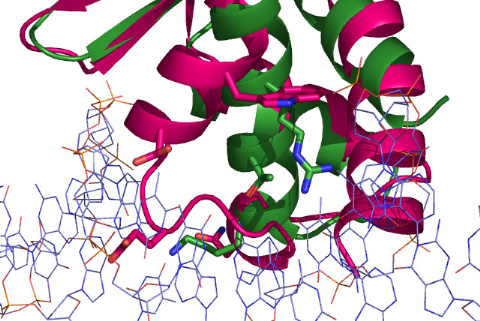
Figure 5.5 : Comparaison
structurale des hélices H3 de K2 et MecI. Les 2
protéines sont
superposées comme dans la Figure 5.4. Les
chaînes latérales des résidus mis en évidence sont
représentées par des sticks violets pour K2, et verts pour MecI.
Les hétéroatomes N, et O sont différenciés et
sont respectivement représentés par des sticks bleus, et
oranges. Le double brin d'ADN est représenté par des traits
bleus et oranges pour le squelette phosphate.
Nous avons également superposé la structure de K2
avec celle des protéines BlaI et
DP2 en complexe avec un fragment d'ADN. Ces 2 domaines
présentent une forte homologie
de structure avec le domaine K2 : la déviation moyenne
rmsd est de 2.5 Å sur 62 carbones á
pour DP2, et de 2.9 Å sur 67 carbones á
pour BlaI. De manière très analogue à MecI, la
structure de K2 est très proche de celle de BlaI et DP2 au
niveau de l'hélice H2, du feuillet â,
de la boucle W1, et aussi au niveau de l'hélice
H1 pour DP2 (Figure 5.6). La plus grande divergence structurale se situe
au niveau de l'hélice H3 dont la longueur dans K2 (10 résidus)
est plus courte que dans BlaI et DP2 (respectivement
15 et 21 acides aminés). De plus, l'orientation plus ouverte de
l'hélice H3 de K2 ne semble pas très favorable à une
interaction avec les bases du sillon majeur de l'ADN et comme
précédemment, celui-ci apparaît principalement
occupé par la large boucle entre H2 et H3 de K2. Ainsi, les
résidus majeurs de l'hélice H3 de BlaI et DP2 qui
établissent des contacts spécifiques avec les bases de l'ADN
appartiennent à une région qui ne se superpose pas avec
l'hélice H3 de K2, mais plutôt avec
l'hélice 310 H2.5.
W1 H3
H3
H1 H1
H2
W1
K2
H1
H2
H1
H2.5
T47
R51
BlaI
DP2
H3
H3
R121
R122
H2.5
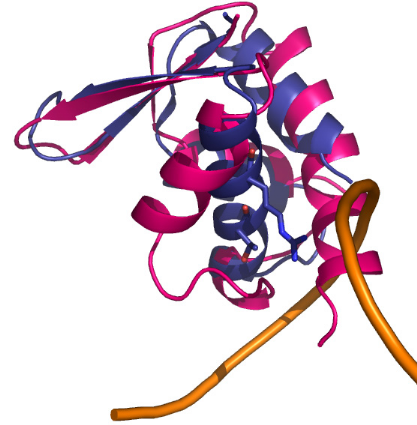
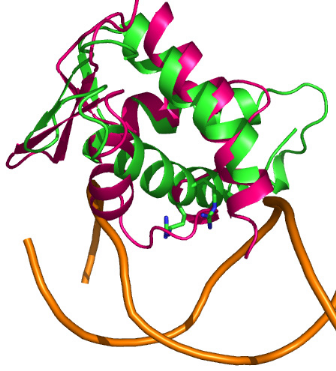
Figure 5.6 : Superposition de
structure du domaine K2 avec le motif Winged Helix des
protéines BlaI et DP2 en complexe avec l'ADN. Les
chaînes latérales des résidus de l'hélice
H3 qui interagissent avec les bases du grand sillon
sont représentées par des sticks. Le squelette phosphate de
l'ADN est coloré en orange. A) Superposition de K2 avec BlaI liée
à
un double brin de 25 paires de bases (PDB : 1XSD). Seul un
des deux brins est représenté. B) Superposition de K2 avec la
protéine DP2 du complexe hétérodimère
DP2-E2F4-ADN contenant un fragment de 15 paires de bases (PDB : 1CF7).
La superposition de structure du domaine K2 avec les
motifs Winged Helix des protéines MecI, BlaI, et DP2 fait
donc apparaître des divergences structurales significatives au niveau de
l'hélice de reconnaissance H3. Contrairement à ces 3 motifs, K2
présente une large boucle de 15 résidus entre H2 et H3 (Figure
5.7) dont la présence semble réduire la taille de l'hélice
H3 et ne lui permet pas d'adopter une orientation favorable pour
interagir avec le grand sillon de l'ADN selon le mode de reconnaissance
classique des Winged Helix. Pour de
nombreux motifs Winged Helix, la liaison à
l'ADN s'accompagne d'une déformation ou
d'une réorientation de l'hélice H3 qui
peut atteindre une quinzaine de degrés (Gajiwala & Burley,
2000). Dans le cas du domaine K2, la réorientation de l'hélice H3
dans une position plus favorable nécessiterait une modification majeure
de l'organisation de la boucle et de la position de l'hélice 310 H2.5.
Or, il apparaît, d'après les données RMN, que cette
hélice 310 est stabilisée par un réseau de liaisons
hydrogène et qu'elle incorpore le corps rigide de la
protéine tout autant que la boucle. Par conséquent, la
réorientation de l'hélice H3 pour adopter
le mode de reconnaissance classique nécessiterait une
modification structurale importante du domaine K2 dont l'envergure, à
notre connaissance, n'a jamais été observée dans un
motif Winged Helix. Il est toutefois important de noter que
ce n'est pas la présence même d'une hélice
additionnelle dans la boucle entre H2 et H3 qui compromet l'interaction
potentielle de
H3 avec le sillon majeur. La protéine Genesis, dont la
structure en complexe avec un fragment d'ADN a été résolue
par RMN (Jin et al., 1999), contient une hélice á additionnelle
H4 de 5 résidus entre H2 et H3 (Figure 5.7). Dans la structure, cette
hélice est positionnée à l'extérieur
du sillon majeur et l'hélice de reconnaissance
H3, dont l'orientation et la longueur sont comparables à H3 de
MecI et BlaI, pénètre et interagit avec le sillon majeur
de manière classique. Par conséquent, il s'agit bien de
la position de l'hélice 310 de K2, et non de sa
présence, qui distingue le plus le motif Winged Helix
du domaine K2 du Winged Helix de BlaI, MecI, DP2, et
Genesis. Il est d'ailleurs intéressant de noter que Genesis
n'apparaît pas comme un homologue structural de K2 d'après
l'analyse DALI, et ceci en dépit d'une certaine
homologie de topologie dans la région H2-H3.
BlaI : SANEIVVEIQKYKEVSDKTIRTLITRLYKKEII
DP2 : SYNEVADELVSEFTNSNNHLAADSAYDQKNIRRRVYDALNVLVAMN
MecI : SANNIIEEIQMQKDWSPKTIRTLITRLYKKGFI
Genesis : LSGICEFISNRFPYYREKFPAWQNSIRHNLSLNDC
K2 : HNNIVYNEYISHREHIHMNATQWETLTDFTKWLGREG
Figure 5.7 : Définition
des éléments de structure secondaire entre les hélices H2
et H3 du motif Winged Helix des protéines BlaI, DP2, MecI, Genesis, et
K2 (KIN17). Les résidus de l'hélice H2 sont surlignés en
vert, et de l'hélice H3 en orange. Les hélices additionnelles
de
K2 (H2.5) et Genesis (H4) apparaissent en rouge.
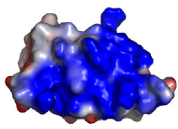
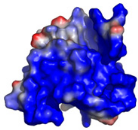
Le calcul des potentiels électrostatiques de surface
met également en évidence des différences majeures
entre le domaine K2 et les motifs Winged Helix structuralement
proches
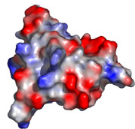
capables de lier l'ADN. Comme le montre la Figure 5.8,
le potentiel électrostatique de la
surface H3-W1 est largement positif chez les protéines
MecI, BlaI, et DP2. C'est également le
cas pour la protéine Genesis. Cette caractéristique
structurale est typique des protéines à motif
Winged Helix qui adoptent un mode de reconnaissance de
l'ADN de type classique.
A) B)
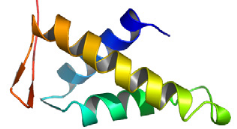
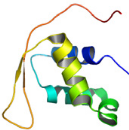
K2
KIN17 humaine
MecI


BlaI
DP2
Genesis
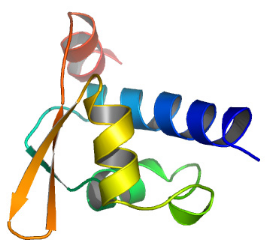
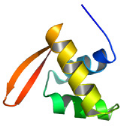
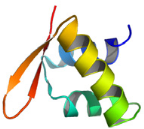
Figure 5.8 : Comparaison des
potentiels électrostatiques de surface des protéines
K2
(KIN17), MecI, BlaI, DP2 (du complexe E2F4-DP2) et
Genesis. A) Représentation de la structure de ces protéines.
L'hélice H1 apparaît dans les tons bleus, H2 dans les tons
verts,
H3 dans les tons jaunes, et la boucle W1 et les
brins S2 et S3 dans les tons oranges.
B) Représentation du potentiel électrostatique
de surface calculé avec le programme APBS (Baker et al., 2001) dans la
même orientation que A). Les régions chargées
négativement sont
en rouge et celles chargées positivement en bleu. Les
régions blanches correspondent à des régions peu
chargées ou hydrophobes.
Dans ces protéines, l'hélice H3 et la boucle
W1 contiennent plusieurs résidus lysine et
arginine dont les chaînes latérales
positives sont projetées vers l'extérieur du domaine et
établissent de nombreux contacts électrostatiques avec le
squelette phosphate de l'ADN chargé négativement. Dans le
cas du domaine K2, seuls 2 résidus lysine et arginine sont
dénombrables au niveau de l'hélice H3, ainsi qu'une lysine dans
la boucle W1. Il en résulte une surface électrostatique H3-W1 peu
polaire et très faiblement chargée positivement.
2.1.3) Comparaison avec la protéine
ADAR1
Comme nous l'avons évoqué
précédemment, la protéine qui présente la
plus forte homologie structurale avec K2 est le domaine Zá de ADAR1
humaine. La protéine ADAR1, impliquée dans la régulation
de l'ARN (Herbert et al., 1995), est capable de lier l'ADN de type Z via son
domaine Winged Helix Zá. L'ADN Z est une forme rare de l'ADN
dont la fonction est encore mal connue. Ce polymère de bases
nucléotidiques se structure en une double hélice gauche riche
en bases guanine et cytosine, et dont l'alternance de conformation anti
et syn a pour conséquence la formation d'un sillon
unique et profond. De manière intéressante, les bases
moléculaires de la reconnaissance de l'ADN Z par le domaine Zá
sont très différentes de celles de la reconnaissance de l'ADN B
(forme classique) par les motifs Winged Helix, mais elles impliquent
les mêmes éléments de structure secondaire (Schwartz et
al., 1999). Ainsi, Zá utilise principalement son
hélice de reconnaissance H3 et sa boucle W1 pour établir des
contacts avec le squelette phosphate et le cycle furanose des sucres.
Cependant, cette protéine n'établit aucun contact direct
avec les bases nucléotidiques. Par conséquent, la
reconnaissance de l'ADN Z par le domaine Zá de ADAR1 n'est pas
spécifique
de la séquence mais plutôt de la conformation Z.
Aussi, à l'image des motifs Winged Helix
qui lient l'ADN classique de type B, la majorité des
interactions avec l'ADN Z sont de nature électrostatique et impliquent
des résidus à chaîne latérale chargée
positivement. Il en résulte que les domaines Winged Helix qui
lient l'ADN Z comme ADAR1 présentent un potentiel de surface H3-W1
largement positif (Figure 5.9B), ce qui n'est pas le cas du domaine K2.
La superposition de structure de K2 avec Zá de ADAR1
fait apparaître une très forte homologie de structure entre ces 2
protéines : la déviation moyenne rmsd est de 2.0 Å sur 61
carbones á (Figure 5.9C). Les hélices H1 et H2 sont d'une
longueur quasiment identique et s'alignent presque parfaitement l'une sur
l'autre. Zá contient une boucle W1 un peu plus longue et 2 brins
â S2 et S3 un peu plus courts, mais le nombre de résidus dans la
région S2-
W1-S3 est sensiblement identique dans les 2 domaines (une
quinzaine de résidus). La plus
grande différence structurale concerne une
nouvelle fois la région de l'hélice H3 et de la boucle
entre H2 et H3. L'hélice H3 de K2 est plus courte d'environ un tour et
la plupart des résidus de Zá qui interagissent avec l'ADN
Z appartiennent à une région de H3 qui ne se superpose
pas sur H3 de K2 mais plutôt sur la boucle ou l'hélice 310.
K2
Zá
A)
H1
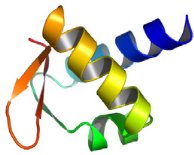
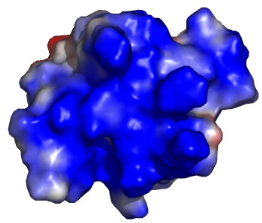
Zá de ADAR1
B)
W1
C)
Y177
K169
H3 H3
K170
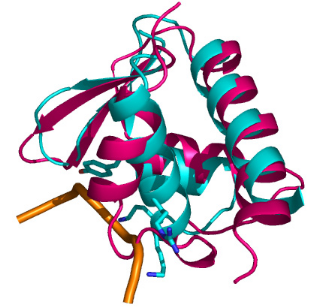
H2
R174
H2.5
Figure 5.9 : Comparaison
structurale du domaine K2 de KIN17 et de Zá de ADAR1
en
complexe avec un fragment d'ADN Z de 6 paires de bases. A)
Structure tridimensionnelle de
Zá. Le code couleur des éléments de
structure secondaire est relatif à la figure
précédente.
B) Surface électrostatique de Zá
calculée avec le programme APBS dans la même orientation que A).
C) Superposition de structure de K2 avec Zá. Les chaînes
latérales des résidus de l'hélice H3 de Zá qui
interagissent avec l'ADN de type Z sont représentées par des
sticks. Le squelette phosphate de l'ADN est coloré en orange. Seul un
brin est représenté.
La protéine ADAR1 humaine comporte un second domaine
Winged Helix (Zâ) situé
en aval de Zá à environ 80 acides aminés
dans la séquence primaire et dont la fonction est inconnue. La
récente résolution de structure de ce domaine par
radiocristallographie (Athanasiadis et al., 2005) montre une très forte
homologie structurale entre Zâ et Zá (rmsd
de 1.3 Å sur 62 résidus). Par conséquent, la
structure de Zâ est également très proche de K2
de KIN17 et les résultats de l'analyse DALI
font apparaître que ce domaine présente la seconde plus
forte homologie structurale avec K2 (rmsd de 2.7 Å sur 67
résidus). De manière intéressante, contrairement à
Zá, le Winged Helix de Zâ n'est pas capable de lier l'ADN
de type Z. En comparant ces 2 structures de manière fine,
Athanasiadis et al., ont mis en évidence des divergences
structurales au niveau de l'hélice de reconnaissance H3 qui
expliquent cette différence fonctionnelle. Ainsi, les 3
résidus conservés K169, R174, et Y177
de Zá qui établissent des contacts majeurs avec
l'ADN ne sont ni présents, ni conservés, chez
Zâ, et sont respectivement remplacés par A327,
A332, et I335 qui n'ont pas la capacité de former de liaison
hydrogène avec leur chaîne latérale. Aussi, les
résidus K169 et R174 de Zá appartiennent à une
région de l'hélice H3 qui ne se superpose pas avec H3 du domaine
K2.
De plus, le potentiel de surface H3-W1 de Zâ est
similaire à celui de K2 et apparaît peu chargé et
plutôt neutre contrairement à Zá qui présente
un potentiel de surface largement positif. Par conséquent, en se
basant sur les relations structure-activité des motifs
Winged Helix de ADAR1, il apparaît que le domaine K2 de KIN17
n'a pas la capacité structurale pour lier l'ADN de type Z selon le mode
de reconnaissance de Zá, et cela en dépit d'une très forte
homologie structurale. Aussi, il n'existe à ce jour aucune donnée
fonctionnelle qui suggère
que la protéine KIN17 est capable de lier l'ADN de type
Z.
2.1.4) Reconnaissance de l'ARN par les protéines
à motif Winged Helix
La publication récente de nouvelles structures de motif
Winged Helix a élargi l'horizon des fonctions adoptées
par ce type de domaine. Ainsi, il a été montré en 2002 par
Selmer et
al., que le domaine de liaison à l'ARN
du facteur d'élongation SelB est un motif Winged Helix
de topologie canonique (Selmer & Su, 2002). Trois
années plus tard, l'institut de Chimie des Substances Naturelles
de Gif-sur-Yvette a caractérisé pour la première
fois les bases moléculaires de la reconnaissance de l'ARN par un motif
Winged Helix en cristallisant
le domaine de liaison à l'ARN de la protéine SelB
avec un fragment de 16 bases (Yoshizawa
et al., 2005). Dans cette structure, plusieurs résidus
des hélices H2, H3, et de la boucle W1 forment une surface
conservée qui interagit de manière quasi exclusive avec les
groupements phosphate de l'ARN chargés négativement. Ce mode de
reconnaissance des acides nucléiques tout à fait unique
implique par conséquent une surface H2-H3-W1 fortement
chargée positivement, ce qui n'est absolument pas le cas du domaine K2
de la protéine KIN17 dont la structure n'apparaît pas homologue
à celle de SelB d'après l'algorithme DALI.
A ce jour, la structure du complexe SelB-ARN est la seule
structure connue d'un motif Winged Helix lié à
l'ARN. Cependant, il existe à notre connaissance au moins 2
autres domaines capables de lier l'ARN qui adoptent un repliement de type
Winged Helix. Il s'agit
du domaine N-terminal LM de la phosphoprotéine La
(Dong et al., 2004) et du domaine
U2AF du facteur d'épissage U2 (Kielkopf et al., 2004). La
topologie du domaine LM, de type
H1-H1'-H1''-B1-H2-H2'-H3-B2-B3, est très
différente de celle des motifs Winged Helix
canoniques, et donc de celle de K2. H1', H1'', et H2' sont des
hélices additionnelles insérées
entre les hélices du tonnelet orthogonal ancestral
formé par H1, H2, et H3. Des expériences de mutagenèse
montrent que des résidus des hélices H1, H1', et H1'' et de la
boucle entre H2 et H2' forment une surface conservée, aromatique, et
hydrophobe susceptible d'interagir avec l'ARN. Il apparaît donc qu'il
existe au moins 2 modes de reconnaissance de l'ARN par des motifs Winged
Helix radicalement différents. La découverte de ces
nouveaux modes de reconnaissance montre la grande versatilité avec
laquelle ce type de repliement est capable de reconnaître les acides
nucléiques. Toutefois, la capacité des domaines Winged
Helix à lier l'ARN reste à ce jour une fonction
atypique de ce genre de motif. Il est donc difficile d'émettre
une hypothèse quant à la capacité structurale du domaine
K2 à lier l'ARN.
2.2) Approche fonctionnelle
Parallèlement à l'étude structurale par RMN,
des études fonctionnelles d'interaction et
de recherche de partenaires biologiques ont été
menées pour les domaines K2 et K3 de KIN17 humaine par Albane le
Maire du Laboratoire de Structure des Protéines. Comme je l'ai
évoqué dans le premier chapitre, le domaine K3 correspond aux 160
premiers résidus de la protéine KIN17 humaine. Il est donc
principalement constitué du domaine K2 (51-160) et du motif
prédit structuré en « doigt de zinc » (28-50).
Deux techniques ont été utilisées pour tester
l'interaction in vitro de K2 et K3 avec l'ADN et l'ARN : il
s'agit de l'hybridation Southwestern et Northwestern.
2.2.1) Interaction avec l'ADN par hybridation
Southwestern
La technique de Southwestern consiste à
incuber des protéines avec un fragment d'ADN radiomarqué
sur une membrane de nitrocellulose afin de révéler les
protéines qui fixent le ligand marqué. Les protéines
d'intérêt sont dans un premier temps séparées sur
un
gel d'acrylamide de type SDS-PAGE, puis
transférées sur une membrane de nitrocellulose. La coloration au
rouge Ponceau de la membrane permet de vérifier la présence et
les quantités des protéines déposées sur le gel. La
membrane de nitrocellulose est ensuite incubée avec une solution
d'hybridation contenant 50 mM de NaCl, 10 mM de Tris-HCl (pH 7.4),
1 mM d'EDTA, du tampon Denhardt 1X, et le fragment d'ADN cible marqué
radioactivement au
phosphore 32P. Après plusieurs lavages de
la membrane, les protéines qui lient la cible
radioactive sont révélées sur plaques
radiosensibles phosphorescentes (phosphoimager).
Pour tester l'interaction de K2 et K3 avec l'ADN, la
cible utilisée est une sonde
d'ADN double brin de 500 nucléotides provenant d'une
origine de réplication reconnue par la protéine KIN17 humaine. La
Figure 5.10 présente les résultats obtenus par Southwestern
de l'interaction de cette sonde avec K2, K3, et KIN17 humaine.
La révélation de la membrane de
nitrocellulose sur plaques radiosensibles fait apparaître un signal
au niveau des bandes protéiques de KIN17 entière et du
domaine K3. Cette expérience confirme donc dans un premier temps
la capacité de la protéine KIN17 humaine à lier
l'ADN cible de 500 paires de bases in vitro. La forte
intensité de la bande correspondant à K3 met en
évidence le rôle important de ce domaine dans la liaison à
cette sonde d'ADN. En revanche, aucun signal de radioactivité n'est
détecté au niveau de la bande protéique relative au
domaine K2. Par conséquent, il semble d'une part, que la région
1-50 de KIN17 humaine contenant le « doigt de zinc » joue un
rôle majeur dans cette liaison à l'ADN
et d'autre part, que le motif Winged Helix de KIN17 n'a
pas la capacité, ou n'est pas suffisant,
pour lier ce fragment d'ADN de manière autonome.

(kDa)
75
50
37
25
20
15
K2 K3 Kin17

·
·

·
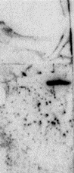

32P-ADN
K2 K3 Kin17
« rouge ponceau » « phosphoimager »
Figure 5.10 : Test d'interaction
de K2, K3, et KIN17 avec une sonde d'ADN de 500 paires de bases par
Southwestern. Sur la membrane colorée au rouge ponceau,
les bandes correspondant à K2, K3, et KIN17 sont indiquées par
des points. Les bandes révélées par le phosphoimager sont
mises en évidence par des flèches.
2.2.2) Interaction avec l'ARN par hybridation
Northwestern
Le principe de l'hybridation Northwestern
est tout à fait similaire à celui de
l'hybridation Southwestern et permet de tester l'interaction d'une
protéine avec un fragment d'ARN radiomarqué au phosphore
32P. Les trois protéines KIN17 ont été
séparées sur gel
SDS-PAGE, transférées sur membrane de
nitrocellulose, puis incubées avec une sonde
d'ARN radiomarquée de 1200 nucléotides.
Les résultats de cette expérience
d'hybridation Northwestern sont tout à fait
comparables à ceux obtenus précédemment. La
révélation de la membrane de nitrocellulose
sur plaques photosensibles fait apparaître les
bandes protéiques correspondant à KIN17 entière et
au domaine K3, mais pas à celle de K2 (Figure 5.11). Par
conséquent, ce test d'interaction in vitro montre une
implication de la région 1-50 contenant le motif « doigt de zinc
» dans la liaison à l'ARN. Cette expérience suggère
également que le motif Winged Helix
de KIN17 humaine n'est pas suffisant pour lier l'ARN de
manière autonome dans ces
conditions d'analyse.
Kin K3 K2
Kin K3 K2
·
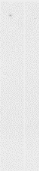

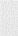
32P-ARN

·

·
« rouge ponceau »
« phosphoimager »
Figure 5.11 : Test d'interaction
de K2, K3, et KIN17 avec une sonde d'ARN de 1200
nucléotides par Northwestern. Sur la membrane
colorée au rouge ponceau, les bandes correspondant à K2, K3,
et KIN17 (Kin) sont indiquées par des points. Les bandes
révélées par le phosphoimager sont mises en
évidence par des flèches.
| 


