III)-La cristallisation des protéines
Pourquoi cristalliser une protéine
?
Pour résoudre par les méthodes biophysiques des
structures 3D à résolution atomique il faut utiliser
un rayonnement dont la longueur d'onde est proche de la
distance inter-atomique, c'est-à-dire de l'ordre de l'?, donc les
rayons X.
Si on voulait résoudre la structure 3D d'une
protéine isolée en solution en l'exposant aux rayons
X : les ondes diffusées seraient
de faible intensité et l'échantillon se dégraderait
sous l'effet du rayonnement X ; sous l'effet du mouvement Brownien la
protéine n'aurait ni position, ni orientation définies.
D'où l'intérêt d'obtenir un cristal
protéique : manipulable et contenant une quantité énorme
de molécules (1015) ayant la même orientation. Chacune
contribue au signal (interférences constructives entre toutes les
molécules) et la dégradation due aux rayons X est partagée
entre elles.
A-Aspects généraux
1-Le cristal : arrangement périodique
ordonné dans l'espace 3D
Le cristal, dont la taille est de quelques dizaines de microns (u
), est un état solide ordonné : les protéines qui le
composent sont empilées d'une façon périodique et
régulière dans les trois directions
de l'espace. La maille cristalline est
répétée par translation à travers tout le cristal
(Figure 12). La cohésion entre les molécules
protéiques voisines dans le cristal est assurée par des
interactions non covalentes : interactions hydrophobes, liaisons
hydrogènes, de Van der Waals,... La protéine dans le cristal a
la même structure qu'en solution, car étant volumineuses
et globulaires, les protéines, ne peuvent pas s'empiler
très étroitement. Les espaces ménagés par
l'empilement des protéines sont comblés par le solvant
(généralement l'eau) qui représente en moyenne 50 %
du volume du cristal
[cette caractéristique est à l'origine de la
fragilité des cristaux] (Mathews, 1985). Les protéines sont donc
dans un état comparable à celui de la solution.
2-Principe de la cristallogénèse : de la
solution au cristal
La solubilité d'une protéine est fonction de
nombreux paramètres tels que : sa concentration, le pH, la
température, la force ionique, l'effet d'additifs....
Lorsque le cristal protéique est obtenu, se pose
la question de l'évaluation de sa qualité ; l'aspect
visuel peut apporter une première indication mais le véritable
test est l'analyse aux rayons X (Mullin, 1993). parmi les défauts
rencontrés dans les cristaux protéiques figure la
mosaïcité (Figure
13) : structure mosaïque du cristal
qui consiste en une juxtaposition de blocs monocristallins
(Helliwell, 1993 ; Chayen et al, 1996 ; Chernov,
1999).
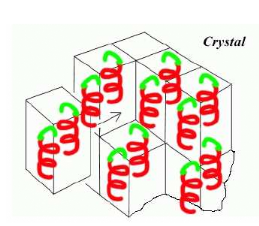
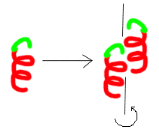
Figure 12 : La cristallisation /
Transition de phases (passage d'un état désordonné
liquide à un état ordonné
solide).
La protéine quitte la solution et passe d'un
état soluble à un état de solide ordonné,
le cristal. Le motif moléculaire
répété par translation le long du réseau cristallin
s'appelle maille cristalline. Elle correspond à une molécule
protéique ou à une sous unité protéique.
L'arrangement périodique des protéines est à
l'origine du phénomène de diffraction observé lorsque le
cristal est placé dans

un faisceau de rayons X. La résolution des
données et la précision finale de la structure
dépendent, directement, de la régularité de
l'empilement cristallin (Sauter et Giegé, 2001).
Figure 13 : Mosaïcité d'un
cristal
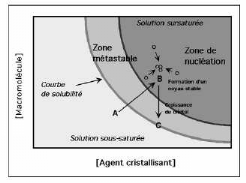
Diagramme de phases
Remplacement des interactions Solvant-Protéines
par des interactions Protéines-Protéines
La cristallisation est une transition de phase : la
protéine cristallisée passe d'un état de soluté
[protéine soluble] à une phase
solide [le cristal]. La cristallisation est le
résultat d'un compromis entre les facteurs thermodynamiques
(solubilité) et cinétiques
(nucléation et croissance cristalline).
Le comportement d'une protéine en fonction des variations
de son environnement peut être décrit sous forme d'un
diagramme de phases (Figure 14) (Saridakis et al, 1994).
Sur le diagramme est reportée la
courbe de solubilité qui définit la limite
entre la phase soluble et la phase solide [cristal,
précipité microcristallin ou amorphe]. Pour qu'une
protéine cristallise, elle doit dépasser cette courbe et entrer
dans une zone hors équilibre thermodynamique, elle
se trouve alors dans un état de sursaturation qui lui
permet d'initier la cristallisation : la nucléation. Il existe de ce
fait une courbe de supersaturation, qui sépare
deux zones sursaturées du diagramme : la zone de nucléation
où la sursaturation élevée conduit à la
nucléation du cristal et la zone métastable où
la sursaturation plus faible est juste suffisante pour
que le cristal existant croisse (Sauter et Giegé,
2001).
Le passage de la zone insaturée à la zone
sursaturée est réalisé en faisant varier un ou plusieurs
paramètres physico-chimiques : la concentration protéique et
de l'agent cristallisant [sel, polymères, alcool], le pH et plus
rarement la température (McPherson, 1982 ; McPherson, 1998).
3-Etapes de la cristallisation
3.1-La nucléation : La
nucléation est le point de départ de la cristallisation,
elle correspond à la constitution d'amas moléculaires qui,
lorsqu'ils dépassent une taille critique, donnent naissance au
cristal.
3.2-La croissance cristalline : La
croissance cristalline est le processus physique qui va suivre la
nucléation et permettre l'augmentation de la taille des cristaux.
a)-Cinétique : elle
dépend directement du degré de sursaturation. Pour les
protéines : une couche de protéines s'ajoute toutes les 3
secondes, soit une croissance d'environ 30 ?.s-1. Le
transport des molécules en solution vers le cristal dépend
de la diffusion, la convection et de l'agitation de la solution
(McPherson et al, 1999). Mais aussi des concentrations de la
protéine et de l'agent précipitant,
du pH, de la force ionique, de la température et de la
pureté des agents chimiques et de la protéine
utilisée (Stelter, 2003).
Agents précipitants (cristallisants) usuels
:
Sels : sulfate d'ammonium, phosphate de
potassium et chlorure de sodium
Polymères :
polyéthylène glycérol
Paramètres de la cristallogenèse
:
Concentration protéique
Contaminants non macromoléculaires
Température, pH et Force ionique et nature du
tampon Densité, viscosité, pression, temps d'évaporatoire
se Vibrations, gravité et mouvements de diffusion,
Convection
Nature de l'agent cristallisant
Solvants organiques : non volatil
(méthyl-2,4-pentadiol), volatils (éthanol)
Figure 14 : Diagramme de phase
bidimensionnel d'une macromolécule en fonction de sa propre
concentration et de celle d'un
agent cristallisant. Lors de la
cristallisation, la solubilité de la macromolécule diminue
jusqu'à atteindre l'état de supersaturation favorable
à l'obtention de cristaux, sans toutefois tomber dans la zone de
précipitation.
Au niveau de la courbe de solubilité,
le cristal et la solution sont en équilibre thermodynamique.
Au-delà de cette courbe, la solution est dite
sursaturée. Plus la molécule s'enfonce dans
cette zone A---B plus la sursaturation est élevée. En B, il y'a
formation d'un noyau stable à partir du quel un cristal va
croître. Dans la zone métastable il y'a la croissance
cristalline.
La courbe de supersolubilité
sépare la zone métastable de la
zone de nucléation.
b)-Equilibre et arrêt de la croissance
: l'arrêt de la croissance (mûrissement d'Oswald)
intervient
lorsque le système atteint le point
d'équilibre où la concentration de la molécule est
égale à la solubilité : le nombre de molécules
qui se détachent du cristal est égal au nombre de
molécules qui s'y lient. La présence d'impuretés peut
induire l'arrêt irréversible de la croissance cristalline
avant d'atteindre le point d'équilibre (Sauter et Giegé, 2001 ;
Stelter, 2003).
B-Méthodes de cristallisation :
Plusieurs méthodes sont développées
pour cristalliser les protéines. Elles ont toutes
pour principe « d'amener les protéines sans les
dénaturer en conditions de sursaturation propices à la
nucléation, puis à la croissance du cristal». La
méthode la plus utilisée est la diffusion de vapeur en goutte
suspendue, sa simplicité et son adaptation aux volumes
réduits de solutions expliquent sa popularité (McPherson,
1998). Dans une enceinte close, l'équilibre s'établit entre
la goutte (contenant
la protéine, le tampon et l'agent cristallisant)
et le réservoir (contenant le tampon et
l'agent cristallisant à des concentrations plus
élevées) par diffusion des espèces volatiles de la
goutte (principalement l'eau) jusqu'à ce que la tension de
vapeur soit la même dans la goutte et dans le
réservoir.
Ce processus d'équilibration concentre l'agent
cristallisant et la protéine dans la goutte, ce qui diminue la
solubilité de la protéine et si les conditions sont favorables
l'amène à cristalliser (Sauter et Giegé, 2001). La
cinétique de la diffusion dépend des différences de
concentrations dans la goutte et dans le réservoir mais également
de la géométrie du système, du volume de la goutte et de
sa distance
au réservoir. Dans la Figure 15, est
représentée la méthode de la goutte assise utilisée
dans les tests au
robot.
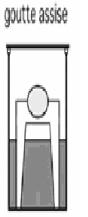

Figure 15 : Technique de la goutte assise
B- Matériel et méthodes
1-Clonage, surexpression et
purification
A-UMPKeco, double mutant D159N D93A [en position
159 : Asp(D) remplacé par Asn (N) / en 93 :
Asp(D) remplacé par Ala(A)] :
Le variant UMPK D159N D93A est construit par la méthode de
double PCR. La souche transformée est cultivée à 37°C
jusqu'à l'obtention d'une absorbance de 1.5 à 600 nm. Puis la
production de protéine est induite par l'ajout de 1 mM
d'isopropyl-thiol--D-galactosidase (IPTG) pendant 3 h à 37°C. Les
bactéries sont ensuite centrifugées. Le culot bactérien
est soit traité immédiatement, soit conservé à
-20°C pour une utilisation ultérieure (Serina et al,
1996).
La purification s'est faite en deux étapes :
-la première met en jeu une chromatographie
d'affinité sur résine de l'acide Ni-Nitrile
Triacétique agarose ;
-la deuxième une chromatographie d'exclusion sur
Séphacryl S-300.
La concentration protéique est
évaluée par la méthode de Bradford. L'enzyme
purifiée est alors conservée dans du Tris-HCl pH 8,5 50
mM et du NaCl 100 mM à 20°C plusieurs semaines. La
pureté du variant UMPKeco D93A est vérifiée par
électrophorèse sur gel de polyacrylamide (12.5 %)
en présence de SDS (SDS-PAGE). A l'issue de cette
étape, l'enzyme doit être très pure, au moins à
97
%, sinon on risque de n'obtenir aucun cristal du fait
des impuretés qui pourraient compromettre la cristallisation. Il
est aussi nécessaire de disposer de quantités suffisantes de
protéines.
B-CKOm sauvage et mutants D169E et D192L :
L'amplification est effectuée par la méthode PCR en
utilisant un plasmide d'E. coli. Le vecteur contenant le gène
d'intérêt : pINA1267,
est ensuite cloné et surexprimé dans la
levure Yarrowia lipolitica. La purification de la
protéine obtenue (CKOm) est effectuée par deux chromatographies
sur colonne échangeuse d'anions. La pureté
de la CKOm est vérifiée par la SDS-PAGE
(Kopeèny et al, 2004).
2-Cristallisation
Avant de procéder à la cristallisation proprement
dite, on a procède à chaque fois à la
vérification
de la concentration protéique en mesurant
l'absorbance à 280 nm (Encadré 1), la
concentration minimale requise pour la cristallogenèse est
d'environ 5 mg/ml. Si la concentration protéique est
inférieure à cette valeur on procède dès lors
à la concentration de la protéine (Encadré 2).
Une fois
que la solution protéique est très pure et
suffisamment concentrée, on peut passer à la phase de
cristallisation qui comprend deux cribles (Figure 16).
A-UMPKeco, double mutant D159N D93A :
Premier crible : tests au robot / essais
préliminaires / goutte assise : La recherche des
conditions prometteuses de cristallisation s'est effectuée en utilisant
les kits Crystal Screen I et II (Annexes 1 &
2) (Hampton Research).
Encadré 1 : Vérification de la
concentration protéique par absorption dans l'UV
La mesure de la concentration protéique s'est
effectuée en utilisant un spectrophotomètre UV/VISIBLE
(BECKMAN DU 640B).
Un rayon lumineux traverse la solution absorbante contenant 2
u l de la solution protéique et 498 u l du
tampon
Tris-HCL pH 8,5 50 mM pour l'UMPK et pH 7,0 100 mM pour la
CKOm, après lecture des absorbances à 280
nm et en appliquant la loi de BEER-LAMBERT on détermine la
concentration en mg/ml de la protéine.
Loi de BEER-LAMBERT : A= log (I / Io) = å.
l . C
A : absorbance, I : intensité de la
lumière incidente, Io : intensité de la
lumière transmise, l est l'épaisseur (en cm)
de la cuve appelé aussi chemin optique, C est la
concentration en mg/ml et å : coefficient d'extinction molaire
caractéristique de chaque protéine à une longueur d'onde
donnée (å est lié à la teneur de la protéine
en Trp, Tyr
et en Phe)
Pour l'UMPKeco : 1/å =2,33 & pour la CKO 1/å =
0,777
Encadré 2 : Concentration de la
protéine
On a utilisé des centricons 10 à contenance
maximale de 2 ml, leur porosité permet à certaines
molécules de passer : sels, petites molécules et retient les
protéines dont la MM est supérieure à 10 kDa. La solution
protéique
est déposée dans la partie supérieure du
tube et repose sur la membrane, le tube est alors centrifugé pour forcer
le liquide et les petites molécules à traverser cette
dernière. Les protéines trop grosses ne peuvent pas
passer et
restent dans la partie supérieure, on peut ensuite les
récupérer pour mesurer la concentration.
Détermination des conditions
prometteuses de la cristallisation
Tests préliminaires
Robot de cristallisation
96 conditions différentes
Robot de cristallisation


![]()
![]()
![]()
![]()




![]()
![]()
![]()
![]()


96 conditions différentes
Tests manuels approfondis dans
les conditions prometteuses
Boite Linbro
Boite Linbro
24 puits
24 puits
Figure 16 : Photos du robot de cristallisation et de la
boite Linbro
Les kits contiennent 96 conditions
différentes : ensemble de précipitants, additifs
et
tampons...le plus fréquemment testés avec
succès dans la PDB. Quoique empiriques, ces kits permettent
d'échantillonner systématiquement un grand nombre de
conditions. Le robot de cristallisation permet d'obtenir des cristaux
rapidement, en utilisant de faibles quantités de la protéine (0,5
u l par test = économie de protéine par rapport aux essais
manuels : 2 u l) ce qui rend possible la réalisation de beaucoup de
tests. Dans les tests au robot, on a testé l'UMPK D159N D93A
seule (enzyme brute), avec l'UMP
(substrat accepteur de phosphate) et avec le GTP
(activateur allostérique). Chaque goutte contient 1 u l de
solution (0,5 u l de solution protéique -enzyme seule ou avec ligand- et
0,5u l du puit).Au total, 288 essais sont
réalisés avec le robot ; la boite est
incubée dans
une enceinte thermostatée à 20 °C.
L'observation des résultats se fait à la
loupe biloculaire le lendemain, après 3 jours puis chaque
semaine. A l'issue de ces essais on détermine et on
retient les conditions prometteuses de cristallisation.
Deuxième crible : tests manuels approfondis
/ méthode de diffusion de vapeur / technique de
la
goutte suspendue (Figures 17) : On
utilise la technique de la goutte suspendue (hanging drop) sur des boites
Linbro. Les essais ont été réalisés de la
façon suivante : un volume A de la solution
SE
(solution de l'enzyme) [UMPK D159N
D93A, ligand GTP et additif ( Glucoside, Glycérol)] est
mélangé avec un volume B de solution
SR (solution du réservoir) [PEG
6000, le NaCl, l'additif et le tampon], A + B = 4ul. Ce
mélange constituant la goutte est déposé sur une
lamelle en verre [ronde, 22 mm de diamètre, siliconé avec
l'aquasil (Pierce) 2,5 % v/v] placée avec précaution au dessus du
puits contenant 1 ml de la SR.
L'étanchéité étant indispensable, on graisse
préalablement le pourtour du puits avec une graisse à vide
à l'aide d'une seringue. Les boîtes ont ensuite été
placées dans l'enceinte thermostatée à une
température de 20°C. L'observation des résultats se
fait se fait comme pour le
robot.
Les travaux menés dans l'UMR de Chimie
Biologique par Briozzo et al, ont montré que l'UMPKeco
cristallise mieux au contact de la graisse, pour chaque paramètre requis
on a effectué des essais en présence et en absence de la graisse.
Le pH est maintenu constant grâce au tampon Tris-HCl
pH 8,5, 50 mM. 266 essais ont étés
effectués manuellement. Au total 554 essais
sont effectués avec
l'UMPK D159E D93A.
B-CKOm sauvage et mutants D169E et
D192L
On n'a pas effectué d'essais au robot pour cette enzyme
car :
(1) nous avons que des petites
quantités de la protéine ;
(2) les mutants de la CKO
cristallisent en général dans les conditions
déterminées en 2004 par Briozzo et al
(précipitant : PEG 1500 à 30% - condition 43 du Crystal
Screen I-, pas de sel, tampon : Tris-HCl pH 7,0 50 mM). Pour les essais
manuels approfondis nous avons employé la méthode de
diffusion de vapeur les boites Linbro. Nous avons varié
dans le puits la concentration du PEG 1500 et
dans la goutte la concentration protéique. Des tests sont
effectués avec la CKOm sauvage brute ( pour
infiltration), avec le mutant D169E-IP et avec le mutant D492L.
Au total 96 essais ont étés effectués avec la CKOm : 42
avec la CKOm native, 30 avec le mutant D169E et 6 avec le mutant L492A. Pour
la CKOm, les concentrations de départ étant
assez importantes (> à 13 mg/ml), on ne l'a pas
concentrée
3- Etapes ultérieures
3.1-Montage des cristaux (Figure : 17 b))
: Les cristaux les plus beaux ont été
montés délicatement dans des boucles en nylon de 0,01 mm de
diamètre pour l'UMPKeco et 0,1 mm pour la CKOm, et serviront à la
diffraction des rayons X. Cette étape est réalisée sous
la loupe biloculaire. Avant de monter les cristaux, une solution correspondant
à la solution de la goutte ou a été
préparée. Cette solution est ensuite déposée
sur la goutte, le cristal a été pêché
soigneusement. La boucle est ensuite montée sur une tête
goniométrique orientable, ce qui permet de centrer et d'orienter
correctement le cristal dans le faisceau de rayons X. Avant l'enregistrement
des données de diffraction,
les cristaux ont été congelés sous flux
d'azote gazeux à 100 K. On augmente ainsi leur durée de vie et
on réduit leur dégradation [modification
chimique et formation de radicaux libres] par les rayons X (faisceaux
très puissants) pendant l'enregistrement. Comme un cristal
contient près de 50 % de solvant, qui est principalement l'eau, la
congélation sans cryoprotectant cristalliserait cette eau sous forme
de microcristaux qui détériorent le cristal et donnent des
anneaux parasites sur les clichés de diffraction. Il est
nécessaire de trouver un cryoprotectant adapté qui
transforme l'eau en solide amorphe (empêchant la formation de cristaux
de glace), sans détériorer le cristal.
On a pêché le cristal avec une boucle de
taille adaptée et on l'a transféré dans la
solution cryoprotectante (pendant environ 1 min). Dans notre cas, le
cryoprotectant utilisé été le glycérol à
20
%. Ces étapes ont lieu au laboratoire d'enzymologie et
biochimie structurales de Gif sur Yvette (91).
3.2-Collecte des données de diffraction
(Figure 17 c)) : Cette étape consiste à soumettre
les cristaux à un faisceau monochromatique de rayons X issu
d'un rayonnement synchrotron intense permettant d'enregistrer à haute
résolution. On choisit la longueur d'onde appropriée et on fait
tourner
le cristal autour d'un axe perpendiculaire au faisceau
incident de rayons X (1° dans notre cas). Les intensités
diffractées son enregistrées par un détecteur placé
lui aussi perpendiculairement au faisceau incident.
3.3-Détermination des paramètres
de maille des cristaux : Les données de diffraction sont
collectées sur la ligne ID14-1 à l'ESRF (European Synchrotron
Radiation Facility, Grenoble) équipée d'un détecteur CCD
(charged coupled device). L'enregistrement étant terminé,
il est nécessaire de déterminer pour chaque
réflexion : son indice hkl, son intensité et l'incertitude
sur sa mesure. Le programme DENZO fait l'indexation sur une image
puis sur toutes les images ; le programme SCALEPACK de HKL est
utilisé pour mettre à l'échelle les intensités de
toutes les réflexions dans un
fichier unique.
![]()
.
Figure 17 a) : Méthode de cristallisation en
goutte suspendue : La méthode de diffusion de vapeur est la
méthode le plus couramment utilisée dans les
cristallisations manuelles. Elle est basée sur la saturation atteinte
par évaporation des solvants volatils dans une enceinte
fermée sans contact entre les solutions liquides.
L'équilibre
![]()
s'établit progressivement par diffusion de la vapeur
d'eau, l'eau passe de la goutte vers le puits et ceci jusqu'à ce qu'on
atteigne les conditions de succès (obtention des cristaux) ou les
conditions de précipitation / agrégation.
![]()
Figure 17 b) : Montage des cristaux sur des
boucles en nylon
Figure 17 c) : Collecte des données de
diffraction
C- Résultats et discussion
A-Mutant D93A non régulé par le GTP de
l'UMPKeco
1-Concentration de l'échantillon
purifié : Comme le montre la figure
18, la purification
réalisée par les collaborateurs de l'Institut
Pasteur est très concluante : le matériel protéique
prêt à être utilisé pour la cristallisation est
très pur.
Les pertes en protéines lors de la concentration,
evaluées par la mesure de l'A280 nm du filtrat, sont
négligeables. Le procédé utilisant les centricons 10 est
adapté à l'UMPKeco.
2-Cristallisation :
---Tests au robot : Les essais de Crystal
Screen I & II ont donné les résultats suivants:
? Pour l'enzyme seule (7,41 mg/ml dans la solution
départ et de 3,7 mg/ml dans la goutte): de nombreuses aiguilles
fines sont apparues dans la condition 1 du Crystal Screen II, en
présence de
10 % p/v de PEG 6000 / de NaCl à 2 M et sans tampon (donc
le pH dans la goutte était de 8,5 :
celui de la solution protéique).
? Pour l'enzyme (3,7 mg/ml dans la goutte) avec le GTP (20
mM) : il y a apparition de belles et nombreuses aiguilles dans la
même condition (condition 1 du Crystal Screen II).
? Pour l'enzyme (3,7 mg/ml dans la goutte) avec à
la fois GTP (20 mM) & UMP (10 mM) : aucune forme cristalline n'est
apparue, il y a dans certaines conditions apparition d'agrégats
et de précipités.
Les conditions les plus favorables à ce mutant pour
cristalliser sont donc : PEG 6000 à 10 % et NaCl 2,0 M, avec GTP
à 20 mM, pH 8,5. Par ailleurs, la structure avec GTP est plus
intéressante que celle de l'enzyme brute.
---Tests manuels approfondis : Afin
d'améliorer les formes cristallines, nous avons fait varier :
Dans le réservoir :
-la concentration : du PEG, du NaCl [exercent un effet
salting out (recherché pour la cristallisation] et rendent
les protéines moins solubles en fixant les molécules d'eau) et du
GTP ;
-la concentration et la nature de l'additif (glycérol et
le â-octyle-glycoside).
Dans la goutte : nous avons fait varier
la concentration protéique et le volume de A &
B.
Des aiguilles sont obtenues dans 14 cas
correspondants à des conditions différentes
(Tableau 3
& Figure 19). Le temps de cristallisation de
l'UMPKeco D159N D93A en présence du GTP est à peu près le
même que celui de l'UMPKeco D159N D93A seule (environ une semaine).
L'étude cristallographique (par cocristallisation) de
complexes enzyme/substrat est aléatoire et présente de grandes
difficultés. En effet, il nous est impossible de savoir si le
GTP (ligand) est
réellement présent dans le cristal avant la
résolution de la structure, qui peut prendre plusieurs mois.
![]()
![]()
1 2 3 4 5 6 7
Figure 18 : Suivi de la pureté de
l'UMPKeco D159ND93A par SDS - PAGE
1 - marqueurs
2 - extrait brut
3 - Pass - through colonne de Ni
4 - lavage
5 - Pool après élution colonne de Ni
6 - Pool après colonne Séphacryl S-
300
7 - Pool après concentration
Matériel prêt à être
donné pour la cristallisation
Figure 18 : Suivi de la pureté de
l'UMPKeco D159ND93A par SDS - PAGE
1 - marqueurs
2 - extrait brut
3 - Pass - through colonne de Ni
4 - lavage
5 - Pool après élution colonne de Ni
6 - Pool après colonne Séphacryl S-
300
7 - Pool après concentration
Matériel prêt à être
donné pour la cristallisation
Tableau 3 : Cristaux de l'UMPKeco D159N D93A :
Conditions d'obtention et dimensions.
|
|
PEG 6000
(%)
|
GTP (mM)
|
Graisse
|
NaCl
(M)
|
â-Glucoside
(%)
|
Dimensions (mm)
|
|
|
10
|
0
|
Présence
|
2
|
0
|
0,016 x 0,32
|
|
7
|
0,016 x 0,96
|
|
12
|
20
|
0,016 x 0,8
|
|
10
|
20
|
Absence
|
0,016 x 0,72
|
|
18
|
20
|
Présence
|
0,016 x 0,32
|
|
22
|
20
|
0,25
|
Non Déterminé (ND)
|
|
12
|
20
|
0
|
0,016 x 0,32
|
|
9,5
|
20
|
0,016 x 0,32
|
|
10
|
20
|
ND
|
|
16,5
|
20
|
0,016 x 0,32
|
|
17
|
20
|
0,016 x 0,32
|
|
19,5
|
20
|
0,016 x 0,32
|
|
18,5
|
20
|
0,016 x 0,32
|
|
19
|
20
|
0,016 x 0,32
|
|
|
|
PEG
6000 (%)
|
NaCl
(M)
|
GTP
(mM)
|
MPD
(%)
|
ß glucoside
(%)
|
Glycérol
(%)
|
Concentration protéique
(mg/ml)
|
|
|
3 à 22
|
1, 6 à 2,
6
|
20
|
5
|
0,25 à 0,5
|
0,25
|
3.7 à 7,4
|
|
Tableau 2 : Résumé de la variabilité
des conditions de cristallisation de l'UMPKeco D159N D93A
|
|
Sans Additif
Avec Graisse
Sans GTP
PEG 6000 10 % PEG 6000 7 %
![]()
![]()
![]()
![]()


![]()

![]()

GTP 20mM
PEG 6000 10 % / Sans graisse
PEG 6000 22 % / âGlucoside
à 0,25 %
/ Avec graisse
Figure 19 : Cristaux de l'UMPKeco D159N D93A
0,2 mm
En revanche, le nombre de cristaux obtenus, pour un
nombre d'essais comparables, en
présence du GTP est plus important que celui obtenu avec
l'enzyme brute (12 cas en présence du GTP
contre 2 avec l'enzyme seule) ; le GTP aurait un
effet favorable sur la cristallisation de cette enzyme.
Les meilleurs résultats sont obtenus dans des conditions
variables de PEG (entre 7 et 22 %) et
la même concentration du sel (NaCl à 2M), les
aiguilles les plus belles sont obtenues au voisinage de
10 % de PEG. La variation de la concentration protéique
n'a pas d'effet significatif sur la vitesse et le rendement de la
cristallisation de l'UMPKeco D159N D93A. Sauf pour les concentrations
élevées du précipitant (22 % du PEG), l'ajout d'additifs
(â-octyl-glucoside à 0,25 %) n'influe pas
nettement sur
le processus de cristallisation de cette enzyme.
Le tableau 3 montre que cette enzyme cristallise mieux au contact
de la graisse (1 cas de succès sans graisse contre 13 avec graisse),
comme pour l'enzyme D159N dont la structure a été
publiée.
3-Enregistrement et traitement des données
(Tableau 6) :
Les données de diffraction des rayons X ont
été enregistrées jusqu'à 3 ? de
résolution.120
images sont enregistrées, les taches sont très
rares et peu intenses et la mosaïcité est importante : le jeu
de données semble extrêmement difficile à
traiter.
B-CKOm sauvage et mutants D169E et
D192L
Pour la CKOm, nous avons fait varier dans le
réservoir la concentration du PEG 1500 et le volume du
tampon Tris-HCl pH 7,0. Dans la goutte nous avons fait
varier la concentration protéique
et le volume de A & B (Tableau
4). De gros cristaux colorés en jaune
(à cause du FAD lié covalemment à l'enzyme par
l'His 87) sont obtenues dans 12 cas correspondant
à des conditions différentes: 5 cas avec l'enzyme native, 7
cas avec le mutant D169E, aucun cristal n'a été obtenu avec
le mutant D492L (Tableau 5 & Figure 20). Le
temps de cristallisation de la CKOm est comparable à celui de
l'UMPKeco (environ une semaine). Plusieurs formes cristallines sont
obtenues (maclées, rectangulaires...). Comme on pouvait s'y attendre,
la CKOm cristallise mieux que l'UMPKeco. Enregistrement et
traitement des données de diffraction X : Le
Tableau 6 indique quelques
paramètres de l'enregistrement.
Les données de diffraction de la CKOm sauvage
complexée avec un inhibiteur suicide : HA-
8 et du mutant D169E avec le substrat : IP ont été
traitées pendant ce stage. Plusieurs paramètres ont
été utilisés pour le suivi et la
validation de l'affinement des résultats des diffraction : Rsym
(Rsym maximal toléré est 40 % dans la dernière
tranche de résolution et 10 % pour l'ensemble des
résolutions), complétude (doit être au moins de 80 %)...,
les statistiques obtenues à l'issue de plusieurs
séries d'affinements sont indiqués dans le
Tableau 7.
Tableau 4 : Résumé de la
variabilité des conditions de cristallisation de la CKOm
|
|
CKOm
|
PEG
1500 (%)
|
Tampon
Tris-HCl pH =7,0
|
â-octyl-glucoside
(%)
|
Concentration
protéique
(mg/ml)
|
VB dans la goutte de 4ul le
reste est complété par VA de la
SE
|
|
|
Native
|
16 à 33
|
+
|
0,5
|
7
|
1,92 à 2,92
|
|
Mutant D169 E
|
28 à 33
|
+
|
0,5
|
8
|
1,92 à 2,92
|
|
Mutant D492L
|
28 à 33
|
+
|
0,5
|
5
|
1,92
|
Tableau 5 : Cristaux de la CKOm : Conditions
d'obtention et dimensions.
|
|
Enzyme
|
PEG 1500 (%)
|
â-Glucoside
(%)
|
Dimensions (mm)
|
|
|
Enzyme Native
|
16
|
0,5
|
0,032 x 0,24
|
|
20
|
0,048 x 0,24
|
|
22
|
0,032 x 0,24
|
|
24
|
0,048 x 0,24
|
|
30
|
0,032 x 0,24
|
|
Mutant D169E
|
22
|
0,032 x 0,24
|
|
26
|
0,13 x 0,24
|
|
28
|
0,13 x 0,24
|
|
30
|
0,13 x 0,24
|
|
31
|
0,13 x 0,24
|
|
32
|
0,032 x 0,24
|
|
33
|
0,032 x 0,24
|
|
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()
![]()

![]()


CKOm native Mutant D169E
Figure 20 : Cristaux de la CKOm (native et mutant
D169E)
Figure 20 : Cristaux de la CKOm (native et mutant
D169E)
Tableau 6 : Enregistrement des données de
diffraction
|
|
Nombre de
cristaux montés sur des boucles
|
Nombre de cristaux qui ont diffracté
|
Conditions retenues
|
Résolution / Ligne
|
Glycérol
(Cryoprotectant)
|
|
|
UMPKeco D93A avec GTP ?
|
3
|
1
|
-18 % de PEG 6000
-2,0 M NaCl
- Protéine à 3,7
mg/ml
|
3 ? / ID 14-1
|
20 % pendant
1 min
|
|
CKOm, mutant D169E avec IP
|
3
|
3
|
-29 % PEG 1500
-0,5 % â-Glucoside
-Protéine à 8 mg/ml
-Tris HCl pH 7,0
100 mM
|
1,9 ? / ID 14-1
|
|
Tableau 7 : Résultats du traitement des
données
|
Jeu de données
|
CKOm sauvage avec HA-8,
Résultats Définitifs
|
CKOm D169E avec IP,
Résultats Préliminaires
du
traitement en cours
|
|
Longueur d'onde des rayons X (?)
|
0,934
|
0,934
|
|
Nombre d'images enregistrées
|
200
|
190
|
|
Angle de rotation lors de
l'enregistrement (°)
|
1
|
1
|
|
Distance Cristal -Détecteur (mm)
|
162,62
|
177,09
|
|
Système cristallin
|
Système monoclinique
|
Système monoclinique
|
|
Groupe d'espace
|
C2
|
C2
|
|
Paramètres de maille
|
a = 254,09 / b = 50,87 / c= 52,26 (?)
á = ã = 90,00 / â = 93,19 (°)
|
a = 251,08 / b =51,27 / c = 51,58 (?)
á = ã = 90,00 / â = 93,27 (°)
|
|
Résolution retenue pour résoudre
la structure 3D (?)
|
Résolution max 25,00
Résolution min 2,4
|
Résolution max 25,00
Résolution min 2,00
|
|
Nombre de réflexions observées
|
287781
|
720276
|
|
Nombre de réflexions uniques
|
22648
|
43460
|
|
Rodandonce
|
12,7
|
16,6
|
|
Mosaïcité
|
1,2
|
2
|
|
Complétude %
|
85,8
|
96,8
|
|
R fact (%) (< 10 %)
|
12,2
|
13,2
|
|
I/ä (> ou = à 2)
|
9,94
|
9,74
|
Les résultats obtenus pour la CKOm-HA-8 sont moins bons
que ceux de la CKOm-D169E-IP.
La structure 3D avec HA-8 ne serait pas assez précise
pour permettre de comprendre et d'expliquer clairement le mécanisme
d'inhibition. En plus, une structure avec un inhibiteur comparable a
été résolue (article en cours de rédaction).
Par contre, celle du mutant D169E-IP, vu les résultats
préliminaires du traitement des données, va certainement
expliquer le rôle de l'Asp 169 dans la catalyse enzymatique. Le
tableau 8 résume les résultats de l'étude
cristallographique des autres mutants de la CKOm cristallisés et
montés par Briozzo et al. La compréhension fine
du rôle et du fonctionnement de la CKOm sera améliorée
si la structure 3D de ces mutants, dont les cristaux sont
déjà obtenus, est résolue.
D- Projets
1-UMPKeco D159N
Notre travail nous a permis d'obtenir des cristaux avec le GTP
d'un mutant du résidu impliqué dans les interactions de maintien
de l'oligomérisation de l'enzyme. La suite du projet consistera d'une
part à poursuivre les études cristallographiques d'autres mutants
ponctuels non régulés par le GTP et d'autre part à
approfondir l'étude des mécanismes de régulation. Cela
pourrait avoir des applications dans le domaine de la santé publique.
2-CKOm
Le traitement des données étant entrepris, il est
nécessaire de résoudre la structure du mutant
D169N avec l'IP et de l'enzyme native avec l'HA-8. Il existe
d'autres structures de mutants ponctuels
en cours d'affinement avec des accepteurs d'électrons. Des
données ont été enregistrées, le même jour
sur la ligne ID14-1, avec les mutants L492A (deux jeux de
données avec des inhibiteurs différents) et E288Q (avec
inhibiteur). L'ensemble de ces structures permettra une meilleure
compréhension des mécanismes de fonctionnement de la
CKOm et cela aura éventuellement des applications
agronomiques.
| 


