|
Etude du polymorphisme génétique du
virus de l'enroulement des feuilles (TYLCV) de tomate (Lycopersicum
esculentum) dans la zone de Baguinéda au Mali.
Mama Théra,
theramama@yahoo.fr
Ousmane Koita, oakoita@yahoo.com
Introduction:
Le Mali est un pays à vocation agro-sylvo-pastorale, ou
plus de 80% de la population vivent du secteur du développement rural
où l'agriculture occupe la première place (72% avec un taux de
croissance autour de 1,5%) [3]. L'agriculture est pratiquée aussi bien
en saison pluvieuse qu'en contre saison. La culture de contre saison est
dominée par le maraîchage et surtout la culture de la tomate qui
constitue un élément très important dans la consommation
alimentaire quotidienne.
D'après une étude récente [9] les petits
exploitants utilisent en moyenne 0,2 hectares pour leur consommation
personnelle et la vente locale.
Le rendement moyen de production est de 17,4 tonnes par
hectare [9] contre 39,2 tonnes par hectare en Amérique, 19.4t/ha en
Amérique Latine D'après le Comité Inter état de
Lutte contre la Sécheresse au Sahel (CILSS), le rendement moyen est de
20t/ha [10].Cette différence peut être due à beaucoup de
facteurs : nutrition minérale, les semences, les insectes, les
agents pathogènes et autres. L'un des agents pathogènes est le
virus de l'enroulement des feuilles jaunes en cuillère de la tomate
(TYLCV), constituant un des problèmes majeurs pour les producteurs de
tomates au Mali, et cités comme une des raisons de la fermeture de
l'usine de transformation de tomate concentrée (SOCAMO) [9] Selon l'IER
, cette virose est plus importante dans la zone traditionnelle de production
(Baguinéda) que dans la zone plus récente de production (Office
du Niger) [9]
Sur la base des symptômes, l'incidence de cette virose
peut atteindre 80-100% de perte production au Burkina, Sénégal
et au Mali [10]. La perte de production varie de 80-100% selon les zones et la
précocité de l'infection.
Les méthodes de luttes classiques telles que les luttes
chimique, biologique et les pratiques culturales sont insuffisantes contre le
TYLCV.
La lutte chimique a pour corollaire la pollution de
l'environnement et des intoxications néfastes pour la santé
humaine.
Des recherches sont entreprises sur des méthodes de
lutte biologique, mais pour trouver un ennemi naturel adéquat sera une
tâche difficile [1].
La lutte par les pratiques culturales comprend le choix des
dates de plantation de manière à éviter les populations
élevées du vecteur, l'élimination des sources primaires et
secondaires du virus, l'utilisation de protection pour exclure le vecteur et
l'utilisation de transplants sains. Des recherches sont menées sur la
résistance [11] et l'on a découvert des cultivars plus
résistants. Des pieds de tomate contenant la protéine de capside
de TYLCV sont résistants au virus [19].
La manipulation génétique est une piste
à explorer pour le contrôle du TYLCV car le génome du virus
peut être cartographié et des gènes qui seraient à
la base de la résistance de la tomate au virus identifiés. Cette
résistance de la tomate serait liée au gène de la capside
du TYLCV.
La technique d'amplification de l'ADN par PCR et la
détermination de la séquence nucléotidique par
séquençage permettent d'examiner le polymorphisme du
génome du virus. Ainsi, nous nous sommes proposés
d'étudier la relation entre les génotypes de TYLCV et les
symptômes de cette virose.
Généralités :
Etudier le polymorphisme génétique des virus de
TYLCV sur la tomate par la technique PCR.
Identifier les virus TYLCV de la tomate par la technique de
PCR.
Déterminer les fréquences des différentes
souches de TYLCV présentes dans la zone d'étude.
Evaluer le polymorphisme génétique des virus
TYLCV dans la zone d'étude.
Etablir la relation entre les symptômes et le type de
virus.
Matériels et Méthodes
Echantillonnage : La collecte des
feuilles sera faite dans toutes les zones d'étude et les feuilles sont
stockée à -20°C avant l'extraction de l'ADN.
Extraction : l'ADN sera extrait
à partir des feuilles infectées de tomate selon la méthode
CTAB 3%).
A chaque tube, contenant 1 ou 2 centimètres de feuille
de tomate infectée, 500ul du tampon de CTAB (100mM de Tris PH 8.0, 50mM
d ; EDTA, 500mM de Na Cl) sera ajoutée puis incubée à
65 ° C pendant 10 minutes. Ajouté 400ul de chloroforme pour centrifuger
à 10000rpm durant 10 mn. Après centrifugation
récupérer le surnageant dans un autre tube contenant de
l'isopropanol pour précipiter l'ADN. Apres lavage avec l'éthanol
70%, 200ul de TE est ajouté pour la PCR et sa conservation.
Amplification par PCR des feuilles collectées.
Les conditions du thermocycleur sont optimisées pour la
détection des souches Israélite et Sardingue avec les amorces
spécifiques aux trois isolats (TYLCV, X15656, TYLCSV-ES [2], L27708
etTYLCV-M1d, X76319). Les paramètres pour la réaction de PCR sont
optimisés pour un volume de 25 ul. La concentration finale des
composantes de réaction : 2ul dNTPs, 2.5ul Buffer 10X PCR, 0.25ul
de Taq Polymerase, 1ul de l'amorce sens et 1ul de l'anti-sens et 1ul
d'ADN. Les paramètres de cycle sont comme suit : un cycle à
94° C pendant 10 minutes; 30 cycles à 94°C pendant pour1 minute, 58°C
pour 1minute, 72°C pour 1minute, 72°C pendant 10 minutes et enfin 4°C pour
toujours .
Résultats Sur un total de
quarante cinq échantillons, vingt quatre ont été
traités par la technique de multiplex PCR. Chaque échantillon a
été amplifié par les trois paires d'amorces, quinze se
sont révélés positifs et neufs négatifs.
Tableau IV : distribution de la taille des bandes aux
isolats et la répartition des virus
|
TYLCV
|
TYLCSV-ES [2]
|
TYLCV Mld
|
|
N
|
Présence
|
6
|
15
|
6
|
|
Absence
|
18
|
9
|
18
|
|
Moyenne de la taille des bandes
|
629.1667
|
386.4667
|
284.5000
|
|
Minimum de la taille des bandes
|
618.00
|
360.00
|
276.00
|
|
Maximum de la taille des bandes
|
638.00
|
401.00
|
290.00
|
Le tableau IV montre la répartition des
échantillons et la taille des fragments observés aux trois
isolats ainsi que les variations de la taille des bandes par les
différentes PCR.
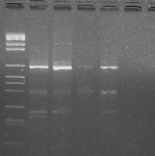
MVI 1 2 3 4
629pb
TYLCV
Figure 2: gel d'agarose présentant les bandes
amplifiées par les marqueurs de TYLCV
La figure2 présente les bandes de TYLCV sur gel
d'agarose dont la taille est estimée à 629 pb. Chacun des
échantillons au niveau des puits 1, 2, 3 et 4 présentent une
bande unique. Le premier puits contient le marqueur de poids moléculaire
VI (Roche Diagnostics Corporation, Mannheim, RFA)
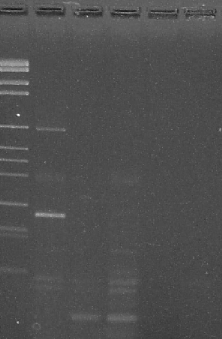
MVI 1 2 3
284pb
TYLCVMld
Figure 3: gel d'agarose montrant la bande de
l'amplification attendue par les marqueurs de TYLCVM1d
La figure3 présente une bande de TYLCVMld sur gel
d'agarose dont la taille est estimée à 284 pb. Le premier puits
contient le marqueur de poids moléculaire VI (Roche Diagnostics
Corporation, Mannheim, RFA)
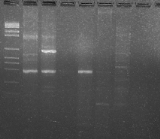
MVI 1 2 3
4 5 6
386pb
TYLCSV-ES [2]
Figure 4: gel d'agarose montrant l'amplification de
trois échantillons avec les marqueurs TYLCSV-ES [2]
La figure4 présentes les bandes de TYLCSV-ES [2] sur
gel d'agarose dont la taille moyenne est estimée à 386 pb. Les
échantillons 1, 2, 4 et 5 ont produit des bandes qui correspondent au
génotype TYLCSV-ES [2]. Le premier puits contient le marqueur de poids
moléculaire VI (Roche Diagnostics Corporation, Mannheim, RFA)
Tableau V : La fréquence relative des
génotypes obtenus par PCR
|
souches
|
Isolats
|
Positivité des échantillons par PCR
|
Fréquences
|
|
Souche Israélite
|
TYLCV
|
9
|
37.5%
|
|
TYLCVMld
|
|
Souche Sardinia virus
|
TYLCSV-ES [2]
|
15
|
62.5%
|
Le tableau V présente la fréquence des
génotypes obtenus par amplification par PCR par les amorces TYLCV ;
TYLCVMld (Souche israélite) et TYLCSV-ES [2] (souche Sardingue). Nous
avons observé que la souche Sardingue était la plus
fréquemment retrouvée (62,5) soit 15/24.
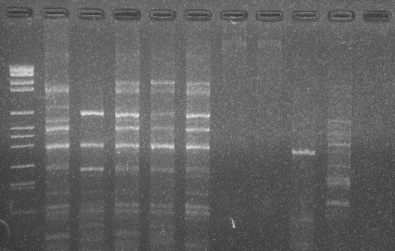
MVI 1 2 3 4 5
6 7 8 9 10
TYLCV
Non Spécifique
TYLCSV-ES [2]
TYLCVMld
Figure 5: Polymorphismes du TYLCV sur gel d'agarose. Cette
photo présente les bandes des trois isolats sur gel d'agarose
amplifiées simultanément avec les 3 paires d'amorce dans le
même test tube. Il faut noter que bandes non spécifiques sont
visibles sur le gel d'agarose. Le premier puits contient le marqueur de poids
moléculaire VI (Roche Diagnostics Corporation, Mannheim).
Tableau VI : fréquence et pourcentage des types
d'infections
|
Type d'infection
|
Fréquence
|
Pourcentage
|
|
Mono infection
|
9
|
37,5
|
|
PCR négative
|
9
|
37.5
|
|
Poly infection
|
6
|
25,00
|
|
Total
|
24
|
100,00
|
Dans le tableau VI, nous avons reparti les échantillons
selon qu'une seule (mono - infection) ou plusieurs bandes (poly-infection) sont
observées avec les trois amorce données.
Nous n'avons pas pu examiner des échantillons à
cause de la négativité des PCR.
Un quart de l'infection est multiple avec la présence
de plus d'un génotype alors que la mono infection était de 37,5%.
Nous notons que neuf échantillons sont restés
indéterminés parce que leur amplification a été
négative.
Tableau VII : Distribution des mono et poly infections
aux différentes souches.
|
souches
|
Isolâtes
|
Mono infection
|
Poly infection
|
fréquence
|
Pourcentage
|
|
Israélite
|
TYLCV
|
0
|
6
|
6
|
40%
|
|
TYLCV Mld
|
0
|
6
|
|
Sardingue
|
TYLCSV-ES [2]
|
9
|
6
|
9
|
60%
|
|
Total
|
|
9
|
6
|
15
|
100%
|
Le tableau VII montre qu'avec l'amorce TYLCV, plusieurs
génotypes ont été observés simultanément sur
les pieds de tomate avec une fréquence de 40%, aucun cas d'infection
mono spécifique n'est observer. Par contre, l'amorce TYLCV-ES [2]
présente des infections monoclonales (60%) et poly clonales (40%).
Tableau VIII : Relation entre les symptômes et les
types de virus
|
Souches
|
Isolats
|
Symptômes
|
|
Enroulement de la feuille
|
Rabougrissement
|
Variation de couleur des feuilles
|
|
Israélite
|
TYLCV
|
-
|
+
|
+
|
|
TYLCVMld
|
-
|
+
|
+
|
|
Sardingue
|
TYLCSV-ES [2]
|
+
|
+
|
-
|
Le tableau VIII donne la relation entre les symptômes et
les souches virales.
L'examen de la relation entre les symptômes et les
génotype montre que les génotypes Sardingue ne présentent
pas de variation de couleur mais des feuilles enroulées et rabougries et
cela de façon statistiquement significative (X2
= 13,333 et p = 0.001) tandis que les génotypes
israélites ne présentent pas de feuilles enroulées mais
des plants rabougris dont les couleurs varient. Cette relation n'était
pas statistiquement significative (X2 = 4,126 et p =
0,078).
DISCUSSION
La maladie de l'enroulement des feuilles jaunes en
cuillère de tomate est la plus dévastatrice des maladies de la
tomate. Beaucoup de ces espèces virales appartiennent au genre
begomovirus de la famille des geminiviridaes qui ont été
associées à la tomate [14]. Deux espèces sont connues
comme agent causal de la maladie au niveau de l'hémisphère ouest,
TYLCV (type Israélite) et TYLCSV-ES [2] (type Sardingue).Ces deux
espèces se retrouvent en Espagne et en Italie [5]. Ces virus sont
transmis pour la mouche blanche de l'espèce Bemisia
tabaci.
L'identification moléculaire est nécessaire pour
évaluer la diversité génétique des souches qui
circulent afin de développer des mesures de prévention contre les
isolats qui s'avèrent néfastes pour la culture des tomates.
Au cours de cette étude nous avons testé 24
échantillons. Ils ont été prélevés à
Baguinéda (Zone traditionnelle de production de tomate au Mali) et en
périphérie de la ville de Bamako.
Cette étude serait une contribution à la
connaissance des virus circulants dans la zone de Baguineda zone par excellence
de maraîchage.
. La limite de l'utilisation de nos amorces est que la mise en
évidence de la diversité est strictement fonction des souches
israélite et Sardingue.
Parmi les vingt quatre échantillons de tomate
traités, quinze ont pu être amplifiés soit un total de
62.5% et neuf non amplifiés soit un total de 37,5%. Cet échec
serait dû probablement soit à des délétions ou
ajouts de nucléotides qui changeraient les séquences sur
lesquelles les amorces s'apparieront [5] ,ou probablement à l'existence
des formes hybrides qui ont étés observés par d'autres
auteurs ([16] ; [17]), que nos amorces ne pouvaient pas reconnaître
afin d'initier les réactions d'amplification par PCR La
négativité de l'amplification par PCR peut s'expliquer aussi par
l'absence du génome viral dans les échantillons de feuilles
collectées.
Selon la taille des produits de multiplex PCR observé
chez ces échantillons, nous avons trouvé trois types de
fragments. Le premier fragment correspondant au génotype TYLCVMld a
été retrouvé au Sénégal et au Cape Vert [2].
Ce génotype est lié sur le plan génétique à
la souche retrouvée au Moyen Orient [2]. Quant au second, il correspond
au génotype TYLCSV-ES [2] (type Sardingue), il se retrouve en Espagne et
en Italie ou il cause le changement de coloration des feuilles (qui deviennent
aussi jaunes) et le rabougrissement des plants de tomate. Ce génotype a
été retrouvé au Mali au cours de notre étude. Le
troisième génotype TYLCV est une souche cosmopolite
retrouvée sur tous les continents [5]. Cependant ce génotype
serait lié à la souche TYLCV de type Israélite.
L'examen des fragments de PCR a montré que
l'amplification par une même paire d'amorce montre la présence de
plusieurs fragments dont les tailles varient sur le gel d'agarose.
Pour le cas de TYLCV (type israélite), les tailles ont
varié de 638 à 618 paires de bases sur le même gel
g'agarose. Nous pensons que chaque fragment représente un type de TYLCV
(Israélite). Pour le TYLCSV-ES [2] (type Sardingue), la taille des
fragments ont varié de 401 à 360 paires de bases sur le
même gel. Et pour le TYLCVMld (type Méditerranéen), de 290
à 276 paires de bases sur le même gel d'agarose. Cette observation
donne une autre dimension à notre analyse, c'est-à-dire la notion
de polymorphisme de taille généré soit par addition ou
délétion de base sur la région cible de l'amplification
par les amorces (thèse de Koita).
Ainsi pour la souche TYLCV la taille moyenne était 629
Pb, ce qui diffère de 5 Pb de la taille attendue (figure2.) En utilisant
les marqueurs pour le génotype TYLCSV-ES [2], la moyenne de la taille
des fragments était 386 paires de bases ce qui diffère de 48
paires de bases par rapport à la taille du fragment de
référence (figure 4). Quant au génotype TYLCVMld, il y a
une différence de 32 paires de bases entre la moyenne des fragments
obtenus au cours de notre étude et le fragment de
référence (figure 3).
Le génotype TYLCV (type Israélite) et TYLCVMld
(type Méditerranéen) sont associés au rabougrissement de
la plante (p = 0,03). Mais nous n'avons pas retrouvé une relation
statistiquement significative entre l'enroulement des feuilles et les deux
génotypes, quoiqu'il y ait des plantes infectées avec ces deux
génotypes présentaient des feuilles enroulées. Le fait que
ces deux génotypes produisent les mêmes symptômes est
compréhensible, ils appartiennent à la même espèce
TYLCV (type Israélite)
Par contre l'enroulement des feuilles était
associé d'une manière statistiquement significative avec la
présence de TYLCSV-ES [2] (type Sardingue) p = 0,001.
Ainsi, nous pouvons suggérer que la
sévérité de la maladie dépend de la présence
simultanée des trois génotypes qui appartiennent à deux
espèces.
La conséquence de cette multiple infection par les
trois génotypes est que la mouche blanche (B tabaci) peut
être infectée par les trois génotypes. La présence
de ces trois génotypes pourrait entraîner la formation des formes
recombinantes à la suite de brassage génétique ([4] ;
[13]).
Nous avons observé que le TYLCV et TYLCVMld sont
associés et ils représentent la souche Israélite avec
37,5% de fréquence tandis que le TYLCSV-ES [2] révèle la
présence de la souche Sardiniavirus avec 62,5% de fréquence. Cela
était attendu car la même amorce aller permet à la fois
d'amplifier TYLCV et TYLCVMld (tableau III).
Les produits amplifiés par multiplex PCR ont
montré des fragments de taille differentes.Trois types de fragments ont
été obtenus.
Le fragment de 629 paires de bases se rapprocherait du
génotype TYLCV qui présente 634 paires de bases selon
l'étude Anfoka et al [6] (figure2). Le second fragment a une
taille de 386 paires de bases cette taille serait proche de la taille du
génotype TYLCSV-ES [2] avec 434 paires de bases que Anfoka et
al auront observés à leur étude (figure4).
Enfin le troisième fragment observé au cours de
notre étude aurait 284 paires de bases qui se rapprocheraient du
génotype TYLCVMld de type Méditerranéen qui a une taille
de 316 paires de bases (figure3). Il faudra noter que cette différence
entre les tailles de nos génotypes avec ceux obtenus par Anfoka et
al peuvent avoir pour cause ; 1] les conditions de migration
electrophoretique de notre laboratoire (notre temps de migration était
différent de celui de Anfoka et al ;2] aux réarrangement,
insertion ou délétion qui pourraient joués sur la longueur
des segment d'ADN venant des souches locales, indiquerait des virus
différents.
La technique de séquençage serait l'outil
idéal pour confirmer cette observation.
Donc notre étude montre que les trois génotypes
trouvés ailleurs [11] se retrouve au Mali Mais la fréquence des
trois génotypes est différentes ainsi TYLCSV-ES [2] était
le plus présent avec 62.5%(15/24) suivi des deux autres qui se trouvent
dans un système de co-infection (infection simultanée de la
tomate par les deux génotypes), de l'ordre de 37,5%.
Cependant à l'intérieur des génotypes
TYLCSV-ES [2], nous avons un polymorphisme de taille. Nous avons une variation
de taille de 401 à 360 paires de bases, suggérant l'existence des
sous types de cette souche. Ce phénomène est observé dans
d'autres systèmes viraux tels que le VIH. Il en est de même chez
les deux génotypes (TYLCV et TYLCVMld).
L'enroulement des feuilles et le changement des couleurs sont
des paramètres qui n'ont pas données une relation statistiquement
significative chez le TYLCV et le TYLCVM1d (X2 = 4.126 et p =
0.078), par contre, le rabougrissement a donné une relation
statistiquement significative chez ces deux isolats (X2 = 9.455 et
p= 0.003).
Chez le TYLCSV-ES [2], l'enroulement des feuilles (X2
= 13.333 et p = 0.001) et le rabougrissement (X2 = 12.185 et p
= 0.001) ont donnés une relation statistiquement significative. Le
changement de couleur n'a pas présenté une relation
statistiquement significative (X2 = 3.789 et p = 0.118).
L'analyse par le test de Fisher montre qu'il existe une
relation statistiquement significative entre les symptômes et
génotype. Cette variabilité de ce génome viral peut
conduire à la formation de formes hybrides [8,12] (résultats des
recombinaisons entre deux génotypes) comme constaté par
Sánchez-Campos, S [15]. La conséquence de cette
réarrangement génétique est d'accroître le
répertoire antigénique des ces virus empêchant le
développement normal de la plante. Plusieurs symptômes sont
associées à la présence des Begomovirus chez la
tomate (enroulement des feuilles, coloration des feuilles et le
rabougrissement générale de la plante. Ces observation ont
été aussi faites par Konaté [10].
Il y a plusieurs méthodes de typer le génome,
nous avons utilisé la technique de multiplex PCR [9] qui permet de
détecter simultanément différents fragments d'ADN d'un
microorganisme donné. Elle a l'avantage d'être économique
(moins de réactifs), avec un temps assez court pour l'amplification. Par
contre, elle requiert que les amorces aient la même température
d'appariement. Aussi il était possible d'utiliser la RFLP, cette
technique demande une grande quantité d'ADN et ce qui n'est pas le cas
lorsqu'on à faire des souches sauvages collectées sur le
terrain.
Référence bibliographique
1] Astier S., Albouy J., Maury Y., Lecoq H.,
(2001). Principes de Virologie végétale : génome,
pouvoir pathogène, écologie des virus.INRA ed. Versailles,
444p.
2] Czosnek H, Laterrot H. (1997). A
worldwide survey of tomato yellow leaf curl viruses. Arch Virol 1997; 142(7);
1391-406.
3] Direction nationale de la statistique,
2000.
4] Faten GORSANE, Hatem FAKHFAKH, 2002.
Virus Detection: Tomato yellow leaf curl virus (TYLCV).Methods:
Variability analysis of TYLCV species (strains)
5] Fouad Akad and Hanokh Czosnek, 2002. The
Hebrew University Address (Email): akad @agri.huji.ac.il and
czosnek@agri.huji.ac.il Date: Oct. 8, 2002
6] Ghandi ANFOKA, Mohamed AHAARY and Mark
NAKHLA, (2005), Detection of tomato yellow leaf curl virus isolates by
multiplex Polymerase Chain Reaction.
7] Gharsallah Chouchane S, Gorsane F, Louzir
H, Nakhla MK, Marrakchi M, Fakhfakh H , (2006) Developpment of a real-time PCR
for tomato yellow leaf curl Sardinia virus. Acta Virol.2006 ;
50(2) : 101-5.
8] Idris AM, Brown JK. Evidence for
interespecific-recombination for three monopartite begomoviral genomes
assaciated whith the tomato leaf curl disesease from central sudan. Arch Virol
(2005) May; 150 (5); 1003-12. epub (2005) Feb 10.
9] IER-Mali: K. Gamby, M. Noussourou
USAID-Mali: R. Shetty, D. McCarthy UC Davis: R.Gilbertson, M.Rojas, Y. Zhou, L.
F. Chen Purdue University: R. Foster (2004)
10] Konaté, G., Otoidobiga, LC. Et
PALE, S., (2004),. Situation du TomatoYellow leaf curl virus au Sahel.
11] Kunik, T.; Salomon, R.; Zamir, D.; Navot,
N.; Zeidan, M.; Michelson, I.; Gafni, Y.; Czosnek,-H. (1994) Transgenic tomato
plants expressing the tomato yellow leaf curl virus capsid protein are
resistant to the virus. Bio/Technology 12, 500-504.
12] L.Ge, J.Zhang, X.Zhou, andH.Li, (2007),
Genetic Structure and Population Variability of TomatoYellowLeafCurlChinaVirus
J. Virol.2007 0: JVI.02431-06v1-
13] Navas-Castillo, J.,
Sánchez-Campos, Noris, E., Louro, D., Accotto, G. P., and Moriones, E.
2000. Natural recombination between Tomato yellow leaf curl virus-Is and Tomato
leaf curl virus. J. Gen. Virol. 81:2797-2801.
14] Rybicki EP., (1994).A phylogenetic and
evolutionary justification for three genera of Geminiviridae. Achieves of
Virology 139: 49-7.
15] Sánchez-Campos, S.; Díaz,
J.A.; Monci, F.; Bejarano, E.R.; Reina, J.; Navas-Castillo, J.; Aranda, M.A.;
Moriones, E. (2002) High genetic stability of the begomovirus Tomato yellow
leaf curl Sardinia virus in Southern Spain over an 8-year period.
Phytopathology, 92(8), 842-849
16] Susana Garcia-Andrés, Gian Paolo
Accotto, Jesus Navas-Castillo and Enrique Moriones (2006). Founder effect,
plant host, and recombination shape the emergent population of begomoviruses
that causes the tomato yellow leaf curl desease in the Mediterranean basin.
Instituto di Virologia Vegetale,Consiglio Nazionale delle Ricerche, 1-10135
Torino Italy.
17] T Kon, K Kuwabara, SH Hidayat, M Ikegami
-Arch Virol,(2007) - ncbi.nlm.nih.gov.A begomovirus associated with ageratum
yellow vein disease in Indonesia: evidence for natural recombination between
tomato leaf curl Java virus and Ageratum yellow vein virus-[Java].".
| 


