|
UNIVERSITE FRANCOIS RABELAIS
TOURS
|
|
RAPPORT DE STAGE DE MASTER 1
INFECTIOLOGIE CELLULAIRE ET
MOLECULAIRE, VACCINOLOGIE
Mémoire présenté par AIDOUD Nacima
Expression d'une protéine non structurale du
virus de l'hépatite
C (la NS4B) en système
dérivé du virus de la Forêt de Semliki et
étude
de son implication dans la formation du membranous web
Travail réalisé au laboratoire de recherche sur
les virus et pseudovirus :
Morphogenèse et
Antigénicité
ERI 19 CHRU Bretonneau TOURS
Sous la direction de Dr BLANCHARD Emmanuelle Dr ROINGEARD
Philippe
Année Universitaire 2007-2008
_Remerciements
J'adresse tout d'abordma reconnaissance d _Philipe
_ROIW~_T~_RD et _Tmmanuelle 11W 9~_RD pour m'avoir accueillie dans le
laboratoire et m'avoir con#iée cepro%et:
Je remercie du fonddu coeur toute l'$quipe _T_RI ig, pour
leur soutien et leur disponibilité ainsi quepour leur
attentionparticulière portée d monpro%et:
Merci d'avoir cru en moi et de m'avoir_aitpartager cet
engouement acharnepour la recherche:
SOMMAIRE
INTRODUCTION
A. Virus de l'hépatite C p.1
a) Structure et organisation du génome viral
b) Les protéines virales
B. Objectif du travail de recherche p.3
MATERIEL ET METHODES p.4
A. Construction plasmidique en système pSFV p.4
a) Le système d'expression pSFV
b) Les constructions vectorielles
B. Amplification des vecteurs d'expression recombinants p.5
a) Transformation bactérienne
b) Maxipréparation classique et vérification des
clones recombinants
C. Transcription in vitro et transféction des
ARNs p.6
a) Linéarisation des vecteurs recombinants par
SpeI
b) Transcription in vitro des vecteurs
linéarisés
c) Culture cellulaire des BHK2 1
d) Transféction des BHK21 par électroporation
D. Expression de la protéine NS4B ...p.7
a) Immunofluorescence des BHK-21 transfectées
b) Western Blot sur les lysats BHK-2 1 transfectées
c)Microscopie Electronique des cellules BHK-2 1 transfectées
RESULTATS p.9
A. Linéarisation . p.9
B. Transcription in vitro p.9
C. Immunofluorescence . p.10
D. Western blotting p.11
E. Microscopie Electronique .. p.12
DISCUSSION p.12
BIBLIOGRAPHIE
INTRODUCTION
L'existence d'un nouveau virus responsable de 90% des
hépatites post-transfusionnelles dites « non-A, non-B » fut
soupçonnée dès les années 70. Grâce à
l'essor des technologies de biologie moléculaire, le virus de
l'hépatite C (VHC) a pu être caractérisé en 1989
suite au clonage et séquençage de l'intégralité de
son génome (Choo et al, 1989) [1]. L'infection par le VHC
représente un intérêt médical majeur puisque 170
millions de personnes à travers le monde et environ 600 000 en France,
sont des porteurs chroniques de ce virus. Dans la majorité des cas,
cette infection chronique conduit à des complications hépatiques
sévères, pouvant évoluer vers une cirrhose associée
au développement d'un hépatocarcinome. Les perspectives de vaccin
sont lointaines, les thérapies anti-virales utilisées
actuellement ont une efficacité limitée.
Bien que le VHC ait été identifié depuis
près de 20 ans et que son génome et ses protéines soient
désormais bien connus, le cycle infectieux de ce virus reste encore
énigmatique par bien des aspects. Ceci résulte principalement de
la difficulté, jusqu'à très récemment, de propager
le VHC sur des systèmes de culture cellulaire in vitro.
Finalement, l'année 2005 aura été un tournant dans la
recherche sur le VHC, avec la mise au point du modèle du virus JFH-1
permettant de reproduire, pour la première fois, un cycle infectieux
complet du VHC in vitro (Wakita et al., 2005) [2]. Ce clone JFH-1 a
été isolé chez un patient atteint d'une hépatite C
fulminante, de génotype 2a.
A- LE VIRUS DE L'HEPATITE C
a Structure et organisation du génome viral
Le VHC est un virus appartenant à la famille des
Flaviviridae et au genre hepacivirus dont il est le seul
membre. Si le génome et les protéines du VHC sont connus depuis
plusieurs années, la structure et la morphogenèse des particules
virales demeurent encore hypothétiques. Cet échec s'explique
d'abord par la difficulté à visualiser avec certitude les
particules virales dans le sérum ou dans des biopsies hépatiques
de patients infectés chroniquement par le VHC. Par analogie avec
d'autres virus de la même famille, le VHC est un petit virus
enveloppé de 50 à 60 nm de diamètre avec un génome
ARN de polarité positive d'environ 9500 nucléotides (nt) contenu
dans une capside protéique de symétrie icosaédrique
entourée d'une enveloppe lipidique empreintée à la cellule
hôte dans laquelle sont enchassées les glycoprotéines
d'enveloppe virale (Figure 1). Le génome est constitué par un
cadre unique de lecture ouvert encadré de deux régions non
codantes (NC) aux extrémités 5' et 3 'intervenant respectivement
dans la réplication et dans la stabilisation du génome viral lors
de sa réplication.
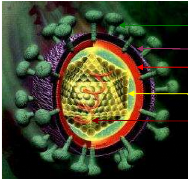
Glycoprotéines d'enveloppe E1/E2
hétérodimériques
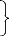
Couche lipidique externe
Couche lipidique interne Enveloppe
Capside protéique icosaédrique
ARN génomique monocaténaire de polarité
positive (ARN+)
Figure 1 : Représentation
schématique de la structure du VHC
b. Les protéines virales
La traduction de l'ARN génomique via le cadre unique de
lecture aboutit à la synthèse d'une polyprotéine
précurseur clivée en protéines structurales et non
structurales par les protéases cellulaires et virales, respectivement
(cf Figure 2). Les protéines structurales situées dans le premier
tiers N-terminal de la polyprotéine précurseur comprennent la
capside qui s'autoassemble pour former la nucléocapside et les
glycoprotéines d'enveloppe E1 et E2, impliquées dans la
reconnaissance cellule-virus. Les protéines structurales et non
structurales sont séparées par une petite protéine p7
encore mal connue, soupçonnée jouer le rôle de canal
calcique. A l'extrémité C-terminale, se trouvent les
protéines non structurales (NS2, NS3, NS4A, NS4B, NS5A et NS5B) qui
interviennent dans la réplication du virus. La protéine NS2
(associée à la protéine NS3 a une fonction
d'autoprotéase, responsable de la coupure NS2/NS3. La protéine
NS3 possède deux fonctions : (i) une activité protéase
à sérine qui associée au co-facteur NS4A (dont le
rôle va être de stabiliser cette enzyme) est impliquée dans
le clivage NS3/NS4A et NS4B/NS5A et (ii) une activité
hélicase/NTPase permettant à l'ARN viral d'adopter une
conformation stable pour sa réplication. NS4B reste une protéine
encore mal caractérisée. La région NS5 code deux
protéines : NS5A qui est une phosphoprotéine et NS5B qui est
responsable de la réplication de l'ARN viral avec une activité
ARN polymérase ARN dépendante.
p7
E1
C
NS2
E2
environ
3000 AA
NS5a
NS4a NS4b
NS5b
NS3
Figure 2 : Organisation génomique du
VHC
5 'non-codant
3 'non-codant
Polyprotéine
Précurseur
(9500 nt)
341nt
protéines structurales protéines
non-structurales
40nt
(U)n
peptidases cellulaire du signal
auto-protéase NS2/NS3
clivage par le complexe NS3-NS4a
98nt
SPP
KDa 22 32-36 70 7 21 70 6 27 56 68
|
capside
recrutement
de l 'ARN
|
protéines
d'enveloppe
|
?
|
protéase à ser
NTPase
hélicase
|
Formation du
membranous
web ?
|
ARN polymérase
ARN-dépendante
|
|
|
|
|
|
|
métalllo-protéase cofacteur
phosphoprotéine
zinc-dependante protéase NS3 co-facteur de
NS5B
(stabilise NS3)
B- OBJECTIF DU TRAVAIL DE RECHERCHE
L'étude du cycle infectieux du VHC a longtemps
été considérablement limitée par l'absence de
système de culture de ce virus in vitro. Toutefois, il est
généralement admis que dans les cellules répliquant le
VHC, une partie de ces protéines non structurales s'organisent entre
elles pour constituer un complexe multiprotéique appelé complexe
de réplication. De plus, une des principales caractéristiques des
virus à ARN positif, en plus de contenir un ARN messager simple brin
dans leur particule virale, est l'association de ce complexe de
réplication avec des cytomembranes cellulaires qui se réarrangent
au cours de l'infection, en structure vésiculaire. De telles membranes,
hébergeant vraisemblablement le complexe de réplication du
génome viral, ont été mises en évidence dans des
coupes de cellules (Egger et al, 2002 [3] ; Gosert et al,
2003 [4]), mais ces structures définies sous le terme de
membranous web sont encore mal caractérisées. Des
études préalables dans un contexte autre que le JFH1 ont
proposé que la protéine NS4B, une protéine exclusivement
membranaire, pourrait jouer un rôle majeur dans la formation de ce
membranous web (Egger et al., 2002 [3]).
Dans ce contexte, l'équipe ERI 19 avait choisi de
cloner et exprimer la protéine NS4B seule, issue du clone JFH-1,
à l'aide d'un vecteur d'expression original et puissant,
dérivé des propriétés de réplication du
virus de la forêt de Semliki (SFV).
Ainsi l'objectif de mon travail consistait à essayer
d'exprimer la protéine NS4B issue du clone JFH-1, munie d'une
étiquette hémagglutinine (HA) en système SFV. En effet, il
n'existe pas d'anticorps disponible dirigé contre la protéine
NS4B de la souche JFH-1. L'expression des cellules transfectées avec les
ARN recombinants codant la protéine NS4B a été
analysée par western blotting et immunofluorescence à l'aide d'un
anticorps anti-HA. Des coupes de cellules transfectées ont
également été réalisées et observées
en ME, dans le but de mieux comprendre le
rôle putatif de la protéine NS4B dans le
mécanisme de remaniement membranaire observé lors de la
morphogenèse du complexe de réplication.
MATERIEL ET METHODE
A- Constructions plasmidiques en système
pSFV
a. Le système d'expression pSFV
Le Virus de la Forêt de Semliki se compose d'un
génome ARN + monocaténaire circulaire appelé ARN 42S(+)
qui possède un fort potentiel d'auto-réplication
conféré par ses protéines non structurales dont les
séquences sont situées en amont du promoteur SFV (P26S)
contrôlant les séquences codant les protéines structurales.
L'utilisation du SFV comme vecteur d'expression consiste à remplacer les
séquences des protéines structurales par un gène
d'intérêt. Ce vecteur nécessite une linéarisation et
une transcription in vitro puisqu'il ne possède pas de
promoteur eucaryote, ainsi l'ARN 42S(+) obtenu est transfécté
dans des cellules permissives (telles que les BHK-2 1 qui permettent une
expression satisfaisante de ce vecteur). Les protéines non structurales
traduites par les ribosomes, s'assemblent et vont fixer l'ARN 42S(+) en 3' et
synthétiser un ARN 42S de polarité négative (ARN 42S(-))
qu'elles ensuite vont lier en 5' afin de former un nouvel ARN 42S(+) qu'elles
vont à son tour fixer en 3' et produire un nouvel ARN 42S(-). Ceci va
aboutir à l'accumulation d'ARN 42S(-), matrice pour la synthèse
de la protéine d'intérêt, et les protéines non
structurales se désassemblent et fixe alors le promoteur SFV (P26S) afin
de transcrire le gène cloné qui sera alors traduit. Finalement on
obtient une production élevée de protéine
d'intérêt.
b. Les constructions vectorielles
A mon arrivée, l'équipe avait déjà
cloné spécifiquement la protéine non structurale NS4B de
la souche JFH-1 du VHC dans le système SFV et disposait de deux
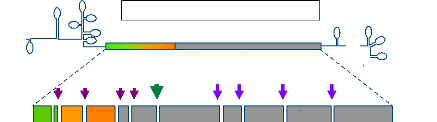
constructions contrôles SFV. Ainsi, 4 constructions étaient
disponibles. (Figure I)
La construction PSFV-NS4B/HA, contenant la protéine
NS4B du VHC avec une étiquette HA (oligopeptide issu de
l'hémagglutinine du virus de la grippe, séquence PYDVPDYA). En
effet, du fait de l'absence d'anticorps dirigés contre la
protéine NS4B de la souche JFH-1 nous avons choisi de marquer cette
protéine en C-terminale avec une étiquette HA. Toutefois, la
présence de l'épitope HA pouvant modifier la topologie
membranaire de la protéine, une autre construction sans étiquette
nommé pSFV-NS4B avait été générée
afin de préserver l'ultrastructure membranaire des cellules exprimant la
protéine NS4B native. Le vecteur pSFV-âgal, qui contient le
gène codant la â-galactosidase (enzyme cellulaire cytosolique et
ubiquitaire) constitue un
témoin négatif de transfection. Enfin un vecteur
pSFV1-C191 contenant le gène codant la protéine de capside du
VHC, ayant la propriété de s'auto assembler pour former des
pseudo-particules visualisable en microscopie électronique (Blanchard
et al, 2003 [11]), est utilisé comme témoin positif.
pSFV-NS4B/HA
11893 N
pb
pSFV- -gal
pSFV_C191
pSFV-NS4B
13078
11869
1 1572
pb
pb
pb
Protéines non-tructurales du pSFV
Insert cloné dans le pSFV
NS1 NS2 NS3 NS4 NS4B
NS1 NS2
NS1 NS2 NS3 NS4 C191
S1 NS2
NS3 NS4 NS4B HA
NS3 NS4 -gal
pSFV 11O33 pb
NS4B 836 pb
NS4B-HA 860 pb
-gal 2045 pb
C191 539 pb
Témoin (+)
Témoin (-)
Figure I. Représentation schématiques des
constructions vectorielles réalisées.
B- Amplification des vecteurs d'expression recombinant
a. Transformation bactérienne
Cinquante microlitres de bactéries compétentes
(Escherichia coli DH5á rendues chimiocompétentes par le
chlorure de rubidium, 108 bactéries/ml) ont été
transformées par incubation de 30 minutes dans la glace en
présence de 1ìl de plasmide, suivie d'un choc thermique par
incubation de 30 secondes à 42°C. Les bactéries ont ensuite
été étalées sur gélose nutritive LB (Luria
Bertani), contenant 10 mg/mL d'ampicilline et incubées à
37°C pendant 16 heures. Ainsi seules les bactéries
transformées avec les constructions pSFV pourront pousser et former des
colonies puisqu'elles contiennent une cassette de résistance à
l'ampicilline.
b. Maxipréparation classique et vérification
des clones recombinants
Des maxipréparations d'ADN plasmidique
ont ensuite été réalisées sur un clone
bactérien ayant
poussé sur LB pour chaque construction selon
un protocole classique. Une colonie provenant de
chaque construction a
été ensemencée dans 500 mL de milieu LB/ampicilline et
incubée pendant
16 heures à 37°C sous agitation. La
culture bactérienne a été culottée par
centrifugation de 30
minutes à 4 000 g à 4°C
(Centrifugeuse Jouan GR 4.11), et le culot a été resuspendu dans
8 mL de
Tampon Glucose/Tris EDTA (GTE) (Glucose 500 mM, Tris 30 mM [pH 8],
EDTA 10 mM)
contenant 50 mg/mL de lysozyme (Sigma). Après une
incubation de 15 minutes dans la glace, 16
mL de tampon de lyse SDS/NaOH
(SDS 35 mM, NaOH 0,2M) ont été ajouté suivi
d'une
incubation de 5 minutes dans la glace. La lyse a été
arrêtée par adjonction de 12 mL d'acétate de
potassium 3 M. Ce lysat a été incubé 15
minutes dans la glace avant d'être centrifugé 30 minutes
à
4 000 g à 4°C. Un demi volume d'isopropanol a
été ajouté au surnageant préalablement
filtré, et le
tout a été incubé 20 minutes dans
la glace, puis centrifugé 20 minutes à 4 000 g à 4°C.
Le culot a
été resuspendu avec 5 mL de tampon Tris/EDTA (TE)
(Tris/HCl 10 mM [pH 8], EDTA 1mM) et
5 mL de chlorure de lithium (LiCl 0,5 M, Tris/HCl 50 mM [pH
7,5]) incubé 15 minutes dans la glace et centrifugé 20 minutes
à 4 000 g à 4°C. L'ADN présent dans le surnageant a
été précipité à -20°C pendant 30
minutes avec deux volumes d'éthanol absolu, puis centrifugé 30
minutes à 4 000 g à 4°C. Le culot a été
resuspendu dans 2 mL de tampon TE suivi d'une incubation d'une heure à
37°C en présence de 30 j.iL de ribonucléase (10 mg/mL). Le
mélange a été incubé toute la nuit à
37°C avec 40 j.iL de protéinase K (10 mg/mL) (Boerhringer
Mannheim). L'ADN plasmidique a été extrait et purifié par
ajout de 500 j.iL de phénol/chloroforme et centrifugé 10 minutes
à 4 000 g. L'ADN présent dans la phase aqueuse a
été précipité à -20°C durant 30 minutes
avec 300 j.iL d'éthanol absolu en présence de 30 j.iL
d'acétate d'ammonium. Après une centrifugation de 15 minutes
à 16 000 g, le culot d'ADN a été lavé à
l'éthanol 70% et a été repris dans 300 j.iL de TE, et
quantifié par spectrophotométrie (SmartSpec, Biorad) à 260
nm. La présence des inserts a été vérifiée
par digestion enzymatique grâce à XmaI puisque deux sites
de restriction XmaI sont présents de part et d'autres du
gène d'intérêt. Le sens des inserts a été
également contrôlé par une deuxième réaction
enzymatique avec l'enzyme BamHI, qui coupe une seule fois dans
l'insert (à la fin de la séquence NS4B) et une seule fois dans le
vecteur pSFV1 (en amont du site multiple de clonage du pSFV1).
C- Transcription in vitro et transfection des ARNs
a. Linéarisation des vecteurs
Dix microgrammes de plasmide pSFV recombinant ont
été linéarisés avec 50 unités d'enzymes
SpeI pendant 90 minutes à 37°C sous un volume
réactionnel de 100 j.iL. L'ADN linéaire a été
purifié par extraction au phénol-chloroforme, suivi d'une
précipitation à l'éthanol et après séchage
le culot d'ADN a été solubilisé dans 40 j.iL d'eau
stérile. La linéarisation a été
vérifiée grâce à migration
électrophorétique en gel d'agarose 1% en tampon Tris
Acétate/ EDTA (Tris/acétate 40 mM, EDTA 1mM) contenant 0,5 j.ig
de bromure d'éthidium (BET). L'utilisation de SpeI s'explique
par le fait qu'elle possède un site de restriction unique dans le pSFV
en aval du site multiple de clonage.
b. Transcription in vitro des vecteurs d'expression
linéarisés par SpeI
Un microgramme d'ADN linéaire a été
transcrit in vitro lors d'une incubation pendant 90 minutes à
40°C, dans un milieu réactionnel de 60 j.iL contenant 1 mM d'un
mélange de rNTP (Biolabs), 1 mM d'analogue de coiffe m7G(5)ppp(5')G
(Biolabs), 90 unités d'inhibiteur de
ribonucléase (Rnasin, Biolabs) et 72 unités de
SP6 RNA polymérase (Biolabs). Après transcription, la
qualité de l'ARN a été vérifiée sur gel
d'agarose. L'ARN a été aliquoté sous 27 j.iL et
conservé à -80°C jusqu'à son utilisation.
c. Culture Cellulaire des BHK21
Les cellules BHK-21 (Baby Hamster Kidney) ont
été utilisées en raison de l'efficacité de
transfection par électroporation obtenue avec ces cellules qui sont
habituellement utilisées avec le système d'expression SFV. Elles
ont été cultivées à 37°C sous 5% de CO2 en
milieu GMEM (Invitrogen) supplémenté avec 5% de sérum de
veau foetal décomplémenté (ATGC), 12% de tryptose
phosphate (Sigma) et 1% de pénicilline/streptomycine (Invitrogen).
d. Transfection des cellules BHK-21 par
électroporation,
A confluence du tapis cellulaire, les cellules BHK-2 1 ont
été trypsinées, lavées au PBS et
centrifugées 10 minutes à 200 g puis resuspendues dans du tampon
PBS à la concentration de 12. 106 cellules/mL. La
transfection a été effectuée en transférant 1 mL de
la suspension cellulaire en présence de 27 j.iL d'ARN SFV dans une
cuvette d'électroporation. L'électroporation a été
réalisée par impulsion électrique de 3 50V, 750 j.iF. Les
cellules ainsi transfectées ont été rapidement mises en
culture pendant 16h à 37°C sous 5% de CO2 dans une flasque T75
(Falcon) et 15 mL de milieu GMEM complet.
D- Expression de la protéine NS4B
a. Immunofluorescence des cellules BHK-21
transfectées
Vingt heures post transfection, les cellules BHK-21 ont
été trypsinées, lavées au PBS, comptées et
reprises dans du PBS contenant 10% de sérum de veau foetal, à
raison de 2,5.105 cellules/mL. Cent microlitres de cette suspension
cellulaire ont été déposés dans une cupule et
cytocentrifugés sur des lames (Cytospin 3, Shandon) pendant 5 minutes
à 250 g. Après séchage et fixation, les lames ont
été fixées dans un bain d'acétone à
-20°C pendant 10 minutes, les cellules transfectées avec les
constructions pSFV-NS4B, -NS4B/HA et SFV-âgal, ont été
incubées en chambre humide pendant 30 min avec 40 j.iL d'un anticorps
monoclonal de rat anti-HA (Sigma) dilué au 1/250e en PBS. Les cellules
transfectées avec la construction pSFV-C191 ont été
incubées avec un anticorps monoclonal murin anti-capside (C7-50, Abcam)
dilué au 1/200e en PBS. Après trois lavages de 10 min
dans du PBS, les cellules ont été incubées comme
précédemment avec 40 j.iL d'un anticorps secondaire de
chèvre anti-rat ou chèvre anti-souris conjugué à
l'Alexa Fluor 488 (molecular Probes®) dilué au
1/1000e. Après trois nouveaux lavages, les lames ont
été montées en milieu PVA/DABCO (25 mM Tris/HCl 1.5M [pH
8.8],
glycérol 5%, DABCO 2,5%, alcool polyvinylique (PVA) 10%),
puis observées au microscope confocal à balayage (Microscope
Confocal à Balayage Laser, Olympus).
b. Western-blot des lysats des cellules BHK-21
transfectées
Après transfection, les cellules BHK-2 1 ont
été trypsinées, lavées au PBS, et lysées
avec 1 mL de tampon de lyse (Tris/HCl 10 mM, NaCl 140 mM, igépal 1%)
supplémenté avec des inhibiteurs de protéases
(phénylméthylsulfonyl fluoride (PMSF) 1mM, aprotinine et
leupeptine 2 ìg/mL). Vingt microgrammes de protéines contenues
dans les lysats cellulaires ont été précipitées et
soumises à une électrophorèse en gel d'acrylamide 12%
(Euromedex) pendant 1h30 à 20 mA (Mini Protean III, BioRad), suivies
d'un transfert actif (Mini Trans-Blot Cell, BioRad) en tampon
TG/éthanol(Tris 0.25M, Glycine 1 .92M, éthanol 20%) sur une
membrane de nitrocellulose (Hybond-C Extra, Amersham Biosciences) durant 1h30
à 90V. Cette membrane a ensuite été saturée pendant
45 minutes en tampon TBS (Tris-HCl 50 mM [pH8] NaCl 150mM, igépal 0.1%)
contenant 5% de lait en poudre. Ensuite, la membrane a été
incubée en présence d'un anticorps monoclonal de rat anti-HA ou
de souris anti-actine (AC-15, Sigma) dilués au 1/5000e ou
d'un anticorps monoclonal humain anti-capside (B12F8, fourni par le Dr
Mondelli, Université de Rome, Italie) dilué au
1/2000e, pendant un nuit à 4°C. Après 3 lavages
de 10 minutes en tampon TBS, la membrane a été incubée
durant une heure, à température ambiante et sous agitation douce,
avec un anticorps secondaire couplé à la peroxydase (anti-rat,
-souris et -humain, Biosource) dilués au 1/10 000e en tampon
de blocage. Enfin, après 2 lavages de 10 minutes et un lavage de 20
minutes en tampon TBS, la révélation a été
réalisée avec un système de détection
chimioluminescent ECL (Pierce) et le signal a été
transféré sur un film d'autoradiographie (Pierce).
c. Microscopie électronique des cellules BHK-21
transfectées
Un tapis cellulaire d'une flasque T75 de cellules BHK-2 1
transfectées a été directement fixé dans la flasque
par ajout de 5 ml d'un tampon tampon phosphate 0,1 M [pH 7.2] (Sigma) contenant
4% de paraformaldéhyde et 1% de glutaraldéhyde, et les cellules
ont ainsi été incubées pendant 48 heures à
4°C. Après un rinçage en tampon phosphate 0,15 M, les
cellules ont été post- fixées à l'aide de
tétroxyde d'osmium 1% en tampon phosphate 0,3 M pendant 1 heure, avant
d'être déshydratées par trempage dans des bains
d'éthanol de titres croissants. Après un lavage dans l'oxyde de
propylène, les cellules ont été incluses dans un
mélange oxyde de propylènerésine/Epon, et incubées
24 heures à 60°C pour permettre la polymérisation de la
résine. Les coupes ultra-fines de blocs ont été
réalisées à l'aide d'un ultramicrotome (Reichert
Scientific Instruments), et contrastées à l'acétate
d'uranyl et au citrate de plomb, avant d'être observées en
microscopie électronique à transmission (MET) (Jeol 1230,
Jeol).
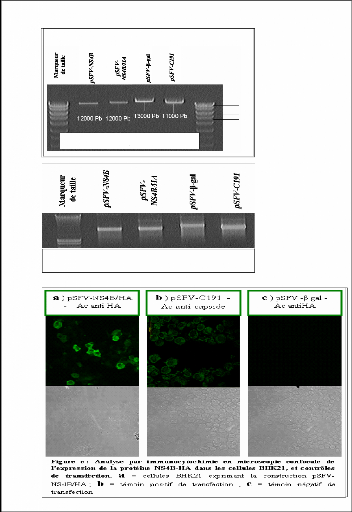
Marqueur de taille
(Pb)
10 000
8 000
6 000
5 000
4 000
Figure a. Migration électrophorétique sur
gel d'agarose (1%BET)
des vecteurs d'expression
linéarisés
Figure b : Migration électrophorétique sur gel
d'agarose 1% (+BET) des vecteurs d'expression transcrits
RESULTATS
L'objectif de ce travail était d'étudier le
rôle potentiel de la protéine NS4B de la souche JFH-1 dans la
formation des remaniements membranaires viro-induits. Cette protéine a
été sortie du contexte de la polyprotéine et a
été exprimée de façon isolée. Deux
constructions, l'une correspondante à la protéine NS4B seule et
l'autre correspondante à cette protéine accompagnée d'une
étiquette HA, ont été exprimées en système
SFV.
A- Linéarisation
La vérification sur gel d'agarose des constructions
après linéarisation par SpeI (Figure a.) mettent en
évidence une bande à 12 000 pb environ pour pSFV-NS4B et
pSFV-NS4B/HA (qui en théorie font respectivement 11 869 pb et 11 893
pb); pour pSFV-â-gal une bande à 13 000 pb (13 078 pb en
théorie) et 11 000 pb pour pSFV-C 191 (11 572 pb). La
linéarisation effectuée avec SpeI a été
optimale puisque non seulement les résultats obtenus sur gel d'agarose
corrèlent avec les résultats théoriques mais surtout parce
nous ne trouvons qu'une seule bande.
B- Transcription in vitro
La transcription a été réalisée
grâce à une polymérase SP6 permettant d'aboutir à la
synthèse d'ARN recombinant. Après transcription in
vitro, les ARN obtenus ont été vérifiés
qualitativement par migration sur gel d'agarose afin de s'affranchir d'une
éventuelle dégradation de ces ARN. Le profil de migration des ARN
a permis de confirmer l'intégrité de ces derniers (figure b).
C- Immunoflorescence
Afin de contrôler l'efficacité de transfection
des cellules BHK-21, l'expression des protéines d'intérêts
a été évaluée par immunocytochimie. Les cellules
exprimant les constructions pSFV-NS4B/HA, pSFV-NS4B et pSFV-âgal ont
été soumises à une réaction immunocytochimique
à l'aide d'un anticorps monoclonal anti-HA. Les cellules exprimant la
construction, pSFV-C191 ont elles été testée à
l'aide d'un anticorps monoclonal anti-capside. La détection de la
protéine de capside du VHC dans les cellules transfectées avec la
construction contrôle pSFV-C 191 (figure c) montre que les transfections
ont été efficaces. La protéine de capside est
présente à un haut niveau dans plus de 70% des cellules. La
réaction d'immunofluorescence avec l'anti-HA a
révélé également un haut niveau d'expression de la
protéine NS4B/HA dans les cellules transfectées par la
construction pSFV-NS4B/HA, avec l'observation d'une fluorescence cytoplasmique
intense (figure c). Cette fluorescence était spécifique puisque
aucun marquage n'a été relevé dans les cellules exprimant
les constructions pSFV-NS4B et pSFV-âgal.
D-
Western Blotting
Après transfection, une partie des cellules BHK-2 1 a
été mise en présence d'un tampon de lyse auquel des
anti-protéases ont été ajoutées. Les lysats
cellulaires ont ensuite été analysés en western-blotting.
Chaque lysat a été soumis à une migration en SDS/PAGE et
transféré sur membrane de nitrocellulose. La présence des
protéines d'intérêts a été mise en
évidence par incubation d'anticorps monoclonal anti-HA, anti-capside ou
anti-actine, suivis d'anticorps secondaires adaptés marqués
à la péroxydase. Les résultats montrent une expression
similaire de l'actine dans toutes les pistes, permettant ainsi de valider
l'analyse (Figure c). En effet, l'actine est une protéine cellulaire
ubiquitaire, et sert de contrôle pour vérifier que tous les puits
ont été chargés en protéines de manière
équitable. La protéine NS4B-HA a été bien
détectée via l'étiquette HA avec une bande à une
taille attendue de 30 kDa, uniquement pour la construction pSFV-NS4B/HA. De
même, la protéine de capside a été
détectée avec une bande à une taille attendue de 21 kDa,
uniquement pour la construction pSFV-C191. Aucune bande n'a été
observée pour les constructions pSFV-âgal et pSFV-NS4B,
puisqu'elles n'expriment ni la protéine de capside ni une
protéine avec une étiquette HA (Figure d). Ceci confirme bien la
spécificité des anticorps utilisés pour ces
réactions.
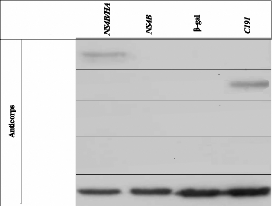
Lysat cellulaire issu des BHK21 Transfectées
par les ARN
Anti-actine
anti-HA
Anti-C191
Anti-E 1
Anti-E2
40 KDa
30 KDa
21 KDa
Figure d : Western Blotting sur les lysats cellulaires
( 20 ng des protéines totales) issues des BHK21 transfectées par
les vecteurs d'expression.
E- Microscopie électronique
Après avoir vérifié l'expression des
protéines, le culot cellulaire de chaque construction pSFV-NS4B,
pSFV-NS4B/HA, PSFV-C191 et pSFV-âgal ont été inclus en
résine et des coupes ultra-fines de blocs ont été
réalisées à l'aide d'un ultramicrotome. Les observations
en microscopie électronique des différentes constructions sont
actuellement en cours d'analyse. En
effet, cette technique d'inclusion en résine et l'analyse
fine qui en découle nécessite environ trois semaines de
travail.
DISCUSSION
L'absence de système de culture in vitro
efficace durant plus de 15 ans a nécessité le
développement d'outils mimant la structure du virions ou la
réplication virale pour étudier les différentes phases du
cycle infectieux du VHC. Différentes équipes se sont notamment
intéressées à développer un modèle de «
réplicon subgénomique » du VHC permettant une
réplication stable d'ARN subgénomique du VHC dans des cellules en
culture (Lohmann et al., 1999 [5]). Ce réplicon, unité minimale
de réplication, constitué par un génome viral où
les protéines structurales sont délétées et
remplacées par un gène de résistance à la
néomycine a été un formidable outil pour l'étude de
la réplication virale et l'évaluation d'inhibiteurs potentiels de
cette réplication (Bartenschlager, J Hepatol 2005 [6]). Cependant, ce
modèle de réplicon ne permet pas de former des particules virales
(puisque les protéines structurales ne sont pas exprimées) et ne
reflète que partiellement la réplication du vrai virus. Ainsi, le
développement du système JFH-1 permettant de
générer de réelles particules virales infectieuses et
réplicatives représente une avancée majeure pour
étudier les étapes clés du cycle infectieux du VHC en
culture cellulaire.
Grâce au modèle réplicon, quelques
études réalisées sur des coupes de cellules
analysées en MET ont suggéré que la réplication du
VHC pouvait induire des remaniements membranaires, qui ont été
appelés membranous web (Egger et al, 2002 [3] &
Gosert et al, 2003 [4]). A l'image de ce qui est connu pour d'autres
virus à ARN positifs (Uchil.P.D et al, 2003 [7]), il a
été suggéré que ces remaniements membranaires
pourraient héberger les complexes de réplication de l'ARN du VHC.
Ces membranous web ont essentiellement été
décrits avec les premiers modèles d'étude de la
réplication du VHC, basés sur des réplicons
subgénomiques d'un génotype particulier 1b. De plus, une
étude a suggéré que la protéine NS4B était
à l'origine de la formation des membranous web dans des
cellules hébergeant un réplicon de type 1b (Egger et al, 2002
[3]). Avec l'avènement du modèle du clone JFH-1 (Wakita et
al, 2005 [2]), il était donc pertinent de s'intéresser plus
particulièrement à l'expression de cette seule protéine
issue d'un génotype 2a. Nous avons réussi à obtenir des
cellules qui expriment la protéine NS4B seule ou avec une
étiquette HA. En absence d'anticorps anti-NS4B disponible, cette
étiquette HA nous a permis de visualiser par immunofluorescence le bon
niveau d'expression de cette protéine dans plus de 70% de cellules
transfectées. Par ailleurs, l'électrophorèse suivie d'une
immuno-empreinte des protéines contenues dans le lysat des cellules
transfectées a montré la présence de la protéine
NS4B grâce à l'étiquette HA à la masse
moléculaire attendue de NS4B. En effet, l'épitope HA
représente 9 acides aminés. Il est largement admis qu'un acide
aminé représente environ 110 kDa
|
ainsi la masse moléculaire de l'épitope HA est
inférieure 1 kDa. Par ailleurs il serait intéressant de
déterminer si l'expression de la protéine NS4B, après
transfection, permet bien la formation de remaniements membranaires au niveau
des membranes du réticulum endoplasmique. En effet, la protéine
NS4B est totalement ancrée dans la membrane du RE, possédant au
moins quatre passages transmembranaires avec des extrémités N- et
C- terminales localisées dans le cytosol (Hugle et al, 2001
[8], Lundin et al, 2003 [9]). Cette topologie particulière peut
dans un contexte de surexpression de cette protéine induire des
structures renflées et vésiculeuses au niveau de la membrane du
RE comme l'a suggéré Egger et al. L'originalité
de ce travail repose sur l'utilisation du vecteur SFV extrêmement
puissant qui nous a déjà permis d'étudier la
morphogenèse de pseudo-virons du VHC (Blanchard et al., 2002 [10]). Les
analyses de microscopie électronique sont actuellement en cours et
devraient nous permettre de comprendre l'implication de cette protéine
sur l'ultrastructure des cellules.
Pour aller plus loin, l'équipe cherche également
à comprendre la morphogenèse du complexe de réplication en
se focalisant sur la purification et l'analyse structurale de ce complexe. Nous
pensons que l'ensemble de ces travaux vont permettre de mieux comprendre
l'interaction entre les différentes protéines non structurales
entre elles ou entre d'autres partenaires cellulaires dans la formation de ces
complexes de réplication. En effet, l'identification de co-facteurs
cellulaires indispensables pour la réplication du VHC pourrait
favoriser, à terme, la conception de nouvelles stratégies
antivirales contre le VHC ou d'autres virus à ARN+ induisant des
remaniements membranaires.
|
BIBLIOGRAPHIE
1. Choo QL, Kuo G, Weiner AJ, Overby LR, Bradley DW, Houghton
M. 1989. Isolation of a cDNA clone derived from a blood-borne non-A, non-B
viral hepatitis genome. Science; 244: 359-62.
2. Wakita T, Pietschmann T, Kato T, et al. 2005.
Production of infectious hepatitis C virus in tissue culture from a cloned
viral genome. Nature Med.; 11 :791-6
3. Egger, D., Wolk, B., Gosert, R., Bianchi, L., Blum, H. E.,
Moradpour, D. & Bienz, K. 2002. Expression of hepatitis C virus proteins
induces distinct membrane alterations including a candidate viral replication
complex. J Virol 76, 5974-5984
4. Gosert, R., Egger, D., Lohmann, V., Bartenschlager, R.,
Blum, H. E., Bienz, K. & Moradpour, D. 2003.
Identification of the hepatitis C virus RNA replication complex in Huh-7 cells
harboring subgenomic replicons. J Virol 77,
5487-5492
5. Lohmann, V., Korner, F., Koch, J., Herian, U., Theilmann,
L. & Bartenschlager, R. 1999. Replication of subgenomic hepatitis C virus
RNAs in a hepatoma cell line. Science 285,
110-113.
6. Bartenschlager R. 2005. The hepatitis C virus replicon
system: from basic research to clinical application. J Hepatol.;
43 :210-6
7. Uchil, P. D., and V. Satchidanandam. 2003. Architecture of
the flaviviral replication complex. Protease, nuclease, and detergents reveal
encasement within double-layered membrane compartments. J. Biol. Chem.
278:24388-24398.
8. Hugle T, Fehrmann F, Bieck E, Kohara M, Krausslich HG,
Rice CM, Blum HE, Moradpour D. 2001. The hepatitis C virus nonstructural
protein 4B is an integral endoplasmic reticulum membrane protein.
Virology; 284(1):70-81.
9. Lundin, M., M. Monne, A. Widell, G. Von Heijne, and M. A.
Persson. 2003. Topology of the membrane-associated hepatitis C
virus protein NS4B. J. Virol. 77:5428-5438.
10. Blanchard E., Brand D., Trassard S., Goudeau A., Roingeard
P. 2002. Hepatitis C virus-like particles morphogenesis.
J. Vi rol. 76 : 4073-4079.
11. Blanchard E, Hourioux C, Brand D, Ait-Goughoulte M,
Moreau A, Trassard S, Sizaret PY, Dubois F, Roingeard P. 2003. Hepatitis C
virus-like particle budding: role of the core protein and importance of its Asp
111. J virol.; 77 :10131-10138.
RESUME
Lors du cycle réplicatif du virus de l'hépatite
C (VHC), les protéines non-structurales (N S) du virus s'organisent en
complexe de réplication à proximité du réticulum
endoplasmique. Cette organisation particulière semble être
responsable d'un réarrangement de membrane en structure
vésiculaire appelé « membranous web ». Des
études antérieures ont suggéré que la
protéine transmembranaire NS4B du VHC pourrait intervenir directement
dans la formation de ces structures membranaires. Toutefois, l'implication de
cette protéine n'a toujours pas été clairement
démontrée.
Dans ce contexte, le travail réalisé dans le
laboratoire d'accueil a consisté à cloner en système
Semliki la séquence codant la protéine non-structurale NS4B issu
d'un clone complet et réplicatif du VHC nommé JFH1. Dans un
premier temps, la production en cellule BHK-21 de la protéine NS4B a
été vérifiée par immunofluorescence et
Western-blot. Des analyses en microscopie électronique à
transmission encore en cours permettront par la suite de caractériser
directement l'implication de la protéine NS4B dans la
morphogenèse de ces remaniements membranaires intracellulaires. Si
l'intervention de la protéine NS4B dans la formation du «
membranous web » devait se confirmer, le développement de
nouvelles stratégies thérapeutiques pourrait être
envisagé.
Mots-clés : hépatite C,
VHC, vecteur SFV, réplication, membranous web
ABSTRACT
During the replicative cycle of the hepatitis C virus (HCV),
non-structural proteins (NS) are organized in a replication complex near the
endoplasmic reticulum. This organization seems to be responsible for a
rearrangement into vesicular membrane structure called «
membranous web ». Previous studies have suggested that
the HCV transmembrane protein NS4B could directly act in the formation of these
particular membrane structures. However, the involvement of this protein has
still not been clearly demonstrated.
In this context, the work undertaken in the host laboratory
has been to clone in a Semliki system the sequence encoding the NS4B
non-structural protein derived from a complete and replicative HCV clone called
JFH1. In a first step, the production of NS4B in BHK-21 cells was verified by
immunofluorescence and Western-blot. Analysis by electron microscopy still
underway will directly characterize the involvement of the NS4B protein in the
intracellular membrane structures morphogenesis. If the involvement of NS4B for
the « membranous web » formation should be confirmed, the
development of new therapeutic strategies could be considered.
Key-words : Hepatitis C, HCV, SFV vector, replication,
membranous web
| 


