|
Université Bordeaux 1
UMR PACEA 5199
Laboratoire d'Anthropologie des Populations du Passé
Mémoire de Master 2 Sciences et Technologies
Mention
Anthropologie Biologique, Paléoanthropologie et
Préhistoire
Spécialité : Anthropologie Biologique
LA NECROPOLE MEROVINGIENNE « LA CHAPELLE » DE
JAU-
DIGNAC ET LOIRAC (GIRONDE) : DETERMINATION DE
LIENS
DE PARENTE PAR APPROCHE PALEOGENETIQUE
ETUDE DE
FAISABILITE
Diane THIBON
Sous la direction de Dominique CASTEX et de Marie-France
DEGUILLOUX.
Année 2008-2009
Remerciements
Je tiens à remercier toutes les personnes sans lesquelles
cette étude n'aurait pu être menée à bien ;
Messieurs les Professeurs J. JAUBERT et P.MURAIL pour m'avoir
permis de suivre les enseignements du Master Anthropologie biologique,
Paléoanthropologie et Préhistoire.
Marie-France DEGUILLOUX et Dominique CASTEX pour m'avoir
dirigée et conseillée dans ce travail ;
Marie-Hélène PEMONGE pour sa gentillesse et sa
très grande participation, sans laquelle ce mémoire n'aurait pu
être réalisé.
Yannick pour sa patiente et son soutien ;
Et enfin, ma famille pour leurs encouragements et leur
réconfort.
Table des matières
Introduction 6
I Présentation du site archéologique de
« La chapelle » 9
1. Environnement du site 9
2. Les quatre phases d'occupation du site 11
2.1. Le temple antique 12
2.2. La nécropole mérovingienne 12
2.3. Habitats de la fin du Haut Moyen-âge 14
2.4. Chapelle et cimetière médiéval
à moderne 15
3. La nécropole mérovingienne 16
3.1. Pratiques funéraires 16

Les tombes à l'intérieur du bâtiment 16
Les tombes de l'extérieur 17
3.2. Le recrutement par âge et par sexe 18
3.3. Le cas particulier des sarcophages du secteur occidental
18
II Matériel et Méthode 22
1. Généralités techniques sur
l'étude de l'ADN ancien 22
2. Matériel 23
2.1. Présentation du matériel 23
2.2. Choix des régions étudiées 24
3. Méthodes 24
3.1. Précautions 25
3.2. Préparations des échantillons 25
3.3. Extraction de l'ADN 26
3.4. Amplification de l'ADN 26
3.5. Vérification de PCR 28
3.6. Clonage 28
3.7. Analyse des séquences 29
3.8. Typage des fouilleurs 29
III Synthèse bibliographique 31
IV Résultats 33
1. Résultats de la synthèse bibliographique 33
2. Résultats des analyses génétiques 39
2.1. Les fouilleurs 39
2.2. Les séquences des individus 39
Sep 169 40
- 1er inhumé : 40
- 2ème inhumé : 41
- 3ème inhumé : 41
Sep 170 42
- 1er inhumé. 42
- 2ème inhumé : 43
- 3ème inhumé : 43
Sep 171 44
- 1er inhumé : 44
- 2ème inhumé : 44
- 3ème inhumé : 45
2.3. Synthèse des résultats 48
V Discussion 49
1. Authenticité des séquences : conservation de
l'ADN et contaminations 49
2. Haplogroupes retrouvés 51
3. Liens de parentés 54
VI Conclusion et perceptives 57
Annexe A : protocoles d'analyses d'ADN ancien 59
Bibliographie 62
Liste des figures :
Figure 1 : Localisation de Jau-Dignac en Gironde (in
Cartron et Castex 2009) 9
Figure 2 : Localisation de Jau-Dignac en Gironde (in
Cartron et Castex 2009) 9
Figure 3 : Vue aérienne de la zone 1 du site
archéologique (in Cartron et Castex2009). 10
Figure 4 : Plan général du site
archéologique (in Cartron et Castex2008). 11
Figure 5 : Plan de la nécropole
mérovingienne (Phase 2) (in Cartron et Castex 2009). 12
Figure 6 : Plan des structures d'habitats (phase 3)
(in Cartron et Castex 2009). 14
Figure 7 : Plan de la chapelle et du cimetière
médiévaux et modernes (phases 4 et 5) (in Cartron et
Castex 2006). 15
Figure 8 : Sépulture 169, 170 et 171 (in
Cartron et Castex 2006). 19
Figure 9 : Schéma d'une amplification d'ADN par
PCR (Vierstraete 1999). 27
Figure 10 : Fréquence actuelle de l'haplogroupe J
en Europe d'après Logan (2009). 52
Figure 11 : Fréquence de l'haplogroupe X en
Europe d'après Logan (2009). 53
Figure 12 : Arbre généalogique montrant
quels individus sont issus d'une même lignée
mitochondriale. 55
Figure 13 : Informations acquises en termes de relations
de parenté maternelles. 56
Liste des tableaux :
Tableau 1 : Détail des individus inhumés
dans les sarcophages. 20
Tableau 2 : Détail des dents utilisées
lors de l'extraction 24
Tableau 3 : Amorces utilisées pour chaque
fragment (Jehaes et al. 2001). 28
Tableau 4 : Synthèse bibliographique.
Critères techniques et résultats. 36
Tableau 5 : Synthèse bibliographique des
résultats d'études paléogénétique de liens
de parenté. 38
Tableau 6 : Mutations présentes sur la
région HVS 1 des fouilleurs. 39
Tableau 7 : Clones obtenus pour chaque fragment et
individu. 39
Tableau 8 : Détail des mutations présentes
sur tous les clones obtenus. 47
Tableau 9 : Mutations authentiques présentes sur
chaque fragment des individus. 48
Introduction
Durant l'antiquité, on observe en Occident une
séparation stricte entre le monde des vivants et celui des morts. Selon
une tradition romaine, il était interdit d'inhumer les défunts
intra muros ainsi qu'au sein des sanctuaires. Du point de vue des
pratiques funéraires, on observe à cette époque une
gestion quasi exclusive des funérailles par la famille. Les
défunts étaient ainsi inhumés aux cotés de leurs
ancêtres, comme on peut le voir encore aujourd'hui (Treffort 1996).
C'est durant la période du Haut-Moyen-Age que des
changements progressifs vont s'effectuer sur plusieurs niveaux. Ceux-ci sont en
grande partie dus à l'installation du christianisme en Gaule. Durant
cette période transitoire, entre Antiquité et Moyen-Age, on
observe alors des remaniements s'appliquant aux espaces ainsi qu'aux pratiques
funéraires. La tradition romaine de séparation se perd peu
à peu et l'on assiste à des regroupements de tombes autour de
lieux sacrés, prémices du cimetière chrétien
retrouvé quelques siècles plus tard. Peu après, on assiste
au passage d'une gestion familiale antique des défunts à une
gestion plus communautaire menée par l'église (Treffort 1996).
Les défunts chrétiens ne sont donc plus
préférentiellement inhumés aux cotés de leurs
ancêtres mais au sein d'un lieu funéraire regroupant une autre
grande famille : la communauté chrétienne.
Le sujet de ce mémoire concerne le site
archéologique de Jau-Dignac et Loirac, situé en Gironde,
fouillé depuis 2001 par I. Cartron (MC Ausonius, Bordeaux3) et D. Castex
(CR, UMR PACEA-LAPP). Ce site est marqué par plusieurs occupations
successives dont une nécropole mérovingienne, présente
à l'intérieur et aux alentours d'un édifice religieux.
Fréquentée du Ve au VIIIe siècle, elle s'inscrit donc
parfaitement dans la période de transition des pratiques
funéraires énoncées précédemment.
Elle se compose majoritairement de sépultures
(sarcophage et autres structures funéraires) primaires individuelles
mais quelques sépultures plurielles sont également
retrouvées et préférentiellement dans le secteur ouest du
site.
Ces sarcophages regroupant 2 à 4 individus posent la
question inévitable des liens entre les inhumés. S'agit-il de
regroupements familiaux ? Ou bien le regroupement était-il un moyen
simple et rapide d'acquérir de la place au sein de la nécropole ?
Peut-être doit-on aussi y voir une volonté de rapprochement d'un
lieu de culte.
Comme nous l'avons vu précédemment, la
composante familiale tend à disparaître au Moyen-Age au profit de
l'aspect communautaire. Mais, toutefois celle-ci semble perdurer dans les
pratiques funéraires du Haut Moyen-Age (Cartron 2009). C'est donc ce
caractère familial que nous souhaitons tester sur plusieurs individus
retrouvés groupés au sein des sarcophages de la nécropole
de Jau-Dignac. Précédemment, une étude visant à
caractériser et comparer des populations du site par l'apport de
variations non métriques dentaires a été
réalisée (L aforest 2008). Malheureusement aucune information n'a
pu être apportée en termes de relation de parenté entre ces
individus.
Le mémoire présenté ici s'inscrit dans le
cadre d'une étude paléogénétique de liens de
parenté. Le principe général de cette étude est
d'analyser l'ADN d'individus afin d'observer si ceux-ci présentent des
séquences identiques et partagent donc des liens biologiques. Lorsque
l'ADN est obtenu, ces analyses paléogénétiques apportent
des informations beaucoup plus fiables et plus précises que l'analyse
des caractères discrets. On peut ainsi parfois avoir une idée du
type de lien (mère-enfant, frère-soeur etc....) qui unit deux ou
plusieurs individus, ce qui est une information très intéressante
pour la compréhension des pratiques funéraires (Cappellini et
al. 2004 ; Haak et al. 2008, etc).
On observe depuis quelques années une multiplication de
ce type d'étude. Néanmoins, ces analyses sont compliquées
et limitées car elles se heurtent à plusieurs obstacles
importants qui sont entre autres : la conservation de l'ADN et la
détection de contaminations. Le nombre d'études
paléogénétique de liens de parenté publiées
est à ce jour assez réduit et la plupart de ces articles ne
mentionnent pas toutes les précautions connues aujourd'hui comme
nécessaires à l'obtention de résultats fiables. Les
résultats sont d'ailleurs généralement fragmentaires, une
grande majorité de ces études ne permettant pas d'obtenir d'ADN
nucléaire et devant se cantonner à l'ADN mitochondrial.
Nous avons jugé opportun de faire une brève
synthèse de travaux publiés pour ce type d'approche (neuf
articles), afin de faire le point sur leurs potentialités et leurs
limites.
L'ADN mitochondrial, ADN seulement transmissible par voie
maternelle, est d'ailleurs le premier marqueur moléculaire
utilisé dans ce type d'étude. Il est en effet plus abondant,
mieux conservé et donc plus facile à obtenir. Il va en quelques
sortes, donner la « tonalité » d'une étude
paléogénétique. De mauvais résultats obtenus
à partir de cette molécule circulaire ne présage rien de
bon concernant l'ADN nucléaire.
L'ADN mitochondrial sera l'unique marqueur analysé dans
le cadre de ce mémoire car le sujet de celui-ci ne concerne
véritablement que la première étape de cette étude
de liens de
parenté, à savoir la faisabilité. Il
s'agit donc en premier lieu d'évaluer la présence ou non d'ADN
analysable et authentique. Cette étude de faisabilité sera
réalisée sur un groupe de neuf individus regroupés par
trois au sein de trois sarcophages.
Ce mémoire pourra également apporter des
informations méthodologiques. Les analyses présentent-elle
beaucoup de contaminations ? D'où proviennent-elles ? Les
prélèvements d'échantillons n'ont pas toujours
été effectués dans les mêmes conditions ni avec les
même précautions. Observe-t-on des différences en termes de
contaminations entre les différents prélèvements ? Peut-on
identifier des gestes contaminants ?
Si nous parvenons à obtenir de l'ADN mitochondrial
authentique chez plusieurs de ces individus, il sera alors possible d'apporter
dans ce mémoire des réponses en terme de relations de
parenté maternelle. De plus, si cette étude de faisabilité
s'avère fructueuse, d'autres analyses plus poussées portant sur
l'ADN nucléaire (STR, Chromosome Y) pourront être
envisagées afin de repérer d'éventuels liens de
parenté paternels et de type parent / enfant.
I Présentation du site archéologique de
« La chapelle »
Les données suivantes sont extraites des rapports de
fouille (I .Cartron et D .Castex 2006).
1. Environnement du site
Le site archéologique de « La Chapelle » se
situe sur la commune de Jau-Dignac et Loirac, sur la rive gauche de l'estuaire
de la Gironde et à environ 950 m des berges du fleuve (fig. 1). La rive
gauche de l'estuaire se caractérise par une alternance de terrasses
argilograveleuses sur lesquelles sont implantés des vignobles et de
vastes zones marécageuses comblés par des sédiments
détritiques apportés par l'estuaire. Le site archéologique
apparait sur un ancien îlot de l'estuaire : le « hameau de
Goulée » (fig.2). Les vestiges mis au jour attestent d'une
occupation du site depuis l'antiquité, ce qui témoigne d'une mise
hors de l'eau de cette partie de l'îlot depuis cette période.
Certains documents historiques attestent que dans les années 1630, avant
l'assèchement des marais, le rivage de l'estuaire devait être
très proche du site archéologique.
Figure 2 : Localisation de Jau-Dignac en Figure
1 : Localisation et environnement du site
Gironde (in Cartron et Castex
2009). de Jau-Dignac (in Cartron et Castex
2006).
Ce site archéologique a été
découvert en 2000 à la suite de travaux agricoles. Durant
l'été 2000, une opération de sauvetage permettant
d'estimer le potentiel archéologique du terrain a été
menée par une équipe de l'INRAP sous la responsabilité de
C. Scuiller. Cette intervention, comprenant une série de sondages et
tranchées, s'est réalisée sur une superficie de 6840
m2 environ. Un décapage de surface, mené aux alentours
d'un sarcophage mis au jour à la
découverte du site, a permis de reconnaître
l'emprise d'un bâtiment funéraire. Par la suite, d'autres
tranchées ont été ouvertes dans plusieurs directions afin
de mesurer l'étendue du site. Deux zones sur le site ont ainsi pu
être caractérisées :
La zone 1, située sur une butte, compose la majeure
partie du site, c'est l'endroit où la grande majorité des
vestiges a été retrouvée (fig.3). Cette zone semble avoir
été occupée de manière assez importante depuis le
Ier siècle après J.-C et l'on peut y observer 4 grandes phases
d'occupation attestées, allant de l'Antiquité jusqu'à nos
jours.

Figure 3 : Vue aérienne de la zone 1 du
site archéologique (in Cartron et
Castex2009).
La zone 2, située au sud de la première, semble
être plus instable et n'a livré que peu de vestiges,
essentiellement quelques tombes laissant penser à un ensemble
funéraire secondaire. Contrairement à la zone 1, plusieurs
indices archéologiques suggèrent que cette zone fut
régulièrement inondée. La zone 2 et le site semblent se
limiter au sud par la présence d'un fossé d'une cinquantaine de
mètre de long qui devait être un drain ou bien pouvait accueillir
une palissade. Au delà, de cette limite sud, la terre semble
stérile.
En 2001, une campagne de fouille dirigée par I. Cartron
et D. Castex a permis de mettre au jour une surface de 300m2
correspondant à l'emprise d'une chapelle (Zone 1). La fouille de ce
site, prolongée de 2003 à 2005 sous forme de d'opération
programmée triennale et de 2007 à 2009, fait l'objet d'un
chantier école, permettant ainsi à de nombreux étudiants
de venir chaque année acquérir des techniques de fouilles propres
à l'archéologie et à l'anthropologie.
La problématique de se site s'insère dans un
contexte de compréhension de genèse des espaces funéraires
et des lieux de cultes pour l'Antiquité tardive et le Haut Moyen-Age.
La
fouille relève de méthodes propres au domaine de
l'archéothanatologie, discipline dont l'objectif est de reconstituer les
modes de dépôts des défunts et les pratiques
funéraires qui en découlent. Il s'agit donc d'une fouille fine
avec des prélèvements et des enregistrements précis des
ossements et du mobilier (Duday 2005). Ce site possède l'avantage
d'avoir été occupé sur une très longue durée
(de l'Antiquité à l'époque moderne), ce qui permet donc de
pouvoir être attentif aux questions de transition, de changement de
fonction du site (habitat, lieu de culte, espace funéraire, passage d'un
espace privé à public ou l'inverse) en même temps
qu'à sa chronologie (continuité d'occupations, ruptures) (Cartron
et Castex 2007). La compréhension de l'environnement naturel et de son
évolution au cours du temps revêt également une grande
importance dans l'étude de ce site puisqu'il permettra de comprendre
assurément la longue occupation d'un ilot des rives de l'estuaire.
L'estuaire est depuis très longtemps une voie fluviatile de
pénétration et d'échanges primordiaux en Aquitaine, ce qui
pourrait donc être une piste pour expliquer cette très longue
occupation.
Nous allons maintenant résumer brièvement les
quatre grandes phases d'occupation de ce site (fig.4)
2. Les quatre phases d'occupation du site
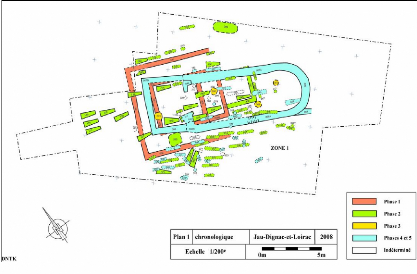
Figure 4 : Plan général du site
archéologique (in Cartron et Castex2008).
2.1. Le temple antique
La phase d'occupation la plus ancienne du site se manifeste
par la présence d'un fanum. Ce petit temple antique, constitué
d'une cella carré, d'une galerie périphérique et d'un
pronaos, aurait été utilisé autour du Ier siècle
après J.-C, puis abandonné à la fin du IVe siècle
(fig. 4). Les pièces de monnaie retrouvées au sein du temple,
permettraient de dater certains niveaux d'occupation du temple mais
malheureusement la période de sa fondation reste obscure. De plus,
malgré la découverte de nombreux vestiges au sein de cet
édifice (différents types de monnaies, nombreuses clochettes en
bronze) aucun culte lié au fanum n'a pu être identifié.
2.2. La nécropole mérovingienne
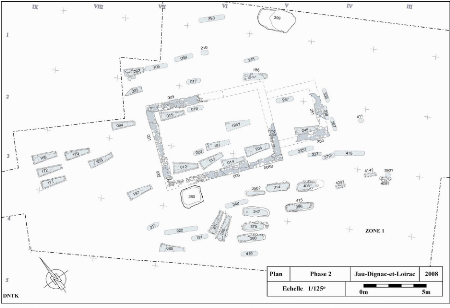
Figure 5 : Plan de la nécropole
mérovingienne (Phase 2) (in Cartron et Castex
2009).
Après un hiatus d'occupation pour les Ve et VIe
siècles, le temple et ces alentours sont transformés en espace
funéraire (fig.5). Les ruines du temple sont aménagées
pour servir de bâtiment funéraire autour duquel une petite
nécropole s'est développée au fil du temps. A ce jour, une
cinquantaine de tombes ont été découvertes. Cet espace
funéraire se compose de
plusieurs regroupements plus ou moins évidents dont les
sépultures ont -pour une grande partie- la caractéristique
commune d'être alignées selon un même axe nord- est/sud-
ouest.
Le groupe le plus apparent est retrouvé au sein
même du bâtiment funéraire réaménagé
sur l'ancienne cella. Ce bâtiment, une église semblerait-il,
rassemble un groupe de 11 sépultures plus ou moins alignées
contre les parois nord et sud de l'édifice dans un sens
nord-est/sudouest. L'alignement de ces sépultures suggère un
fonctionnement de circulation au sein de l'église selon un axe ouest/est
avec une ouverture principale à l'ouest. On compte parmi ces
sépultures six sarcophages ainsi que deux coffrages de bois.
Le deuxième groupe de sépulture marqué
est situé dans la partie occidentale de la nécropole. Il semble
presque en décalage avec les autres groupes car les sépultures
(uniquement des sarcophages) semblent moins nombreuses et mieux
organisées. Ce petit ensemble funéraire se compose de sept
sarcophages dont cinq sont regroupés distinctement. Parmi ces cinq
tombes, trois d'entre elles forment une rangée parfaite. Toujours
à l'Ouest mais plus proche de l'église, les deux derniers
sarcophages ont causés l'arasement du mur Ouest des anciennes galeries
du fanum. Le même phénomène est observé au niveau de
la galerie sud ou deux sépultures se sont installées sur le mur
et à l'intérieur de la galerie.
Dans cette même région sud, une quinzaine de
sépultures sont regroupées de manière
désordonnée. Ces tombes, beaucoup de coffrage de bois et quelques
sarcophages, sont toujours orientées sommairement dans la direction
nord-est/sud-ouest, hormis deux sarcophages dont l'orientation est radicalement
différente (sud-ouest/nord-est). On observe des calages en pierre autour
de la majorité des fosses situées dans ce secteur. Une
étude plus précise des ces tombes est en cours.
La partie nord comprend globalement 9 sépultures, dont
3 sont des sarcophages presque détruits. Sept d'entre elles semblent
disposées parallèlement aux parois intérieure et
extérieure de la galerie du fanum.
La zone la plus à l'Est est un peu plus confuse et se
compose approximativement de 12 sépultures ne comprenant aucun
sarcophage. Trois fosses sont disposées parallèlement à la
paroi de l'édifice et sont donc orientées dans une direction
nord/sud, orientation perpendiculaire à la majorité des autres
tombes. Pour finir, quatre petites fosses entourées de calage de pierre
ont été trouvées plus ou moins regroupées à
l'extrême Est de la nécropole.
A l'issue de cette description, nous pouvons remarquer qu'il
existe donc une certaine hétérogénéité non
seulement dans la disposition des sépultures au sein des pôles et
entre pôles
mais également dans la nature même des ces tombes
mérovingienne (sarcophages, contenants en bois avec ou sans calage de
pierre, avec ou sans aménagement particulier).
Selon les responsables de la fouille et des études, cet
espace funéraire privé, de type oratorium, aurait
constitué un lieu de mémoire familiale à la fin de
l'époque mérovingienne. L'utilisation de cet oratorium
se serait achevée autour du XIe siècle, période à
laquelle cette petite église fut détruite.
2.3. Habitats de la fin du Haut
Moyen-âge
A la nécropole mérovingienne succède une
occupation qui suggère la présence de petits habitats, de type
cabane, dont subsistent surtout des structures en creux (fosses, silos, trous
de poteaux) (fig.6). Ces structures laissent penser que l'église aurait
été totalement détruite et de nombreuses cuves de
sarcophage auraient également été arasées sans
doute pour la récupération des matériaux. Ces destructions
forment une bande ouest-est sur laquelle se concentrent les vestiges
d'habitats. Iles concernent les restes d'une première cabane
située à l'angle sud ouest, formé par les anciens murs du
fanum ainsi que d'autres éléments (silo, structure en tuiles)
dans la zone est. Plusieurs petites fosses et trous de poteau sont
également observés de manière assez éparse sur le
site. On peut dénombrer aujourd'hui quatre petits habitats
dispersés. Malheureusement, il n'a pas été possible de
dater précisément ces vestiges en raison du peu de
matériel retrouvé. Il a été proposé une
large fourchette chronologique comprenant une période s'étalant
du IXe au XIe siècle.
Figure 6 : Plan des structures d'habitats (phase
3) (in Cartron et Castex 2009).
2.4. Chapelle et cimetière médiéval
à moderne
C'est entre le XIIIe et XVe siècle, et fort
probablement par hasard, que le site est de nouveau utilisé. Une
chapelle est alors construite à peu près au même
emplacement que les précédents bâtiments (fig. 7). Ce
dernier type d'occupation est assez mal représenté sur le site
car il se compose de niveaux presque totalement arasés par les labours
successifs. De plus, la totalité des matériaux de construction de
la chapelle ont été récupérés, y compris les
fondations, ce qui explique également que seuls les négatifs du
bâtiment aient été mis à jour. Les quelques
informations retirées de ces négatifs indique une chapelle
à nef unique avec une abside semi circulaire et deux piliers à
l'entrée. Par la suite, un porche fut adjoint à la partie
occidentale de la chapelle. Les sources écrites nous apprennent que cet
édifice fut fréquenté et maintenu en bon état
jusqu'en 1737 puis laissé à l'abandon dans les années
1780. La chapelle fut fermée en 1787 puis vendue en 1792.
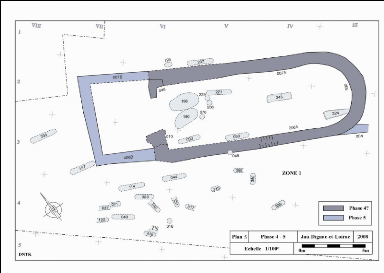
Figure 7 : Plan de la chapelle et du
cimetière médiévaux et modernes (phases 4 et 5)
(in Cartron et Castex 2006).
Autour et à l'intérieur de cette chapelle, de
nombreuses sépultures ont été mises à jour.
Ce cimetière, en grande partie localisé sur la zone
1 (32 sépultures) mais aussi sur la zone 2 (7 sépultures), fut
utilisé du bas Moyen-Age à l'époque moderne.
Malheureusement, pour ce
dernier stade d'occupation, il a été difficile de
bien séparer chronologiquement les vestiges médiévaux
(phase 4) des vestiges modernes (phase5).
3. La nécropole mérovingienne
L'organisation, la disposition spatiale des sépultures
et des différents pôles de cette petite nécropole ont
été en partie détaillée dans la première
partie du mémoire, nous nous attarderons donc ici plus
particulièrement sur les pratiques funéraires mises en oeuvres au
sein de celle-ci.
3.1. Pratiques funéraires
Le groupe mérovingien présent au sein de la
nécropole peut sommairement être scindé en deux : les
sépultures présentes à l'intérieur du
bâtiment et toutes celles à l'extérieur. Ces deux zones
funéraires se distinguent sur plusieurs aspects :
- - le type de sarcophage utilisé. Cette
différenciation se base sur la morphologie
des cuves, le matériau utilisé, ainsi que la
technique de taille des sarcophages.
- la présence ou non de mobilier.
- le nombre et le mode de dépôts des
défunts.
|
/ Hs Wo& bHs à ieli2WéaiHua du
bliWi& H2W
|
L'intérieur de l'édifice abrite exactement 11
tombes dont une grande majorité est représentée par des
sarcophages. Fabriqués en calcaire coquiller, ces sarcophages
trapézoïdaux qualifiés de type 1 ou de « Bordeaux
» (Cartron et Castex 2006), se divisent en deux types de décors.
Dans la majorité des cuves, un riche mobilier comprenant plaques boucle,
boucles de chausse et autre est associé au défunt. Selon les
auteurs, les individus enterrés dans ce bâtiment rectangulaire
constitueraient un groupe sélectif d'inhumations plus prestigieuses. En
témoigne en particulier un sarcophage (sep 014), contenant une femme
d'âge entre 20 et 29 ans qui se distingue par la présence d'une
bague en or massif datant du IIIe --IVe siècle. Cet espace
funéraire constituerait une petite nécropole aristocratique
familiale du haut Moyen Âge (Cartron et Castex 2009). Le bâtiment
pourrait correspondre plus particulièrement à une
mémoria, c'est-à-dire à un monument
funéraire en relation avec la mémoire d'un groupe d'individus.
Toutefois, il semble difficile d'y attester un culte chrétien (Cartron
et Castex 2006).
Concernant le nombre et le mode de dépôt des
défunts au sein de ces sarcophages ; seul
deux d'entre eux semblent avoir fait l'objet de
dépôts primaires individuels (sep 013 et 014) perturbés
postérieurement. Les autres sarcophages de l'édifice, semblent
quant à eux avoir fait l'objet de réutilisations avec
superposition d'un seul individu associé à quelques manipulations
d'ossements pratiquées sur le 1 er inhumé. Notons que
le mauvais état général de certains individus
retrouvés au sein de ces sépultures réutilisées,
n'a pas permis de diagnose sexuelle et d'estimation fiable de l'âge. En
conséquence, aucun regroupement fondé sur ces critères n'a
été mis en évidence.
La mémoria, se compose majoritairement
d'adultes, mais abrite néanmoins deux sépultures individuelles
d'immature. Contrairement aux adultes, aucun mobilier n'est associé
a ces enfants, inhumés dans ce qui pourrait être un
contenant en bois.
|
/ IKINP Hifielieff
té1IIA1
|
La nécropole entourant l'édifice comporte une
quarantaine de sépultures. Ces tombes, plus ou moins regroupées
en pôle aux quatre points cardinaux de l'église, semblent se
démarquer de celles retrouvées pour la même période
à l'intérieur du bâtiment. Ces sépultures, des
sarcophages mais aussi des coffrages en bois et des cercueils, ne contiennent
que très
rarement du mobilier. L'unique exception étant un
sarcophage isolé situé à l'Est du bâtiment
ou une fibule a été mis au jour au coté
du défunt. Cet unique mobilier ainsi que les datations C14 sur les
ossements permettent de dater l'installation de ces tombes au milieu du VIIe
siècle (Cartron et Castex 2006).
Au sein de cet ensemble, un nouveau type de sarcophage,
qualifié de type deux est retrouvé dans la partie ouest. Dans ce
secteur, les sarcophages sont taillés dans un calcaire blanc et fin et
semblent former un groupe assez homogène et différent de ceux du
type 1. Au sud ainsi qu'au nord du bâtiment, quelques sarcophages sont en
calcaire coquiller mais il semblerait que seul deux d'entre eux (sep 092 et sep
093) soit assimilable à ceux de la mémoria, de par le
matériau et la technique de taille utilisée. Les sarcophages au
sud et à l'est de la nécropole n'ont pas pu être
rapprochés d'un des deux types car mal conservés, ou encore non
fouillés. Certains d'entres eux pourraient également appartenir
à un troisième type. Les
sarcophages de la zone extérieure ont, pour une grande
majorité, été réutilisés. Parmi eux, on
observe des cas de superpositions avec quelques manipulations
d'ossements (sep 092, 094 et
169), des cas de superpositions multiples (jusqu'à 4
individus) accompagnés de réductions
pour les
sépultures du secteur ouest (sep 170, 171, 172) et quelques
regroupements
d'ossements issus de gestes indéterminés. Les 17
autres sépultures en contenant en bois ne semblent pas avoir fait
l'objet de réutilisation (Gleize 2006).
La position des individus lors de leur dépôt a pu
être en grande partie reconstituée. Il semblerait que la
majorité des individus, à l'intérieur comme à
l'extérieur de l'édifice, ait été couchée
sur le dos, les membres inférieurs en extension. Dans les cas, ou elle a
pu être déterminée, la position des avants bras est basse,
le long du corps ou ramenée vers le bassin ou l'abdomen. Quelques
exceptions confirment néanmoins la règle ; certains jeunes
enfants peuvent être retrouvés en position foetale, comme dans le
cas des sépultures 210 et 170, mais il serait cependant imprudent de
rapprocher ces deux cas. L'individu de la sépulture 217 se
démarque également par la position de ces pieds, semble t-il
croisés.
Dans plusieurs cas, les observations taphonomiques ont permis
d'émettre l'hypothèse de l'existence de brancard (sep 122) ainsi
que des supports en matière périssable sous la tête (sep
191).
La plupart des jeunes enfants sont retrouvés en
dépôt primaire individuel dans des contenants en bois, ainsi que
nous avons pu l'observer à l'intérieur de l'édifice (sep
210, 243 et 217). Cependant, seul l'un d'entre eux, un périnatal
retrouvé dans le secteur ouest (sep 170), semble
bénéficier d'un sarcophage, fait rare à cette
époque.
3.2. Le recrutement par âge et par sexe
La nécropole extérieure, comme
intérieure, se compose majoritairement d'adultes et, comparé
à une population archaïque, il existe un déficit
général pour les individus de moins de quinze ans. La prise en
compte des taux de mortalité montre que cette différence est du
à un biais important chez les immatures de moins de dix ans (Cartron et
Castex 2006). Il est par ailleurs important de noter que lors de la post
fouille de 2005, il a été mis en évidence dans les niveaux
archéologiques de cette zone de nombreux ossements erratiques,
généralement immatures. Il est bien entendu difficile de savoir
si ces sépultures se rattachent à cette période mais de ce
fait, il faut donc considérer cette population mérovingienne
comme relativement biaisée.
3.3. Le cas particulier des sarcophages du secteur
occidental
Comme nous l'avons vu précédemment, l'ensemble
des sarcophages situés dans le secteur occidental de la chapelle forme
un groupe relativement défini et homogène au sein de la
nécropole extérieure. En effet, cet ensemble de sept sarcophages
taillés dans un même type de
calcaire se démarque des autres sur certains aspects.
Outre le type de sarcophage, ces sépultures ont en majorité
été réutilisées de multiples fois, allant de deux
à quatre fois, ce qui est une pratique spécifique à cette
zone. A côté de cela, le groupe est topographiquement bien
identifiable du fait que quatre des sept sépultures sont très
resserrées et alignées de façon organisée. De plus
la position du groupe dans la nécropole, face à l'édifice,
et la présence d'une agrafe tardive pourraient suggérer une
datation postérieure aux inhumations réalisées dans le
bâtiment. L'élément le plus notable reste encore le fait
qu'un de ces sarcophages renferme un périnatal, fait qui soulève
un certain nombre de questions. Quelle est la raison de la présence de
cet individu au sein de ces sarcophages ? Est-il apparenté à l'un
des individus inhumé à ses côtés ?

Figure 8 : Sépulture 169, 170 et 171
(in Cartron et Castex 2006).
Des prélèvements de mandibule, en vue d'analyse
génétique, ont été effectués sur chacun des
13 individus des sépultures 169, 170, 171 et 172 (La figure 8
présente uniquement les sépultures des individus analysés
dans le cadre de cette étude). Ces prélèvements
présentent un double intérêt. Ils pourraient tout d'abord
permettre la compréhension du fonctionnement de ces sarcophages, groupe
archéologiquement pertinent, ainsi qu'une discussion sur un
éventuel lien de parenté entre les individus d'une même
structure (Castex et Cartron 2007). Ils
pourraient dans un second temps apporter des informations
d'ordre méthodologiques. Ces prélèvements ont
été effectués avec plus ou moins de précautions
suivant les sépultures. Il s'agirait donc d'observer si ces
différences ont eu des répercutions sur les analyses
génétiques.
Pour ces quatre sépultures, il convient de noter que les
couvercles des sarcophages étaient absents mais les cuves, bien
conservées, présentaient un remplissage homogène.
Le tableau ci-dessous (tabl .1) détaille l'âge et
le sexe des individus inhumés dans chaque sarcophage. Seuls les
sarcophages analysés en lors de notre étude
paléogénétique sont présentés dans ce
tableau.
Tableau 1 : Détail des individus
inhumés dans les sarcophages.
La sépulture 169, accueillait 3 individus dont les
dépôts superposés et différés dans le temps,
n'ont entrainé que très peu de manipulations des premiers
inhumés.
La sépulture 170 est un peu plus complexe. Lors de la
réutilisation de la cuve, le squelette du 1er inhumé a
été en partie réduit et une grande majorité de ces
os ont été retirés puis redéposés sur le
deuxième inhumé. La position décentrée de la jeune
femme ainsi que l'enchevêtrement de ces os avec ceux du périnatal
laissent penser à un éventuel dépôt simultané
de ces deux corps. Une relation mère-enfant unirait-elle ces deux
derniers inhumés ? Cette information pourra peut-être être
apportée par des analyses génétiques. Par ailleurs,
l'hypothèse d'un support en matière périssable type
brancard ou civière a été émise face à la
présence d'un espace vide situé sous la jeune femme. Pour finir,
signalons également la découverte d'une agrafe à double
crochet dans le remplissage de la cuve qui renvoie à la période
du mérovingien récent 3 (entre 660 et 750 ap. J.-C).
La sépulture 171 a fait l'objet de plus de
remaniements. En effet, les deux premiers inhumés, au départ
superposés, ont subi plusieurs manipulations avant le dépôt
du 3ème individu. Ainsi, certains de leurs ossements auraient
été sortis puis redéposés dans les angles
du sarcophage, sans doute de manière à
ménager le plus d'espace pour la superposition du dernier
inhumé.
D'un point de vue paléopathologique, aucune
lésion importante n'a été décelée chez ces
treize individus. Bien au contraire, on note une absence d'hypoplasie
linéaire chez six d'entre eux alors que cette pathologie de
l'émail dentaire est fréquente à 68 % (12/ 19) dans
l'effectif de la population de la nécropole (Gleize 2006).
La recherche de caractères discrets osseux s'est
montrée infructueuse (Cartron et Castex 2006 ; Gleize 2006). Une
étude menée sur les caractères discrets dentaires des
individus de la nécropole n'a malheureusement pas permis d'apporter
d'information intéressante laissant penser à un quelconque lien
de parenté entre certains de ces individus (Laforest 2008). La
présence d'un même caractère est cependant observé
(entocunulide sur une M3 inférieur) sur deux individus du sarcophage
172.
II Matériel et Méthode
1. Généralités techniques sur
l'étude de l'ADN ancien
Depuis une vingtaine d'année, la
paléogénétique a permis d'apporter de nombreuses
réponses aux anthropologues et paléoanthropologues. Ainsi, elle
peut permettre aujourd'hui : l'identification d'individus, de relations de
parenté, mais aussi la confirmation des origines et des migrations
humaines ou encore la reconstruction des phylogénies d'espèces
éteintes. Malheureusement, les études sur l'ADN ancien sont
limitées par plusieurs facteurs importants.
La dégradation de la molécule d'ADN est le
premier obstacle aux analyses génétiques. En effet, l'ADN est
dégradé, de manière plus ou moins importante suivant les
conditions de conservation, par plusieurs mécanismes chimiques et
biologiques comme l'autolyse, l'hydrolyse, l'oxydation ou encore la lyse par
des microorganismes. L'ADN récupéré se trouve bien souvent
fragmenté (taille < 250pb) et chimiquement modifié
(présence de déaminations).
Le deuxième obstacle rencontré est la
présence d'inhibiteurs de PCR. En effet, des impuretés,
présentes dans les extraits, peuvent venir inhiber la réaction
d'amplification de l'ADN.
Pour finir, le troisième obstacle à ces analyses
est le risque important de contamination par de l'ADN exogène moderne.
Il existe en effet plusieurs sources possibles de contaminations. Celles-ci
peuvent provenir des fouilleurs, des manipulateurs, des produits
utilisés pour les analyses ou encore des analyses antérieures
(séquences précédemment amplifiées au laboratoire)
(Yang 2005 ; Cooper 2000).
Même si différentes précautions sont
prises (clonage, multiples contrôles), afin de repérer ces
contaminations, il apparaît souvent difficile d'isoler et d'identifier la
séquence authentique, surtout quand il s'agit d'ADN humain. Afin
d'éviter au maximum ce phénomène de contamination, il
convient donc de prendre des précautions et de suivre rigoureusement les
protocoles appropriés.
Les analyses sur l'ADN ancien visant, entre autres,
l'étude de lien de parenté ou de migration de population, se
concentrent en premier lieu sur l'ADN mitochondrial. Cette
molécule circulaire de petite taille (16569 Pb chez
l'homme - Anderson 1981), située dans les mitochondries, présente
plusieurs caractéristiques intéressantes pour ce genre de
problématique. La première est que cette molécule est
présente en grande quantité au sein de la cellule vivante,
environ plus de mille copies par cellule, alors que l'ADN nucléaire
n'est présent que par deux copies. Cela facilite donc son obtention sur
des vestiges anciens. De plus, grâce à ça taille
réduite, cette molécule circulaire à pu être
entièrement séquencée (séquence de
référence de Cambridge) et l'on y observe la présence
d'une région non codante (D-loop) hypervariable dont le taux de mutation
est particulièrement élevé. Ce dernier aspect permet ainsi
de pouvoir reconstituer l'histoire évolutive ainsi que les mouvements
migratoires sur un laps de temps de quelques milliers d'années. Le
dernier élément important à noter est que cette
molécule est transmise uniquement par voie maternelle et ne subit pas de
recombinaison, ce qui bien entendu simplifie grandement les études
phylogénétiques. Néanmoins, l'héritage
génétique n'étant que maternel, seulement la moitié
de l'histoire évolutive et des liens de parenté pourra être
reconstruite.
2. Matériel
2.1. Présentation du matériel
Le matériel osseux utilisé pour cette
étude est constitué des 9 mandibules d'individus retrouvés
dans les sarcophages 169, 170 et 171 de la nécropole. Ceux-ci ont
été prélevés lors de la campagne de fouille
menée par D. Castex et I. Cartron en 2005. Le choix du matériel
se justifie essentiellement par le fait qu'il a été montré
que les dents en place et intactes présentent un taux de conservation de
l'ADN plus important que les os (Shultes 1999). Des dents ont donc
été prélevées chez 8 individus sur 9. Aucun type de
dent n'a été privilégié, le choix
privilégiant surtout les dents non endommagées et non
cariées. Le tableau n° 2 présente le type de dent
utilisé pour l'extraction de l'ADN. Concernant le périnatal, il
n'a bien sur pas été possible d'utiliser de dent, une hémi
mandibule a donc été utilisée à la place.
|
N°Sépulture
|
169
|
170
|
171
|
|
1er inhumé
|
M3
|
P1
|
C
|
|
2ème inhumé
|
M2
|
M2
|
M3
|
|
3ème inhumé
|
M3
|
X
|
P1
|
Tableau 2 : Détail des dents
utilisées lors de l'extraction.
Les conditions de prélèvement des
échantillons sur le site ont été assez variables.
Les prélèvements des individus du sarcophage 169
ont fait l'objet de précautions tout à fait rigoureuses, tel que
le port de masque, de gants, nettoyage systématique des outils à
la javel puis mis en congélation immédiate des
échantillons. Pour plusieurs auteurs (Poinar et al. 2003 ; Gilbert
2005), toutes ces précautions prises lors des prélèvements
sont essentielles et permettent de limiter sérieusement les
contaminations exogènes.
Les individus des sarcophages 170 et 171 n'ont pas fait
l'objet de si grands soins. En effet, les prélèvements ont
été effectués sans gant. Néanmoins, les
échantillons ont été mis en sachet immédiatement
après avoir été prélevés, n'ont pas
été lavés et ont été conservés
à -20°C jusqu'aux analyses. Bien que ce dernier aspect soit
regrettable, il sera néanmoins intéressant, d'un point de vue
méthodologique, de comparer les résultats d'analyse ADN issus de
prélèvements avec et sans port de gant.
2.2. Choix des régions
étudiées
Pour tous les arguments énoncés
précédemment, notre analyse s'est portée en premier lieu
sur l'ADN mitochondrial. Nous avons décidé de nous concentrer
uniquement sur la région de contrôle (D-Loop) même si la
région codante est parfois indispensable pour la confirmation de
certains haplogroupes. Cette région hypervariable, comme nous l'avons
dit, est partagée en 2 segments de taille réduite : HVRI (421pb)
et HVRII (383pb). Seul la région HVRI a été
analysée car c'est la région pour laquelle les bases de
données sont les plus riches. L'ADN ancien étant très
souvent dégradé, cette région sera fractionnée lors
de l'amplification. Nous obtiendrons donc, lors des résultats, quatre
fragments chevauchants et courts A, B, C et CC reconstituant la région
HVR1.
3. Méthodes
L'analyse génétique se divise en deux grandes
étapes importantes.
La première est l'obtention des séquences ADN, par
les méthodes d'extraction, d'amplification de l'ADN, de clonage et de
séquençage qui vous seront décrites.
La seconde est l'analyse de ces séquences: alignement
des séquences de clones, détection de contaminations et de
déaminations, authentification des séquences, comparaison avec
d'autres séquences issues de base de données, et identification
d'haplogroupe. Le détail des protocoles est présenté dans
les annexes (annexe A).
3.1. Précautions
Bien entendu, un certain nombre de précautions
vis-à-vis des contaminations doivent être prises tout au long de
la première étape et notamment avant l'amplification ADN, puisque
tout contaminant intégré est préférentiellement
amplifié par la PCR. Neuf critères d'authenticité de l'ADN
ont été élaborés (Gilbert 2005 ; Poinar 2003). Les
plus importants d'entre eux ont été respectés dans le
cadre de cette étude :
- Échantillonnage propre
- Port de gants, charlotte, combinaison, blouses. - Utilisation
de matériel et ustensiles stériles.
- Nettoyage régulier du matériel et de la paillasse
à la javel et aux UV.
- Séparation physique des laboratoires ADN ancien - Salle
blanche en surpression, avec sas et UV
- Séparation des salles d'extraction et de
préparation des PCR
- Séparation des laboratoires pré et post-PCR
- Plusieurs contrôles négatifs d'extraction et de
PCR - Clonage puis séquençage des produits PCR
Il est important d'insister sur le fait que la
détection de contaminations est essentielle et plus encore dans cette
étude. En effet, de par sa provenance géographique ainsi que sa
faible ancienneté (seulement 1500 ans), ce matériel
génétique pourrait être tout à fait comparable et
similaire aux contaminations exogènes modernes potentielles
(manipulateurs, réactifs), rendant ainsi l'authentification plus
ardue.
3.2. Préparations des
échantillons
Les échantillons, après avoir été
prélevés, ont été placés au
congélateur dans des sachets stériles empêchant tout
contact avec l'extérieur.
En laboratoire, des dents en place non cassées sont
été extraites des alvéoles, raclées à l'aide
d'un scalpel puis nettoyées à la javel et rincées à
l'eau stérile (le tout sous hotte stérile UV).
L'échantillon préparé est ensuite mis en sachet
stérile double marqué.
3.3. Extraction de l'ADN
Cette étape se déroule sur une
demi-journée en salle blanche. Il convient de signaler une fois pour
toute que tout le matériel contenu au sein de cette salle est
stérile. La stérilisation des locaux et matériels est
obtenue par nettoyage à la javel et exposition aux UV.
En premier lieu, la dent est broyée
grossièrement à l'aide d'un marteau puis la poudre en
résultant est mélangée à un tampon de lyse (EDTA +
NaOH + N-Lauryl sarcosyl + Protéinase K). Le tout est placé en
étuve à 37°C toute la nuit. Cette étape de «
digestion » va permettre la lyse des cellules et la libération des
acides nucléiques de l'échantillon au sein de la solution. Le
deuxième jour, la solution subit une étape de
déprotéinisation, par séparation sur gradient de
phénol-chloroforme. Cette étape va permettre la séparation
de l'ADN et des constituants lipidiques et protéiques. Cette
deuxième journée s'achève avec la filtration et
purification de l'extrait grâce à des colonnes (Centricons). Cette
dernière étape permet la concentration de l'ADN ainsi que
l'élimination de diverses particules inhibitrices de PCR.
Parallèlement, un tube témoin extraction (sans ADN) suit
l'étape d'extraction.
3.4. Amplification de l'ADN
Cette étape débute avec le mélange des
réactifs PCR ou mix réalisé en salle blanche mais au sein
d'une petite pièce spécifique. Ce mélange contient des
dNTP (désoxyribonucléiques tri phosphates) qui permettront
l'étape d'élongation, l'enzyme Taq polymérase qui va
réaliser cette polymérisation ainsi que les amorces
spécifiques qui servent de point d'ancrage et de départ à
la polymérisation. Associé à cela d'autres constituants
comme du tampon 2x, du BSA, du Mgcl2 et de l'eau sont incorporés au
mélange. Une fois l'ADN ajouté, le mélange est prêt
pour l'amplification.
La technique de la PCR (Polymerase Chain Reaction) permet
l'amplification de fragments d'ADN à partir de très petite
quantité d'ADN. Ce système, indispensable en
paléogénétique, va ainsi nous permettre d'obtenir des
millions de copie de notre région cible HVRI. Comme il est possible de
le voir sur la figure 9, cette PCR se déroule en 3 étapes et est
réalisée par une seule et même machine destinée
à cette fonction (thermocycler).
La dénaturation, étape de séparation des
deux brins d'ADN, s'effectue en une minute à une température de
94 O.
Lors de l'étape suivante, l'hybridation, un couple
d'amorce spécifique va venir s'hybrider sur les brins d'ADN (voir
détail des amorces spécifiques à chaque fragment tableau
3). Cette étape dure 45 secondes et la température d'hybridation
est spécifique au couple d'amorce utilisé.
Pour finir, l'ADN est synthétisé lors de la phase
d'élongation ou polymérisation. Cette étape ne dure qu'une
minute et est réalisée à une température de
72°C.
L'ADN est ainsi synthétisé de manière
exponentielle durant un nombre de cycle prédéfinis. Au sein du
LAPP, 45 cycles sont estimés nécessaires pour une bonne
amplification. Le risque étant qu'un nombre de cycles trop important
rende la PCR plus sensible aux contaminations, aboutissant ainsi à la
détection quasi-systématique d'ADN contaminant (liés aux
réactifs) au sein des témoins (Yang 2003).

Figure 9 : Schéma d'une amplification d'ADN par PCR
(Vierstraete 1999).
De la même façon que lors de l'extraction, un
témoin négatif PCR est réalisé. Une fois l'ADN
transporté dans une autre salle, un autre témoin aérosol
contenant le mix PCR reste ouvert pendant tout le temps de la manipulation afin
de vérifier la pureté de l'air de la salle blanche.
|
Fragment
|
Amorce
|
séquences des amorces
|
ille (pb)
|
T°
|
|
HVSI complet
|
HVSIcompU
|
CCCAAAGCTAAGATTCTAAT
|
421
|
56
|
|
HVSIcompL
|
GAGGATGGTGGTCAAGGGAC
|
|
|
|
HVSI A
|
HVSIaU
|
CCCAAAGCTAAGATTCTAAT
|
186
|
56
|
|
HVSIaL
|
TGGATTGGGTTTTATGTA
|
|
|
|
HVSI B
|
HVSIbU
|
TGACCACCTGTAGTACATAA
|
174
|
56
|
|
HVSIbL
|
GGAGTTGCAGTTGATGT
|
|
|
|
HVSI C
|
HVSIcU
|
CCCCATGCTTACAAGCAAGT
|
261
|
56
|
|
HVSIcL2
|
GAGGATGGTGGTCAAGGGAC
|
|
|
|
HVSI CC
|
HVSIccU
|
CCCCATGCTTACAAGCAAGT
|
133
|
56
|
|
HVSIccL
|
TGGCTTTATGTACTATGTAC
|
|
|
Tableau 3 : Amorces utilisées pour chaque
fragment (Jehaes et al. 2001).
3.5. Vérification de PCR
Cette étape consiste tout simplement à
vérifier que l'ADN a bien été amplifié en
quantité suffisante. Pour cela, une électrophorèse est
réalisée. En premier lieu, un gel d'agarose est fabriqué
puis l'ADN y est incorporé par l'intermédiaire de puits. Du bleu
de bromophénol est ajouté à l'ADN afin d'alourdir le
mélange et de permettre de visualiser le front de migration. Un marqueur
de taille est également déposé dans certains puits afin de
servir d'échelle. Le gel, une fois chargé, est introduit dans
l'électrophorèse ou il est immergé dans un tampon qui
permettra la conduction du courant électrique. Une fois le courant
établi les échantillons vont migrer différentiellement
selon la taille et la charge des fragments d'ADN. Les petits fragments
migreront plus vite que les gros qui seront ralentis dans les mailles du gel.
Le gel est ensuite trempé dans du BET (bromure d'éthidium),
intercalant qui permettra ensuite la révélation des bandes d'ADN
sous UV. La taille des fragments est enfin estimée par comparaison au
marqueur de poids moléculaire.
3.6. Clonage
Les échantillons pour lesquels on a obtenu un signal
sont clonés. Cette étape de clonage permet de visualiser
l'ensemble des séquences ADN présentes dans un même produit
PCR, permettant ainsi de séparer les potentielles séquences
contaminantes des séquences authentiques. En effet, comme nous l'avons
vu précédemment, la PCR va préférentiellement
amplifier l'ADN le plus abondant et le moins dégradé, ce qui
n'est pas souvent compatible avec l'ADN ancien. La première étape
de ce clonage est la ligation : elle permet l'insertion du fragment ADN
d'intérêt dans la bactérie, par l'intermédiaire de
vecteurs. Ces bactéries sont
ensuite mises en culture dans un milieu contenant de
l'ampicilline, un antibiotique, puis mises à incuber. Seules les
bactéries ayant inséré le fragment d'intérêt,
sont résistantes à l'ampicilline et peuvent se développer.
Une fois les colonies formées, celles-ci seront
récupérées, formant ainsi un stock d'ADN. Afin de
vérifier que les bactéries ont bien intégré le
fragment de taille attendue, une PCR ainsi qu'une vérification sur gel
d'agarose sont réalisées. Dans le cas d'un clonage réussi,
les échantillons sont envoyés à séquencer
(entreprise Co-genics), ce qui nous permettra d'obtenir la succession des bases
des séquences d'intérêt.
3.7. Analyse des séquences
Après le retrait des vecteurs et des amorces des
séquences, la première étape est l'alignement des
séquences (clones, témoins, ADN des manipulateurs et fouilleurs,
contaminations répertoriées au sein du laboratoire), à
l'aide du logiciel Bioedit.
La comparaison directe des séquences entres elles ainsi
qu'avec la séquence de référence d'Anderson va nous
permettre de repérer les mutations sur les clones mais aussi de
détecter les séquences contaminantes, les erreurs de
réplication lors de la PCR et les phénomènes de
déamination. Il est particulièrement important de comparer les
séquences obtenues dans les témoins et les séquences des
tubes contenant des extraits d'ADN ancien. En effet, Lorsque de l'ADN est
retrouvé dans les témoins, celui-ci est issu de contamination
provenant de plusieurs sources (réactifs d'extraction ou
d'amplification, manipulateurs du laboratoire ou encore séquences
étudiées précédemment dans le laboratoire). Par
conséquent, toute séquence d'ADN ancien identique à une
séquence retrouvée au sein des témoins sera
considérée comme contamination.
Lorsqu'une séquence présentant des mutations est
authentifiée, il est ensuite possible d'aller chercher des informations
concernant ces mutations dans les bases de données du net (Blast NCB I,
http://www.ncbi.nlm.nih.gov/):
ont-elles été répertoriées,
référencées, sont-elles caractéristiques d'un
haplogroupe ? Ces informations sont acquises à partir du site
http://www.stats.gla.ac.uk/~vincent/founder2000/
présentant une base de données de séquences
mitochondriales obtenues pour un grand nombre de populations Européennes
et proche-orientales (Richards 2000).
3.8. Typage des fouilleurs
Le typage des fouilleurs s'effectue de la même
manière que les analyses réalisées sur de l'ADN actuel.
Nous avons envoyé à chacun des fouilleurs un kit de
prélèvement buccal afin de pouvoir récupérer l'ADN
issu des cellules épithéliales de l'intérieur de la joue.
En une journée, l'extraction, l'amplification et la vérification
de la PCR sont réalisées.
Les étapes d'extraction et d'amplification ne
s'effectuent pas en salle blanche car la quantité d'ADN, normalement
assez abondante, permet à la PCR d'être insensible aux
contaminations extérieures. Pour cette même raison, l'étape
de clonage n'est pas non plus réalisée. L'étape
d'extraction se trouve très réduite par rapport à celle
effectuée en ADN ancien. La partie concernant le broyage des
échantillons est bien sûr absente pour ces analyses ainsi que
l'étape de purification. Les produits utilisés lors de
l'extraction sont un peu différents de ceux retrouvés pour l'ADN
ancien. Ceux-ci sont utilisés sous forme de kit ce qui réduit
encore un peu le temps passé pour la réalisation de cette
étape.
L'ADN étant normalement en très bon état
il est alors inutile de fragmenter la région HVR1. En
conséquence, les amorces utilisées pour la PCR vont amplifier
toute cette région (voir tableau 4).
III Synthèse bibliographique
Les études génétiques de parenté sur
de l'ADN ancien peuvent apporter des informations essentielles quant à
l'organisation des sociétés et le mode de vie des populations du
passé.
Dans le cas de résultats positifs, ces études
permettent parfois de nous renseigner sur le statut de la famille au sein de la
société, le fonctionnement marital (endogamie, exogamie) ou
permet encore de déterminer si un système matrilocal ou
patrilocal était adopté par ces populations.
Afin d'obtenir toutes ces informations, plusieurs marqueurs
génétiques sont utilisés.
L'ADN mitochondrial (ADN mt) est presque toujours le premier
marqueur analysé pour les raisons que nous avons
énumérées précédemment. Il permet de
repérer les liens maternels. Très souvent, lorsque des
résultats sont obtenus à partir de l'ADN mt, des analyses sur les
STR autosomaux sont réalisées. Les STR (short tandem repeat) sont
de petites séquences de quelques paires de bases se
répétant de manière variable au sein du génome
nucléaire et présentant un polymorphisme important entre
individus. Pour ces marqueurs, nous disposons chacun d'un allèle
transmis par nos deux parents et il est donc possible de repérer des
liens de parenté biparentaux à partir de la taille de ces STR. Le
même concept est utilisé pour le chromosome Y et permet alors de
déterminer uniquement les liens paternels.
Sur les chromosomes sexuels XY, les analyses peuvent
également porter sur un marqueur spécifique du sexe : le
gène de l'amélogénine, afin de confirmer le sexe
morphologique de l'individu.
On recense actuellement très peu d'articles (moins d'une
quinzaine) portant sur les liens de parenté génétiquement
établis à partir d'ADN ancien.
Cette réalité résulte sûrement du
fait que les études sur l'ADN ancien se heurtent à de nombreux
obstacles (ADN dégradé, contaminations...) ne permettant pas
d'aboutir à des résultats exploitables. Néanmoins, une
grande majorité de ces études datent des années 2000, et
l'on peut donc espérer qu'elles se multiplient dans un avenir proche
grâce aux progrès méthodologiques qui seront
réalisés quant à l'extraction et l'amplification de
l'ADN.
Nous avons souhaité établir, dans le cadre de ce
mémoire, un bref bilan (non exhaustif) des études
paléogénétique de liens de parenté publiées
à ce jour. Cette synthèse bibliographique présente
plusieurs intérêts. Il s'agit en premier lieu de présenter
le contexte
archéologique des sites étudiés ainsi que
les motivations principales conduisant à ces études de liens de
parenté. Quelles données importantes pourraient ressortir de ces
liens de parenté établis génétiquement ? Les
informations qui seront obtenues pourront-elles être
généralisées à une population, une époque...
?
Elle permet ensuite de faire un point sur les techniques et
protocoles utilisés pour effectuer ces analyses
génétiques. Retrouve-t-on, de manière systématique
dans ces études, toutes les précautions reconnues aujourd'hui
comme essentielles à la conservation de l'ADN, à la limitation
des contaminations ainsi qu'à la détection de celles-ci ?
Quels résultats génétiques, ces
études obtiennent t'elles pour l'ADN mitochondrial et l'ADN
nucléaire ? Peut-on faire un rapprochement entre les précautions
prises et la qualité des résultats obtenus ?
Pour finir, qu'obtiennent ces études en terme de liens de
parenté ? Et que peut-on en conclure ?
Neuf publications seront présentées dans le
cadre de cette synthèse bibliographique non exhaustive : (Haak et
al. 2008 (art n°2) ; Cappellini et al. 2003 (art n°4) ;
Bouwman et al. 2008 (art n°3) ; Keyser-Tracqui et al.
2003 (art n°6) ; Dudar et al. 2003 (art n°9) ; Mooder
et al. 2005 (art n°1) ; Rudbeck et al. 2005 (art
n°7) ;.Clisson et al. 2002 (art n°5) ; Adachi et al.
2004 (art n°8) (tabl. 4 et 5).
IV Résultats
1. Résultats de la synthèse
bibliographique
Le tableau ci-dessous (tabl .4) présente neuf
publications portant sur liens de parenté, entre individus d'une
même sépulture ou d'une même nécropole,
établis génétiquement. Ce premier tableau détaille
certains aspects techniques essentiels : les précautions prises lors de
ces études ainsi que les résultats obtenus en termes de
séquences authentiques d'ADN mt et nucléaire.
Le premier point à remarquer est que ces études
portent sur des vestiges de toute époque et l'on constate que la
qualité des résultats obtenus concernant l'ADN mitochondrial ne
semble pas dépendre de l'âge des vestiges mais de leur milieu de
conservation. Ce constat est d'ailleurs reconnu dans la littérature
(Schultes 1999).
L'étude de Haak, (2008) est un parfait exemple de bonne
conservation d'ADN sur des vestiges très anciens, puisque cette
étude, menée sur de l'ADN datant de plus de 4000 ans, fournit
malgré tout des résultats très satisfaisants pour l'ADN mt
et l'ADN nucléaire.
Plus de la moitié des articles mentionnés dans
ce tableau portent sur des vestiges retrouvés dans des milieux froids
voir gelés (permafrost) et l'on constate aisément à partir
des résultats que ces milieux sont idéaux à la
conservation des vestiges et de l'ADN.
Parallèlement, ce tableau illustre assez bien les
difficultés rencontrées aujourd'hui concernant l'analyse du
génome nucléaire et cela même lorsque l'ADN mitochondrial
sort relativement bien. Pour toutes les raisons énoncées
précédemment, les marqueurs nucléaires sont en effet plus
difficiles à obtenir que l'ADN mitochondrial. L'analyse des STR a
été réalisée seulement dans six des ces
études et quatre d'entre elles aboutissent à des
résultats, globalement satisfaisants.
Malgré le caractère récent de ces
publications, il convient de signaler que beaucoup d'entre elles ne respectent
pas les protocoles conseillés et reconnus concernant la conservation de
l'ADN, la détection de contamination et l'authentification des
séquences.
Pour ce qui est de la conservation, plusieurs études
méthodologiques ont montré que la mise en congélation
immédiate des restes après leur prélèvement
était une précaution essentielle afin de limiter la
dégradation de l'ADN (Pruvost 2008). Ce protocole n'a
cependant pas toujours été respecté dans le
cadre des ces études puisque seulement trois articles sur les neuf font
mention de cette congélation immédiate des restes à
-20°C.
Comme nous l'avons vu précédemment, le
deuxième élément important dans les études portant
sur l'ADN ancien est la détection des contaminations. Or, dans ce
tableau, plus de la moitié des publications ne mentionne pas
d'étape de clonage pour l'ADN mitochondrial, laissant ainsi planer un
doute non négligeable sur l'authenticité des séquences
obtenues. En effet, comme nous l'avons expliqué
précédemment, le clonage est une étape clef de
repérage d'éventuelles contaminations modernes. Sans elle,
l'authentification des séquences obtenues peut être
sérieusement remise en question.
Concernant l'ADN nucléaire, plusieurs extractions et
amplifications sont nécessaires afin de permettre l'authentification des
allèles obtenus. On estime que huit répétitions (donnant
des résultats positifs) est un nombre tout à fait acceptable pour
cela (Taberlet 1996). Dans les études précédentes, le
nombre de répétitions pour les STR est assez variable et
s'étale de deux à dix-huit en fonction des individus et des
articles. Le pourcentage de résultats authentiques parmi ces
répétitions est une information qui n'a pas pu être obtenue
car non mentionnée dans les publications. Les résultats
concernant le gène de l'Amélogénine ont été
notés dans ce tableau. Ce marqueur permet de vérifier le sexe de
l'individu et de s'assurer également de la présence d'ADN
nucléaire. Dans une grande majorité des études, les
résultats concernant ce marqueur sont meilleurs que ceux obtenus pour
les STR autosomaux ce qui est pour le moins étrange.
On constate donc de manière générale dans
ce tableau une certaine hétérogénéité entre
les articles vis-à-vis des protocoles et précautions
effectuées. Il nous montre néanmoins clairement que les
précautions sont indispensables à une bonne analyse
paléogénétique.
L'étude de Bouwamn (2008) illustre assez bien
l'importance des précautions dans ce type d'analyses car les
résultats obtenus dans cette étude sont assez décevants
(seulement 18% d'ADN authentique pour l'ADN mt et 0% pour d'ADN
nucléaire) et ceci pourrait s'expliquer entre autre par l'absence de
précautions prises lors des prélèvements ainsi que
l'absence de congélation immédiate des échantillons
(fouille ancienne).
Qu'en est-il maintenant des résultats obtenus en termes de
liens familiaux ?
Des liens de parenté entre individus regroupés ont
été mis en évidence dans cinq de ces études (Haak
et al. 2008 ; Cappellini et al. 2003 ; Keyser-Tracqui et
al. 2003 ; Bouwamn et
al. 2008 ; Keyser-Tracqui et al. 2003 et Dudar
et al. 2003) (tabl .5). Les regroupements familiaux retrouvés
sont de deux sortes.
On distingue en premier lieu les regroupements familiaux mis
en évidence au sein d'une même structure funéraire
réduite de type sépulture plurielle, chambre mortuaire etc. On
retrouve ce type de regroupement au sein du cimetière néolithique
d'Eulau en Allemagne (Haak 2008) ainsi qu'au sein de la nécropole
Etrusque de Monterozzi (Cappellini 2003). Bien que quelques milliers
d'années séparent ces deux sociétés, dans les deux
cas, des relations de parenté très proches, de type
parents-enfants unissent les individus regroupés. Ces types de
regroupement sont très informatifs puisqu'ils permettent de montrer
l'importance que tiennent les liens biologiques dans ces
sociétés. Ce type de regroupement est également
retrouvé au sein de la tombe circulaire mycénienne (Bouwman
2008). En effet, une relation de type frère- soeur est retrouvée
entre deux individus issus d'une même sépulture. Outre l'influence
importante des liens biologiques, cette relation de parenté apporte une
nouvelle information qui est le statut de la femme dans cette
société. Ici, nous sommes face à une tombe royale ou
seules quelques rares femmes sont inhumées. Les liens biologiques
unissant ces deux individus attestent de l'autorité de cette femme,
autorité acquise par droit de naissance et non par relation
conjugale.
Le deuxième type de regroupement familial mis en
évidence dans ces études est le regroupement intra sectoriel au
sein de cimetière ou nécropole. Les études portant sur la
nécropole antique de Gol Egyin en Mongolie (Keyser-Tracqui 2003) ainsi
que le cimetière moderne de Harmony road au Canada (Dudar 2003) mettent
en évidence des regroupements familiaux sectoriels. Dans le cas du
cimetière de Harmony road, les liens maternels figurent sur deux
générations d'inhumations ce qui laisse penser qu'un
système de patrilocalité était adopté par ces
pionniers canadiens. Pour ce qui est de la nécropole de Gol Egyin,
quelques familles regroupées formant des secteurs dans la
nécropole ont été identifiées mais quelques
individus apparentés sont également retrouvés
dispersés eu sein de celle-ci.
|
chronologie
|
N°art
|
conditions de prélèvements et de stockage.
état
conservation des restes.
|
échantillon
|
régions étudiées
|
% de séq
authentiques
|
critères d'authentification.
|
|
Néolithique
|
ADN mt (HVR1)
Bonne conservation du reste dû au climat froid. Pas de 83%
pas de clonage
7000 BP, Sibérie. 1 précautions lors des
prélèvements ni de congélation. Fouille 37 individus ADN
mt (SNP)
très ancienne. Amélogénine 75% concorde
avec sexe morpho
|
|
ADN mt (HVR1) 75%
Clonage
ADN mt (HVR2) 22%
4600 BP, Allemagne. 2 Bonne conservation générale
des restes. Prélèvements des
échantillons avec précautions puis
congélation. 13 individus Amélogénine 75% concorde avec
sexe morpho
STR autosomaux 45%
3 à 9 répétitions selon les individus
STR chrom Y 23%
|
|
ADN mt (HVR1)
18% Clonage
ADN mt (HVR2)
3500 BP, civilisation
Mycéenne, Grèce. 3 Fouilles des sépultures
très anciennes, pas de précautions
prises lors des prélèvements ni de
congélation 22 individus Amélogénine 0%
STR autosomaux 0%
STR chrom Y 0%
|
|
Antiquité
|
IVe- IIIe siècle av J.-C,
Italie.
|
4
|
Mauvaise conservation des restes. Pas de précautions
lors
des prélèvements, ni congélation.
|
4 individus
|
ADN mt (HVR1)
|
100%
|
Clonage
|
|
Amélogénine
|
100%
|
concorde avec sexe morpho
|
|
STR autosomaux
|
0%
|
|
|
IIIe siècle av J.-C,
Kazakhstan.
|
5
|
Très bonne conservation dû au gel.
Précautions prises lors
des prélèvements et
congélation des échantillons.
|
2 individus
|
ADN mt (HVR1)
|
100%
|
pas de clonage
|
|
Amélogénine
|
100%
|
concorde avec sexe morpho
|
|
STR autosomaux
|
100%
|
15 répétitions
|
|
IIIe - IIe siècle av J.-C,
Mongolie.
|
6
|
Conservation des os moyenne. Pas de précautions lors
des
prélèvements mais congélation des
échantillons.
|
62 individus
|
ADN mt (n=56)
|
82%
|
pas de clonage
|
|
Amélogénine
|
100%
|
concorde avec sexe morpho
|
|
STR autosomaux
|
79%
|
18 répétitions
|
|
STR chrom Y (n= 35)
|
27%
|
18 répétitions
|
|
Moyen-Age
|
1000-1250 ap J.-C,
Danemark.
|
7
|
Précautions lors des prélèvements mais pas
de congélation
des échantillons, stockage à
température ambiante.
|
10 individus
|
ADN mt (HVR1)
|
90%
|
Clonage
|
|
ADN mt (reg codante)
|
90%
|
Clonage
|
|
Epoque
Moderne
|
XVIIIe - XIXe siècle
ap J.-C, Japon.
|
8
|
Pas de précautions lors des prélèvements
ni congélation des
échantillons.
|
41 individus
|
ADN mt (HVR1)
|
73%
|
pas de clonage
|
|
ADN mt (HVR2)
|
n.d
|
pas de clonage
|
|
XIXe - XXe siècle ap
J.-C, Nord Canada.
|
9
|
Pas de précautions lors des prélèvements
ni de congélation
des échantillons.
|
38 individus
|
ADN mt (HVR2)
|
60%
|
pas de clonage
|
|
Amélogénine
|
34%
|
concorde avec sexe morpho
|
|
STR autosomaux
|
47%
|
???
|
|
n°
article
|
contexte archéologique et Problématique
de l'étude.
|
résultats
|
discussion
|
|
1
|
Cimetière Néolithique de Lokomotiv, Lac
Baïkal, Sibérie (7000 BP). Plus
grand cimetière
Néolithique du nord asiatique. Grande variabilité dans
les
pratiques funéraires (architecture des tombes, dépôt
des défunts, traitement
des corps...). Cette variabilité
indique t-elle une absence d'affinité biologique
entre les individus
?
|
Profil ADN mt de 31 individus sur 37. haplogroupe F
dominant
à 70%. Haplogroupes ? de ceux d'aujourd'hui dans la
région.
Existence de très peu de groupes
matrilinéaire.
|
Les quelques liens matrilinéaire retrouvés n'ont
pas façonné
l'organisation spatiale du cimetière, mais
ils ont influencé le
type de tombe et dans certains cas le type de
traitement
donné au défunt.
|
|
2
|
Cimetière d'Eulau (Allemagne), Néolithique (4600
BP). Découverte de 4
sépultures collectives regroupant adultes
et enfants. Proximité entre
individus apparente. Individus
sûrement victimes de mort violente. Approche
multidisciplinaire
[archéologique, anthropologique, géochimique
(isotope
radioactif) et paléogénétique] dans le cadre
de l'étude. Analyses
géochimiques et
paléogénétiques effectuées sur 13 individus
regroupés dans
les 4 tombes.
|
Mise en évidence d'un noyau familial dans 2 des 4
tombes. Dans
l'une, père, mère, fils, fille. Dans la
deuxième, 3 frères et soeurs
associés à une
femme qui n'est pas leur mère. Présence
d'haplogroupe X2, I,
K, H, J. Mort violente pour 5 individus sur
13. Les Isotopes du strontium
montrent que les hommes et les
enfants mâles ont grandit dans la
même région mais pas les
femmes.
|
Mise en évidence de l'importance des liens biologiques
au
sein de l'organisation de cette société
Danubienne
Néolithique. Système patrilocal et exogame
adopté par ces
populations.
|
|
3
|
Tombe circulaire mycénienne, Grèce (3500 BP). Les
preuves archéologiques
montrent que les 35 individus inhumés
au sein de cette structure ont un statut
d'élite. Il est
supposé que les groupes de sépulture représentent
différentes
dynasties ou différents membres d'une même
famille. Des reconstructions
faciales réalisées semblent
départager les individus en trois groupes en
fonction de la forme de
leur visage. Etude paléogénétique en vue de
localiser
d'éventuels regroupements familiaux au sein de cette
tombe.
|
Echec des analyses portant sur l'ADN nucléaire.
Profil
mitochondrial obtenu pour 4 individus sur 22. Mise en
évidence
d'une même lignée maternelle unissant deux
individus, masculin
et féminin. Les deux individus apparentés
étaient regroupés dans
la même catégorie de
visage.
|
Les liens unissant ces deux individus pourraient être de
type
frère - soeur. Les sujets féminins sont très rares
au sein de
cette tombe. Ces femmes semblent disposer d'un
statut
particulier au sein de la société. L'implication est
que le
sujet féminin en question n'est pas inhumé dans cette
tombe
pour cause de relation conjugale mais parce qu'elle a
occupé
une position d'autorité par droit de naissance.
|
|
4
|
Nécropole de Monterozzi (Latium, Italie) (IVe-I I Ie
siècle BC). Sépulture
collective de 4 individus
retrouvés dans une chambre mortuaire étrusque [2?
(1 mature +
1 imamat) + 2 c' (1 mature + 1 imamat)]. S'agit-il d'un
regroupement
familial?
|
Mise en évidente d'une même séquence HVRI
chez la ? adulte
et les 2 immatures. STR non reproductibles.
|
Regroupement familial dans cette chambre
mortuaire.
Présence d'une mère et ces deux enfants. L'homme
adulte
pourrait être le père.
|
|
5
|
Tombe de prince Saka retrouvé dans un tumulus en pierre
(Kazakhstan, IIIe
siècle BC). Sépulture double: 1 d' (30-35
ans), le prince ? Et 1 ? (+ 60 ans).
Liens de parenté proche entre
les 2 individus?
|
Aucune lignée maternelle mise en évidence avec
l'ADN mt.
Comparaison des STR ambigus mais surement aucun lien
de
parenté.
|
Hypothèse d'un couple. Le prince et sa femme ?
|
|
6
|
Nécropole de Gol Egyin, Nord de la Mongolie (IIIe - IIe
siècle BC, période
Xiongu). Découverte d'une centaine
d'individus. Cimetière partagé en 4
secteurs. Sépulture
simple pour une grande majorité mais quelques
sépultures
double. Regroupement familial au sein des secteurs ainsi
qu'au sein des
sépultures doubles?
|
49 profils génétiques obtenus. 27 individus
typés pour le chromosome Y : 18 haplotypes ?. Profil mitochondrial de 46
individus sur 56. 89% d'haplogroupes asiatiques (A, C, D) et quelques
haplogroupes européens. Présence de sépultures
doubles avec individus sans liens de parenté
apparente.
|
Mise en évidence de liens familiaux maternels et
paternels
au sein des secteurs majoritairement et intersecteur pour
une
minorité. Plusieurs familles identifiées. Chronologie
des
phases des secteurs établis à partir des résultats
génétiques
et des datations.
|
|
7
|
Cimetière de Kongemarken, Danemark (1000-1250 AC). Un
des plus vieux
cimetières paléochrétiens du Danemark.
Ségrégation hommes-femmes. d'
inhumés au Sud du
cimetière et ? au Nord. Cependant quelques d' avec les ?
au Nord et
quelques ? avec les d' au Sud. Liens de parenté entre d' et
?
regroupés ? Analyses ADN effectuées sur 9 individus issus de
2 zones (1d' +
4 ? et 1 d' + 3 ?).
|
Aucune lignée matrilinéaire commune entre les
individus.
Présence de 4 haplogroupes différents (U7, I, J,
T2). Grande
diversité de la population inhumée. Haplogroupe U7
absent dans
la région actuellement et haplogroupe I très
rare.
|
Ces observations suggèrent que les individus vivant dans
la
région de Roskilde il ya 1000 ans ne sont pas tous
membres
d'une population local mais composé de personnes ayant
des
liens génétiques avec des populations beaucoup plus
lointaine
(Sibérie, Inde...)
|
|
8
|
Site d'Hatanai, N-E Japon (XVIIIe- XIXe siècle,
période Edo). Découverte de
45 individus. Sépultures
simples. Mise en évidence de 3 cimetières majeurs.
1
cimetière regroupe des femmes et des enfants uniquement et un
autre
regroupe des individus pathologiques. Regroupement familial au sein
des
deux premiers cimetières?
|
ADN de 30 individus exploitable, 8 haplogroupes et 14
lignées
maternelles sont observées. Nombreuses lignées
maternelles ?.
|
Différences généalogiques importantes dans
ces cimetières,
peut-être dû à des effets de
remplacement de la population
dû à des périodes de
famine. Cimetières datant de 3
périodes différentes.
|
|
9
|
Cimetière de Harmony road, Nord du Canada (XIXe- XXe
siècle). Mise en
évidence de 3 périodes d'inhumation
distinctes dans le cimetière. Analyse
génétique de lien
de parenté entre individus effectuée sur 28 individus.
|
Utilisation du cimetière sur 1 ou 2
générations. Aucune preuve
de liens de parenté
établie mais certains haplogroupes sont
retrouvés
regroupés dans une même zone. Les STR ne
renseignent pas.
|
Diversité de l'ADN mt dû à la
mobilité et aux migrations
au XIXe siècle. L'ADN mt
démontre des haplotypes
matrilinéaire figurant pour deux
générations
d'Inhumations, suivie de remplacement par de
nouvelles
lignées : ceci suggère un système
patrilinéaire / patrilocal
adopté par ces pionniers du nord du
Canada.
|
Tableau 5 : Synthèse bibliographique des
résultats d'études paléogénétique de liens
de parenté.
2. Résultats des analyses
génétiques
2.1. Les fouilleurs
Le tableau ci-dessous (tabl.6) présente les
séquences HVSI des fouilleurs. Ces séquences ont
été systématiquement confrontées à celles
obtenues sur les échantillons anciens.
|
fragment A
|
fragment B
|
fragment C
|
fragment CC
|
|
Fouilleur 1
|
0
|
0
|
0
|
0
|
|
Fouilleur 2
|
0
|
0
|
0
|
16362 TC.
|
|
Fouilleur 3
|
16079 CA, 16085 CG.
|
0
|
0
|
16298 TC.
|
|
Fouilleur 4
|
16126 TC.
|
0
|
0
|
0
|
|
Fouilleur 5
|
16147 CA, 16172 TC.
|
16217 AG, 16223 CT,
16248 CT.
|
16320 CT.
|
16355 CT.
|
Tableau 6 : Mutations présentes sur la
région HVS1 des fouilleurs.
2.2. Les séquences des individus
Dans le cadre de cette étude de faisabilité, une
seule extraction par individu a été réalisée. La
majorité des fragments n'a été amplifiée qu'une
seule fois, cependant une deuxième PCR a été
effectuée dans certains cas problématiques.
Le tableau ci-dessous (tabl .7) présente le nombre
d'amplifications réalisées ainsi que le nombre de clones obtenus
pour chaque fragment et chaque individu. Le tableau 8 présente le
détail des mutations observables sur chacun des clones obtenus.
|
|
Nombre de clones obtenus
|
|
|
HVSIA
|
HVSIB
|
HVSIC
|
HVSICC
|
|
Sep 169
|
1er inhumé
|
6 (1)
|
6 (1)
|
6 (1)
|
|
|
2ème inhumé
|
|
13 (3)
|
3 (1)
|
|
|
3ème inhumé
|
6 (1)
|
|
|
6 (1)
|
|
Sep 170
|
1er inhumé
|
7 (2)
|
6 (1)
|
6 (1)
|
|
|
2ème inhumé
|
|
6 (1)
|
3 (1)
|
3 (1)
|
|
3ème inhumé
|
|
|
|
6 (2)
|
|
sep 171
|
1er inhumé
|
3 (1)
|
3 (1)
|
3 (1)
|
|
|
2ème inhumé
|
|
6 (1)
|
|
6 (1)
|
|
3ème inhumé
|
|
2 (1)
|
|
3 (1)
|
Tableau 7 : Clones obtenus pour chaque fragments
et individus.
Les chiffres entre parenthèses correspondent au
nombre de PCR réalisées.
Avant toute chose il convient de signaler que les clones
obtenus présentent, pour une grande majorité, des cas de
déamination. Ce phénomène de dégradation de l'ADN,
très fréquent sur l'ADN ancien, est une réaction chimique
d'hydrolyse des acides nucléiques. Ces « fausses » mutations
sont relativement repérables et nous ne les énumérerons
donc pas dans cette partie.

Sep 169
- 1er inhumé :
Des séquences ont été obtenues pour les
fragments A, B et C.
Pour le fragment A, les mutations 16069 CT et 16126 TC sont
retrouvées sur les six clones obtenus. Lors de ces analyses, aucun des
témoins n'a rendu de séquences contaminantes sur ce fragment. La
mutation 16126 TC est également retrouvée chez un fouilleur ainsi
qu'au sein de séquences répertoriées au laboratoire comme
potentiellement contaminante. Néanmoins, l'association des deux
mutations n'est retrouvée nulle part. Au vu des résultats dont
nous disposons, les séquences présentant les mutations 16069 CT
et 16126 TC semblent donc authentiques. Toutefois, une deuxième PCR
serait nécessaire afin de le confirmer.
Les séquences obtenues pour le fragment B ne
présentent aucune mutation récurrente. Il s'agit donc de la
séquence de référence. Les séquences d'Anderson
obtenues ne peuvent pas être authentifiées car des contaminations
provenant de fouilleurs, de manipulateurs et autres ne peuvent pas être
exclus. Néanmoins l'individu pourrait tout à fait
présenter une séquence d'Anderson pour ce fragment.
En ce qui concerne le fragment C, on constate la
récurrence de la mutation 16300 AG chez cinq clones sur six. Cette
mutation n'est retrouvée ni dans les témoins, ni chez les
fouilleurs et manipulateurs et n'est pas non plus observée au sein des
séquences répertoriées au laboratoire comme
potentiellement contaminantes. Par conséquent, celle-ci est sans doute
authentique. Toutefois, là aussi, une deuxième PCR serait
nécessaire pour l'authentification.
En recherchant dans les bases de données, l'association
des trois mutations 16069 CT, 16126 TC et 16300 AG est connue et nous renvoie
à l'haplogroupe J. Le blast de l'association des trois fragments
consensus (mutations 16069 CT, 16126 TC et 16300 AG) est réalisé
et nous renvoie à une seule séquence homologue à celle-ci,
séquence retrouvée dans une étude encore non
publiée (sans info sur la provenance géographique ?!).
- 2ème inhumé
:
Pour cet individu, des séquences ont été
obtenues seulement pour les fragments B et C.
Pour le fragment B, deux PCR ont été
réalisées. La première amplification ne présente
aucune mutation pour huit séquences sur neuf. Il s'agit donc de la
séquence de référence. Malgré le nombre important
de clones, ces séquences d'Anderson ne peuvent pas être
authentifiées car nous ne pouvons pas écarter un
phénomène de contamination. La deuxième amplification
donne des séquences différentes. Les trois clones obtenus
présentent la mutation 16223 CT. Cette même séquence a
été retrouvée au sein des témoins de cette PCR ;
celle-ci est donc potentiellement une contamination.
L'analyse du fragment C, nous fournit des séquences
d'Anderson pour les trois seuls clones obtenus. Là aussi, les
contaminations ne peuvent pas être exclues mais il se pourrait
également que l'individu ne possède pas de mutation sur ce
fragment. Ces séquences ne peuvent donc pas être
authentifiées.
En conclusion, aucune des séquences obtenues pour cet
individu ne peut être authentifiée. -
3ème inhumé :
Des clones sont obtenues pour les fragments A et CC.
Les séquences du fragment A fournissent trois mutations
récurrentes. On observe ainsi les mutations 16069 CT, 16093 TC et 16126
TC chez trois clones ainsi que la mutation 16037 AG sur 2 clones. Les trois
premières mutations sont répertoriées et retrouvées
fréquemment mais la mutation 16037 AG est totalement inconnue. Les
témoins réalisés pour ce fragment ne fournissent aucune
séquence contaminante. De plus, cette association n'est retrouvée
ni dans les séquences répertoriées au laboratoire comme
pouvant être contaminante ni dans les séquences des fouilleurs et
manipulateurs. Il est alors tout à fait possible que les
séquences présentant les mutations 16069 CT, 16093 TC et 16126 TC
soient endogènes. Toutefois, une seconde PCR serait nécessaire
afin de confirmer ce résultat.
Les séquences obtenues pour le fragment CC, nous
montrent la présence d'une mutation présente chez trois clones
sur 6. Cette mutation 16298 TC a déjà été
rencontrée au sein de témoins aérosol dans le cadre de
cette étude mais non au sein des témoins propres à cette
PCR. Malgré cela, il n'est pas possible d'authentifier cette
séquence car il peut tout à fait
s'agir d'une contamination. Un des fouilleurs présente
également cette unique mutation sur le fragment CC. Une contamination
par l'ADN de cet individu ne peut donc pas être exclue. Cependant les
autres mutations présentes chez cet individu ne sont retrouvées
nulle part. On observe également la présence d'une mutation
isolée 16258 AG sur un seul clone. Cette mutation est issue de
contamination puisque retrouvée au sein des témoins extraction de
cette PCR.
L'association des mutations 16069 CT, 16093 TC et 16126 TC a
déjà été répertoriée. Celle-ci
renvoie à l'haplogroupe J. Il s'agit toutefois d'un haplotype
différent de celui rencontré précédemment. Le blast
le la séquence comportant les mutations 16069 CT, 16093 TC et 16126 TC
nous renvoie à plusieurs études non publiées dans
lesquelles des séquences homologues à 100% sont
retrouvées. D'après les titres de ces articles on constate que
cet ensemble de mutation est plus particulièrement retrouvé dans
le nord de l'Europe ainsi qu'en Hongrie.
Sep 170
- 1er inhumé.
Des séquences ont été obtenues pour les
fragments A, B et C.
Le fragment A a été amplifié deux fois.
Il ne fournit que des séquences d'Anderson sur les 7 clones
analysés. Ces séquences peuvent être des contaminations
mais cet individu pourrait tout aussi bien ne présenter aucune mutation
sur ce fragment. Ces séquences ne peuvent donc pas être
authentifiées dans le cadre de cette étude de
faisabilité.
Le fragment B fournit des clones présentant un nombre
important de mutations. La plupart de ces mutations ne sont pas
récurrentes et résultent essentiellement de
phénomène de déamination. La mutation 16224 TC semble
néanmoins authentique car présente chez cinq clones sur six et
absente des témoins et des séquences potentiellement
contaminantes (séquences répertoriées au laboratoire,
séquences des fouilleurs et manipulateurs).
La mutation 16224 TC est également retrouvée sur le
fragment C chez six clones sur six. Ceci confirme l'authenticité de
cette mutation
En recherchant dans les bases de données, la mutation
16224 TC nous renvoie aux haplogroupes H ou K. Dans les deux cas, ces
haplogroupes sont originaires d'Europe de l'Ouest.
Le blast des séquences nous renvoie à un grand
nombre d'articles dans lesquels sont répertoriés des
séquences identiques présentant uniquement cette mutation 16224
TC.
- 2ème inhumé
:
Des clones ont été obtenus pour les fragments B, C
et CC.
Le fragment B est sans ambigüité ; la mutation
16224 TC retrouvée chez le premier inhumé est également
présente chez cet individu. La présence de cette mutation chez
cinq clones sur cinq ainsi que son absence des témoins et des
séquences répertoriées comme potentiellement contaminantes
permet d'authentifier cette mutation.
On constate la présence d'une mutation 16220 AG
très récurrente au sein des témoins. Cette séquence
est donc à rajouter aux séquences contaminantes du
laboratoire.
Le fragment C confirme le caractère authentique et
endogène de la mutation 16224 TC. Cette mutation est retrouvée
sur les trois clones obtenus. Trois mutations successives sont également
observées dans la zone (16374-16376) mais celles-ci sont surement dues
au dérapage de la Taq polymérease au niveau du poly C
situé à cet endroit.
Les témoins montrent trois séquences
présentant une nouvelle mutation : 16298 TC. Cette séquence est
à ajouter comme issue de contamination.
Le fragment CC atteste définitivement du
caractère endogène de la mutation 16224 TC car celle-ci est
présente sur les trois clones obtenus pour ce fragment. Aucune autre
mutation n'est présente sur ces séquences.
Tout comme pour le 1er inhumé, la recherche
de cette mutation fournit les haplogroupes H ou K. De la même
façon, le blast nous apprend que de nombreuses séquences
présentant cette unique mutation sont retrouvées au sein
d'étude de génétique portant sur des populations
européennes.
- 3ème inhumé
:
Des séquences ont pu être obtenues seulement sur le
fragment CC. Deux amplifications ont été
réalisées.
La première amplification fournit trois clones sur
lesquels sont observés uniquement des séquences d'Anderson. La
deuxième fournit le même résultat.
Malgré les deux répétitions
réalisées sur ce fragment, ces séquences ne peuvent pas
être authentifiées car peuvent tout à fait être
issues de contaminations (séquence retrouvée de
façon récurrente comme contaminante) vu le mauvais
état de conservation de l'ADN de cet individu.
Sep 171
- 1er inhumé :
Pour cet individu, des séquences ont été
obtenues pour les fragments A, B et C.
Le fragment A comporte une seule mutation présente une
seule fois sur les 3 clones. Cette mutation non répertoriées est
sans doute le résultat d'un phénomène de
déamination.
Le fragment B comporte trois mutations récurrentes en
position 16183 AC, 16189 TC et 16223 CT sur les trois clones obtenus. Ces
séquences pourraient être considérées comme
contaminantes car les mutations 16189 TC et 16223 CT sont retrouvées
dans les séquences potentiellement contaminantes du labo. Elles sont
néanmoins absentes des témoins.
Le fragment C comporte également la mutation 16223 CT
vu sur le fragment B. S'ajoute à celle-ci la mutation 16278 CT
présente chez trois clones sur trois. L'association de ces deux
mutations n'est pas retrouvée au sein des témoins ni des
séquences potentiellement contaminantes (manipulateurs, fouilleurs)
Lorsque que l'on recherche dans les bases de données
l'association des mutations 16189 TC, 16223 CT et 16278 CT, celle-ci nous
renvois à un haplogroupe bien définis, l'haplogroupe X. Le blast
d'une séquence comprenant les mutations 16189 TC, 16223 CT et 16278 CT,
nous renvoie à un grand nombre d'articles dans lesquels sont
figurés des séquences similaires d'haplogroupe X. Ces
publications ne seront pas énumérées car les
séquences qui y sont répertoriées sont issues d'origines
géographiques différentes.
- 2ème inhumé
:
Pour cet individu, des clones ont été obtenus pour
les fragments B et CC.
Le fragment B comprend six clones présentant tous la
même mutation 16223 CT. Aucune autre mutation n'est observée sur
ces clones.
Le fragment C présente également la mutation
16223 CT chez 3 clones sur 6. Malheureusement cette unique mutation est
retrouvée parmi les témoins PCR de cette amplification.
Les séquences obtenues pour cet individu ne semblent pas
être authentiques ou du moins il n'est pas possible de le prouver
à ce stade des analyses.
- 3ème inhumé
:
Des séquences ont été obtenues seulement
pour les fragments B et CC.
Pour le fragment B, seul deux clones ont été
récupérés. Ces deux clones présentent la mutation
16223 CT retrouvée également chez le deuxième
inhumé de ce même sarcophage. Aucune autre mutation n'est
observée sur ces clones. Cette mutation retrouvée
également chez les témoins ne peut pas être
authentifié sur cet individu comme sur le précédent.
Toutefois, les résultats obtenus pour ces deux individus ne sont pas
tous issus d'une même amplification et il paraît donc assez
difficile à ce stade d'affirmer avec certitude que cette mutation est
issue d'une contamination. Ces individus pourraient éventuellement
partager cette mutation mais ceci serait à vérifier par des
analyses complémentaires.
Cependant, le fragment C indique tout autre chose. La mutation
16223 CT n'est pas retrouvée sur ce fragment la mais on observe à
la place l'association des mutations 16222 CT et 16261 CT sur trois clones.
Cette association de mutation n'est pas retrouvée dans
les séquences potentiellement contaminantes (témoins,
séquences des fouilleurs et manipulateurs, séquences du labo).
Ces séquences sont donc peut être authentiques. De la même
façon que pour les séquences précédentes, des
analyses complémentaires sont nécessaires afin de confirmer
cela.
L'association des deux mutations 16222 CT et 16261 CT donne l'
haplogroupe H retrouvé dans le Nord-Ouest de l'Europe. Celles-ci sont
également retrouvées associé aux mutations 16069, 16126,
16145 et forme l'haplogroupe J1b1 retrouvé en Grande-Bretagne, Irlande
et Scandinavie. Le blast des séquences du fragment C comportant ces deux
mutations nous renvoie à un grand nombre d'articles portant sur des
origines géographiques très variées. Ces études ne
seront donc pas énoncées.
|
n°
sép
|
|
|
HVSIa
|
HVSIb (1 61 59-16236)
|
HVSIc
|
HVSIcc
|
|
169
|
|
extrait
|
tube
|
nb
clones
|
mutations
|
Tube
|
nb
clones
|
mutations
|
tube
|
nb
clones
|
mutations
|
tube
|
nb
clones
|
mutations
|
|
1er
inhumé
|
T118
|
A1
|
3
|
2 séq 16069 CT, 16126 TC
; 1
séq 16069 CT, 16089
GA, 16126 TC, 16129 GA,
16130 GA.
|
A1
|
3
|
3 séq Anderson.
|
B1
|
3
|
1 séq 16300 AG; 1 séq 16260
CT, 16282
CT, 16300 AG; 1 séq
16294 CT, 16304 TC, 16362
TC.
|
|
|
|
|
A2
|
3
|
1 séq 16034 GA, 16069
CT, 16126 TC ; 2
séq
16069 CT, 16126 TC,
16142 CT.
|
A2
|
3
|
1 séq 16204 GA; 2
séq
Anderson.
|
B2
|
3
|
1 séq 16300 AG; 1 séq 16300
AG, 16390
GA; 1 séq 16250
CA, 16300 AG, 16336 GA.
|
|
|
|
|
2ème
inhumé
|
T122
|
|
|
|
B1
|
3
|
2 séq 16179 CT, 16186
CT; 1 séq
Anderson.
|
C1
|
3
|
3 séq Anderson.
|
|
|
|
|
|
|
|
B2
|
3
|
1 séq 16187 CT; 1 séq
16179 CT, 16186
CT; 1
séq Anderson.
|
|
|
|
|
|
|
|
|
|
|
|
B7
|
3
|
3 micro séq.
|
|
|
|
|
|
|
|
3ème
inhumé
|
T130
|
A11
|
3
|
3 séq d'Anderson
|
|
|
|
|
|
|
A12
|
3
|
3 séq 16298 TC.
|
|
|
A13
|
3
|
2 séq 16037 AG, 16069
CT, 16093 TC, 16126 TC ;
1
séq 16069 CT, 16093 TC,
16126 TC.
|
|
|
|
|
|
|
A13
|
2
|
1 séq d'Anderson ;
1 séq 16258 AG.
|
|
170
|
1er
inhumé
|
T119
|
A3
|
3
|
3 séq d'Anderson
|
A4
|
3
|
1 séq 16224 TC; 1 séq
16179 CT, 16184
CT,
16190 CT, 16197 CT,
16201 CT, 16211 CT,
16224 TC; 1
séq 16168
CT, 16184 CT, 16190
CT, 16191 CT, 16193
CT.
|
B4
|
3
|
1 séq 16221 CT, 16224 TC;
1 séq 16224
TC, 16266 CT;
1 séq 16221 CT, 16224 TC,
16266 CT.
|
|
|
|
|
|
|
|
A5
|
3
|
2 séq 16224 TC; 1 séq
16196 GA, 16213
GA,
16224 TC.
|
B5
|
3
|
2 séq 16224 TC; 1 séq 16224
TC, 16286
CT, 16287 CT,
16291 CT.
|
|
|
|
|
2ème
inhumé
|
T123
|
|
|
|
B2
|
2
|
1 séq 16224 TC ; 1 séq
16204 GA, 16208
GA,
16224 TC, 16227 AG.
|
C3
|
3
|
3 séq 16224 TC, 16375 CT,
16376 CT, 16377 CT.
|
D1
|
3
|
3 séq 16224 TC.
|
|
|
|
|
|
|
B3
|
3
|
1 séq 16224 TC ; 1 séq
16179 CT, 16224 TC ;
1
séq 16193 CT, 16201
CT, 16222 CT, 16225
CT, 16232 CT.
|
|
|
|
|
|
|
|
3ème
inhumé
|
T124
|
|
|
|
|
|
|
|
|
|
D6
|
3
|
3 séq d'Anderson
|
|
|
|
|
|
|
|
|
|
|
|
A12
|
3
|
3 séq d'Anderson
|
|
171
|
1er
inhumé
|
T120
|
A13
|
3
|
2 séq d'Anderson ; 1 séq
16071 CT.
|
A7
|
3
|
1 séq 16167 CA, 16173
CT, 16183 AC,
16189
insertion A, insertion C
en 16194, 16189 TC,
16223 CT;
1 séq 16183
AC, 16189 TC, insertion
c en 16194 16223 CT;
1
séq 16175 AC, 16183
AC, 16189 TC, 16223
CT.
|
B9
|
3
|
3 séq 16223 CT, 16278 CT.
|
|
|
|
|
|
|
|
|
A 8
|
5
|
1 séq 16183 AC, 16185 CT, 16191 CT, insertion C en
16194, 16223 CT ; 1 séq 16183 AC, 16190 CT, 16191 CT, 16193
CT, insertion C en
16194, 16223 CT,
16324 CT; 3 micro séq
|
|
|
|
|
|
|
|
2ème
inhumé
|
T131
|
|
|
|
B9
|
3
|
3 séq 16223 CT
|
|
|
|
A16
|
4
|
3 séq 16223 CT; 1 séq
16216 AG, 16220 AG.
|
|
|
|
|
|
B10
|
3
|
3 séq 16223 CT
|
|
|
|
A17
|
3
|
2 séq d'Anderson
|
|
3ème
inhumé
|
T132
|
|
|
|
B12
|
2
|
2 séq 16223 CT
|
|
|
|
A20
|
3
|
2 séq 16222CT, 16261
CT; 1 séq 16261 CT.
|
|
|
|
|
|
|
|
|
|
|
|
A21
|
3
|
3 séq d'Anderson
|
Tableau 8 : Détail des mutations
présentes sur tous les clones obtenus.
2.3. Synthèse des résultats
Le tableau ci-dessous (tabl.9) présente les
séquences endogènes de ces individus à ce stade des
analyses. Ces séquences ont été authentifiées
suivant plusieurs critères.
- La mutation ou l'ensemble des mutations présentes sur
ces séquences
n'a pas été retrouvée parmi les
témoins ni au sein des séquences des manipulateurs et fouilleurs.
Elle n'a pas non plus été retrouvée parmi les
séquences répertoriées au laboratoire comme
potentiellement contaminantes.
- Ces mutations étaient répétées
sur un minimum de deux ou trois clones issus d'une même PCR ou de PCR
différentes, sur un même fragment ou sur des fragments
différents.
|
|
Séquences consensus
|
|
|
|
HVSIA
|
HVSIB
|
HVSIC
|
HVSICC
|
|
Sep 169
|
1er inhumé
|
16069 CT, 16126 TC.
|
Contam ?
|
16300 AG
|
n.d
|
|
2ème inhumé
|
n.d
|
Contam ?
|
Contam ?
|
n.d
|
|
3ème inhumé
|
16069 CT, 16093 TC,
16126 TC.
|
n.d
|
n.d
|
Contam ?
|
|
Sep 170
|
1er inhumé
|
Contam ?
|
16224 TC
|
16224 TC
|
n.d
|
|
2ème inhumé
|
n.d
|
16224 TC
|
16224 TC
|
16224 TC
|
|
3ème inhumé
|
n.d
|
n.d
|
n.d
|
Contam ?
|
|
Sep 171
|
1er inhumé
|
n.d
|
16183 AC ?, 16189 TC,
16223 CT.
|
16223 CT, 16278 CT
|
n.d
|
|
2ème inhumé
|
n.d
|
16223 CT ?
|
16223?
|
n.d
|
|
3ème inhumé
|
n.d
|
?
|
n.d
|
16222 CT, 16261 CT ?
|
Tableau 9 : Mutations authentiques
présentes sur chaque fragment des individus.
n.d = non déterminé. Cases vertes :
séquences authentifiées ; cases roses : doutes sur
l'authenticité des séquences.
V Discussion
Dans cette partie, nous nous attarderons tout d'abord sur les
aspects techniques (authenticité et conservation de l'ADN) qui
étaient les principaux objectifs de cette étude de
faisabilité. Nous apporterons par la suite quelques résultats en
termes de relation de parenté maternelle entre les individus
étudiés.
1. Authenticité des séquences :
conservation de l'ADN et contaminations - Conservation de l'ADN
sur le site de Jau-Dignac :
Au vue des résultats que nous obtenons, la conservation
de l'ADN des individus analysés apparaît très
satisfaisante. Les résultats génétiques obtenus sont
toutefois fragmentaires (tabl.9). Les neufs individus exhumés
présentaient un état de conservation macroscopique relativement
bon. Cette dernière information conforte bien l'idée qu'il existe
une corrélation entre la conservation macroscopique des restes et leur
conservation moléculaire, comme cela a été décrit
dans la littérature (Cipollaro et al. 1998).
Le caractère récent des
prélèvements ainsi que la mise en congélation
immédiate des échantillons à - 20°C sont deux
facteurs qui ont dû jouer un rôle important de frein à la
dégradation de l'ADN des individus ; ceci pourrait entre autre expliquer
la qualité des résultats obtenus. En effet, il a
été démontré dans la littérature qu'il est
préférable d'analyser un échantillon juste après
son prélèvement sinon les processus de dégradation se
remettent en route (Pruvost et al. 2008). La congélation des
échantillons s'avère également être une
précaution essentielle aux analyses paléogénétiques
puisque celle-ci permet de limiter voir stopper la dégradation de l'ADN
(Barriel 1997). Cette mesure, reconnue depuis longtemps en médecine
légale, n'est cependant pas souvent prise lors d'études portant
sur l'ADN ancien. En effet, comme nous l'avons vu dans la synthèse
bibliographique, les études de (Cappellini et al. 2003 ; Dudar
et al. 2003 ; Rudbeck et al. 2005 ; Adachi et al.
2004) portent sur des vestiges récemment fouillés mais qui n'ont
pas été mis en congélation des leurs
prélèvements.
Les études portant sur des échantillons
prélevés anciennement comme les articles de Bouwman et
al. (2008) et Mooder et al. (2005) n'ont malheureusement pas non
plus bénéficié de cette précaution, ce qui est
encore plus regrettable.
L'état de conservation de l'ADN est souvent
associé à un autre phénomène qui est la
contamination des échantillons par de l'ADN moderne. En effet, plus
l'ADN est dégradé plus
les contaminations exogènes présentes
apparaîtront lors des résultats. Il convient donc, afin de
réaliser des analyses correctes, d'éviter au maximum ces
phénomènes de contamination mais également de pouvoir les
repérer.
- Contamination des échantillons
Comme nous l'avons dit précédemment, les
prélèvements de mandibule n'ont pas tous été
réalisés dans les mêmes conditions.
La sépulture 170 est la seule à avoir
bénéficié de précautions particulièrement
rigoureuses (port de gant, charlotte, mise en sachet puis congélation
immédiate). Les deux autres sépultures, quant à elles,
n'ont pas fait l'objet de port de gants. Toutefois, les autres
précautions ont été prises.
L'analyse de la séquence du fouilleur n°2 (seul
individu ayant prélevé les échantillons des
sépultures 169 et 171) nous permet d'affirmer que cet individu n'a pas
contaminé les échantillons. En effet, son unique mutation en
position 16362 n'a jamais été retrouvée seule dans le
cadre de cette étude. Les quatre autres fouilleurs, qui n'ont jamais
été en contact direct avec les échantillons,
présentent également des séquences absentes des analyses,
ils n'ont donc pas contaminé les échantillons. En
conséquence, l'absence de port de gant lors des
prélèvements des sépultures 169 et 171 n'a eu aucune
conséquence sur les résultats de ces analyses et le fait de
manipuler mains nues, de façon brève, un échantillon ne
s'avère pas un geste contaminant. Cette information confirme que la
manipulation des échantillons à mains nues n'est pas le geste le
plus contaminant, comme précédemment évoqué dans la
littérature (Yang et al. 2005 ; Deguilloux et al.
2009). Cependant, il convient de signaler que les extractions ADN ont
été réalisées à partir d'échantillons
très particuliers (dents intactes en place) et que cette information ne
sera peut-être pas valable pour des prélèvements d'os
(Deguilloux et al. 2009).
Les contaminations observées lors de nos analyses sont
alors issues d'autres sources (réactifs, séquences
préalablement analysées au laboratoire, manipulateurs du
laboratoire).
La séquence associant les mutations 16223 CT, 16256 CT
et 16270 retrouvées dans les derniers témoins a été
identifiée et répertoriée au labo comme contaminante des
réactifs PCR. D'autres contaminations de ce type
(précédemment répertoriées) n'ont pas
été mises en évidence. Aucune contamination via les
manipulateurs du laboratoire n'a été détectée.
Deux séquences comprenant, la mutation 16220 AG pour
l'une et la mutation 16258 AG pour l'autre, ont été
retrouvées dans plusieurs témoins. Selon les études
menées au labo, ces
contaminants proviendraient des lots de primers. Enfin, la
séquence comportant la mutation 16298 TC observée dans les
témoins aérosol est également à répertorier
comme séquence contaminante retrouvée au sein du laboratoire.
Comme nous l'avons montré dans le chapitre
synthèse bibliographique, beaucoup d'études publiées
portant sur l'ADN ancien ne respectent pas toutes les précautions
nécessaires afin de limiter au maximum la dégradation de l'ADN
ainsi que les contaminations par de l'ADN moderne (précautions lors des
prélèvements, congélation des échantillons,
multiples témoins, répétitions dans quelques cas,
clonage). Toutes ces mesures ont été prises dans le cadre de
cette étude de faisabilité ce qui explique sans doute la
qualité des résultats obtenus.
Pour résumer, l'ADN mitochondrial semble globalement
bien conservé et aucune contamination par des fouilleurs et
manipulateurs n'a été mise en évidence. Par
conséquent, des analyses complémentaires peuvent être
envisagées. Dans un premier temps, les résultats obtenus dans le
cadre de cette étude de faisabilité devront être
authentifiés par des répétitions (autres extractions et
amplifications). Si ces nouvelles analyses s'avèrent fructueuses, nous
pourrons nous tourner vers l'ADN nucléaire avec l'analyse des STR. Comme
nous l'avons dit précédemment, ces marqueurs permettront
d'identifier des relations paternelles et de type parent-enfant.
2. Haplogroupes retrouvés
Ces analyses préliminaires ont permis de
caractériser deux haplogroupes européens différents J et X
chez trois individus au moins. La mise en évidence de mutations
diagnostiques dans ces séquences anciennes nous laisse confiants quant
à l'attribution de ces haplogroupes.
Pour deux individus (présentant la même
séquence), il n'a pas été possible de préciser
l'haplogroupe à partir des séquences HVRI seules, mais il
pourrait s'agir de l'haplogroupe H ou K, tous deux européens.
- L'haplogroupe J
La sépulture 169 présente deux individus
d'haplogroupe J mais d'haplotypes différents.
Celui-ci pourrait également être présent
chez le 3ème inhumé de la sépulture 171 mais
des analyses complémentaires des fragments A et B seraient
nécessaires afin de le confirmer.
Cet haplogroupe J serait apparu au Moyen Orient il y a environ
45 000 ans. Il serait aujourd'hui majoritairement européen. Le graphique
ci-dessous (fig.10) présente les fréquences de cet haplogroupe
retrouvées en Europe de nos jours (Logan 2009).
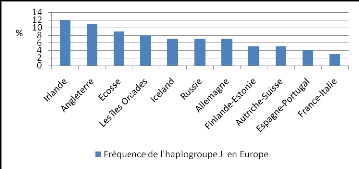
Figure 10 : Fréquence actuelle de
l'haplogroupe J en Europe d'après Logan (2009).
Une étude phylogéographique menée en
France montre que l'haplogroupe J serait aujourd'hui essentiellement
retrouvé à l'Ouest. La fréquence la plus importante de
celui-ci serait observée en Bretagne (Finistère) avec 9%. On le
retrouverait également en Centre Ouest (Corrèze, Dordogne), ou il
serait un peu moins présent, avec une fréquence de 4% (Dubut
2004). Il convient toutefois d'ajouter que les estimations de cette
étude sont très limitées du fait du très faible
échantillonnage réalisé, concernant seulement 3
régions françaises et 50 individus, ces informations sont donc
à prendre avec précautions.
Le blast de séquences endogènes obtenues pour le
troisième individu nous renvoie à plusieurs études dans
lesquelles sont retrouvées des séquences homologues à
100%. Ces séquences sont retrouvées en Europe du Nord
(Royaume-Uni, Islande) ainsi qu'en Hongrie.
- Haplogroupe H ou K ?
La mutation 16224 TC retrouvée chez les deux premiers
inhumés de la sépulture 170 renvoie aux deux haplogroupes K et H
qui sont tous deux majoritairement observés en Europe de l'Ouest et du
Nord (Richards 2000). En Europe, l'haplogroupe K, apparu il y a environ 50 000
ans, est majoritairement présent en Scandinavie. En France, celui-ci est
plus particulièrement observé en Bretagne ainsi qu'en
Centre-Ouest (Dubut 2004). Cependant, là
aussi ces estimations sont limitées par le faible
échantillon concerné. L'haplogroupe H, quant à lui, est
l'haplogroupe le plus présent en Europe actuellement avec une
fréquence variant de 35 à 55 % suivant les pays et les
régions (Richards 2000).
- L'haplogroupe X
L'haplogroupe X retrouvé chez le 1er
inhumé de la sépulture 171 est un haplogroupe assez rare mais
présent dans beaucoup de région du monde. L'haplogroupe X dans
son ensemble représente environ 2 % de la population d'Europe, du
Proche-Orient et d'Afrique du Nord. Le sous-groupe X2,
spécifiquement européen et à priori absent de la majeure
partie de l'Asie continental mais se retrouve également parmi les cinq
haplogroupes majeurs des populations indigènes d'Amérique. Le
graphique ci-dessous présente la fréquence de cet haplogroupe en
Europe (fig.11) (Logan 2009).
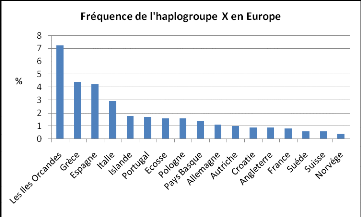
Figure 11 : Fréquence de l'haplogroupe X en
Europe d'après Logan (2009).
En France, l'haplogroupe X est uniquement présent dans
le Nord (Bretagne et Nord-Pas de calais) à une fréquence
d'environ 0,4% (Dubut 2004). Ces estimations se basent sur un
échantillon très réduit, elles n'ont donc qu'une valeur
indicative et très approximative.
Les mutations présentes chez le 1er
inhumé du sarcophage 171 ne permettent pas d'obtenir un haplotype
précis car celles-ci sont présentes au sein des deux sous groupes
X1 et X2. D'autres régions de l'ADN mt seraient nécessaires afin
de pouvoir acquérir cette information.
- Cas des deux derniers inhumés de la sépulture
171
La détermination d'haplogroupe chez les deux derniers
inhumés de la sépulture 171 n'a pas été possible.
Comme nous l'avons dit précédemment, l'hypothèse selon
laquelle le dernier inhumé de ce sarcophage serait d'haplogroupe J1B1
par l'association des mutations 16069 CT, 16126 TC, 16145 CT, 16222 CT et 16261
CT serait également envisageable. On resterait alors dans une
continuité d'haplogroupe J. Des analyses complémentaires portant
sur les fragments A et B seraient nécessaires afin de clarifier cela.
En résumé, nous obtenons dans le cadre de cette
étude, des haplogroupes s'intégrant parfaitement dans le pool
génétique européen, peut-être plus
particulièrement du Nord-ouest du continent. Ceci paraît donc tout
à fait logique et attendu et cela pourrait être un argument
supplémentaire d'authenticité des séquences obtenues.
3. Liens de parentés
Comme nous l'avons dit précédemment, les
séquences obtenues pour les deux premiers inhumés de la
sépulture 170 paraissent authentiques. On observe l'unique mutation
16224 TC chez ces deux individus sur deux ou trois fragments. On peut donc
d'ores et déjà penser que ces deux individus sont
apparentés du coté maternel.
Cependant, la similitude génétique de ces deux
individus pourrait être expliquée par une deuxième
éventualité, jusque là non abordée dans la
littérature. En effet, les os de la 1ere inhumée auraient pu
être contaminés par les « jus organiques » produits par
la deuxième inhumée lors de sa décomposition. Cette
hypothèse ne peut pas être exclue irrévocablement,
néanmoins, la qualité des prélèvements (dents
intactes et en place) ne permet pas d'envisager sérieusement cette
possibilité. La logique privilégie donc l'hypothèse selon
laquelle les deux individus seraient bien apparentés maternellement.
Les deux individus, une adolescente de sexe féminin
ainsi qu'une femme âgée entre 20 et 30 ans, pourraient avoir eu
des relations de plusieurs types, tel que mère-fille, soeurs, tante-
nièce ou encore des relations bien plus éloignées
(fig.12).
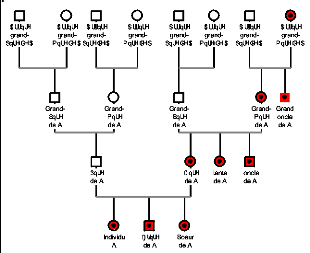
Figure 12 : Arbre généalogique
montrant quels individus sont issus d'une même lignée
mitochondriale. Les individus en rouge présentent le même
haplogroupe mitochondriale que l'individu A.
Les méthodes archéothanalogiques
pratiquées sur le chantier ont démontré que l'adolescente
aurait été inhumée en 1er. Puis ces os auraient
été réduits afin de fournir de l'espace à l'autre
jeune femme. Malheureusement, il n'existe aucun argument taphonomique
permettant d'obtenir un ordre de grandeur quant au temps séparant les
deux inhumations. La décomposition du corps peut s'être faite
aussi bien en quelques mois qu'en quelques années. Il n'est donc pas
possible d'affiner nos hypothèses concernant le type de lien unissant
ces deux femmes. Des analyses STR pourraient permettre de mettre en
évidence des relations de type mère-fille ou soeurs, si cela est
le cas.
La présence du périnatal dans ce sarcophage
reste toujours un mystère, puisque malheureusement les analyses
génétiques n'ont rien fournit pour cet individu.
À cette époque, les enfants n'étaient
baptisés qu'à l'âge de 3 ou 4 ans, lorsqu'on était
certain que l'enfant était en bonne santé et allait vivre, car un
baptême coûtait fort cher. Les enfants décédés
sans être baptisés étaient donc enterrés en tant que
non-chrétiens, hors de l'enclos sacré. Il se pose alors toujours
la question de la présence de ce périnatal au sein de cette
nécropole et surtout au sein d'un sarcophage. L'hypothèse la plus
plausible expliquant cette pratique est que ce bébé aurait
été déposé aux coté de l'un de ses parents.
Au vu des arguments archéothanatologiques énoncés dans la
première partie du mémoire, ce périnatal aurait pu
être l'enfant de la femme (2nde inhumée) située
à ses cotés. Si tel était le cas ce sarcophage
regrouperait donc trois personnes issues d'une même famille.
Cette étude a permis le rapprochement biologique de
deux individus seulement mais certains résultats fragmentaires de ces
analyses ne permettent pas d'écarter catégoriquement d'autres
liens de parenté maternels entre d'autres individus, au sein des
sarcophages et entre les sarcophages. Toutefois, au sein des tombes, nous
pouvons d'ores et déjà exclure tous liens de parenté
maternels entre les premier et troisième inhumés de la
sépulture 169 ainsi qu'entre les premier et troisième
inhumé de la sépulture 171 (fig.13). Nous pouvons donc aussi
écarter tous lien de parenté entre ces quatre individus.
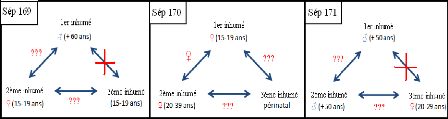
Figure 13 : Informations acquises en termes de
relations de parenté maternelles.
X : aucun lien de parenté maternel ; ??? : Pas de
liens maternels démontrés ; ? : liens maternels
démontrés.
Ces analyses paléogénétiques très
préliminaires permettent de mettre en évidence 2 cas de figures
distincts :
Nous retrouvons des individus apparentés maternellement
déposés dans le même sarcophage mais aussi des individus
non apparentés maternellement déposés dans le même
sarcophage. Cette information nous montre que l'on ne peut pas forcément
globaliser les résultats. En effet, la mise en évidence de
relations de parenté au sein d'un sarcophage ne veut pas dire que cela
est forcément le cas pour le sarcophage voisin. Nous sommes donc peut
être face à des gestes funéraires différents.
Ces dernières informations posent la question de
l'apport des données paléogénétiques : sont-elles
potentiellement généralisables à partir d'un site, d'un
sarcophage, d'un groupe dans la nécropole ? La synthèse
bibliographique présentée précédemment nous montre
bien que dans la plupart des sites étudiés, il n'est pas possible
de généraliser les phénomènes de regroupements
familiaux car ceux-ci sont souvent propres à une seule sépulture
ou à un secteur particulier.
VI Conclusion et perspectives
L'analyse génétique préliminaire de neuf
individus issus de trois sarcophages du site de Jau-Dignac et Loirac a permis
de mener à bien les objectifs principaux de cette étude qui
était de tester la conservation de l'ADN et les contaminations.
Nous avons ainsi mis en évidence une bonne conservation
générale de l'ADN chez ces individus. Dans l'ensemble, peu de
contaminations ont été observées et les rares
retrouvées ne semblent pas provenir des fouilleurs et manipulateurs du
laboratoire. Nous pouvons donc affirmer que l'absence de port de gant lors de
certains prélèvements n'a pas été reconnue comme
geste contaminant dans le cadre de cette étude.
Concernant les résultats obtenus, nous avons
trouvé de façon fiable la présence d'ADN authentique mais
fragmentaire chez au moins cinq de ces individus (tabl. 9).
La bonne qualité des résultats nous laisse donc
envisager la possibilité de poursuivre cette étude de lien de
parenté par des analyses complémentaires. Dans cette
éventualité, des répétitions (multiples extractions
et amplifications) devront être tout d'abord réalisées sur
le génome mitochondrial afin d'authentifier les résultats de la
présente étude. Par la suite, des analyses portant sur l'ADN
nucléaire (STR) pourront être considérés afin de
pouvoir repérer d'éventuels liens de parenté paternels et
de type parents-enfants.
Toutefois, il convient de signaler que l'analyse des relations
de parenté entre vestiges humains anciens reste l'application de la
paléogénétique la plus complexe et la plus
problématique. En effet, l'obtention de marqueurs hautement informatifs
pour les relations familiales (les STR autosomaux) ne reste qu'exceptionnelle
sur des restes humains (Gill et al. 1994 ; Keyser-Tracqui et
al. 2003 ; Deguilloux et al. 2009). Ceci est lié à
une mauvaise conservation du génome nucléaire dans les vestiges
anciens, ainsi qu'à la difficulté de leur analyse du fait de la
dégradation de l'ADN (problème de « slippage » ou
dérapage durant l'amplification de ces séquences
répétées). En conséquence, les analyses
paléogénétiques ne s'arrêtent souvent qu'à
l'information partielle fournie par le génome mitochondrial (Capellini
et al. 2004 ; Mooder et al. 2005 ; Rudbeck et al.
2005 ; Deguilloux et al. 2009).
A ce stade des analyses, nous pouvons toutefois apporter
quelques informations en termes de relations de parenté maternelles. Les
résultats obtenus montrent de façon fiable le partage d'une
lignée maternelle par deux individus issus d'un même sarcophage.
L'haplogroupe de ces individus (deux femmes) ne peut cependant pas être
précisé car, compte tenu des données
dont nous disposons, il pourrait tout aussi bien s'agir de
l'haplogroupe K que de l'haplogroupe H, tous deux présents en Europe du
Nord et de l'Ouest. Le type de relation précis unissant ces deux femmes
est une information inaccessible à ce stade de l'étude mais
celle-ci pourrait peut-être être apportée par les STR, si la
conservation de l'ADN s'y prête.
Compte tenu du caractère fragmentaire des
résultats, certains liens de parenté maternels entre individus ne
peuvent pas être exclus mais nous pouvons d'ores et déjà
affirmer, qu'au sein de deux sarcophages sur trois, deux individus sur trois
présentent des lignées maternelles différentes.
Pour terminer, il convient de souligner la difficulté
de ce type d'étude de liens de parenté où se pose le
problème non négligeable de la généralisation des
résultats partiels. En effet, ce type d'étude se base souvent sur
un nombre relativement restreint d'individus et il apparaît très
imprudent de vouloir généraliser ces résultats à
l'ensemble d'une nécropole, d'une période, d'une culture.
Annexe A : protocoles d'analyses d'ADN ancien
1- PREPARATION DES ECHANTILLONS
Ensacher dans un double sachet stérile les fragments
d'échantillons ou échantillons. Bien laver la hotte à la
javel entre chaque échantillon.
Mettre 15min les UV avant chaque manip/chaque
échantillon
Manipuler avec gants, masque et blouse.
Emmener les sachets dans la salle ADNa, les nettoyer avec un
papier imbibé de javel dans le sas et une nouvelle fois dans la salle
extraction.
2- EXTRACTION PHENOL-CHLOROFORME
LA VEILLE
1- préparation de la salle = lavage paillasse à la
javel, préparation des papiers alu + scalpels + spatules, exposition des
échantillons aux UV et laisser la nuit au UV
2- préparation du tampon de base (volume final 50mL) :
9,3g EDTA O,5M (EDTA = chelateur d'ions, les cations
libérés par le broyage vont se fixer sur l'edta et pas sur
l'adn). de plus, L'EDTA en chelatant les ions Ca2+ va favoriser la disparition
des complexes proteiques impliques dans les contacts cellule-cellule tels que
jonctions serres et autres desmosomes. Les protéines impliques dans ces
complexes sont dépendantes du Ca2+.
1,6g NaOH (pH=8), lyse les cellules par un détergent
en milieu alcalin (SDSNaOH) afin de libérer l'ADN génomique et
plasmidique. L'ADN génomique et les protéines sont
précipitées par de l'acétate de sodium
qsp eau ultra-pure
3- préparation de la solution stock protéi nase K
: 1mL dans 25mg de poudre.
Elle hydrolyse les protéines de toutes origines en
quelques heures avec une préférence pour les liaisons peptidiques
situées après les acides aminés hydrophobes (leucine, par
exemple)
LE JOUR-J
1- allumer le four hybridation (55°C)
2- Préparer le tampon de lyse
(10mL/échantillon) dans un tube Falcon 15mL
Tampon de base 1 0mL
N-Lauryl Sarcosyl 10% 500uL inactive les activités
enzymatiques
Protéinase K (solution stock) 100uL
3- (laver et) broyer l'échantillon
(papier alu + papier + papier alu dans sachet)
4- récupérer 0,5-1g de poudre dans
un tube Falcon 15mL
5- fermer les tubes avec plusieurs
épaisseurs de parafilm
6- incuber toute la nuit à 55°C,
sous agitation (à l'abri des UV)
LE JOUR-J+1
Allumer la hotte chimique !!
1- centrifuger les tubes 10min à 800rpm
2- préparer xX3 tubes Falcon 50mL contenant 10mL de
Phénol -Chlo-IAA chacun
3- transférer le surnageant dans les tubes Falcon
contenant le phénol-chlo, fermer avec du parafilm et
homogénéiser le tout doucement
4- centrifuger les tubes 10-15min à 1200rpm
5- répéter les étapes 2 à 4 deux
fois
0- transférer environ 2mL du surnageant
final sur les centricons (colonnes sur grands tubes fournis) et fermer à
moitié avec du parafilm
7- centrifuger 2-5h à 3000rpm
8- jeter l'éluât, retourner la colonne sur les
petits tubes fournis et ajouter 100uL d'eau sur la colonne
9- centrifuger 10min à 3000rpm
10- récupérer les éluats dans des
eppendorfs
3- AMPLIFICATION
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20
|
|
12 14 16 18 20 22 24 26
r
30 32 34 36 38
U
1,5 1,75 2 2,25 2,5 2,75 3 3,25 3,5 3,75 4 4,25 4,5 4,75
L
H2O d16NT9P2 149 165 182 12235 m30
Tampon 10 X 2,5 5 7,5 10 12,5 15 17,5 20 22,5 25 27,5 30 32,5 35
37,5 40 42,5 45 47,5 50
MgCl2 25 mM 2 4 6 8 10
BSA 20 mg/mL 1,25 2,5 3,75 5 6,25 7,5 8,75 10 11,3 12,5 13,8 15
16,3 17,5 18,8 20 21,3 22,5 23,8 25
dNTP 25 mM chaque 0,25 0,5 0,75 1 1,25
amorce Upper 0,5 1 1,5 2 2,5 3 3,5 4 4,5 5 5,5 6 6,5 7 7,5 8 8,5
9 9,5 10
amorce Louver 0,5 1 1,5 2 2,5 3 3,5 4 4,5 5 5,5 6 6,5 7 7,5 8
8,5 9 9,5 10
Taq 5 U/mL 0,5 1 1,5 2 2,5 3 3,5 4 4,5 5 5,5 6 6,5 7 7,5 8 8,5 9
9,5 10
|


4- VERIFICATION AMPLIFICATION
Faire migrer sur un gel d'agarose 2u L de bleu + 8u L de produit
PCR
Prendre une photo
5- CLONAGE
Préparation des boîtes de culture
- diluer grossièrement le LB agar dans l'eau
- autoclaver la bouteille de mi lieu 15min à 121°C
- ajouter l'ampicilline lorsque le liquide est tiède (pas
trop chaud)
- Couler dans les boîtes (assez épais)... du coup
avec le mélange pour 20 boites, on en fait 12
épaisses
|
nombre de boîtes
|
eau (mL)
|
LB (g)
|
ampicilline (pi.)
|
|
1
|
25
|
0,8
|
12,5
|
|
2
|
50
|
1,6
|
25
|
|
3
|
75
|
2,4
|
37,5
|
|
4
|
100
|
3,2
|
50
|
|
5
|
125
|
4
|
62,5
|
|
6
|
150
|
4,8
|
75
|
|
7
|
175
|
5,6
|
87,5
|
|
8
|
200
|
6,4
|
100
|
|
9
|
225
|
7,2
|
112,5
|
|
10
|
250
|
8
|
125
|
|
11
|
275
|
8,8
|
137,5
|
|
12
|
300
|
9,6
|
150
|
|
13
|
325
|
10,4
|
162,5
|
|
14
|
350
|
11,2
|
175
|
|
15
|
375
|
12
|
187,5
|
|
16
|
400
|
12,8
|
200
|
|
17
|
425
|
13,6
|
212,5
|
|
18
|
450
|
14,4
|
225
|
|
19
|
475
|
15,2
|
237,5
|
|
20
|
500
|
16
|
250
|
- laisser refroidir sur la pà llasse et mettre au frigo
(entourées de papier alu
La solution stock d'ampicilline doit être à 10mg/mL
(20mL sur 200mg) et être conservée au
frigo.
Ligation - clonage
- Préparer la réaction de ligation (volume final
3uL): ingrédients conservés à -20°C 1 uL de
produit PCR
0,5 uL de Salt solution
1 uL eau pure
0,5uL de TOPO vector
- Mélanger doucement et laisser incuber 5-30 min sur la
paillasse
- Pendant l'incubation, préparer les bactéries :
. Préparer la glace
. Allumer le bain-marie à 42°C
. Décongeler dans la glace 1 tube de
bactéries pour 2 réactions (aliquoter 30uL dans
2 eppendorfs et les placer dans la glace)
. Sortir le SOC du frigo (125 uL par
réaction)
- Après incubation de la réaction de clonage,
ajouter 1 uL de réaction de ligation à chaque tube de
bactérie (conservation du rab' du produit de ligation au
congélateur pour réemploi)
- Laisser les tubes de bactéries transformées dans
la glace 5-30 min
- Plonger les tubes 30s au bain-marie à 42°C pour le
choc thermique
- Transférer immédiatement dans la glace quelques
minutes
- Hors glace, ajouter 125 uL de SOC
- Mettre sous agitation 1h, à 37°C, avec les
boites
- Etaler 50uL de produit de transformation sur les boites, autour
du bec bunzen (nettoyage paillasse alcool 70°c)
- incuber les boites retournées, à 37°c, toute
la nuit (max. 18h puis à 4°C)
DECHETS !
- autoclaver les boites pétri dans les sacs biohazard
- stocker les cônes/eppendorfs dans une poubelle remplie de
javel puis dans les sacs biohazard jusqu'à l'autoclave
5- VERIFICATION DES CLONES
- préparer une plaque avec 50uL d'eau ultra-pure dans
chaque puit
- piquer un cône dans une colonie puis le laisser tremper
dans les 50uL d'eau (dans plaque) = produit stock ADN clone. On
récupère 24 clones par produit PCR
- Préparer le mix PCR sous la hotte
Pour 35uL final : 15uL MasterMix 2.5x
0.375uL M13FOR1 (10uM)
0.375uL M13REV1 (10uM)
14.25uL H2O
5uL ADN
- dispatcher le mix dans la plaque/ la barrette (30uL par puits)
et ajouter 5uL de produit stock ADN clone.
- Fermer avec un autocollant alu la plaque PCR. Fermer avec un
autocollant plastique la plaque des stocks ADN clone.
- lancer la PCR : programme M 13
- vérifier la taille des inserts sur gel d'agarose (5uL
produit PCR + 2uL bleu)
- envoyer 30uL de produit PCR + additionnée de 10uL d'eau
à Génome Express, dans des eppendorfs (IECB mardi matin 10h)
Bibliographie
ADACHI, N., UMETSU, K., TAKIGAWA, W. et SAKAUE, K. 2004
-- Phylogenetic analysis of the human ancient mitochondrial
DNA. Journal of Archaeological Science, 31, p. 1339-1348.
BARRIEL,V. 1997 - L'ADN fossile [en ligne]. «
http://www.cnrs.fr/cw/dossiers/dosevol/
decouv/articles/chap7/barriel .html ». 20 Avril 2009.
BOUWMAN, A. S., BROWN, K. A., PRAG, A. J. N. W. et BROWN, T.
A. 2008 -- Kinship between burials from Grave Circle B at
Mycenae revealed by ancient DNA typing. Journal of Archaeological
Science, 35, p. 2580-2584.
CAPPELLINI, E., CHIARELLI, B., SINEO, L., CASOLI, A., GIOIA,
A. D., VERNESI, C., BIELLA, M. C. et CARAMELLI, D. 2004 --
Biomolecular study of the human remains from tomb 5859 in the Etruscan
necropolis of Monterozzi, Tarquinia (Viterbo, Italy). Journal of
Archaeological Science, 31, p. 603-612.
CARTRON, I. et CASTEX, D. 2006 - Jau-Dignac et Loirac
(Gironde) "La Chapelle Saint- Siméon". Rapport final d'opération
(2001/2003-2005). Bordeaux : Service régional de
L'archéologie d'Aquitaine, 373 p.
CASTEX, D. et CARTRON, I. 2006 - L'occupation d'un ancien
îlot de l'estuairede la Gironde : du temple antique à la chapelle
Saint-Siméon (Jau-Dignac-et-Loirac). Aquitania, XXII, p.
253-282.
CASTEX, D. et CARTRON, I. 2009 - Identité et
mémoire d'un groupe aristocratique du haut
moyen âge : le site
de «la chapelle» à jau-Dignac et Loirac (gironde). In ALDUC
LE
BAGOUSSE A.
(Edi,-IIÇKXmEFIRÇs-IdI-ISOEPTUe-IRX-ISOEPTHe-IIe-It'iÇKXPEFIRÇ
? Expressions du SRXYRII-IdEÇs-It'EX-delà. Actes de la table
ronde de Caen (23-24 Mars 2007), Centre de recherches
Archéologiques et Historiques Médiévales, sous presse.
CIPOLLARO, M., DI BERNARDO, G., GALANO, G., GALDERISI, U.,
GUARINO, F., ANGELINI, F. ET CASCINO, A. 1998 -- Ancient DNA
in Human Bone Remains from Pompeii Archaeological Site. Biochemical and
Biophysical Research Communications, 247, p. 901-904.
CLISSON, I., KEYSER-TRACQUI, C., FRANCFORT, H. P., CRUBEZY,
E., SAMASHEV, Z. et LUDES, B. 2002 -- Genetic analysis of
human remains from a double inhumation in a frozen kurgan in Kazakhstan (Berel
site, Early 3rd Century BC). International Journal of Legal Medicine,
116, p. 304-308.
COOPER, A. et POINAR, H. N. 2000 -- Ancient DNA
: Do It Right or Not at All. Science, 289, 1139 p.
DEGUILLOUX, M. F., MARCHET, A., BESSOU, M., PEMONGE, M. H.,
GUILLON, M. et WOSNY, L. 2009 - Analyse paléogénétique des
sépultures du groupe D du site du Peyrat In Le site du Peyrat au Moyen
Âge du Vè au XIVè siècle ap. J.-C. Aquitania.
(Sous presse).
DUBUT, V., CHOLLET, L., MURAIL, P., CARTAULT, F.,
BÉRAUD-COLOMB, E., SERRE, M. et MOGENTALE-PROFIZI, N. 2004 - mtDNA
polymorphisms in five French groups : importance of regional sampling.
European Journal of Human Genetics, 12, p. 293- 300.
DUDAR, J.C., WAYE, J. S. et SAUNDERS, R. 2003 - Determination
of a Kinship SystemUsing Ancient DNA, Mortuary Practice et Historic Records
inan Upper Canadian Pioneer Cemetery. International Journal of
Osteoarchaeology, 13, p. 232-246.
DUDAY H. 2005 - L'archéothanatologie ou
archéologie de la mort. In : DUTOUR O.; HUBLIN J.J ET VETERMEERSCH B.
(Eds.), Objets et méthodes en apléoanthropologie. Paris
: Comité des travaux historiques et scientifiques, p. 153-207.
GILL, P., IVANOV P.L., KIMPTON, C., PIERCY, R., BENSON, N.,
TULLY, G., EVETT, E., HAGELBERG, E. et SULLIVAN, K. 1994 - Identification of
the remains of the Romanov family by DNA analysis. Nature genetics, 6,
p. 130-135.
GLEIZE, Y. 2006 - Gestion des corps, gestion de morts.
Analyse archéo-anthropologique de réutilisations de tombes et de
manipulations d'ossements en contexte funéraire au début du
Moyen-Age (entre Loire et Garonne, V-VIIIème siècle).
Université de Bordeaux 1 : Thèse de doctorat, 632 p.
HAAK, W., BRETT, G., JONG, H. N. D., MEYER, C., GANSLMEIER,
R., HEYD, V., HAWKESWORTH, C., PIKEB, A. W. G., MELLER, H. et ALT, K. W. 2008 -
Ancient DNA, Strontium isotopes, et osteological analyses shed light on social
et kinship organization of the Later Stone Age. P.N.A.S, 105, p.
18226-18231.
KEYSER-TRACQUI, C., CRUBEZY, E. et LUDES, B. 2003 - Nuclear et
Mitochondrial DNA Analysis of a 2,000-Year-Old Necropolis in the Egyin Gol
Valley of Mongolia. American journal of human genetic, 73, p.
247-260.
LAFOREST, C. 2008 - Apport des variations non métriques
dentaires : caractérisation et comparaison des populations du site " La
chapelle" de Jau-dignac et Loirac (Gironde). Université de Bordeaux1 :
Mémoire de Master 2, 72 p.
LOGAN, I. 2009 - mtDNA [en ligne]. «
http://www.ianlogan.co.uk/mtdna.htm
». 20 Avril 2009.
MOODER, K., WEBER, A. W., BAMFORTH, F. J., LIEVERSE, A. R.,
SCHURR, T. G., BAZALIISKI, V. I. et SAVEL'EV, N. A. 2005 - Matrilineal
affinities et prehistoric Siberian mortuary practices : a case study from
Neolithic Lake Baikal. Journal of Archaeological Science, 32, p. 61
9-634.
POINAR, H. N. 2003 - The top 10 list: criteria of authenticity
for DNA from ancient et forensic samples. In). International
Congress Series 1239, p. 575-579.
PRUVOST, M., SCHWARZ, R., BESSA-CORREIA, V., CHAMPLOTA, S.,
GRANGEA, T. et GEIGL, E. M. 2008 - DNA diagenesis et palaeogenetic analysis:
Critical assessment et methodological progress. Palaeogeography,
Palaeoclimatology, Palaeoecology, 266, p. 211- 219.
RICHARDS, M., MACAULAY, V., HICKEY, E., VEGA, E., SYKES, B.,
GUIDA, V., RENGO, C., SELLITTO, D., CRUCIANI, F., KIVISILD, T., VILLEMS, R.,
THOMAS, M., RYCHKOV, S., RYCHKOV, O., RYCHKOV, Y., GÖLGE, M., DIMITROV,
D., HILL, E., BRADLEY, D., ROMANO, V., CALÌ, F., VONA, G., DEMAINE, A.,
PAPIHA, S., TRIANTAPHYLLIDIS, C., STEFANESCU, G., HATINA, J., BELLEDI, M.,
RIENZO, A. D., NOVELLETTO, A., OPPENHEIM, A., NØRBY, S.,
SANTACHIARA-BENERECETTI, S., SCOZZARI, R., TORRONI, A. et BETELT, H.-J. 2000 -
Tracing European founder lineages in the Near Eastern mtDNA pool. American
Journal of Human Genetics, 67, p. 125 1- 1276.
ROUSSELET, F. et MANGIN, P. 1998 - Mitochondrial DNA
polymorphisms: a study of 50 French Caucasian individuals et application to
forensic casework. International Journal of Legal Medecine, 111, p.
292-298.
RUDBECK, L., THOMAS, M., GILBERT, P., WILLERSLEV, E., HANSEN,
A. J., LYNNERUP, N., CHRISTENSEN, T. et DISSING, J. 2005 - mtDNA Analysis of
Human Remains From an Early Danish Christian Cemetery. American Journal of
Physical Anthropology, 128, p. 424-429.
SCHULTES, T., HUMMEL, C. et HERRMANN, B. 1999 - Amplification of
Y-chromosomal STRs from ancient skeletal material. Human genetics,
104, p. 164-166.
SERK, P. 2004- Human mitochondrial DNA haplogroup J in europe
et near East. Université de Tartu: thèse de doctorat, 65
p.
SMITH, D. G., MALHI, R. S., ESHLEMAN, J., LORENZ, J. G. et
KAESTLE, F. A. 1999 - Distribution of mtDNA haplogroup X among Native North
Americans. American Journal of Physical Anthropology, 110, p.
271-284.
TABERLET, P., GRIFFIN, S., GOOSSENS, B., QUESTIAU, S.,
MANCEAU, V. et ESCARAVAGE, N. 1996 - Reliable genotyping of samples with very
low DNA quantities using PCR. Nucleic Acids Research, 24, p.
3189-3194.
THOMAS, M., GILBERT, P., BETELT, H. J., HOFREITER, M. et BARNES,
I. 2005 - Assessing ancient DNA studies. Trends in Ecology &
Evolution, 20, p. 541-544.
TREFFORT, C. 1996 - Du cimiterium christianorum au
cimetière paroissial : évolution des espaces funéraires en
Gaule du VIe au Xe siècle In FÉRACF (ed).
Archéologie du cimetière Chrétien - Actes du
2ème Colloque A.R. C.H.E.A. Orléans, p. 55-63.
WILLIAMS, S. R., CHAGNON, N. A. et SPIELMAN, R. S. 2002 -
Nuclear et Mitochondrial Genetic Variation in the Yanomamo·: A Test Case
for Ancient DNA Studies of Prehistoric Populations. American Journal of
Physical Anthropology, 117, p. 246-259.
YANG, D. Y. et WATT, K. 2005 - Contamination controls when
preparing archaeological remains for ancient DNA analysis. Journal of
Archaeological Science, 32, p. 33 1-336.
YANG, D. Y., ENG, B. et SAUNDERS, S. 2003 - Hypersensitive PCR,
Ancient Human mtDNA et Contamination. Human Biology, 75, p.
355-364.
YANG, H., GOLENBERG, E. M. et SHOSHANI, J. 1997- A Blind Testing
Design for Authenticating Ancient DNA Sequences. Molecular phylogenetics et
evolution, 7, p. 261-265.
| 


