|
|
|
|
République Tunisienne
Ministère de
l'Enseignement supérieur
De la recherche scientifique et de la
technologie
Université de Monastir
Institut supérieur de
biotechnologie de Monastir
Mémoire Bibliographique
Première année Mastère
Génétique et Biodiversité
|
|
|
Les éléments transposables chez
l'Homme
|
|
|
Présenté par :
Abdelbasset Amara
Soutenue en 2008
Année universitaire : 2007-
|
2008
|
|
|
« Nous ne pouvons achever ce travail sans rendre
grâce à dieu pour
son aide sa générosité et pour
la patience qu'il nous a donné pour
aboutir à ce stade »
Dédicace
A mes chers parents :
« Jamais je n'oublierai vos sacrifices pour moi, vous
étiez toujours avec moi par vos
encouragements et vos conseils. Que
vous trouviez dans ce travail une expression
de mon grand amour envers vous
et de ma grande reconnaissance ! Que dieu tout
puisant vous protége
et vous offre la santé et une longue vie ! »
A mes chers frères:
« Vous étiez toujours à mes
cotés. Je ne pourrais jamais imaginer ma vie sans vous.
Que dieu vous
garde, vous protège et vous offre une vie pleine de bonheur et
de
succès ! Que vous trouviez dans ce travail mes vifs sentiments
d'amour et
d'affection ! »
A tous les membres de ma famille et à tous mes
amis :
« Vous êtes toujours dans mon coeur. Je ne vous
oublierai jamais. Veuillez trouver
dans ce travail mon expression d'amour et
d'amitié envers vous ! »
Remerciement
A Messieurs et Mesdames, les membres de jury
:
« Je vous suis très reconnaissant pour avoir
accepté de juger ce travail
modeste. Qu'il me soit permis de vous
exprimer mes sentiments de respect, d'estime
et de gratitude»
Liste des abréviations
A : Adénine G :
Guanine U : Uracile T : Thymine C
: Cytosine ADN: Acide
Désoxyribonucléique
ARN: Acide Ribonucléique
ARNm: ARN messager
ARNt : ARN de transfert
env : enveloppe
ET : Elément Transposable
gag: group associated antigen
IR: Inverted Repeat (Repetition
Inversée)
IS: Insertion Sequence
ITR: Inverted Terminal Repeat
Kb: kilobases
LINE: Long Interspersed repetitive Element
LTR : Long Terminal Repeat
MITE: Miniature Inverted Repeat Transposables
Elements
ORF: Open Reading Frame
PBS : protein binding site
pb : paire de base
pol : polymérase
SAGE : Serial analysis of gene expression
SINE: Short Interpersed Nuclear Element
SVA: SINE VNTR Alu
SOX : sex related box
TIR : Terminal Inverted Repeats
Sommaire
Introduction 1
I. Les différents éléments
transposables 2
1. Définition 2
2. Les différentes classes d'éléments
transposables 2
2.1. Les éléments de classe I . 2
a. Les rétrotransposons avec LTR 3
b. Les rétrotransposans sans LTR . 4
a. 1 les éléments LINE 4
a.2 les éléments SINE 6
2.2. Les éléments de classe II 7
2.3. Les éléments de classe III . 7
II. Les éléments transposables actifs dans
le génome humain 8
III. Les interactions entre les éléments
transposables et le génome humain 9
1. Effets négatifs sur le génome 9
a. Les insertions 9
b. Les recombinaisons 9
c. Implications des ET dans des maladies 10
2. L'effet du génome sur les rétotransposons 11
3. Effets bénéfiques pour le génome 12
a. Rôles des ET dans la régulation de l'expression
des gènes 12
b. Rôles des ET dans la création de nouveaux
gènes . 13
c. Les éléments transposables et le système
immunitaire .. 13
d. Rôles des éléments transposables dans
l'évolution et la spéciation 14
IV. Importance des éléments transposables
dans la génétique .. 15
1. Marqueur de polymorphisme . 15
2. Utilisation dans la thérapie génique 15
Conclusion . 16
Références bibliographiques .. 17
Introduction
Pendant une longue période de temps le
génome était considéré comme une entité
statique. Bien que l'idée du changement brusque des gènes par
mutations soit acceptée, on estimait notamment que la localisation des
gènes sur les chromosomes était fixe sur de longues
périodes d'évolution, tout en admettant la possibilité de
changement par la recombinaison.
Cette approche changea après la découverte des
éléments transposables par BARBARA MCCLINTOCK dans les
années cinquante. Cette découverte n'a pas commencé
à être véritablement admise que lorsque de tels
éléments ont été trouvés tout d'abord chez
les bactéries (Shapiro 1969) puis chez la drosophile (Kidwell et al.
1977) et lorsque l'on s'est aperçu qu'une grande partie de la plupart
des génomes était constituée de séquences
répétées non codantes. Ainsi, la vision statique du
génome était abandonnée pour une vision dynamique recelant
un grand nombre de fluctuations.
Dans cette nouvelle approche, les éléments
transposables ont été considérés au début
comme de simples parasites (Orgel et Crick 1980). En effet, la transposition
leur permet une autoréplication en utilisant les ressources
moléculaires de l'hôte. Ainsi, sous cette hypothèse, seul
l'avantage réplicatif des ETs pourrait expliquer leur augmentation et
leur maintenance dans le génome et selon cette conception, les ET n'ont
que des effets négatifs sur leurs hôtes. Puis des études
ont suggéré qu'ils pouvaient jouer un rôle fondamental dans
l'évolution des génomes et l'augmentation du pouvoir adaptatif de
certains individus. (Orgel et Crick 1980)
Actuellement, de nombreuses études ont pour but
d'étudier les ETs d'un point de vue structural et fonctionnel, ainsi que
leurs effets sur les génomes hôtes car ils apparaissent de plus en
plus comme des composés fondamentaux des génomes.
Les questions principales abordées dans ce travail ont
pour objectif de présenter les différentes familles
d'éléments transposables et d'étudier comment ces
éléments transposables interagissent avec le génome humain
et quelles sont les conséquences qu'ils entraînent ?
I. Les différents éléments
transposables :
1. Définition :
Les éléments transposables sont classiquement
définis comme des séquences moyennement
répétées qui ont la capacité de se déplacer
d'une position à une autre. Ce mouvement est appelé
transposition. Jusqu'à présent, les ETs ont été
trouvés dans tous les organismes vivants (procaryotes et eucaryotes). Au
sein d'un même génome, on peut trouver des éléments
de type différent. Le nombre de copies des ETs peut varier de moins
d'une dizaine à plusieurs centaines de milliers selon le type
d'éléments et l'espèce hôte. (Orgel et Crick
1980)
Par exemple, chez le maïs, ils représentent plus
de 50% du génome total (SanMiguel et al. 1996) alors que chez la levure
ils ne forment que 3% du génome (Kim et al. 1998) et ils
occupent 44% du génome chez l'homme. (Mills et al 2007)
Lors de leurs déplacements, ils peuvent
s'insérer dans les gènes ou dans des régions
régulatrices et être responsables de mutations pouvant être
délétères ou non. Ils sont aussi responsables d'importants
remaniements chromosomiques comme des délétions, des inversions
et des translocations (Evgen'ev et al. 2000). Ils sont donc une source
de variations génétiques importantes. Ainsi, les ETs ont un
impact important sur leur génome hôte. Malgré la
caractéristique commune qui est de dupliquer leur site cible lors de
leur insertion, les ETs ont des structures pouvant être très
différentes. On peut ainsi les caractériser selon leur structure
et selon l'intermédiaire qu'ils utilisent pour se déplacer
(Finnegan 1992).
Les éléments autonomes possèdent des
gènes codant pour des protéines nécessaires à leur
déplacement. On trouve aussi un certain nombre d'éléments
non autonomes qui dépendent des éléments autonomes pour
leur transposition.
2. Les différentes classes
d'éléments transposables :
Selon leur diversité structurale et leur mode de
transposition, Les ET se répartissent en trois principaux groupes : les
éléments de classe I ou rétrotransposons, les
éléments de classe II ou transposons, et les
éléments de classe III.
2.1 Les éléments de classe I :
Les éléments transposables de classe I ou les
rétrotransposons sont des éléments qui se déplacent
via un intermédiaire ARN selon un modèle réplicatif ou un
modèle copier-coller. Ils sont subdivisés en deux sous-classes,
selon qu'ils possèdent ou pas des séquences (LTR) « Long
Terminal Repeat ».
a. Les rétrotransposons avec LTR :
Ce sont des éléments de 5 à 9 kb
encadrés de longues séquences (de quelques centaines de bases)
terminales répétées en orientation directe connues sous le
nom de LTR. Ces séquences contiennent les régions promotrices et
régulatrices des séquences codantes de l'élément et
comportent trois domaines fonctionnels : U3, R et U5 (Figure 1)
Lors de leur transposition, les ARN messager des
rétrotransposons à LTR sont tout d'abord rétrotranscrits
par une transcriptase reverse avant d'être insérés dans le
génome. De point de vue organisation, ces éléments sont
proches des rétrovirus (Capy 2005). Ainsi, on retrouve principalement
deux ORFs (Open Reading Frame) présents chez les rétrovirus : les
gènes gag et pol. Chez les rétrovirus, le
gène gag code pour une polyprotéine qui donne trois
protéines matures : la protéine de la matrice, la protéine
de la capside et la protéine de la nucléocapside qui composent la
capside virale (Warmus et Brown 1989). Chez les rétrotransposons,
l'utilité du gène gag n'est pas encore
élucidée. Le gène pol code pour une
polyprotéine qui, après protéolyse, forme les
protéines requises pour la transposition, notamment une protéase,
une transcriptase inverse nécessaire à la
rétrotranscription, une Rnase H et une intégrase, qui permet
l'insertion de l'élément dans le génome. Les
rétrotransposons à LTR sont subdivisés en deux groupes
principaux suivant l'ordre des domaines protéiques du gène
pol. Chez les éléments de type Ty1/copia,
l'intégrase se trouve du côté 5' de la transcriptase
inverse alors que chez les éléments de type Ty3, elle se
place du côté 3' (voir Figure 1). Chez certains
rétrotransposons à LTR du groupe gypsy/Ty3, on trouve
aussi une troisième ORF, le gène env. (Warmus et Brown
1989). Les rétrovirus possèdent ce gène qui code pour une
polyprotéine formant après protéolyse deux
protéines : une transmembranaire (TM) et une extracellulaire (SU). Ces
protéines permettent l'adsorption et la pénétration dans
la cellule cible du rétrovirus (Warmus et Brown 1989).
Ces différentes études suggèrent un lien
étroit entre rétrovirus et rétrotransposons à LTR.
L'hypothèse la plus couramment avancée est l'apparition de
rétrovirus à partir de rétrotransposons à LTR ayant
acquis un gène d'enveloppe (Temin 1980). Cependant, on ne peut exclure
le phénomène inverse : un rétrovirus perdant son
gène d'enveloppe devient un rétrotransposon.
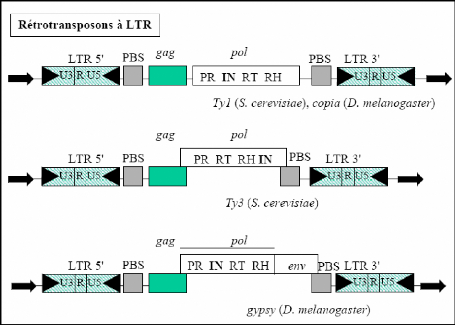
Figure 1 : Structure des
différents types de rétrotransposons à LTR (Warmus et
Brown 1989)
b. les rétrotransposons sans LTR :
Ces éléments sont caractérisés par
une queue poly A ou une région riche en A à leur
extrémité 3' (Finnegan 1992). Ils possèdent à
chaque extrémité de courtes répétitions directes.
La grande diversité des rétrotransposons sans LTR et leur
similitude avec les rétrotransposons à LTR suggèrent que
les rétrotransposons sans LTR seraient à l'origine des
rétrotransposons à LTR (Craig, 2002). La sous-classe des
rétrotransposons sans LTR se divisent en deux familles : les LINE et les
SINE.
b.1 LINE (Long Interspersed Nuclear
Elements):
Les LINE peuvent mesurer 1 à 7 kb et possèdent
généralement deux ORF, correspondant aux gènes gag
et pol. Contrairement aux rétrotransposons à LTR,
le gène pol est dépourvu d'intégrase. La plupart
de ces éléments sont tronqués en 5' et possèdent
des promoteurs internes qui leur permettent d'être transcrits par une ARN
polymérase II (McLean et al. 1993).
Bien qu'ils soient rétrotranscrits, les
éléments LINE ont un système d'intégration
complètement différent des rétrotransposons à LTR.
En effet, la transcriptase inverse reconnaît le transcrit et initie la
rétrotranscription, en même temps que l'intégration au
niveau d'une coupure de l'ADN génomique effectuée par une
endonucléase (Luan et al. 1993).
Il y a plusieurs familles de LINE telles que LINE 1
(L1), L2 et L3. Mais dans le génome humain on
trouve généralement des L1 (figure 2).
· Structure, expression et
répartition des L1.
Un fragment L1 de 6 kb possède une
région non traduite 5' (UTR) de 906 nucléotides, deux phases
ouvertes de lecture (ORF1 et ORF2) et une deuxième région non
traduite 3' de 206 nucléotides qui se termine par une queue poly A. La
région UTR 5' joue le rôle de promoteur interne qui contrôle
la transcription de l'élément L1 par l'ARN
polymérase II (figure 2). ORF1 code pour une protéine de 40 KDa
qui se lie à l'ARN de L1 pour former une
ribonucléoprotéine cytoplasmique (RNP) et le domaine LZ a
probablement un rôle dans la dimérisation de cette
protéine. Plusieurs analyses ont montré qu'ORF1 contribue dans la
rétrotransposition mais son rôle exact n'est pas encore clair. Des
études suggèrent qu'elle a une fonction de chaperonne des acides
nucléiques pour leurs permettre des réarrangements stables. Alors
qu'ORF2 code pour une protéine de 150 kDa qui possède des
activités endonucléase et rétrotranscriptase qui va couper
l'ADN de l'hôte et faire une transcription inverse de l'ARN de
L1 en ADN qui sera inséré dans le site de coupure de
l'ADN hôte formant ainsi un nouvel élément L1.
(Goodier et Kazazian 2005)
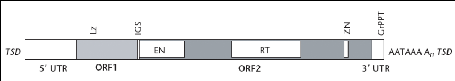
Figure 2 : Structure complète
d'un élément LINE humain (UTR : untranslated region, TSD
: variable length target site duplication; EN : endonuclease
domain; RT: reverse transcriptase domain ; Lz . a putative
leucine zipper domain ; IGS : intergenic spacer region ZN :
putative zinc knuckle motif ; GrPPT : guanosine-rich polypurine
tract) (Goodier et Kazazian 2005)
La famille de L1 peut être divisé en
deux sous-familles : les Ta (transcribed, subset a) et pre-Ta qu'on peut les
distinguer par diagnostic des trinucléotides de leur région UTR
5'. La sous-famille Ta comporte le plus grand nombre d'éléments
actifs puisque 13 des 14 événements de rétrotransposition
récemment découvert et qui sont responsable de maladies chez
l'homme sont dus à des insertions de Ta. La dernière insertion
des quatorze est due à un pre-Ta. Ce qui montre une grande
activité de cette sous-famille. (Goodier et Kazazian 2005)
Les études de Kazazian et Moran en 1998 ont
montré que le génome humain diploïde comporte
entre 80
à 100 éléments L1 actifs. Par contre le
génome de la souris compte des milliers
d'éléments
L1 actifs et qui sont subdivisés en plusieurs sous familles.
Ceci est démontré par le
pourcentage élevé de mutation chez la souris (2.5%)
par rapport à l'homme (0.07%). (Kazazian and Moran 1998)
Les éléments L1 sont rencontrés
dans tous les chromosomes mais préférentiellement sur le
chromosome X où ils représentent 26% de sa composition. Les
analyses cytogénétiques ont montré que les
séquences L1 atteignent une grande densité au niveau de
l'hétérochromatine. Des études supposent un rôle
présumé des L1 dans l'inactivation du chromosome X.
(Goodier et Kazazian 2005)
b.2 SINEs (Short Interspersed Nuclear Elements):
Les SINE font généralement environ 500 pb de
longueur. Ils sont des éléments non autonomes qui ne
possèdent pas de cadre de lecture, et sont composés de deux
«boîtes» A et B consensus du promoteur (deux sites de fixation
à l'ARN polymérase III) et d'une queue polyA à
l'extrémité 3'. Les SINE ne contiennent pas de terminateur de
transcription pour l'ARN polymérase III. Ils dérivent presque
tous des ARN de transfert (Okada 1991) excepté l'élément
Alu des primates et la famille B1 des rongeurs qui
dérivent de l'ARN 7SL (Ullu et Tschudi 1984). Les SINEs
dérivés d'ARNt ont une structure composite comportant une
région homologue à un ARNt, une région « core »
conservée de fonction inconnue, une région non homologue à
un ARNt et une zone de taille variable AT riche en 3'. La zone homologue
à un ARNt contient le promoteur interne d'ARN polymérase III. Ces
éléments non autonomes utilisent vraisemblablement la
transcriptase inverse des LINEs pour se déplacer (Okada et al.
1997). Les SINE représentent 13% du génome humain et les
éléments les plus abondants sont les éléments
Alu et ils sont appelés ainsi car ils contiennent un site qui
est reconnu par un enzyme de restriction qui est Alu I. Cette séquence
est spécifique au génome des primates.
Chez l'homme, les Alu sont responsables de 0,1 à
0,3% des maladies (ROY et al. 1999).
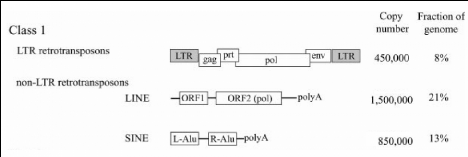
Figure 3 : Structure des
éléments transposables de classe I et leurs nombres de copies
dans le génome humain. (Insuk et al. 2003)
2.2 Les éléments de classe II :
La classe II comporte les éléments se
déplaçant par un intermédiaire ADN. Ils sont directement
excisés puis introduits à un autre endroit du génome
grâce à une transposase. Ce mécanisme est appelé
transposition selon le mode couper-coller. Ces éléments sont plus
petits que les rétrotransposons. Ils possèdent à chacune
de leurs extrémités de courtes séquences
répétées inversées de quelques dizaines à
quelques centaines de paires de base (ITR : Inverted Tandem Repeat). On trouve
généralement dans leur séquence un seul cadre de lecture
qui code pour la transposase (figure 4). Celle-ci se fixe au niveau de
séquences spécifiques près des ITR. Cette classe regroupe
un ensemble de superfamilles hétérogènes dont les
éléments les plus caractéristiques sont
l'élément Ac du maïs et les éléments
P et mariner de la drosophile. Les bactéries peuvent
comporter deux types de transposons : les IS (Mahillon et Chandler
1998) et les Tn, qui sont des éléments composites
pouvant comporter un gène de résistance à un antibiotique
ou à des métaux lourds.

Figure 4 : Structure des
éléments transposables de classe II et leurs nombres de copies
dans le génome humain. (Insuk L et al. 2003)
2.3 Les éléments de classe III :
Cette classe regroupe des éléments dont on
connaît mal le mécanisme de transposition. C'est le cas par
exemple pour les éléments foldback qui possèdent
de grandes répétitions terminales inversées. Ils ont tout
d'abord été détectés chez la drosophile (Truett et
al. 1981) mais ils sont aussi présents chez d'autres organismes comme le
nématode (élément Tc4) (Yuan et al.
1991) et l'oursin de mer (éléments TU) (Liebermann
et al. 1983).
On place aussi dans cette classe la superfamille des MITEs
(Miniature Inverted-repeat Transposable Elements) qui présente des
éléments mal caractérisés et de très petite
taille (100 à 500 pb) et qui sont flanquées par des
répétitions inversés caractéristiques d'environ 15
pb. On les trouve principalement chez les plantes comme le poivre vert ou le
maïs et les champignons. Ils ont cependant été
récemment détectés chez certains vertébrés
(l'homme, le poisson zèbre et le xénope). Certaines
caractéristiques comme des ITR et des duplications
générées lors de l'insertion suggèrent qu'ils
peuvent dériver d'éléments de classe II, il pourrait donc
s'agir d'éléments de classe II
dégénérés (Feschotte et Mouchès 2000).
II. Les éléments transposables actifs dans
le génome humain :
Malgré que les éléments transposables
présentent 44% du génome humain. Une très faible
proportion de ces éléments (moins de 0.05%) demeure active
aujourd'hui. Des études récentes indiquent qu'environ 35 à
40 sous-familles de L1, Alu, SVA (SINE-R, VNTR and
Alu) restent encore activement mobiles dans notre génome. Ces
éléments qui demeurent actifs sont d'importance majeure car se
sont eux qui sont responsables de la diversité génétique
dans les populations humaines et se sont eux aussi qui continuent à
produire des maladies en s'intégrant dans ou à proximité
des gènes. (Mills et al 2007)
Les éléments L1 actifs dans le
génome sont accablés par un grand nombre de copies de L1
inactifs. Généralement tous les L1 sont tronqués
à leurs extrémités 5'. La plupart des L1
possèdent des codons stop au niveau des régions qui codent les
deux protéines ORF1 et ORF2 qui ont un rôle dans la
rétrotranposition. La capacité de L1 à s'exprimer
n'a été découverte que lorsque ce dernier a sauté
pour s'insérer au niveau du gène codant pour le facteur VIII et
causant ainsi l'hémophilie. Donc les L1 actifs sont rares dans
notre génome et ne sont pas facilement reconnaissables dans cette grande
collection d'éléments L1 inactifs. Les copies actives
d'Alu sont quant à eux plus difficile à identifier vu
qu'ils sont des éléments non autonomes et qui dépendent
des protéines de L1 pour leur transposition. (Mills et al
2007)
Plusieurs stratégies ont été
adoptées pour identifier les éléments qui ont
été récemment mobiles comme par exemple la comparaison du
génome de l'homme au génome du chimpanzé pour
détecter les insertions de transposons spécifiques à
chaque espèce (Mills et al 2007). Les copies d'éléments
trouvés dans l'une des deux espèces ont
généralement été mobiles durant les 6 derniers
millions d'années (le temps dans lequel un ancêtre commun entre
l'homme et le chimpanzé existait). Plus de 10000 de ces insertions de
transposons spécifiques à chaque espèce ont
été identifié et la plupart d'entre eux (plus de 95%) sont
des éléments L1, Alu et SVA (un
élément inhabituel composite qui est dérivé de
trois autres éléments qui sont SINE
VNTR et Alu). Des
expériences de transposition dans des cultures cellulaires ont
été effectuées pour démontrer qu'uniquement une
faible proportion des éléments transposables est active. (Brouha
et al. 2003)
III. Les interactions entre les ET et le génome
humain :
1. Effet négatif des éléments
transposables sur le génome :
Les éléments transposables peuvent causer des
mutations au niveau du génome soit par l'insertion à
l'intérieur, ou à proximité d'un gène, et
altérer alors directement son expression ou en causant des aberrations
chromosomiques suites à des recombinaisons homologues.
a. Les insertions : Ils peuvent être :
V' dans une phase codante : interruption de la phase, un
polypeptide tronqué. V' dans un intron et dans ce cas il y a plusieurs
possibilités :
· elles n'auront aucun effet.
· Un arrêt de la transcription dans le transposon :
polypeptide incomplet.
· Un épissage incorrect ou plus ou moins
inhibé : polypeptide tronqué, protéine chimère ou
protéine correcte moins abondante.
V' en amont d'un gène dans une zone régulatrice :
D'où dérégulation
· soit transcription inhibée
.
· éloignement entre promoteur et séquences
activatrices ou destruction de ces dernières.
· soit transcription activée
.
· Le promoteur propre est faible, remplacé par un
promoteur fort présent à l'intérieur du transposon ; ceci
peut conduire à l'induction ectopique (dans un autre lieu) de la
transcription (une dérégulation spatio-temporelle). (kazazian
2004)
b. Les recombinaisons : Les séquences
L1 et Alu peuvent provoquer des mésappariements et des crossing-over
inégaux qui entraînent des délétions ou des
duplications des séquences entre les fragments
répétés. Une implication étonnante des
éléments Alu dans ces événements a
été largement décrite dans la littérature (kazazian
2004). Les éléments L1 sont moins impliqués dans ces
mésappariements à cause de leur faible représentation dans
les régions de forte densité géniques par rapport aux
éléments Alu. L'implication des Alu est étonnante car
normalement leur insertion est dépendante de la machinerie des L1. Donc
cette distribution des L1 et Alu peut refléter une contre
sélection au cours de l'évolution des L1 dans les régions
riches en gènes (kazazian 2004). D'un autre coté la recombinaison
homologue entre les éléments Alu peut entraîner des
duplications de fragments. Ces fragments dupliqués de 200 à 400
Kb représentent environ 5% du génome humain. Quand ces
séquences dupliquées dépassent les 5Mb, ils peuvent
être responsables de la génération de plusieurs maladies.
Ceci en causant des délétions, duplications ou des inversions
suite à des mésappariements et des crossing-over inégaux.
(figure 5)
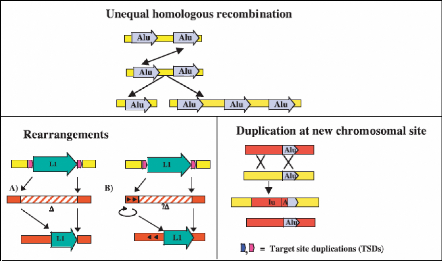
Figure 5 : Quelques insertions et
recombinaisons causés par les éléments transposables dans
le génome humain (kazazian 2004)
c. Implication de ET dans des maladies : Ces
réarrangements causés par les insertions et les recombinaisons
ont de graves conséquences sur le fonctionnement de la machinerie
cellulaire et peuvent ainsi générer des maladies. Les
recombinaisons non homologues des éléments SINEs et la perte de
séquence génomique ont provoqué l'apparition de cancers
dans des lignées cellulaires humaines (Craig et al 2002). Une
étude a montré aussi, après comparaison de tissus sains et
de tissus tumoraux par la méthode SAGE, que les éléments
SINE proches des gènes contribuent à la cascade de
dérégulation de gènes dans les cellules cancéreuses
(Lerat et al 2007). Il y a plus de 30 maladies
génétiques et 16 types de cancer qui ont été
attribués à la recombinaison homologue entre des SINEs (Tableaux
1 et 2 : Deininger et Batzer 1999). L'implication de recombinaison des
éléments LINE a été aussi observée dans le
cas des cancers de l'oesophage et du tractus génital femelle.
L'hypométhylation des LINEs a été associé à
un haut niveau d'expression de gène dans des tissus cancéreux
divers. Il a été suggéré que
l'hypométhylation de ET peut promouvoir l'instabilité
génomique et faciliter ainsi la progression de la tumeur. (Lerat et al
2007).
L'élément L1 en s'insérant au
niveau du gène codant pour le fracteur VIII cause la maladie de
l'hémophilie (Kazazian 1988). L'élément L2, un
membre de la famille LINE2 et qui représente 3% du génome humain,
lui aussi en s'insérant auprès d'un nombre de gènes est
capable de jouer le rôle d'un silencer des gènes codant aux
cellules T. (Donnelly et al. 1999)
Tableau 1 : Quelques types de cancers dus aux
recombinaisons A lu/A lu
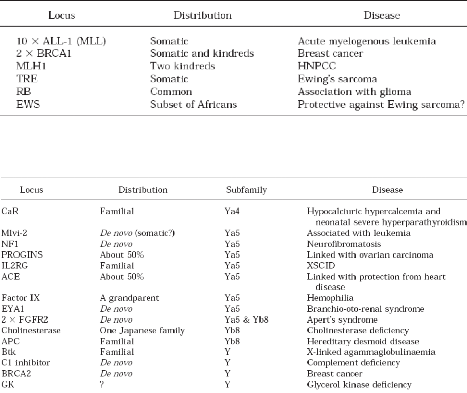
Tableau 2 : Quelques maladies dues aux insertions des
éléments Alu
2. L'effet du génome sur les
réterotransposons :
Il est clair que les éléments transposables ont
la capacité de causer plusieurs dégâts dans le
génome humain par leurs insertions et leurs recombinaisons. Mais ceci
n'empêche pas que plusieurs rétrotranspositions sont
inhibés dans la plupart des cellules. La protéine ORF1 de L1 et
son ARN par exemple ont été détectées dans des
cellules tumorales ou embryonnaires mais rarement dans des cellules
différenciées. On soupçonne que la méthylation des
transposons joue un rôle important dans leurs inactivations. Les
dinucléotides CpG du promoteur L1 sont très
méthylés ce qui bloque sa transcription. Le traitement des
cellules avec 5-azacytidine (un inhibiteur de la méthylation)
entraîne une augmentation de la transcription des L1 (Woodcock et al.
1997).
Les facteurs cellulaires peuvent aussi contribuer à la
régulation de la transcription des
éléments
transposables. Dans le cas de l'élément
L1, la région UTR 5' comporte 2 sites de liaison, un pour
SOX1 1
(facteur de la famille de SRY) et l'autre pour YY (Ying Yang). Un rôle
important de ces
deux facteurs a été démontré dans
l'inhibition de la transcription de cet élément. (Goodier et
Kazazien 2005)
On peut également spéculer un autre moyen
possible pour l'inhibition de la rétrotransposition. C'est le
phénomène de l'ARN interférence (RNAi) qui a
été découvert fortuitement chez le nématode
Caenorhabditis elegans. Par ce
phénomène des molécules d'ARN double brin sont capables de
réduire au silence les gènes de séquences homologues. L1
peut s'insérer au niveau de l'ADN et occasionnellement dans des
gènes, en orientation directe ou inversée, et de ce fait il peut
être transcrit comme étant un fragment interne en un large ARNm
hétérologue. Les RNAi empêchent la traduction de l'ARNm en
protéine par appariement d'un ARN antisens sur l'ARNm, ce qui forme un
ARN double brin reconnu et détruit par la machinerie cellulaire de
l'hôte. (Goodier et Kazazien 2005)
3. Effet bénéfique sur le génome
:
En plus de leur rôle dans la plasticité
structurale du génome, les éléments transposables ont le
moyen de modifier la structure d'un gène par leurs insertions. Ils
pourraient alors intervenir dans l'évolution du génome et
l'expression des gènes. (Kazazian 2004)
a. Rôles des ET dans la régulation de
l'expression des gènes :
Même si les insertions des ET n'ont pas lieu dans les
exons, ces éléments peuvent influencer les gènes
situés à proximité. Les ET peuvent aussi s'insérer
dans les promoteurs, dans les introns ou les parties transcrites et non
traduites du gène. L'insertion d'un élément transposable
dans le promoteur peut modifier la transcription du gène. Cette
insertion peut inhiber les boites de régulation qui augmentent alors la
transcription d'un gène ou plus simplement éteindre le
gène (Conte et al 2002). Mais l'insertion d'un élément
transposable dans le promoteur d'un gène peut aussi donner un avantage
au génome hôte, par exemple en créant des sites de liaison
à des facteurs de transcription précédemment non
présent dans les gènes spécifiques. (Bartley et al. 2006).
Les éléments transposables peuvent modifier la localisation
tissulaire de la transcription d'un gène donné : chez l'humain et
la souris, une dizaine de gènes orthologues ont une transcription
tissulaire différente selon l'insertion ou non d'éléments
transposables dans l'un des deux génomes. D'autre part, les
éléments non autonomes peuvent apporter de nouveaux motifs de
régulation contenus dans leurs séquences internes. Chez l'humain,
la séquence Alu contient plusieurs motifs de liaison aux
facteurs de transcription. Ces motifs vont modifier le profil d'expression du
gène à proximité. L'élément non-autonome
peut s'insérer dans le promoteur et devenir une partie intégrante
de celui-ci ou devenir le nouveau promoteur du gène. Donc les ET ont un
grand potentiel pour influencer la régulation de la transcription des
gènes. (Tempel 2007)
b. Rôles des ET dans la création de
nouveaux gènes :
En plus de la capacité des ETs à
contrôler l'expression d'un gène hôte, ils peuvent aussi se
transformer en nouveaux gènes hôtes. Cette transformation
s'effectue après la perte de mobilité (perte des
extrémités palindromiques et/ou des gènes de transposition
dans le génome) et la mutation de la séquence interne de
l'élément transposable. Certains éléments non
autonomes peuvent capturer des fragments de gènes différents
(Jiang et al 2004) et peuvent être transcrits en un seul ARNm. Ainsi, une
récente étude a démontre que 5 % des exons du
génome humain dérivent des rétrotransposons Alu
(Kreahling et al 2004).
Il parait aussi que la séquence UTR 5' de L1 a
assumé le rôle d'un enhancer des apolipoprotéines dans les
hépatocytes. (Goodier et Kazazian 2005)
Un autre exemple a été également
cité celui du promoteur du gène codant pour le facteur lX. On
pense que ce promoteur réside dans l'extrémité 3' dans un
ancien L1. (Goodier et Kazazian 2005)
c. Les éléments transposables et le
système immunitaire : Un exemple spectaculaire de domestication
moléculaire est fourni par la découverte qu'une des fonctions
clés du système immunitaire des vertébrés,
notamment l'homme, a émergé il y a environ 100 millions,
d'années à partir d'un élément transposable
(Agrawal et al 1998). Dans ce système, deux protéines,
RAG1 et RAG2, sont essentielles pour la recombinaison V(D)J. Elles
possèdent en effet à la fois la propriété de
reconnaître des séquences nucléotidiques spécifiques
correspondant à des signaux de recombinaison placés au voisinage
des segments V, D et J, et celle de cliver de l'ADN immédiatement
à proximité de ces signaux. Ces derniers, composés
d'heptamères et de nonamères très conservés et
séparés les uns des autres par des séquences relativement
homogènes de 1 2 ou 23 nucléotides, évoquent les
répétitions terminales inversées de nombreux
éléments transposables. In vitro, les protéines RAG
purifiées entraînent la transposition intra- et
intermoléculaire de segments d'ADN flanqués par les signaux de
recombinaison dans une grande diversité de sites cibles, en provoquant
généralement une duplication de 5 pb de ces sites. Ces
réactions sont, du point de vue de leurs mécanismes, identiques
à celles provoquées par les transposases d'éléments
mobiles ou les intégrases des rétrovirus. La production
d'antigènes spécifiques par la recombinaison V(D) J ainsi que le
gène apparenté aux gènes RAG sont restreints aux
vertébrés à mâchoires. Les similitudes
fonctionnelles et structurales amènent à penser que les
gènes RAG1 et RAG2 étaient autrefois des
parties d'un élément transposable actif possédant des
répétitions terminales inversées identiques aux signaux
des recombinaisons V(D) J (figure 6). (Agrawal et al 1998)
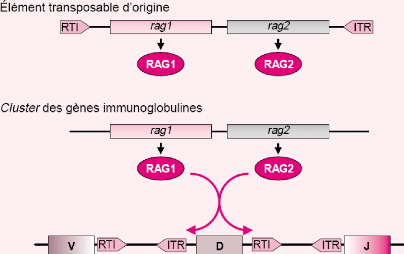
Figure 6 : L'activité des
gènes rag1 et rag2 du système immunitaire des mammifères a
pour origine l`activité transposase d`un élément
transposable. (Agrawal et al 1998)
d. Rôles des éléments transposables
dans l'évolution et la spéciation :
En plus de leurs capacités à créer de
nouveaux gènes, des études suggèrent que les transposons
ont eu un rôle majeur dans l'évolution et la transformation des
espèces. D'après cette suggestion, les insertions des ET et les
réarrangements génomiques qu'ils peuvent créer, ont
probablement conduit à la spéciation de plusieurs organismes.
Notamment la lignée des primates qui a subit une activité intense
de ces transposons (Pace and Feschotte 2007). On suppose que la plupart des
transposons ont été intensivement mobiles après
l'existence d'un ancêtre commun entre l'homme et le chimpanzé
(avant environ 6 million d'années). De ce fait le niveau de
transposition chez l'homme était plus élevé que celui du
chimpanzé durant ces derniers millions d'années. L'homme a donc
amplifié des copies de sous familles de transposons différentes
de celles amplifiées par le chimpanzé, ce qui a permis une
évolution différente des deux sous-espèces. (Mills et al.
2006)
IV. L'importance des éléments
transposables dans la génétique:
1. Marqueur de polymorphisme :
Les intégrations récentes de
l'élément Alu dans le génome humain ont
généré des variants qui sont présents ou absents et
qui ont une grande utilité dans l'étude des populations humaines.
Ces variants peuvent être utilisés comme des marqueurs d'ADN dans
les analyses de paternité et de médecine légale. (Kass et
al. 2006)
2. Utilisation dans la thérapie génique
:
La thérapie génique est une stratégie
très prometteuse dans le traitement de plusieurs maladies
génétiques héréditaires ou acquises. Divers types
de vecteur ont été utilisés pour délivrer l'acide
nucléique thérapeutique aux cellules affectées. Les
vecteurs viraux ont montré des effets génotoxiques et des
complications immunologiques. Alors que les vecteurs non viraux sont moins
efficaces dans l'intégration au niveau du génome. Une solution
était proposée et qui consiste à utiliser les
éléments transposables. Ces éléments ayant la
capacité de s'intégrer facilement dans le génome
permettent donc l'introduction du gène d'intérêt et son
expression. (Ivics and Izsvák 2006)
La reconstruction moléculaire de Sleeping
Beauty, qui est un ancien transposon des poissons, représente une
étape importante dans l'application de la thérapie génique
fondée sur la transposition. (Ivics and Izsvák 2006)
Conclusion
Les éléments transposables ont été
considérés pendant longtemps comme des éléments
sans valeur qui ne participent pas à la machinerie cellulaire. Ils ont
été vus comme des parasites du génome ou de « l'ADN
égoïste » qui ne faisaient qu'amplifier leurs ADN sans aucune
influence sur le développement et le fonctionnement de la cellule. Or,
il s'est apparu que leur existence dans le génome n'est pas due au
hasard. Aujourd'hui, on considère les éléments
transposables comme potentiellement nécessaires au génome de
l'hôte. Bien que leur mobilité soit, à court terme, plus
souvent génératrice de problèmes (source de mutation...)
pour l'organisme, à long terme, il en est tout autrement. Les
éléments transposables permettraient la création de
nouveaux gènes et seraient aussi un facteur d'adaptation face à
l'environnement. Ainsi plusieurs études soulignent l'impact de ces
éléments transposables dans l'évolution des
espèces. Ils seraient, par exemple, à la base du
développement de notre système immunitaire. Il reste donc encore
beaucoup de choses à découvrir sur eux et la présence de
ces éléments dans le génome pose de nombreuses questions
sur leur origine et leur relation avec certains hôtes
indésirables.
Références bibliographiques
· AGRAWAL A, EASTMAN QM, SCHATZ DG (1998) Transposition
mediated by RAG1 and RAG2 and its implications for the evolution of the immune
system. Nature 394:744-751.
· BARTLEY G, THORNBURG A, VALER GOTEA, WOJCIECH M (2006).
Transposable
elements as a significant source of transcription regulating
signals. Gene. 365: 104-110
· BROUHA B, et al. (2003) Hot L1s account for the bulk of
retrotransposition in the human population. Proc. Natl. Acad. Sci.
U. S. A. 100, 5280-5285.
· CAPY P. (2005). Classification and
nomenclature of retrotransposable elements. Cytogenet Genome
Res. 110:457-461.
· CONTE C, DASTUGUE B, AND VAURY C. (2002) Promoter
competition as a mechanism of transcriptional interference mediated by
retrotransposons. The EMBO Journal, 21:3908- 3916.
· CRAIG NL, GRAGIE R, GELLERT M, LAMBOWITZ AM, (2002).
Mobile DNA II second edition. ASM Press,
· DEININGER, P.L, BATZER, M.A, (1999). Alu repeats and
human disease. Mol. Genet. Metab. 67 (3); 183-193.
· DONNELLY SR, HAWKINS TE and MOSS SE. (1999). A conserved
nuclear element with a role in mammalian gene regulation. Human
Molecular Genetics. 8: 1723-1728.
· EVGEN'EV MB, ZELENTSOVA H, POLUECTOVA H, LYOZIN GT et
al. (2000) Mobile elements and chromosomal evolution in the virilis
group of Drosophila. Proc Natl Acad Sci
USA 97: 11337-11342.
· FESCHOTTE C, MOUCHÈS C. (2000) Evidence that a
family of Miniature Inverted-repeat Transposable Elements (MITEs) from the
Arabidopsis thaliana genome has arisen from a pogo-like DNA
transposon. Mol Biol Evol 17:730-737
· FINNEGAN DJ. (1992) Transposable elements.
Curr Opin Genet Dev 2:861-867
· GOODIER J, KAZAZIAN HH. JR. (2005). Long Interspersed
Nuclear Elements (LINEs) ENCYCLOPEDIA OF LIFE
SCIENCES (
www.els.net)
· INSUK L, and HARSHEY R, (2003) Patterns of sequence
conservation at termini of long terminal repeat (LTR) retrotransposons and DNA
transposons in the human genome: lessons from phage Mu. Nucleic
Acids Research Vol. 31, No. 15: 4531-4540.
· IVICS Z, and IZSVÁK Z, (2006).
Transposons for Gene Therapy. Current Gene Therapy:
6, 593-607.
· JIANG N., BAO Z., ZHANG X., EDDY SR., and WESSLER SR.
(2004). Pack-mule transposable elements mediate gene evolution in plants.
Nature, 431: 569-573.
· KASS D, JAMISON N, MELANIE M, et al. (2007).
Identification of a unique Alu-based polymorphism and its use in human
population studies. Gene, 390: 146-152
· KAZAZIAN HH. JR et al. (1988). Haemophilia A resulting
from de novo insertion of L1 sequences represents a novel mechanism for
mutation in man. Nature 332, 164-16
· KAZAZIAN HH. JR, et al. (2004). Mobile Elements:
Drivers of Genome Evolution. Science 303, 1626.
· KAZAZIAN HH. JR and MORAN JV (1998). The impact of L1
retrotransposons on the human genome. Nature Genetics
19: 19-23
· KIDWELL MG, KIDWELL JF, SVED JA (1977) Hybrid
dysgenesis in Drosophila melanogaster: a syndrome of aberrant traits
including mutation, sterility, and male recombination.
Genetics 86:813-833
· KIM JM, VANGURI S, BOEKE JD, GABRIEL A, VOYTAS DF
(1998) Transposable elements and genome organization: a comprehensive survey of
retrotransposon revealed by the complete Saccharomyces cerevisiae
genome sequence. Genome Res 8:464-478.
· KREAHLING J, and GRAVELEY BR. (2004). The origins and
implications of aluternative splicing. Trends
Genet. 20 :1-4.
· LERAT E, SÉMON M, (2007). Influence of the
transposable element neighborhood on human gene expression in normal and tumor
tissues. Gene 396 : 303-311
· LIEBERMANN D, HOFFMAN-LIEBERMANN B, WEINTHAL J, CHILDS
G, MAXSON R, MAURON A, COHEN SN, KEDES LH (1983). An unusual transposon with
long terminal inverted repeats in the sea urchin Strongylocentrotus
purpuratus. Nature 306:342- 347.
· LUAN DD, KORMAN MH, JAKUBCZAK JL, EICKBUSH TH (1993)
Reverse transcription of R2Bm RNA is primed by a nick at the
chromosomal target site: a mechanism for non-LTR retrotransposition.
Cell 72:595-605.
· MAHILLON J, CHANDLER M (1998) Insertion sequences.
Microbiol Mol Biol Rev 62:725-774.
· MCLEAN C, BUCHETON A, FINNEGAN DJ (1993) The
5''untranslated region of the I factor, a long interspersed nuclear
elements-like retrotransposon of Drosophila melanogaster, contains an
internal promoter and sequences that regulate expression. Mol Cell
Biol 13:1042-1050.
· MILLS RE, et al (2006). Recently mobilized transposons in
the human and chimpanzee genomes. The American Journal of human
genetics. 78: 671-679.
· MILLS RE, BENNETT EA, ISKOW RC, DEVINE SE, (2007). Which
transposable elements are active in the human genome? TRENDS in
Genetics. 23 (4): 183-191.
· OKADA N (1991) SINEs. Curr Opin Genet
Dev 1: 498-504
· ORGEL LE, CRICK FH (1980) Selfish DNA: the ultimate
parasite. Nature 284:604-607.
· PACE JK, and FESCHOTTE C. (2007). Genome
Res. Apr; 17(4):422-32.
· ROY AM, CARROLL ML, KASS DH, NGUYEN SV, SALEM A-H,
BATZER MA, DEININGER PL (1999). Recently integrated human Alu repeats: finding
needles in the haystack. Genetica 107:149-161.
· SANMIGUEL P, TIKHONOV A, JIN YK, MOTCHOULSKAIA N,
ZAKHAROV D, MELAKE-BERHAN A, et al. (1996) Nested retrotransposons in the
intergenic regions of the maize genome. Science
274:765-768.
· SHAPIRO JA (1969). Mutations caused by the insertion of
genetic material into the galactose operon of Escherichia coli.
J Mo! Bio! 40:93-105.
· TEMIN HM (1980) Origin of retroviruses from cellular
moveable genetic elements. Ce!! 21:599-600.
· TEMPEL Sébastien (2007). Dynamique des
hélitrons dans le génome d'Arabidopsis thaliana :
Développement de nouvelles stratégies d'analyse des
éléments transposables. Thèse
pour l'obtention du grade de docteur de l'université de Rennes : mention
biologie.
· TRUETT MA, JONES RS, POTTER SS. (1981). Unusual structure
of the FB family of transposable elements in Drosophila. Ce!!
24:753-763.
· ULLU E, TSCHUDI C. (1984) Alu sequences are
processed 7SL RNA genes. Nature 312: 17 1-172.
· WARMUS H, BROWN P (1989) Retroviruses. In BERG
DE, HOWE M (eds) Mobile DNA. American Society for microbio!ogy,
Washington DC, USA, pp. 53-108.
· WOODCOCK DM, LAWLER CB, LINSENMEYER ME, DOHERTY JP AND
WARREN WD (1997) Asymmetric methylation in the hypermethylated CpG promoter
region of the human L1 retrotransposon. Journa! of Bio!ogica!
Chemistry. 272: 7810-7816.
· YUAN JY, FINNEY M, TSUNG N, HORVITZ HR (1991)
Tc4, a Caenorhabditis elegans transposable element with an
unusual fold-back structure. Froc Nat! Acad Sci USA
88:3334- 3338.
| 


