|
REPUBLIQUE DE COTE D'IVOIRE
Union - Discipline - Travail
Ministère de l'Enseignement Supérieur et
de la Recherche Scientifique

Ecole Supérieure d'Agronomie (ESA)
![]()
en vue de l'obtention du
Diplôme d'Agronomie
Approfondie (DAA)
Option : Agronomie et
Productions Végétales
Thème :
DIVERSITE GENETIQUE DES RHIZOBIA ASSOCIES A UN CHAMP DE
POIS D'ANGOLE (Cajanus cajan L.)
A YAMOUSSOUKRO (CENTRE DE LA CÔTE
D'IVOIRE)
Présenté par
FOSSOU Kouakou Romain
Elève Ingénieur Agronome (41e
promotion, ENSA)
Jury de soutenance
Président : Prof. BROU Yao Casimir,
Maître de Conférences à l'INP-HB/DFR ARA
Membres
Maître de stage/Encadreur Pédagogique :
Prof. ZEZE Adolphe, Maître de Conférences à
l'INP-HB/DFR ARA
Assesseurs : Dr AGNEROH ATCHAM
Thérèse, Enseignant-chercheur à l'INP-HB/DFR
ARA
M. KOUAKOU Kouadio Paul-Martial,
Enseignant-chercheur à l'INP-HB/DFR ARA
M. NANDJUI Jacob, Enseignant-chercheur
à l'INP-HB/DFR ARA
(c) Août 2011
RESUME
Le pois d'Angole (Cajanus cajan) est une importante
légumineuse à graines dans les zones tropicales, notamment en
Afrique de l'ouest. Toutefois, dans cette partie de l'Afrique, la
diversité des rhizobia nodulant cette plante est pratiquement inconnue
alors que son exploration peut permettre d'améliorer davantage ses
intérêts, notamment en agriculture. La présente
étude visait à évaluer la diversité
génétique des rhizobia nodulant un champ de pois d'Angole
cultivé à Yamoussoukro (centre de la Côte d'Ivoire). Au
total, 169 souches de rhizobia ont été isolées des nodules
collectés. Le milieu synthétique TY utilisé pour cultiver
et purifier les isolats avant l'étude génétique a permis
de distinguer ceux-ci en deux groupes en fonction de leur vitesse de
croissance. La diversité génétique a été
étudiée à partir du polymorphisme de la longueur des
fragments de restriction (RFLP) de leur ADNr 16S préalablement
amplifié par une réaction de polymérisation en
chaîne (PCR). L'analyse des produits de la digestion de l'ADNr 16S des
isolats avec l'endonucléase Tsp 509I a
révélé trois différents génotypes, dans les
proportions allant de 11,2 % pour le moins représenté à
53,9 % pour le plus important. Ces résultats montrent qu'il y a une
variabilité physiologique et génétique au sein des
rhizobia nodulant le champ de pois d'Angole cultivé à
Yamoussoukro.
Mots clés : Cajanus
cajan ; Diversité ; Rhizobia ; Côte
d'Ivoire ; ADNr 16S ; PCR-RFLP.
ABSTRACT
Pigeon pea (Cajanus cajan) is an important grain
legume in tropical zone, notably in West Africa. However, in this area of
Africa the diversity of rhizobia nodulating this plant is poorly understood
even though its exploration can contribute to increase its interest, notably in
agriculture. This work aimed at determining the genetic diversity of rhizobia
nodulating a pigeon pea field in Yamoussoukro (Center Ivory Coast). One hundred
sixty-nine (169) rhizobia's strains were isolated from field nodules. The use
of TY medium allowed the purification and identification of two groups
according to their growth rate. PCR-RFLP analyses of the 16S rRNA genes with
the endonuclease Tsp 509I enabled the characterization of three main
genotypes within the pigeon pea nodulating rhizobial community. The abundance
of these genotypes are 11,2 % for the least to 53,9 % for the most important.
These studies showed the evidence of physiological and genetic variability
within the pigeon pea noduating rhozobial strains.
Keywords: Cajanus cajan; Diversity;
Rhizobia; Côte d'Ivoire; rDNA16S; PCR-RFLP.
AVANT-PROPOS ET REMERCIEMENTS
Ce mémoire a été élaboré en
vue de l'obtention du Diplôme d'Agronomie Approfondie (DAA). Il marque la
fin d'études des étudiants agronomes de l'Ecole Supérieure
d'Agronomie (ESA) de l'Institut National Polytechnique Félix
Houphouët Boigny de Yamoussoukro (INP-HB). En effet, dans le souci de
donner une formation complète à ses élèves
ingénieurs, l'ESA a initié un stage de fin d'études d'une
durée de six (6) mois portant sur un thème de recherche ou de
développement.
C'est pour bénéficier de cette initiative
pédagogique que nous avons séjourné successivement au
Laboratoire d'Agronomie et Productions végétales de l'INP-HB et
au Laboratoire Central de Biotechnologie (LCB) du Centre National de Recherche
Agronomique (CNRA). Ce stage nous a permis de réfléchir sur le
thème : «Diversité génétique des
rhizobia associés à un champ de pois d'Angole (Cajanus cajan
L.) à Yamoussoukro». Ce fut pour nous une occasion
d'apprentissage d'une technique fondamentale dans l'identification
moléculaire des micro-organismes (la Réaction de
polymérisation en chaîne ou PCR) et aussi, un temps d'initiation
à quelques rudiments de la recherche scientifique.
Ce stage a été proposé par Pr ZEZE
Adolphe, enseignant-chercheur à l'INP-HB, à qui nous voulons
traduire toute notre reconnaissance pour l'opportunité offerte. Pour
nous, il a été plus qu'un encadreur pédagogique et de
terrain durant ce stage. C'est pourquoi, nous voulons lui exprimer toute notre
fierté de l'avoir côtoyé au moment où nous terminons
notre parcours académique à l'INP-HB.
Au Dr AGNEROH née Thérèse ATCHAM, chef
du Laboratoire de Phytopathologie et Biologie Végétale, nous
exprimons toute notre reconnaissance pour avoir ouvert grandement les portes de
son laboratoire pour nos nombreuses et diverses sollicitations durant ce
stage.
Au Dr KOUASSI Nazaire, Directeur du LCB du CNRA, nous voulons
exprimer notre gratitude pour l'accueil au sein de l'unité de biologie
moléculaire et virologie de sa structure pour les manipulations
moléculaires.
Nous remercions également MM. YAO Bognon, AMOAKON
Adou, YOHOU Alain et Mlle DALE, personnel technique des deux laboratoires
sus-cités, pour leur aide précieuse.
Enfin, à notre frère et unique collègue
de fin d'études ZAKO, ainsi qu'à toute la 41ème
promotion ENSA, nous disons merci.
TABLE DES MATIERES
RESUME I
ABSTRACT II
AVANT PROPOS ET REMERCIEMENTS III
LISTE DES TABLEAUX VII
LISTE DES FIGURES VIII
LISTE DES ANNEXES IX
SIGLES ET ABREVIATIONS X
DEDICACES XI
INTRODUCTION 1
PREMIERE PARTIE : REVUE BIBLIOGRAPHIQUE 3
CHAPITRE I : GENERALITES SUR LA SYMBIOSE
LEGUMINEUSES-RHIZOBIA. 4
I.1. FIXATION BIOLOGIQUE DE L'AZOTE 4
I.2. SYMBIOSE LEGUMINEUSES/RHIZOBIA 5
I.2.1. Définition de la symbiose rhizobienne 5
I.2.2. Intérêts de la symbiose
rhizobia-légumineuses 6
I.2.3. Caractérisation des deux partenaires 7
I.2.3.1. Rhizobia 7
I.2.3.2. Légumineuses 7
I.2.3.3. Caractéristiques du couple :
infectivité, efficience et spécificité 8
CHAPITRE II : APERÇU SUR LE PROCESSUS DE
NODULATION CONDUISANT A LA FIXATION AZOTEE 10
II.1. GENETIQUE DE LA NODULATION CHEZ LA BACTERIE 10
II.1.1. Gènes nod 10
II.1.1.1. Gènes nod D 10
II.1.1.2. Gènes nod communs (ABCIJ) 11
II.1.1.3. Gènes nod spécifiques
(nod H, nod FE, nod G, nod MN, nod
Q etc.) 11
II.1.1.4. Autres gènes nécessaires à la
nodulation 12
II.1.2. Gènes nif 13
II.1.3. Gènes fix 14
II.2. GENETIQUE DE LA NODULATION CHEZ LA LEGUMINEUSE 14
II.3. SCHEMATISATION DES ETAPES DU PROCESSUS DE NODULATION
15
CHAPITRE III : TAXONOMIE DES RHIZOBIA
17
III.1. HISTOIRE DE LA TAXONOMIE RHIZOBIENNE 17
III.2. TAXONOMIE POLYPHASIQUE ET PHYLOGENIE DES RHIZOBIA 18
III.2.1. Taxonomie polyphasique 18
III.2.2. Notion de phylogénie chez les Rhizobia
18
III.3. CLASSIFICATION ACTUELLE DES RHIZOBIA 19
CHAPITRE IV : GENERALITES SUR LE POIS D'ANGOLE
(Cajanus cajan L.) 21
IV.1. ORIGINE ET DISTRIBUTION GEOGRAPHIQUE DE LA PLANTE 21
IV.2. ZONES DE CULTURE ET IMPORTANCE DES PRODUCTIONS 21
IV.3. TAXONOMIE DU POIS D'ANGOLE 22
IV.3.1. Nomenclature 22
IV.3.2. Classification 22
IV.4. DESCRIPTION DU POIS D'ANGOLE 23
IV.4.1. Description botanique 23
IV.4.2. Ecologie 24
IV.4.3. Mode de multiplication et systèmes de culture
24
IV.4.4. Croissance, développement et rendement 24
IV.4.5. Ennemis et maladies de Cajanus cajan 25
IV.5. IMPORTANCE DE LA CULTURE DU POIS CAJAN 25
IV.5.1. Importance nutritionnelle du pois cajan 26
IV.5.2. Importance agronomique du pois cajan 26
IV.5.3. Importance du pois cajan en Afrique 27
CHAPITRE V : RHIZOBIA SYMBIOTIQUES DU POIS CAJAN
28
DEUXIEME PARTIE : ETUDE EXPERIMENTALE
29
CHAPITRE VI : PRESENTATION DE L'ETUDE ET DES
LABORATOIRES D'ACCUEIL 30
VI.1. OBJECTIFS DE L'ETUDE 30
VI.2. PRESENTATION DU SITE DE COLLECTE DES ECHANTILLONS 30
VI.3. PRESENTATION DES LABORATOIRES D'ACCUEIL DE L'ETUDE 30
CHAPITRE VII : MATERIEL ET METHODES
31
VII.1. MATERIEL 31
VII.1.1. Matériel vivant 31
VII.1.2. Matériel technique 31
VII.2. METHODES 33
VII.2.1. Récolte des nodules et isolement des souches
bactériennes 33
VII.2.2. Préparation des cellules bactériennes
par effet thermique 34
VII.2.3. Amplification et digestion de la région 16S de
l'ADNr des rhizobia par
PCR-RFLP 34
VII.2.3.1. Principe de la PCR-RFLP et choix du gène de
l'ADNr 16S 34
VII.2.3.1.1. Principe de la PCR-RFLP 34
VII.2.3.1.2. Choix du gène de l'ADNr 16S 35
VII.2.3.2 Réalisation de la PCR-RFLP 36
VII.2.3.2.1. Principe de la PCR 36
VII.2.3.2.2. Choix des amorces et milieu réactionnel de
la PCR 37
VII.2.3.2.3. Programme de la PCR 38
VII.2.3.2.4. Electrophorèse sur gel d'agarose et
visualisation des gels 39
VII.2.3.2.5. Migration de l'ADN et visualisation 39
VII.2.3.2.6. Digestion des amplifiats 40
CHAPITRE VIII : RESULTATS ET DISCUSSION
41
VIII.1. RESULTATS 41
VIII.1.1. Croissance des bactéries sur milieu TY solide
41
VIII.1.2. Amplification par PCR de l'ADNr 16S 41
VIII.1.3. Produits de digestion 42
VIII.2. DISCUSSION DES RESULTATS 44
CONCLUSION ET PERSPECTIVES 46
REFERENCES BIBLIOGRAPHIQUES 47
ANNEXES 57
LISTE DES TABLEAUX
Tableau I : Exemples des
différents types de micro-organismes fixateurs d'azote 4
Tableau II : Classification des
rhizobia 20
Tableau III : Noms communs du
pois d'Angole en quatre langues principales 22
Tableau IV : Amorces choisies pour
l'amplification des fragments d'ADNr 16S 37
Tableau V : Composition du milieu
réactionnel unitaire commun de la PCR 38
Tableau VI : Caractéristiques
de l'enzyme de restriction Tsp 509 I 40
Tableau VII : Composition du
milieu réactionnel unitaire commun de la digestion 40
LISTE DES FIGURES
Figure 1 :
Nodules racinaires (a) et tige portant des nodules aériens
matures (b) 5
Figure 2 : Symbiose fixatrice
d'azote Rhizobia-Parasponia sp. 6
Figure 3 : Diversité des
Légumineuses 8
Figure 4: Différents types de
gènes nod 11
Figure 5 : Nitrogénase et
mécanismes d'action 13
Figure 6 : Etapes du processus de
nodulation lors de la symbiose Légumineuse-rhizobia 16
Figure 7 a : Inflorescences de
Cajanus cajan 23
Figure 7 b : Fruit sec en
déhiscence 23
Figure 8 : Feuillage
rejeté par une culture de pois cajan en jachère
améliorée 27
Figure 9 a : Nodosités sur
racines de C. cajan 31
Figure 9 b : Nodosités
étalées pour l'isolement 31
Figure 10 :
Présentation d'un thermocycleur 32
Figure 11: Appareil de migration 32
Figure 12 : Transilluminateur
relié à un ordinateur 32
Figure 13 : Organisation de
l'opéron de l'ADNr observé chez les procaryotes 35
Figure 14 : Etapes de la PCR
pour un cycle d'amplification 37
Figure 15 : Mode de
croissance sur milieu TY en 24 heures et 48 heures des groupes de rhizobia
isolés du pois cajan 41
Figure 16 : Résultats
d`amplification de l'ADNr 16S des rhizobia isolés de Cajanus cajan
42
Figure 17 : Différents
profils de digestion obtenus avec l'enzyme de restriction Tsp 509I
42
Figure 18 : Proportions
relatives des trois profils obtenus par PCR-RFLP 43
Figure 19 :
Proportions des profils de digestion des souches supposées
à croissance lente 43
LISTE DES ANNEXES
ANNEXE I : Classification des
rhizobia selon les séquences du gène de l'ARN 16 S 58
ANNEXE II : Présentation
des structures et des laboratoires d'accueil de l'étude 60
ANNEXE III : Composition du
milieu TY (Tryptone Yeast Agar) en g/l 63
SIGLES ET ABREVIATIONS
ADNr : Acide
DésoxyriboNucléique
ribosomique
ARNr : Acide
RiboNucléique
ribosomique
ATP : Adénosine
Tri-Phosphate
BET: Bromure
d'Ethydium
CNRA : Centre
National de Recherche
Agronomique
DAA : Diplôme
d'Agronomie Approfondie
dct :
dicarboxylate transport
genes
DFR-ARA :
Département de Formation et de
Recherche Agriculture et
Ressources Animales
dNTP :
désoxyriboNucléotide
TriPhosphate
ENSA : Ecole Nationale
Supérieure Agronomique
ESA : Ecole
Supérieure d'Agronomie
FAO: Food and
Agriculture Organization
GS : Glutamine
Synthétase
hsn : host
specific nodulation
INP-HB : Institut
National Polytechnique
Félix Houphouët
Boigny
ITS: Intergenic
Transcribed Spacer
LCB : Laboratoire
Central de Biotechnologie
MoFe : Molybdène
Fer
pb : paire de base
PCR : Polymerase
Chain Reaction
RFLP : Restriction
Fragment Lenght
Polymorphism
SDS-PAGE: Sodium
Dodecyl Sulfate-
Polyacrilamide Gel
Electrophoresis
Taq : Thermus
aquaticus
TBE: Tris
Borate EDTA
TY: Tryptone
Yeast extract
USDA: United
State Department of
Agriculture
UV: Ultra Violet
YMA: Yeast
Manitol Agar
DEDICACES
Ce Mémoire, conclusion de toute notre formation à
l'INP-HB est dédié à nos parents :
À notre père FOSSOU Félix et à
notre mère POKOU Florentine pour la vie qu'ils nous ont
donnée;
À notre cousin KONAN Georges et sa famille pour le
soutien moral et financier indéfectible ;
Et enfin,
À la famille ETTIEN pour sa grande contribution à
la réalisation de notre cursus scolaire à Yamoussoukro.
FOSSOU KOUAKOU ROMAIN
INTRODUCTION
L'azote, en tant que composant omniprésent dans les
biomolécules (protéines, acides nucléiques, vitamines...),
est l'élément constitutif des végétaux le plus
important après le carbone. Malheureusement, la concentration de ses
formes assimilables dans le sol (ammonium, nitrate, etc.) est souvent limitante
pour la bonne croissance des plantes et constitue de ce fait, très
fréquemment, le facteur clé de la production agricole (ROGER
et al., 1996).
Certaines plantes, notamment les légumineuses, ont
réussi à s'affranchir de cette limitation en établissant
des relations symbiotiques avec des bactéries capables de fixer l'azote
atmosphérique grâce à un complexe enzymatique, la
nitrogénase. Cette symbiose est indubitablement le facteur majeur
expliquant le grand succès de cette famille de plantes en agriculture
(NOEL, 2009). En effet, cultivées pour leurs graines (riches en
protéines) ou pour leurs propriétés fourragères,
les légumineuses occupent 12 à 15% des terres cultivables dans le
monde et représentent 27% de la production mondiale des cultures. Plus
de 35% des huiles végétales proviennent de leurs productions avec
en tête le soja et l'arachide et les légumineuses à graines
contribuent à elles seules pour 33% aux besoins azotés de
l'alimentation humaine (VANCE et al., 2000 ; GRAHAM et VANCE,
2003). Par ailleurs, la rotation des cultures avec ces plantes permet
d'économiser les engrais azotés, très coûteux en
énergie fossile et contribuant à l'effet de serre via
l'émission de grandes quantités d'oxyde nitrique (CRUTZEN et
al., 2007).
L'étude de cette importante symbiose depuis plus de 100
ans a permis de comprendre certains mécanismes de son
établissement et d'explorer la diversité biologique des deux
types de partenaires associés. Dans certains cas, ces études ont
conduit à la sélection de souches très performantes de
bactéries fixatrices d'azote et le développement de leur
inoculum. Les inoculats permettent en effet d'améliorer la
productivité des légumineuses en culture, de mieux réduire
leur dépendance vis-à-vis des engrais azotés et de
s'inscrire par conséquent dans la logique d'un développement
agricole durable.
Pour la légumineuse tropicale Cajanus cajan
ou pois d'Angole (5ème légumineuse à graines en
terme de production), l'étude de la diversité de ses symbiotes a
permis d'identifier aujourd'hui environ une dizaine d'espèces
bactériennes. Celles-ci appartiennent à trois principaux genres
de rhizobia ou Bactéries Nodulatrices de Légumineuses (BNL). Il
s'agit des genres Rhizobium (BENDER et al., 1986;
WOLDE-MESKEL et al., 2004 a), Sinorhizobium (CHEN et
al., 1988 ; DUBEY et al., 2010) et Bradyrhizobium
(RAMSUBHAG et al., 2002 ; WOLDE-MESKEL et al., 2004 a).
Toutefois, ces études taxonomiques ont été principalement
menées en Inde et en Afrique de l'Est, respectivement premier et
deuxième centre de diversité et de culture de cette
légumineuse à graines (SMARTT, 1990 ; VAN DER MAESEN,
1990 ; SINGH, 1991 ; SONGOK et al., 2010).
En Afrique occidentale, quelques études similaires ont
été menées seulement au Nigéria (ABAIDOO et
al., 2000), alors que toute cette partie du continent africain constitue
également une zone de production d'importance de cette plante de grande
utilité (NENE et SHEILA, 1990).
Notre stage se propose d'étudier la diversité
génétique des rhizobia nodulant le pois d'Angole en culture dans
un champ à Yamoussoukro. Il s'agit d'évaluer par PCR-RFLP de
l'ADNr 16S, la variabilité génétique des rhizobia
isolés de ce champ. Une collection locale des partenaires symbiotiques
de cette légumineuse sera également constituée.
Ce document qui présente les résultats de cette
étude, s'articule autour de deux parties. La première fait une
synthèse bibliographique sur le pois d'Angole, les rhizobia et le
processus de nodulation. Quant à la seconde, elle concerne
l'étude expérimentale qui décrit le site de collecte des
échantillons et les manipulations au laboratoire, le matériel et
les méthodes utilisés et enfin, présente les
résultats qui feront l'objet d'une discussion.
PREMIERE PARTIE :
REVUE BIBLIOGRAPHIQUE
CHAPITRE I : GENERALITES SUR LA SYMBIOSE
LEGUMINEUSES-RHIZOBIA
I.1- FIXATION BIOLOGIQUE DE L'AZOTE (FBA)
L'azote moléculaire (N2), constituant majeur de
l'atmosphère mais chimiquement inerte, ne peut être utilisé
que par certains micro-organismes procaryotes appelés fixateurs
biologiques de N2. Ils sont soit libres, soit symbiotiques, c'est à dire
associés à d'autres organismes. La découverte de cette
propriété fixatrice de N2 atmosphérique par les
micro-organismes remonte à la fin du XIXè
siècle avec les travaux de plusieurs scientifiques dont HELLRIEGEL et
WILFART (1888)1(*) et
BEYERINCK (1888)2(*).
Depuis, de nombreux genres et espèces bactériennes ont
été reconnus comme fixateurs de N2 atmosphérique. Ces
organismes présentent pratiquement tous les types de comportements en ce
qui concerne les relations plante-micro-organisme, les relations avec
l'oxygène et les modes trophiques comme le montre le tableau
I:
Tableau I : Exemples des différents
types de micro-organismes fixateurs d'azote (D'après ROGER
et al., 1996)
Micro-organismes libres
· Aérobies
· Hétérotrophes
Azotobacter spp.; Klebsiella pneumoniae;
Beijerinckia indica; Azospirillum lipoferum
· Phototrophes:
Cyanobactéries
·
Hétérocystées Nostoc; Anabaena;
Calothrix; Tolypothrix
· Homocystées
Trichodesmium; Oscillatoria
· Unicellulaires
Gloeothece; Gloeocapsa
· Anaérobies
· Hétérotrophes
Clostridium pasteurianum; Desulfovibrio vulgaris;
Desulfotomaculum spp. ; Methanobacterium spp.
· Phototrophes
Rhodospirillum rubrum; Rhodobacter capsulata;
Chromatium vinosum
Microorganismes symbiotiques
· Légumineuses
· à nodules racinaires
Sinorhizobium meliloti; Rhizobium leguminosarum
Bradyrhizobium japonicum; S. fredii etc.
· à nodules caulinaires
Azorhizobium caulinodans
· Symbioses actinorhiziennes Frankia
· Symbioses à cyanobactéries
· Azolla
Anabaena azollae
· Cycas
Anabaena cycadeae
· Lichens
Nostoc
· Mousses et hépatiques
Nostoc
De tous ces systèmes fixateurs de N2
atmosphérique, les associations symbiotiques sont les plus importantes.
Celles-ci sont responsables chaque année de la réduction de 120
millions de tonnes (Mt) d'azote atmosphérique en ammonium (FREIBERG
et al., 1997), pour un niveau total de réduction estimé
à 175 Mt/an (ROGER, 1996). Parmi ces associations symbiotiques, celle
existant entre les plantes de la famille des Légumineuses et les
Rhizobia est l'une des plus efficaces. En effet, les légumineuses
cultivées en association avec leurs symbiotes fixent 40 à 60 Mt
d'azote par an, tandis que 3 à 5 Mt d'azote sont fixées par les
légumineuses des écosystèmes naturels (GRAHAM et VANCE,
2003). Par ailleurs, cette symbiose est aussi responsable de la plus grande
partie de la fixation d'azote dans les sols agricoles et fait l'objet
d'étude depuis un siècle (NUTMAN, 1981). Les
paragraphes suivants se consacreront à une synthèse des
connaissances importantes actuellement acquises sur elle.
I.2- SYMBIOSE LEGUMINEUSES/RHIZOBIA
I.2.1- Définition de la symbiose rhizobienne
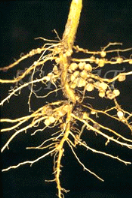
La symbiose rhizobienne est une association entre les plantes
de la famille des légumineuses et des bactéries du type
Rhizobium permettant de réduire l'azote atmosphérique en
des formes assimilables par les plantes. A bénéfice
réciproque, cette association donne lieu à des interactions
multiples entre les deux partenaires. Au cours de ces interactions, un nouvel
organe, le nodule, est formé sur les racines (Figure
1a) ou plus rarement sur les tiges à partir de primordia
racinaires dormants et disposés en rang le long de la tige
(Figure 1b). C'est au sein de cet organe protecteur que
l'azote atmosphérique est fixé par les bactéries.
(a) (b)
Figure 1 : Nodules
racinaires (a) et tige portant des nodules aériens matures
(b)
(DUHOUX et NICOLE, 2004)
A ce jour, les seules plantes non légumineuses capables
de noduler avec des rhizobia (Sinorhizobium et Bradyrhizobium
spp.) sont de petits arbres tropicaux appartenant au genre
Parasponia, de la famille des Ulmacées (LAFAY et
al., 2006 ; NOEL, 2009) (Figure 2).
C
A
B
Figure 2 : Symbiose
fixatrice d'azote Rhizobia-Parasponia sp.
A. Tige de Parasponia.
B. Nodule multilobé de Parasponia rigida.
C. Coupe longitudinale d'un lobe nodulaire de
Parasponia (D'après LANCELLE et TORREY,
1985)
Bien que la symbiose légumineuse-Rhizobium
soit une interaction hautement adaptée et régulée, il ne
s'agit pas d'une interaction obligatoire ou permanente. En effet, les deux
partenaires peuvent vivre indépendamment et de manière autonome,
et chaque nouvelle génération de plante doit être
infectée par de nouvelles bactéries (KNEIP et
al., 2007). Lorsque cette symbiose a lieu, elles présentent de
nombreux intérêts aussi bien pour les deux partenaires que pour la
pratique agricole.
I.2.2- Intérêts de la symbiose
rhizobia-légumineuses
Cette symbiose présente de nombreux avantages pour les
légumineuses. En effet, celle-ci leur permet d'avoir une bonne
croissance sur des sols carencés en azote. A titre d'exemple, le pois
d'Angole (Cajanus cajan) qui est cultivé sous les tropiques
incluant les régions semi-arides peut satisfaire jusqu'à 96% de
ses besoins azotés par le biais de celle-ci (KUMAR RAO et al.,
1986). De ce fait, cette symbiose est indubitablement le facteur majeur
à l'origine du grand succès de la famille des Légumineuses
parmi les végétaux (NOEL, 2009).
A l'opposé, la plante subvient aux besoins
énergétiques de la bactérie au cours de cette symbiose en
fournissant des substances carbonées résultant de la
photosynthèse. Elle lui offre également un microenvironnement
très particulier et nécessaire à la fixation de l'azote.
Par ailleurs, outre l'augmentation au niveau du sol de la population des
rhizobia spécifiques à la légumineuse hôte
après culture, la symbiose fournirait un cadre de reproduction
bénéfique qui favoriserait l'évolution des espèces
bactériennes (NOEL, 2009).
I.2.3- Caractérisation des deux partenaires
I.2.3.1- Rhizobia
Les rhizobia sont des fixateurs symbiotiques de l'azote
atmosphérique dans les nodules des racines ou des tiges des plantes
légumineuses où ils se différencient en
bactéroïdes (GRAHAM, 1991 ; HAUKKA et al.,
1998 ; GAGE, 2004). Ces bactéries du sol sont de formes
bâtonnets, à Gram négatif (paroi
hétérogène, coloration rouge). Elles sont non sporulant,
d'une largeur variant entre 0,5 et 0,9 ìm et une longueur comprise entre
1,2 et 3 ìm. Elles sont mobiles par un seul flagelle polaire (cas de
Mesorhizobium) ou par deux à six flagelles péritriches
(JORDAN, 1984 ; SOMASEGARAN et HOBEN ,1994).
Au niveau de leur matière sèche, les cellules
des rhizobia contiennent 52 à 55 % de carbone et 4 à 5 % d'azote
(ALLEN et ALLEN, 1950).
Les milieux au mannitol et aux extraits de levure sont les
plus utilisés pour la culture de ces bactéries (ALLEN et ALLEN,
1950). Une croissance optimale de la plupart des souches de Rhizobium
a lieu à des températures variant de 25 à 30°C
et un pH compris entre 6,0 et 7,0 (SOMASEGARAN et HOBEN ,1994).
I.2.3.2- Légumineuses
Les Légumineuses représentent une famille
importante et variée des Angiospermes. En effet, il s'agit de la
troisième plus grande famille chez les plantes supérieures avec
plus de 720 genres et 20 000 espèces allant des espèces
herbacées comme la luzerne, aux arbres composant les forêts
tropicales d'Amérique Latine et d'Afrique (CRONK et
al., 2006), à l'image des acacias. Leurs fruits (gousses) les
caractérisent par rapport aux autres végétaux.
Classiquement, les Légumineuses sont divisées en trois
sous-familles à savoir les Caesalpinoideae, les
Mimosoideae et les Papilionoideae (Faboideae). La
plupart des espèces cultivées appartiennent à la
dernière sous-famille citée.
Les Papilionoideae représentent la
sous-famille la plus diverse avec 429 genres et environ 12000 espèces
(YOUNG et al., 2003). Deux groupes majeurs de plantes cultivées
sont présents au sein de cette sous-famille. Il s'agit des
légumineuses tempérées encore appelées
Galégoides, avec les genres Cicer (pois chiche), Lens
(lentilles), Lotus (lotier), Medicago (luzerne),
Melilotus (mélilots), Pisum (pois), Trifolium
(trèfle) et Vicia (vesce) etc. Le deuxième groupe est
celui des légumineuses tropicales ou Phaséolides, avec notamment
les genres Phaseolus (haricot), Glycine (soja),
Vigna (vigne) et Cajanus (pois d'Angole).
Les Caesalpinoideae (150 genres et 2 200
espèces) sont principalement constituées de plantes ornementales
et d'arbres à bois ou alimentaires (Tamarindus etc.) (YOUNG
et al., 2003). Quant aux plantes de la sous-famille des
Mimosoideae (62 genres et environ 2 500 espèces), elles sont
présentes principalement dans les forêts tropicales et
subtropicales, avec notamment les genres Acacia et Albizia
(YOUNG et al., 2003).
La figure 3 ci-après
illustre de façon non exhaustive, la grande diversité biologique
au sein de la famille des Légumineuses.
Figure 3 : Diversité des
Légumineuses
A. Glycine max, le soja
(Papilionoideae);
B. Vigna unguiculata, le
niébé (Papilionoideae) ;
C. Pisum sativum, le pois
cultivé (Papilionoideae) ;
D. Cercis siliquastrum
(Caesalpinoideae) ;
E. Albizia julibrissin
(Mimosoideae);
F. Acacia albida
(Mimosoideae).
(D'après VERNIE, 2008)

I.2.3.3- Caractéristiques du couple :
infectivité, efficience et spécificité
La capacité des bactéries d'infecter les
racines de légumineuses et de former des nodules s'appelle
"'infectivité", alors que le terme "efficience" donne une indication de
la capacité des plantes nodulées à fixer l'azote (BECK
et al., 1993). Selon les mêmes auteurs,
l'infectivité est le résultat de l'interaction
de la légumineuse hôte et la souche envahissante.
Toutes les légumineuses ne sont pas capables de noduler
et l'effectivité de la nodulation concerne 88 % des espèces
examinées (GRAHAM et VANCE, 2003). Par ailleurs, les espèces
déjà étudiées pour leur aptitude à la
nodulation représentent 20 % de l'effectif total des légumineuses
connues (NOEL, 2009). Des trois sous-familles, les Caesalpinoideae
sont les moins nodulantes. Seulement 30 % d'entre elles forment des nodules,
contre 90 % chez les deux autres sous-familles (SOLTIS et
al., 1995). Ces proportions seraient de 10 ; 80 et 90 %
respectivement pour les Caesalpinoideae, les Mimosoideae et
les Papilionoideae selon NOEL (2009).
La spécificité peut être définie
par la diversité taxonomique des partenaires auxquels l'autre symbiote
peut s'associer. La spécificité peut être stricte (une
seule espèce de bactérie pour un seul genre de
légumineuse) ou large, c'est-à-dire concerner plusieurs taxons
bactériens ou de légumineuses (PRIN et al.,
1993). En général, chaque légumineuse ne peut être
infectée que par un nombre restreint de souches de rhizobia. Par
exemple, la légumineuse Medicago truncatula ne peut être
nodulée que par Sinorhizobium meliloti et S. medicae
(VERNIE, 2008). Concernant les rhizobia, si certains ont une
spécificité très restreinte, d'autres par contre ont une
très large spécificité d'hôtes. Ainsi, la souche
Sinorhizobium (Ensifer) sp. NGR 234 peut noduler 353 espèces de
légumineuses appartenant à 112 genres différents et
représentant les trois sous-familles des Légumineuses. Elle peut
même noduler l'Ulmacée Parasponia andersonii (PUEPPKE et
BROUGHTON, 1999 ; NOEL, 2009).
CONCLUSION PARTIELLE
Les légumineuses réalisent avec les
bactéries du sol de type rhizobia une symbiose à
bénéfice réciproque (mutualisme). En général
très spécifique, cette symbiose est à l'origine du grand
succès de la famille des Légumineuses au niveau de l'agriculture
où elles occupent, en termes d'importance, la seconde place
derrière les Graminées. Le chapitre suivant présentera son
mode d'établissement.
CHAPITRE II : APERÇU SUR LE PROCESSUS DE
NODULATION CONDUISANT A LA FIXATION AZOTEE
Le processus d'une symbiose fixatrice d'azote débute
par la formation de nodosités au niveau des racines ou des tiges de la
plante hôte. La formation de ces nodosités survient quand les
rhizobia pénètrent leur hôte d'une manière
coordonnée et contrôlée. De nombreux gènes
appartenant aussi bien aux bactéries qu'à la plante hôte
interviennent au cours de ce processus.
II.1- GENETIQUE DE LA NODULATION CHEZ LA BACTERIE
En général, trois types de gènes
symbiotiques interviennent dans le processus de nodulation et de fixation
azotée chez la bactérie. Il s'agit des gènes nod
nécessaires à la nodulation, des gènes nif codant
pour la nitrogénase et des gènes fix indispensables pour
la fixation de l'azote. Chez la plupart des rhizobia (Rhizobium,
Allorhizobium et Sinorhizobium etc.), ces gènes
symbiotiques sont situés sur un grand plasmide symbiotique appelé
pSym (MERCADO-BLANCO et TORO, 1996 ; NOEL, 2009). Chez les genres
Bradyrhizobium, Azorhizobium et chez l'espèce
Mesorhizobium loti, ces gènes symbiotiques, qu'on
élucidera dans les paragraphes suivants, sont situés sur le
chromosome (SHARMA et al., 1993).
II.1.1- Gènes nod
Les gènes de nodulation ou gènes nod
incluent des gènes communs et des gènes spécifiques de
l'hôte à infecter (gènes hsn) (BROUGHTON et
al., 2000 ; SPAINK, 2000). En 2000, au total 13 gènes de nodulation
étaient déjà connus. Il s'agit des opérons
nod ABCIJ, nod FEL, nod MNT, du nod O et du
nod D (PERRET et al., 2000).
II.1.1.1- Gène nod D
Le gène nod D négocie les
premières étapes de la nodulation chez la bactérie. Il est
par conséquent le premier gène nod transcrit lors du
processus de nodulation. Sa transcription se fait de manière
constitutive (GEURTS et BISSELING, 2002) et l'activation de son produit, le Nod
D, est réalisée par les flavonoïdes qui sont des
molécules émises par les racines des légumineuses. Une
fois activé, le Nod D se lie aux sites régulateurs des
opérons porteurs des autres gènes nod et active leur
transcription. La Figure 4 ci-après illustre ce
rôle régulateur du Nod D et présente les autres
gènes de nodulation.
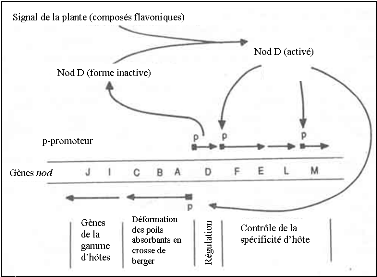
Figure 4 : Différents
types de gènes nod (ANONYME, 2010)
II.1.1.2- Gènes nod communs (ABCIJ)
Les gènes nod communs (nod A, B, C,
I et J) sont des gènes de nodulation communs à tous les rhizobia
jusqu'ici étudiés. Ils sont situés sur un opéron
duquel les nod ABC sont essentiels pour la nodulation. Ces derniers
sont physiquement et fonctionnellement conservés chez toutes les
espèces de rhizobia et leurs mutations provoquent une perte
complète de la capacité à infecter et à noduler les
plantes hôtes. En effet, ces gènes sont indispensables pour
induire la déformation des poils absorbants en forme de crosse
(curling) et inciter les cellules végétales à se
diviser (BREWIN et al., 1992 ; PELMONT, 1995).
II.1.1.3- Gènes nod spécifiques (nod H,
nod FE, nod G, nod MN, nod Q etc.)
Les gènes hsn (host specific
nodulation) sont des gènes spécifiques de la plante
à infecter (PELMONT, 1995). Ils ne sont pas nécessairement
présents ou fonctionnellement conservés chez tous les rhizobia
(SHARMA et al., 1993). Ces gènes sont responsables de
la spécificité d'hôte et de la reconnaissance entre la
bactérie et la plante, étape préalable à
l'infection (DAVET, 1996). Leur mutation provoque un retard, une
réduction de la nodulation ou une modification de leur
spécificité d'hôte (DEBELLE et SHARMA, 1986 ; DEBELLE
et al., 1986; HORVATH et al., 1986 ;
CERVANTES et al., 1989).
Après leur transcription, les gènes
nod produisent des enzymes qui contrôlent la synthèse de
molécules indispensables au processus de nodulation. Ces signaux, qui
sont généralement connus sous le nom de facteurs Nod, sont des
molécules lipo-chito-oligosaccharidiques (LCOs) (TEREFEWORK, 2002). La
synthèse du noyau lipooligosaccharide de ces molécules est
contrôlée par les gènes nod ABCD tandis que les
gènes hsn assurent les diverses substitutions de celles-ci
(DEBELLE et al., 2001).
Les facteurs Nod sont responsables de la
spécificité de la reconnaissance entre les symbiotes et du
déclenchement de l'organogenèse nodulaire. En effet, même
émis à des concentrations minimales, ces signaux peuvent
déclencher des réponses symbiotiques chez la plante telles que la
déformation des poils radiculaires, la division corticale des cellules
et la formation de nodule primordial (DEBELLE et al., 2001).
L'identification du signal Nod est une étape essentielle dans
l'établissement du dialogue moléculaire à l'origine de la
symbiose entre les légumineuses et les bactéries fixatrices
d'azote.
II.1.1.4 - Autres gènes nécessaires
à la nodulation
En plus des gènes nod, les rhizobia
possèdent d'autres gènes essentiels au processus de nodulation.
Il s'agit des gènes exo, lps et ndv qui
codent pour l'expression et la synthèse des molécules de
structure de la surface bactérienne (BROUGHTON et al.,
2000 ; SPAINK, 2000). Ces molécules qui sont successivement les
exopolysaccharides (EPS), les lipopolysaccharides (LPS) et les â-glucans
sont nécessaires pour le développement continu du fil
d'infection. Elles sont par ailleurs considérées comme
d'importants facteurs dans l'efficacité symbiotique (BREEDVELD et
MILLER, 1998). En effet, les â-glucans
sont majoritairement des molécules du périplasme qui permettent
la croissance des bactéries sous des conditions hypoosmotiques (PFEFFER
et al., 1994). Ils jouent également un rôle important
dans la suppression du déclenchement du mécanisme de
défense par les phytoalexines chez l'hôte (BHAGWAT et
al., 1996). Quant aux exopolysaccharides, ils fonctionnent comme des
messagers pendant l'infection (GONZALEZ et al., 1996). Enfin, les
mutants de rhizobia défectifs en la synthèse ou à faible
production de lipopolysaccharides n'ont pas la capacité d'induire la
formation de cordons d'infection. Ils perdent également leur
capacité compétitive ou forment des nodosités
incomplètement développées (LAGARES et al.,
1992).
Faisant suite à l'activité des
gènes d'infection et de nodulation, deux groupes de gènes
interviennent dans les étapes tardives de la symbiose. Il s'agit des
gènes nif et fix.
II.1.2- Gènes nif
Les gènes nif existent chez
plusieurs bactéries dont les rhizobia (YOUNG et HAUKKA, 1996). Chez ces
derniers, ces gènes codent pour la synthèse d'un complexe
enzymatique catalysant la réduction de l'azote et connu sous le nom de
nitrogénase ou dinitrogénase (HOPKINS, 2003). Ce
complexe enzymatique est constitué de deux
métalloprotéines de tailles différentes : le site de la
réduction du substrat correspondant à la MoFe-protéine ou
dinitrogénase (245 KDa) et le donneur d'électrons correspondant
à la Fe-protéine ou dinitrogénase réductase (64
KDa).
La Fe-protéine est un dimère de deux
sous unités polypeptidiques identiques codé par le gène
nif H. Quant à la Mo-Fe-protéine, elle est un
tétramère composée de deux sous unités non
identiques de type á2â2 (WERNER, 1992 ;
HOPKINS, 2003). Celle-ci est codée par les gènes nif D
et nif K (Figure 5).
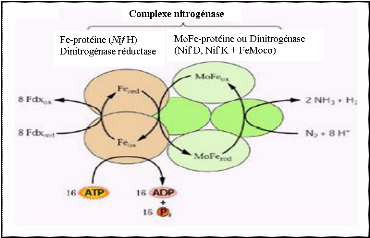
Figure 5 : Nitrogénase et
mécanismes d'action (YANN, 2006)
Sous catalyse de ce complexe enzymatique, la
réduction de l'azote moléculaire se déroule en deux
étapes. Lors de la première, la Fe-protéine est
réduite par un donneur primaire d'électrons, habituellement la
ferrédoxine. Dans la seconde étape, la Fe-protéine
réduite transfère les électrons à la
Mo-Fe-protéine qui catalyse à la fois la réduction du
diazote gazeux et la production d'hydrogène (HOPKINS, 2003)
(Figure 5).
L'ATP dans la réaction provient de la respiration
aérobique des bactéroïdes. Il réagit avec la
Fe-protéine réduite et intervient dans le transfert des
électrons entre la Fe-protéine et la Mo-Fe-protéine. Pour
chaque molécule de diazote réduite, au moins 16ATP sont
nécessaires, deux par électron (HOPKINS, 2003). Au
total, les légumineuses utilisent jusqu'à 22% de l'énergie
issue de leur photosynthèse pour réaliser la fixation
azotée dont l'équation générale est la suivante:
N2 + 8H+ + 8 e- + 16 ATP
2NH3 + H2 + 16 ADP + 16Pi
À la fin de la réaction, l'ammoniac fixé
est converti en glutamine, asparagine, uréides etc. avant d'être
transporté dans la sève du xylème pour son assimilation
par la plante hôte. Cette conversion est possible grâce aux
gènes nif et fix, etc. qui codent en partie pour la
synthèse de différents enzymes catalyseurs comme le glutamate
déshydrogénase, la glutamine synthétase (GS) etc.
II.1.3- Gènes fix
Les gènes fix sont des gènes
additionnels, propres aux fixateurs symbiotiques et impliqués dans les
étapes tardives de développement des nodules lors de la fixation
symbiotique azotée (BREWIN et al., 1992 ; HOPKINS, 2003 ;
NOEL, 2009). Certains de ces gènes (fix L, fix J) sont
des régulateurs de la synthèse du gène nif A,
intervenant dans la régulation de la synthèse de la
nitrogénase (NOEL, 2009). D'autres encore (fix NOPQ)
codent pour la synthèse d'enzymes catalysant la régulation de
l'oxygène lors de la fixation comme la cbb3 cytochrome oxydase.
Quoique n'étant pas habituellement
désignés par le terme de gènes fix, les
gènes dct (Dicarboxylate transport genes)
épousent la définition des gènes fix. Ils sont
nécessaires à l'assimilation par les bactéries des acides
dicarboxyliques (succinate, malate) issus des composés carbonés
(glucose, fructose) apportés par la plante au cours de la fixation
azotée. La mutation de ces gènes chez les bactéries
entraîne une faible prolifération des bactéroïdes et
une déficience de ceux-ci lors de la fixation de l'azote (NOEL, 2009).
II.2- GENETIQUE DE LA NODULATION CHEZ LA
LEGUMINEUSE
Tout comme la bactérie, la plante émet des
molécules essentielles à la réalisation de chaque
étape du processus de nodulation et de fixation. Ainsi, au début
du processus de nodulation, divers composés déterminant une
chimiotaxie positive entre les deux symbiotes sont émis par la racine de
la légumineuse, parmi lesquels des
flavonoïdes.
Les flavonoïdes constituent les premiers signaux de l'hôte qui
déclenchent chez la bactérie l'expression du gène de
régulation de la nodulation nod D et induisent le
mécanisme du chimiotactisme des rhizobia (PETERS et VERMA, 1990). La
production de ces molécules est limitée à la zone de
prolongation des poils racinaires à partir de laquelle la plupart des
nodules se développent plus tard (BROUGHTON et al., 2000).
En plus de ces composés émis seuls, la
légumineuse participe aussi à la synthèse d'un certain
nombre de protéines essentielles au développement de nodules
fonctionnels. Il s'agit par exemple de la production des nodulines en
réponse à des stimulis provenant des bactéries
symbiotiques. Certaines de ces nodulines sont des enzymes du métabolisme
azoté (glutamine synthétase, etc.) ou carboné (saccharose
synthase, etc). La plus connue est une protéine (la globine) qui,
associée à l'hème produit par les bactéroïdes,
constitue la leghémoglobine, protéine fixatrice d'oxygène.
Cette dernière protéine est indispensable à la fixation du
N2 (PELMONT, 1995). En effet, la nitrogénase étant
extrêmement labile en présence de O2, la
leghémoglobine est produite autour des symbiosomes pendant la fixation
de l'azote afin de maintenir une pression partielle basse en oxygène
nécessaire à son bon fonctionnement, tout en assurant aux
bactéroïdes un approvisionnement suffisant en oxygène pour
leur respiration (WERNER, 1992 ; DAVET, 1996 ; OTT et al.,
2005).
En somme l'effet conjugué de tous les gènes et
facteurs de nodulation de la bactérie et de la plante aboutissent
à la formation de nodules fixateurs d'azote. Ce processus complexe est
résumé dans le paragraphe suivant par un schéma.
II.3- SCHEMATISATION DES ETAPES DU PROCESSUS DE
NODULATION
En général, la formation d'une nodosité
fixatrice d'azote suit les étapes de développement suivant :
A - chimiotactisme et attachement des rhizobiums aux racines
de l'hôte ;
B- déformation des poils absorbants ;
C- invasion du cytoplasme des cellules corticales par les
bactéries à travers les cordons d'infection ;
D- division des cellules du cortex aboutissant à la
formation d'un primordium nodulaire ;
E- différentiation des bactéries en
bactéroïdes et du primordium nodulaire en nodosité
F- fixation de l'azote par la bactéroïde
(Figure 6).
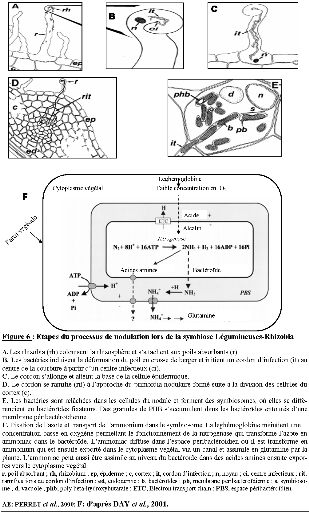
Figure 6 : Etapes du processus de nodulation lors
de la symbiose Légumineuses-Rhizobia
A. Les rhizobia (rh) colonisent la
rhizosphère et s'attachent aux poils absorbants (r).
B. Les bactéries induisent la
déformation du poil en crosse de berger et initient un cordon
d'infection (it) au centre de la courbure à partir d'un centre
infectieux (ci).
C. Le cordon s'allonge et atteint la base de
la cellule épidermique.
D. Le cordon se ramifie (rit) à
l'approche du primordia nodulaire formé suite à la division des
cellules du cortex (c).
E. Les bactéries sont
relâchées dans les cellules du nodule et forment des symbiosomes,
où elles se différencient en bactéroïdes fixateurs.
Des granules de PHB s'accumulent dans les bactéroïdes
entourés d'une membrane péribactéroïdienne.
F. Fixation de l'azote et transport de
l'ammonium dans le symbiosome. La leghémoglobine maintient une
concentration basse en oxygène permettant le
fonctionnement de la nitrogénase qui transforme l'azote en ammoniac dans
le bactéroïde. L'ammoniac diffuse dans l'espace
péribactéroïdien où il est transformé en
ammonium qui est ensuite exporté dans le cytoplasme
végétal via un canal et assimilé en glutamine par la
plante. L'ammoniac peut aussi être assimilé au niveau du
bactéroïde dans des acides aminés ensuite exportés
vers le cytoplasme végétal.
r, poil absorbant ; rh,
rhizobium ; ep, épiderme ; c, cortex ;
it, cordon d'infection ; n, noyau ;
ci, centre infectieux ; rit, ramifications du
cordon d'infection ; ed, endoderme ; b,
bactéroïdes ; pb, membrane
péribactéroïdienne ; s, symbiosome ;
d, vacuole ; phb, poly beta-hydroxybutarate ;
ETC, Electron transport chain ; PBS, espace
péribactéroïdien.
AE: PERRET et al., 2000; F: d'après DAY
et al., 2001
CHAPITRE III : TAXONOMIE DES RHIZOBIA
III.1- HISTOIRE DE LA TAXONOMIE RHIZOBIENNE
La présence de nodosités chez les
légumineuses était historiquement bien connue, mais leur origine
était controversée. WORONIN (1866)3(*) fut le premier à signaler l'observation de
micro-organismes ressemblant à des bactéries dans les
nodosités de Lupinus mutabilis. En 1879, FRANK4(*), un microbiologiste allemand,
rapporta que ces micro-organismes étaient des champignons en leur
affectant le nom de Schinzia leguminosarum. En 1888,
les chimistes allemands HELLRIEGEL et WILFARTH5(*) établissaient un
raccordement important entre les plantes légumineuses, les nodules des
racines et la fixation symbiotique de l'azote. Ils démontrèrent
que l'aptitude à utiliser l'azote atmosphérique par les plantes
légumineuses est liée au développement des nodules suivant
l'infection des racines par des microorganismes du sol. La même
année, la première partie de l'histoire de l'étude des
nodules racinaires connut son apogée, quand BEYERINCK6(*) devint le premier à
isoler une bactérie capable de noduler une légumineuse qu'il
nomma Bacillus radicicola. En 1889, non convaincu par
la nomenclature donnée par BEYERINCK, FRANK7(*) renomma la bactérie
identifiée Rhizobium leguminosarum. Il décida par
ailleurs que toutes les bactéries qui seront ultérieurement
isolées des nodules des légumineuses porteront ce même
nom (NOEL, 2009). Plus tard (1929), BALDWIN et FRED ont
rapporté que la classification des différents rhizobiums devrait
être basée sur la spécificité de l'espèce
bactérienne par rapport à la plante hôte : c'est le
concept de spécificité d'hôte.
Par la suite, FRED et certains collègues
proposèrent le concept d'inoculation croisée qui fut
défini comme un test pour vérifier si une bactérie
isolée d'une espèce de légumineuse donnée peut
induire des nodules chez d'autres espèces. Ce test permit de
définir des groupes de légumineuses dits d'inoculations
croisées, qui d'après FRED et al. (1932) sont
« des groupes de plantes entre lesquelles les organismes
présents dans les nodules sont mutuellement
interchangeables ». Ainsi, FRED et al. (1932) ont pu
identifier six groupes d'inoculations croisées : R.
leguminosarum pour Lathyrus, Pisum, Vicia et
Lens ; R. trifolii pour Trifolium ; R. phaseoli
pour Phaseolus ; R. meliloti pour Glycine max
et R. lupini pour Lupinus.
Si ce concept se révéla très utile en
agriculture, il n'en demeure pas moins qu'il présenta rapidement des
limites dans la classification bactérienne (WILKSON, 1944). En effet, ce
concept se montra très limité dans l'exploration de la
diversité des souches rhizobiennes et dans l'établissement des
relations existantes entre celles-ci (NOEL, 2009).
Par la suite, les bactéries vont être
classées sur la base de leur temps de génération et leur
vitesse de croissance sur milieu de culture, tels que définis par
LÖNHIS et HANSEN en 1921. Ainsi, utilisant ce concept, JORDAN (1982)
classa pour la première fois, les bactéries symbiotiques en deux
genres : le genre Rhizobium correspondant aux souches à
croissance rapide et le nouveau genre Bradyrhizobium pour les souches
à croissance lente.
Toutefois, les observations discordantes entre la notion de
vitesse de croissance des bactéries et la gamme d'hôte ont
jeté le doute sur la validité de cette nouvelle classification.
Celle-ci fera par ailleurs place à des méthodes comparatives
comme la sérologie, le coefficient de Chargaff, l'hybridation ARN / ADN
ou ADN / ADN, l'analyse des plasmides etc. Cette ère marqua le
début d'une nouvelle taxonomie basée sur la comparaison des
résultats de différentes analyses phénotypiques et
biochimiques dans l'identification des bactéries symbiotiques. Cette
taxonomie fut connue sous le nom de taxonomie polyphasique, terme initié
en 1970 par COLWELL.
III.2- TAXONOMIE POLYPHASIQUE ET PHYLOGENIE DES
RHIZOBIA
III.2.1- Taxonomie polyphasique
Originellement basée sur des critères
phénotypiques, l'application de la terminologie polyphasique va
être révisée grâce à la révolution de
différentes techniques moléculaires dont principalement la
Réaction de Polymérisation en Chaîne ou PCR (MULLIS et
FALOONA, 1987). Ainsi, il fut proposé en 1991 les principes standards
de la description de nouveaux genres ou espèces sur la base de
l'analyse numérique qui est la synthèse des résultats de
différents tests phénotypiques et moléculaires
(GRAHAM et al., 1991). Cette nouvelle définition de l'approche
polyphasique a permis l'étude systématique appropriée de
grands groupes bactériens, l'évaluation de la résolution
taxonomique de différentes techniques d'études de
diversité (VANDAMME et al., 1996) et l'établissement de
bases de données comparables entre différents laboratoires de
microbiologie appliquée (GILLIS et al., 2001).
III.2.2 - Notion de phylogénie chez les Rhizobia
La comparaison des données obtenues sur les souches ou
les espèces bactériennes par différentes approches
moléculaires permet l'identification de groupes ou de taxons. Les
différents taxons obtenus peuvent être comparés et
utilisés pour le traçage de lignées de filiation reconnues
communément sous le nom d'arbre phylogénétique. Ainsi, la
phylogénie peut se définir comme l'histoire évolutive qui
révèle les rapports existants entre un groupe d'organismes avec
un ancêtre situé à un plus haut niveau taxonomique.
De nos jours, les relations phylogénétiques
établies entre les rhizobia sont basées principalement sur la
comparaison des séquences de l'ADN ribosomique 16S (OLSEN et
al., 1994 ; YOUNG et HAUKKA,
1996 ; TEREFEWORK, 2002). Par ailleurs, selon
une analyse phylogénétique basée sur le gène de
l'ARN 16S, les rhizobia sont dispersés dans 12 genres parmi neuf groupes
monophylogénétiques (SAWADA et al., 2003). Tous ces
rhizobia caractérisés sont des bactéries
Gram-négatives, présentes dans le sol et appartenant aux
sous-classes alpha (á) et beta (â) des
Protéobactéries. La plupart des espèces se trouvent dans
la sous-classe á, divisée en sept groupes
monophylogénétiques (Annexe 1)
- Rhizobium et Allorhizobium ;
- Sinorhizobium (et Ensifer) ;
- Mesorhizobium ;
- Bradyrhizobium ;
- Methylobacterium (JAFTHA et al.,
2002 ; SY et al., 2001) ;
- Azorhizobium ;
- Devosia (RIVAS et al., 2002).
Dans la sous classe â, les rhizobia sont présents
dans deux groupes monophylogénétiques suivants :
- Burkholderia (MOULIN et al.,
2001) ;
- Ralstonia (CHEN et al., 2003).
En somme, la taxonomie bactérienne moderne vise
l'intégration de toutes les données phénotypiques,
génotypiques et phylogénétiques pour avoir une
classification plus stable (ZAKHIA et DE LAJUDIE, 2001; VANDAMME et
al., 1996). Le paragraphe suivant traitera de la classification actuelle
des rhizobia avec cette nouvelle approche.
III.3 - CLASSIFICATION ACTUELLE DES RHIZOBIA
La combinaison d'études polyphasiques et
phylogénétiques a permis de modifier considérablement la
taxonomie des rhizobia ces dernières années. Le tableau II
présente la diversité des rhizobia identifiés à
partir de cette nouvelle approche en 2006.
Tableau II : Classification des rhizobia
(D'après NOEL, 2009)
|
Protéobactéries
|
Espèces
|
|
Division
|
Genre
|
Nombre
|
Représentatives
|
Hôtes représentatives
|
|
Alpha
|
Rhizobium
|
16
|
R. leguminosarum
R. etli
R. tropici
|
Psivum, Trifolium etc.
Phaseolus
Phaseolus, leucaena
|
|
Bradrhyzobium
|
7
|
B. japonicum
B. elkanii
|
Glycine, Vigna
Glycine
|
|
Sinorhizobium (Ensifer)
|
11
|
S. eliloti
S. fredii
|
Medicago
Glycine, Vigna
|
|
Azorhizobium
|
2
|
A. caulinodans
|
Sesbania
|
|
Mesorhizobium
|
11
|
M. loti
|
Lotus spp.
|
|
(Allorhizobium)
|
1
|
A. undicola
|
Neptunia
|
|
Methylobacterium
|
1
|
M. Nodulans
|
Crotalaria Spp.
|
|
Devosia
|
1
|
D. neptuniae
|
Neptunia
|
|
Ochrobacterium
|
1
|
O. lupinus
|
Lupinus
|
|
Phyllobacterium
|
1
|
P. lupinii
|
Trifolium et Lupinus
|
|
Beta
|
Burkholderia
|
5
|
B. phymatum
|
Mimosa
|
|
Cupriavidus (Ralstonia)
|
2
|
C. taiwanensis
|
Mimosa
|
Au total, 59 espèces de rhizobia réparties en 12
genres et appartenant aux sous-classes alpha (10 genres) et beta (2 genres) des
Protéobactéries ont été identifiées depuis
2006 (NOEL, 2009). Ces espèces se répartissent également
en huit familles (Rhizobiacées, Phyllobacteriacées,
Bradyrhizobiacées, Hyphomicrobiacées,
Methylobactériacées, Brucellaceés, Burkholderiacées
et Ralstoniacées) et deux ordres, à savoir les Rhizobiales et les
Burkholderiales. L'ordre des Rhizobiales renferme les six premières
familles.
CONCLUSION PARTIELLE
La taxonomie des rhizobia a changé significativement
ces dernières années avec le développement des nouvelles
techniques d'études. Toutefois, cette taxonomie restera toujours
dynamique car bien que les rhizobia soient étudiés depuis plus de
100 ans, des symbiotes ont été identifiés pour moins de 10
% des 720 genres de légumineuses. Ainsi, il est probable qu'avec ces
nouveaux outils d'étude, de nouveaux genres de rhizobia soient
découverts parmi les sous-classes alpha et beta des
Protéobactéries et peut-être même parmi d'autres
taxons (VERNIE, 2008). Pour le pois d'Angole qui est `` une culture
négligée en termes de recherche '' (MINJA, 2001), cette
probabilité peut être même élevée.
CHAPITRE IV : GENERALITES SUR LE POIS D'ANGOLE
(Cajanus cajan L.)
Ces généralités portent sur l'origine,
la distribution géographique et la taxonomie de C. cajan. Elles
font également la description botanique de la plante et donnent ses
intérêts.
IV.1 - ORIGINE ET DISTRIBUTION GEOGRAPHIQUE DE LA
PLANTE
Le pois cajan ou pois d'Angole est la seule espèce
domestiquée du genre Cajanus qui en compte 34 (VAN DER MAESEN,
2006). Il est originaire de l'Inde et résulterait de
l'hybridation interspécifique entre ses parents proches sauvages que
sont C. cajanifolia et C. scarabaeoides (NADIMPALLI et
al., 1992). L'Inde serait son centre de domestication qui aurait
lieu un peu plus de 2000 ans avant J.C. Après sa domestication, le pois
d'Angole a été introduit successivement en Malaisie, en Afrique
de l'Est puis en Egypte à travers la vallée du Nil. Par la suite,
il a été introduit en Afrique de l'ouest, en l'an 2000 avant J.C
(VAN DER MAESEN, 1990). On estime que, préalablement à la traite
négrière, sa culture a été exportée de la
République démocratique du Congo et de l'Angola vers le nouveau
monde (Amérique) (VAN DER MAESEN, 1986). Plus tard, la traite
négrière l'a introduite aux Antilles où il a
été nommé pois des pigeons en 1962 (VAN DER MAESEN,
1986).
IV.2 - ZONES DE CULTURE ET IMPORTANCE DES
PRODUCTIONS
Aujourd'hui, le pois d'Angole est présent sous tous
les tropiques et occupe plus de 4 millions d'hectares dans le monde. Mais, sa
culture est mieux adaptée et plus productive sous les tropiques
semi-arides. Il est particulièrement cultivé dans le
sous-continent indien où sa production représente plus de 70% de
la production mondiale (FAOSTAT, 2007). Le Sud et l'Est du continent africain,
particulièrement le Kenya, le Malawi, la Mozambique, la Tanzanie et
l'Ouganda, constituent sa deuxième plus grande zone de culture (SINGH,
1991). Les autres zones de production d'importance sont le sud-est du
continent asiatique, l'Afrique centrale et de l'ouest et enfin le continent
américain (NENE et SHEILA, 1990).
Selon les statistiques de la FAO, la production mondiale de
pois cajan entre 1999 et 2003 s'élevait à 3,1 millions de tonnes
par an sur 4,3 millions d'hectares (ha) de culture. L'Inde (principal pays
producteur) avait une production de 2,5 millions de tonnes (t) sur 3,4 millions
d'ha. Les principaux pays producteurs d'Afrique ont été le Malawi
(79 000 t), l'Ouganda (78 000 t), le Kenya (59 000 t), la
Tanzanie (47 000 t) (VAN DER MAESEN, 2006).
IV.3 - TAXONOMIE DU POIS D'ANGOLE
IV.3.1 - Nomenclature
Le nom scientifique du pois d'Angole est Cajanus
cajan (L.). Plusieurs synonymes de ce nom existent. Ce sont Cajanus
bicolor DC., Cajanus flavus DC., Cajanus indicus Spreng et Cystisus
cajan L. Il existe aussi une multitude de noms communs
utilisés pour désigner la plante selon les pays et les langues
(tableau 3).
Tableau III : Noms
communs du pois d'Angole en quatre langues principales
|
Langues
|
Noms communs
|
|
Anglais
|
Congo pea, pigeon pea, redgram, yellow dahl
|
|
Français
|
ambrevade, pois d'Angole
|
|
Inde
|
arhar, tuver
|
|
Espagnol
|
cachito, gandul
|
IV.3.2- Classification
Cajanus cajan est la seule espèce
domestiquée dans la sous-tribu des Cajaninae de la tribu des
Phaseolae Benth., appartenant à la sous-famille des
Papilionoidae et à la famille des Leguminosae
(BENTHAM, 1865). Sa classification entière est ci-dessous
donnée:
Règne : Végétaux
(Plantae)
Embranchement :
Spermaphytes (Spermaphyta)
Sous-embranchement : Angiospermes
(Magnoliophyta)
Classe : Dicotylédones
(Magnoliopsida)
Ordre : Fabales (Léguminosales)
Famille : Fabacées
(Leguminosae)
Sous-famille : Papilionoideae
(Faboideae),
Tribu : Phaseoleae Benth.
(Phaséolidées)
Sous-tribu : Cajaninae
Benth.
Genre : Cajanus
Espèce: C. cajan
(L.) Millspaugh.
Il existe deux variétés «traditionnelles''
au sein de l'espèce C. cajan (NIYONKURU, 2002). Il s'agit d'une
part de Cajanus cajan var. bicolor. Cette variété est
pluriannuelle et caractérisée par des fleurs jaunes et rouges
avec de longues gousses (5-7 graines). La deuxième
variété, Cajanus cajan var flavus, est à
floraison précoce et cultivée en plante annuelle. Elle est
identifiable par ses fleurs jaunes et ses gousses plus courtes (3 graines).
De nombreuses variétés
sélectionnées par l'ICRISAT (Institut international de recherche
sur les plantes cultivées des zones tropicales semi-arides) sont
également cultivées. Ce centre est basé en Inde (VAN DER
MAESEN, 2006).
IV.4- DESCRIPTION DU POIS D'ANGOLE
IV.4.1- Description botanique
Le pois d'Angole est un arbuste dont la durée de vie en
culture varie entre 3 et 5 ans. Son port érigé atteint 4 m de
hauteur avec des racines atteignant 2 m de profondeur (DUKE, 1981 a).
Sa tige, qui peut avoir 15 cm de diamètre dans sa
partie basale, porte de nombreuses branches et brindilles. Ces dernières
supportent un feuillage abondant de couleur vert-clair ou vert-jaune et
formé de feuilles trifoliolées. Les feuilles
sont disposées en spirales sur les tiges.
Les inflorescences sont des racèmes de 4 à 12 cm
de long situés aux extrémités des branches et comportant 6
à 12 fleurs. Les fleurs ont quatre lobes de calices
séparées. Les pétales ont une couleur qui évolue du
jaune au rouge-pourpre (Figure 7 a) (BORGET, 1989).
Le fruit est une gousse linéaire-oblongue de 4 à
10 cm et renfermant des graines arrondies. Les gousses, de couleur
crème, sont comestibles, déhiscentes à maturité et
ont un hile foncé avec une extrémité en bec. Le nombre de
graine par gousse varie de 2 à 8, avec la couleur du tégument
allant du blanc, blanc-brun, beige, marron, rougeâtre au tacheté
(Figure 7 b) (SEDGA, 1997; NIYONKURU, 2002).


Figure 7 a : Inflorescences de Cajanus
cajan Figure 7 b : Fruit sec en
déhiscence
IV.4.2 - Ecologie
En culture, le pois cajan peut supporter des
sécheresses allant jusqu'à cinq (5) mois, tolère les sols
salés ainsi que les sables aux argiles lourds. Il
préfère un pH de 5 à 7, mais peut tolérer un
pH allant de 4,5 à 8,4 (DUKE, 1981a ; VAN DER MAESEN, 2006).
Ses pluviométries optimales sont comprises entre 600 et
1000 mm de pluie par an. Toutefois, il se développe bien dans les zones
semi-arides et les zones humides avec des pluviométries atteignant
respectivement 400 et 2500 mm par an (BARTHOUX, 1975).
Cajanus cajan L. se développe mieux
en pleine lumière du soleil mais peut tolérer un faible ombrage.
Cependant, il est très sensible au bas rayonnement qui engendre une
production médiocre de graines (SPORE, 2010).
La plante se développe dans des conditions
chaudes avec températures optimales comprises entre 17 et 28°C.
Lorsque la température dépasse 35°C, elle souffre et l'on
constate la chute des fleurs et des gousses (SPORE, 2010).
IV.4.3 - Mode de multiplication et systèmes de
culture
La culture du pois cajan se fait
généralement à partir des grains et par semis direct
(BORGET, 1989). Les traitements de pré-germination des grains avant le
semis ne sont pas nécessaires et la profondeur de semis varie de 2,5
à 5 cm. Quant à la densité de semis, il dépend du
système de culture. Pour une jachère améliorée, il
faut observer un espacement de 1 m x 1,50 m, contre 3 m x 3 m si la culture
vise la production de grains (SKERMAN, 1982). Le nombre de grains
recommandé par poquet est d'au moins deux.
En Afrique et en Inde, le pois cajan se cultive souvent en
association, avec des céréales, mais également avec le
manioc et le cotonnier. Il est bien adapté aux systèmes de
cultures associées en raison de sa croissance initiale lente, qui
réduit la compétition avec la plante qu'il accompagne, et de sa
maturité tardive, qui permet d'étaler les besoins de main
d'oeuvre au moment de la récolte (VAN DER MAESEN, 2006).
IV.4.4 - Croissance, développement et rendement
La germination des grains de pois cajan semés a lieu 2
à 3 jours après semis (JAS), à des températures
comprises entre 19 et 43°C. La levée est complète 2 à
3 semaines après le semis et le développement
végétatif, qui débute lentement, s'accélère
après 2 à 3 mois.
En général, la floraison (de 50% des plantes)
commence 56 à 210 jours après le semis et la maturité de
la graine est atteinte entre 95 et 260 jours. Toutefois, la durée totale
du cycle de production est fonction des cultivars. En Inde par exemple, on
distingue 10 groupes de cultivars, que l'on combine d'ordinaire en quatre
catégories : les cultivars à maturité extra
précoce, précoce, moyenne et tardive (120, 145, 185, et plus de
200 JAS respectivement).
Dans des conditions optimales de culture et en culture pure,
les rendements en grains du pois cajan peuvent atteindre 5000 kg/ha. Toutefois,
les rendements sont en général faibles (600kg/ha en moyenne en
Afrique) et cela est dû en partie qu'une part considérable des
grains est récoltée et consommée avant la maturité.
Les rendements en fourrage de la plante sont de l'ordre de 3-8 t/ha. Comme
combustible, on obtient généralement 7-10 t/ha, mais des
rendements atteignant 30 t/ha ont été déjà
enregistrés (VAN DER MAESEN, 2006).
IV.4.5 - Ennemis et maladies de Cajanus cajan
La maladie la plus importante du pois d'Angole est le
flétrissement fusarien causé par Fusarium udum
et présente dans toutes les grandes régions de culture
(NIYONKURU, 2002). La forte humidité des sols favorise l'installation de
l'agent pathogène qui infecte les vaisseaux des racines et des tiges et
cause leur dessèchement progressif. Le virus de la mosaïque et de
la stérilité du pois cajan (PPSMV) constitue tout de même
sa principale maladie en Inde (VAN DER MAESEN, 2006). Les plantes atteintes par
cette maladie présentent sur les feuilles, des symptômes
caractéristiques de la mosaïque avec réduction ou absence de
floraison (stérilité). Les baisses de rendement observées
peuvent atteindre dans certains cas, plus de 90% (KANNAIYAN et al.,
1984).
Parmi les ravageurs, les borers des gousses
restent de loin les plus dangereux. Ces chenilles foreuses
(Helicoverpa armigera, Maruca testutalis et Laspeyresia ptychora)
peuvent dans certaines circonstances provoquer jusqu'à 80% de pertes.
Elles sont d'ailleurs souvent précédées de punaises
suceuses (Clavigralla tomentosicolis et Nezara
viridula).
D'autres dégâts sont également
causés par les nématodes, notamment les nématodes à
galles (Meloidogyne spp.) et les nématodes réniformes
(Rotylenchus spp.).
La lutte contre ces nuisibles repose sur l'utilisation de
variétés résistantes, les rotations culturales ainsi que
l'utilisation de produits chimiques (VAN DER MAESEN, 2006).
IV.5 - IMPORTANCE DE LA CULTURE DU POIS CAJAN
Le pois cajan est une importante légumineuse à
grains de la tribu des Phaseoleae tout comme les genres Phaseolus et
Vigna (VAN DER MAESEN, 1990). Les intérêts de sa culture
se situent à plusieurs niveaux : alimentation humaine et animale,
agriculture, médecine etc.
IV.5.1 - Importance nutritionnelle du pois cajan
Le pois cajan est principalement cultivé pour ses
grains dont la valeur nutritive est comparable à celle du haricot commun
(Phaseolus vulgaris) (NIYONKURU, 2002). En effet, connus pour
être une excellente source de protéines (21,7%), les grains
mûrs du pois cajan sont une bonne source d'énergie (343 kcal par
100 g de partie comestible), de vitamines (A 28 UI, B6) et d'acides
aminés essentiels (lysine, phénylalanine, valine, leucine et
isoleucine). Les grains sont également riches en acides gras dont les
principaux sont l'acide linoléique et l'acide palmitique (USDA,
2004 ; VAN DER MAESEN, 2006). La consommation de ces grains est surtout
importante dans les pays tropicaux en développement (Inde, Kenya
etc.).
En alimentation animale, le feuillage coupé constitue,
frais ou conservé, un bon fourrage servant à nourrir le
bétail. En effet, les feuilles sont riches en protéines
(21-25%/Matière Sèche (MS)) et en fibres (30-35% de cellulose
brute/MS) (GRIMAUD, 1988). Pour les animaux de pâturage, l'on peut
réaliser des coupes tous les deux ou trois mois avec une
productivité de 1,5 à 3,5 t de MS/ha/par coupe et une valeur
azotée de 100 à 120 g de Matière Azotée Digestible
(MAD)/100 kg de matière sèche (ANONYME, 2002). Par
ailleurs, des essais, conduits en station, ont montré qu'une
introduction de graines de pois cajan ayant subi un broyage comme seule
transformation dans l'aliment des porcs et des volailles n'entraînait
aucun trouble de croissance chez ceux-ci. Les pourcentages d'incorporation
pouvant être atteints sont de 15% chez le porc à l'engrais et 25%
à 30% chez le poulet de chair (GRIMAUD, 1988).
IV.5.2 - Importance agronomique du pois cajan
Le pois cajan offre de nombreux avantages aux agriculteurs
à faibles ressources y à savoir du combustible, du
matériel de clôture, l'amélioration de la fertilité
et le contrôle de l'érosion des sols (SIAMBI et
al., 1992). Il peut aussi être
utilisé comme brise-vent et comme plante de couverture dans certaines
plantations de cultures industrielles, notamment celles du caféier
(BARRIOS et al., 1997 ; BASHIR et al., 1998). Egalement,
grâce à son système racinaire étendu, à
l'azote atmosphérique qu'il fixe et au mulch que procurent ses feuilles
rejetées durant sa culture (Figure 8), le pois cajan
améliore significativement la fertilité des sols (VAN DER MAESEN,
2006). A titre d'exemple, la plante peut fixer jusqu'à 235 kg d'azote
par hectare de culture et s'avère de ce fait très
intéressante pour une agriculture protectrice de l'environnement
(PEOPLES et al., 1995). Quant aux résidus d'azote
laissés par culture, ils avoisinent 40 kg/ha (VAN DER MAESEN, 2006) ;
ce qui renforce la fertilité des sols pour les cultures en rotation.

Figure 8 : Feuillage
rejeté par une culture de pois cajan en jachère
améliorée
(TA BI, 2010)
IV.5.3 - Importance du pois cajan en Afrique
En Afrique, le pois d'Angole est une plante de très
grande importance notamment en alimentation humaine et animale. Dans le
Nord-est de la Côte d'Ivoire, ses grains constituent pour les populations
locales, en l'occurrence les Koulango et les Abrons, un aliment de soudure
(NDABALYSHE, 1995). Elle est utilisée pour la complémentation
animale (bovins, caprins etc.), surtout en saison sèche et sert de
matériaux pour la reconstitution des pâturages dans les îles
du Cap Vert (LEPAPE, 1980). Cajanus cajan est par ailleurs
utilisé comme plante améliorante des jachères en
agroforesterie en Zambie (BOEHRINGER et CADWEL, 1989). Toujours sur le
continent, son association avec le maïs dans plusieurs programmes
d'expérimentation, a donné des résultats
intéressants. Au Malawi par exemple, le rendement du maïs,
cultivé en rotation avec le pois cajan, a accru de 2,8 kg/ha par rapport
à sa culture continue recevant 35 kg N/ ha (MACCOLL, 1989). Au Nigeria,
des études similaires ont montré une augmentation de 50% du
rendement du maïs par rapport à sa culture sans engrais (HULUGALLE
et LAL, 1986). Par ailleurs, C. cajan fait partie des plantes de
couverture les mieux adoptées au Bénin dans la lutte contre
Imperata cylindrica, en raison de l'exploitation supplémentaire de
son bois, ses graines et ses feuilles comestibles (VISSOH et
al., 2004).
CONCLUSION PARTIELLE
En somme, le pois cajan est une légumineuse de grand
intérêt aussi bien pour l'alimentation humaine, animale que pour
la pratique agricole. Le chapitre suivant fait une synthèse des rhizobia
identifiés jusqu'à ce jour comme ses symbiotes pour la fixation
azotée.
CHAPITRE V : RHIZOBIA SYMBIOTIQUES DU POIS
CAJAN
Les travaux de recherche effectués sur les
bactéries nodulant le pois d'Angole ont permis d'identifier trois genres
symbiotiques. Il s'agit premièrement du genre Rhizobium. En
effet, on a isolé Rhizobium IHP 100 des nodules de
Cajanus cajan (BENDER et al., 1986). Par l'étude du
polymorphisme de la longueur des fragments de restriction (RFLP) des
gènes de l'ARNr 16S, de l'ARNr 23S, de l'espace intra-génique
transcrit (ITS) 16S-23S amplifiés ainsi que par le
séquençage partiel de l'ARNr 16S, il a été
confirmé en Ethiopie que ce genre est effectivement symbiote du pois
cajan (WOLDE-MESKEL et al., 2004 a).
En 1988, deux espèces bactériennes du genre
Sinorhizobium (S. fredii et S. xinjiangense) furent
également identifiées comme symbiotes du pois cajan à
partir de travaux menés en Chine (CHEN et al., 1988).
Sinorhizobium fredii fut identifié à partir d'un test
d'hybridation ADN-ADN et de quelques tests physiologiques. Quant à
S. xinjiangense, il fut identifié à partir
d'une analyse numérique de 240 différents caractères (CHEN
et al., 1988).
En 1981, il a été observé que des
souches de Bradhyrizobium isolées des nodules du
niébé (Vigna sp.) au Nigeria nodulaient le pois cajan
(AHMAD et al., 1981 a, 1981 b). Par ailleurs, des travaux
plus récents ont permis de confirmer que le pois cajan est l'hôte
de certaines souches de Bradyrhizobium. La
technique du polymorphisme de la longueur des fragments de restriction de
l'ADNr 16S a été couplée à une amplification
aléatoire des fragments d'ADN digérés pour obtenir ces
résultats. Il s'agit de la souche B. spp. TAL 1039
isolée au Kenya (ABAIDOO et al., 2000) et des
souches B. spp. TAL 36 et B. spp. TAL 191 isolées au
Nigeria (ABAIDOO et al., 2000). Egalement, l'étude de la
diversité de 30 isolats de rhizobia à croissance lente nodulant
le pois d'Angole, a montré que 16 d'entre eux étaient
phylogénétiquement proches du rhizobia à croissance lente,
Bradyrhizobium elkanii, nodulant le soja (RAMSUBHAG et al.,
2002). L'analyse numérique de 80 traits phénotypiques et le
séquençage partiel de l'ARNr 16S ont permis d'aboutir à
ces résultats. Toutefois, seulement 3 de ces 16 isolats ont
été capables de noduler le soja. Enfin, la PCR-RLFP du
gène de l'ARNr 16S et 23S, de l'ITS 16S-23S ainsi que le
séquençage partiel de l'ARNr 16S ont permis d'identifier
Bradyrhizobium japonicum comme symbiote de Cajanus cajan
(WOLDE-MESKEL et al., 2004 a).
En somme, l'émergence de techniques moléculaires
d'identification telles que la PCR-RFLP et le séquençage de
l'ADNr 16S, ont permis ces dernières années de mieux explorer la
diversité génétique des rhizobia symbiotes du pois cajan
à travers le monde.
DEUXIEME PARTIE :
ETUDE EXPERIMENTALE
CHAPITRE VI : PRESENTATION DE L'ETUDE ET DES
LABORATOIRES D'ACCUEIL
VI.1- OBJECTIFS DE L'ETUDE
Dans cette étude, on évalue la diversité
génétique des rhizobia associés à des plants
de pois d'Angole issus d'un champ cultivé à Yamoussoukro.
Il s'agit à partir de nodosités
prélevées :
- d'isoler les bactéries symbiotiques ;
- de les mettre en culture et les purifier ;
- de révéler la diversité
génétique de ces bactéries à partir d'une PCR-RFLP
;
- et enfin, de constituer une collection de celles-ci et les
conserver.
VI.2- PRESENTATION DU SITE DE COLLECTE DES
ECHANTILLONS
Le matériel vivant utilisé (rhizobia) a
été collecté à partir de jeunes plants de
Cajanus cajan (4 à 14 mois) issus d'un champ cultivé
à l'INP-HB. Ce champ, d'une superficie totale d'environ 1,5 ha, est la
propriété du directeur des études du cycle des
ingénieurs agronomes de l'Ecole Supérieure d'Agronomie (ESA). Il
l'a mis en place en vue de produire des grains de pois cajan pour
l'alimentation de sa volaille. Le champ est localisé à droite du
chemin menant à l'ESA via le garage de l'INP Centre,
précisément à 100 m dudit garage.
VI.3- PRESENTATION DES LABORATOIRES D'ACCUEIL DE
L'ETUDE
Le principal laboratoire d'accueil de l'étude est
celui d'Agronomie et Productions Végétales de l'Institut National
Polytechnique Félix Houphouët Boigny (INP-HB) de Yamoussoukro
(Annexe 2). Ce laboratoire a servi à l'isolement, la
culture et la purification des souches rhizobiennes. L'analyse
moléculaire de ces isolats a eu lieu au Laboratoire Central de
Biotechnologie (LCB) du Centre National de Recherche
Agronomique (CNRA) d'Abidjan-Adiopodoumé, précisément au
sein de l'unité de virologie et de biologie moléculaire
(Annexe 2).
CHAPITRE VII : MATERIEL ET METHODES
VII.1- MATERIEL
VII.1.1 - Matériel vivant
Le matériel vivant utilisé est constitué
de 169 souches de rhizobia isolées d'environ 200 nodosités
collectées. Les figures 9 a et
9 b suivantes présentent respectivement quelques
nodosités sur racines et des nodosités préparées en
vue de l'isolement des bactéries.
 
Figure
9 a: Nodosités sur racines de
C. cajan Figure
9 b: Nodosités
étalées pour l'isolement
VII.1.2 - Matériel technique
Le matériel technique utilisé dans cette
étude est constitué de plusieurs équipements de
laboratoire et des kits de réactifs. En effet, pour l'isolement, la
culture et la purification des bactéries au Laboratoire de Productions
Végétales de l'INPHB, un kit de réactif pour milieu TY
(Tryptone Yeast Extract), des tubes Eppendorf et plus d'une centaine
de boîtes de pétri ont été utilisés. Au LCB
du CNRA d'Adiopodoumé, la constitution de la collection locale de
rhizobia a nécessité 2 plaques à 96 puits chacune et du
glycérol. La PCR a été possible grâce à un
kit PCR contenant des amorces spécifiques à l'ADNr 16S des
rhizobia, une enzyme de polymérisation (la Taq DNA polymérase) et
divers appareils de laboratoire. Le thermocycleur est le plus important de tous
ces appareils (figure 10 a). Celui-ci est muni d'un bloc
thermique de 96 puits (figure 10 b) où l'on peut
insérer les tubes contenant le mélange réactionnel de la
PCR. Il est aussi équipé d'un couvercle qui pressant sur les
capuchons des tubes, permet d'éviter l'évaporation du
mélange réactionnel. Un clavier composé de quelques
touches et un écran à cristaux liquides permettent d'entrer des
programmes PCR dans la mémoire de la machine. Dans notre cas, nous avons
utilisé un thermocycleur de type BIOMETRA UNO II.
 
(a) : Vue
générale (b) : Bloc
thermique
Figure 10 :
Présentation d'un thermocycleur (Photos AVO, 2010)
Les autres appareils de laboratoire sont constitués
d'une centrifugeuse utilisée pour séparer la phase aqueuse (le
surnageant) du culot des bactéries cultivées sur milieu TY
liquide. Il y a également un bain-marie servant à faire le choc
thermique des bactéries afin d'avoir accès à quelques
fragments d'ADN pour l'amplification. On cite aussi une machine à glace
nécessaire pour la conservation des réactifs lors de la
préparation de la solution réactionnelle, une micro-onde
idéale pour la préparation de gel d'agarose et un appareil
Mupid-One (figure 11) pour les migrations
électrophorétiques. Un transilluminateur à caméra
relié à un ordinateur (figure 12) muni du
logiciel AlphaDigiDoc a permis de visualiser les fragments d'ADN après
un temps de migration.
 
Figure 11: Appareil de migration
Figure 12 : Transilluminateur relié à un
ordinateur
(Photo AVO, 2010)
(Photo AVO, 2010)
Les fragments amplifiés ont
été digérés dans le bain-marie grâce à
une enzyme de restriction, la Tsp 509I -R0576S. Pour la migration et
la visualisation des résultats de cette digestion, les mêmes
appareils ayant permis d'observer les résultats de la PCR ont
été utilisés.
VII.2- METHODES
Les méthodes évoquées
ici sont celles de la récolte des nodules, de l'isolement, la culture et
la purification des bactéries, de l'amplification du gène cible
ADNr 16S ainsi que de sa digestion pour évaluer la diversité
génétique des bactéries. La méthode de constitution
de la collection locale des bactéries est également
exposée.
VII.2.1 - Récolte des nodules et isolement des
souches bactériennes
Les nodules ont été récoltés au
champ à l'aide d'une tarière. Des tubes Ependorf de 15
ml, remplis d'eau à moitié, ont servi à maintenir
hydratés les nodules afin d'assurer la survie des bactéries. Au
laboratoire, leur isolement a suivi le protocole suivant:
- les nodules ont été lavés avec de l'eau
distillée pour enlever toute trace de terre ;
- chaque nodule lavé superficiellement avec l'eau
distillée a été désinfecté par trempage dans
de l'eau de javel pendant une trentaine de secondes ;
- après ce bref temps, l'eau de javel a
été immédiatement éliminée par une
série de rinçages des nodules avec de l'eau distillée
stérile ;
- les nodules ont été ensuite
étalés un à un, sur des gouttes d'eau dans des
boîtes de pétri ;
- emmenés sous une hotte, ils ont été
écrasés au moyen de cônes préalablement
stérilisés à 120°C pendant 2 heures;
- l'isolement des bactéries a été
réalisé par prélèvement d'une quantité du
broyât de chaque nodule à l'aide d'un cure-dent
stérilisé, puis son ensemencement sur du milieu TY solide
(BERINGER, 1974) (Annexe 3) coulé dans des boîtes
de pétri et divisé en quartiers;
- les milieux ensemencés puis identifiés ont
été ensuite incubés à l'étuve à
30°C ;
- Après 24 heures ou 48 heures passées à
l'étuve, chaque culture bactérienne a été
retirée et conservée au réfrigérateur.
Par la suite, les bactéries ont
été cultivées continuellement sur des boîtes de
pétri, à intervalles de temps variables, en vue de les purifier.
Après trois cycles de purification à l'INP-HB, les souches ont
été cultivées sur milieu TY solide, coulé en
inclinaison dans des tubes Eppendorf stériles de 15 ml. Cette
dernière culture a servi pour les manipulations au Laboratoire Centrale
de Biotechnologie (LCB) du CNRA à Adiopodoumé.
Au LCB, les souches issues de cette dernière
culture ont été cultivées une fois sur milieu TY liquide
pour l'étude de la diversité, à raison de 3 ml du milieu
par souche. Une réplique de cette dernière culture a
été également réalisée pour une longue
conservation de toutes les souches à -80°C. Les deux plaques de 96
puits chacune ont été utilisées à cet effet et
chaque puits a eu comme contenu, 100 ul de suspension bactérienne +
quelques gouttes de glycérol.
Cette réplique constitue l'ébauche de la
collection locale des rhizobia symbiotes du C. cajan.
VII.2.2 - Préparation des cellules
bactériennes par effet thermique
La PCR permettant d'amplifier à partir de petits
fragments le gène ciblé sur le génome, on a
effectué un choc thermique sur les bactéries afin d'avoir
accès à quelques fragments d'ADNr 16S. Pour chaque
bactérie, ce choc thermique a eu lieu sur 1 ml de sa suspension de 3 ml
préalablement cultivée pendant 2 jours dans un
incubateur-agitateur à 30°C. Les différentes
étapes de ce choc thermique sont les suivantes :
- passage sur glace de chaque prélèvement
bactérien (1 ml) contenu dans un tube de 1,5 ml ;
- centrifugation à 12000 t/min pendant 10 min de toutes
les souches prélevées;
- recueil du culot bactérien pour chaque
souche centrifugé;
- reprise du culot dans 100 ul d'eau distillée
stérile et homogénéisation au vortex;
- passage sur glace des différentes souches ;
- chauffage des souches au bain-marie à 100°C
pendant 10 min ;
- enfin, retour sur glace jusqu'à refroidissement total
des souches.
Chaque bactérie ainsi préparée est
prête pour la réalisation de la PCR-RFLP sur l'ADNr 16S.
VII.2.3 - Amplification et digestion de la région
16S de l'ADNr des rhizobia par PCR-RFLP
VII.2.3.1 - Principe de la PCR-RFLP et choix du
gène de l'ADNr 16S
VII.2.3.1.1 - Principe de la
PCR-RFLP
L'approche PCR-RFLP conduit à comparer la longueur des
fragments de restriction d'une région choisie du génome et
préalablement amplifiée par PCR, afin de déterminer le
polymorphisme. Cette région est utilisée comme substrat pour les
enzymes de restriction. Les enzymes de restriction sont des
endonucléases qui reconnaissent spécifiquement une
séquence courte (4 à 8 bases) et coupent la chaîne d'ADN
chaque fois qu'elles reconnaissent cette séquence
élémentaire. L'ADN se retrouve ainsi fragmenté en morceaux
de différentes longueurs séparés en fonction de leur
taille par électrophorèse sur un support physique. Un fragment va
migrer d'autant plus loin qu'il est court. Un polymorphisme de la longueur des
fragments de restriction est ainsi mis en évidence. Les profils
observés permettent l'analyse de la diversité des souches
par :
- la caractérisation de chaque isolat ;
- l'estimation des divergences de séquences entre
isolats et l'établissement de leur proximité
phylogénétique (NDOYE, 1998).
VII.2.3.1.2 - Choix du gène de l'ADNr
16S
Les gènes ribosomiques sont utilisés comme
marqueurs moléculaires dans l'étude phylogénétique
de différents organismes en raison de leur universalité, leur
abondance, leur taille convenable aux analyses comparatives (LUDWIG et
SCHLEIFER, 1999). Les ADNr contiennent par ailleurs des régions de
séquences hautement conservées, très utiles pour la
désignation des amorces (HILLIS et DIXON, 1991 ; STAHL et AMMAN, 1991)
et d'autres régions de séquences suffisamment variables pour
servir comme un excellent moyen taxonomique (GRIMONT et GRIMONT, 1986).
Chez les procaryotes particulièrement, les gènes
codant pour les ARN ribosomaux sont organisés en opérons qui
contiennent également des espaces intra-géniques ainsi que
d'autres gènes codant pour les ARN de transfert (ARNt) (figure
13).
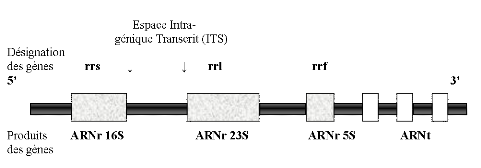
Figure 13 : Organisation de
l'opéron de l'ADNr observé chez les procaryotes
(D'après EL HILALI, 2006)
Comme le montre la figure 13, il existe
trois types d'ADN ribosomique : le 5S, le 16S et le 23S (JENSEN et STRAUS,
1993). L'ADNr 5S est très peu utilisé dans les études de
diversité vu sa petite taille d'environ 120 nucléotides.
Contrairement à ce dernier, l'ADNr 16S codant pour la petite sous
unité ribosomique d'environ 1500 pb (opéron rrs : ribosomal
RNA small subunit) et l'ADNr 23S codant pour la grande sous unité
d'environ 2500 à 3000 pb (opéron rrl : ribosomal RNA large
subunit) (GÜRTLER et STANISICH, 1996) sont très
utilisés. L'espace entre ces deux opérons est transcriptionnel
d'où la désignation ITS (Internal Transcribed Spacer)
(NORMAND et al., 1996). Il est aussi utilisé dans ces
études. Toutefois, depuis que WOESE (1987) a montré que le
gène de l'ADNr 16S est présent chez toutes les bactéries,
qu'il a la même fonction et que sa structure est conservée,
plusieurs chercheurs l'ont préférentiellement utilisé
comme une approche rapide pour évaluer la variabilité
génétique entre les souches de rhizobia (LAGUERRE et
al., 1994 ; NOUR et al., 1994b).
VII.2.3.2- Réalisation de la PCR-RFLP
VII.2.3.2.1 - Principe de la PCR
La PCR (SAIKI et al., 1985 ;
MULLIS et FALOONA, 1987) se réalise dans un mélange
réactionnel contenant de faibles quantités d'ADN possédant
la séquence à amplifier et utilisées comme matrice, des
paires d'amorces nucléotidiques complémentaires des
séquences qui encadrent la cible à amplifier et un ensemble de
quatre dNTP (dATP, dTTP, dCTP et dGTP) nécessaires à la
synthèse de nouveaux brins d'ADN. A ce mélange, s'ajoute l'enzyme
de polymérisation, la Taq DNA polymérase. C'est une enzyme
thermorésistante qui fut isolée à l'origine de Thermus
aquaticus, une bactérie thermophile.
Classiquement, la PCR consiste en la répétition
de trois étapes thermiques réalisées successivement et
connu sous le nom d'étapes d'un cycle d'amplification. Chaque
étape est caractérisée par un temps et une
température de réalisation qui déterminent
l'efficacité de toute la PCR. Dans notre cas, le cycle d'amplification
se présente comme suit :
· Première
étape : le double brin d'ADN est d'abord
chauffé à 94°C pendant 30 secondes ; ce qui permet de
rompre les liaisons entre les deux brins. C'est l'étape de
dénaturation ;
· Deuxième
étape : la température est abaissée
à 55°C pendant 30 secondes. Au cours de cette étape les
amorces s'apparient spécifiquement sur leurs séquences
complémentaires aux bornes de la région à amplifier,
chacune sur son brin. C'est l'étape d'amorçage ou
d'hybridation, annealing. La spécificité de la PCR
repose sur la qualité de cet appariement lié à la taille
des amorces et à leurs séquences ;
· Troisième
étape : la température est
élevée à 72°C pendant 1 minute 30 secondes. A cette
température, la polymérase allonge les nouveaux brins d'ADN
à partir des amorces en assemblant des dNTP. On parle d'étape de
polymérisation, d'extension des amorces ou d'élongation
des brins d'ADN (Figure 14).
À la fin de chaque cycle d'amplification, la
séquence définie par les amorces initiales est dupliquée.
Les brins d'ADN néoformés ainsi que les brins initiaux servent de
matrice dans les cycles successifs d'amplification. C'est la
répétition des cycles de
dénaturation-hybridation-élongation qui conduit finalement
à une accumulation exponentielle de l'ADN, c'est-à-dire à
la réalisation de la PCR. La figure 14
ci-après présente le cycle d'amplification de la PCR que
nous avons réalisée au LCB du CNRA à
Adiopodoumé.
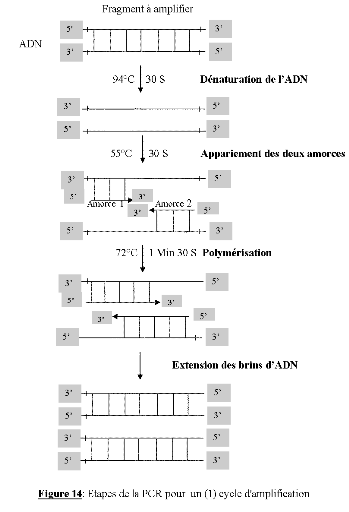
VII.2.3.2.2 - Choix des amorces et milieu
réactionnel de la PCR
Le tableau 4 présente la liste et les
caractéristiques de la paire d'amorces utilisées.
Tableau IV :
Amorces choisies pour l'amplification des fragments ADNr
16S
|
Amorces
|
Séquences 5'3'
|
Sites
|
Références
|
|
FGPS 6
|
GGAGAGTTAAGATCTTGGCTCA
|
ADNr 16S
|
NORMAND et al., 1992
|
|
FGPS1509
|
AAGGAGGGGATCCAGCCGGA
|
ADNr 16S
|
NORMAND et al., 1992
|
Ces deux amorces utilisées sont spécifiques de
l'ADNr 16S des rhizobia. Elles furent mises au point en 1992 (NORMAND
et al., 1992) et utilisées dans plusieurs études de
diversité des rhizobia basée sur cette région (NDOYE,
1998 ; SY et al., 2001 etc.).
Avec ces amorces et les autres réactifs, le milieu
réactionnel de la PCR se présente comme suit (Tableau
5) :
Tableau V : Composition du milieu
réactionnel unitaire commun de la PCR
|
Concentration de travail
|
Volume utilisé pour un échantillon
|
|
Dntp
|
2 ,5 Mm
|
2 ul
|
|
Amorce 1 (FGPS 6)
|
2 uM
|
2,5 ul
|
|
Amorce 2 (FGPS 1509)
|
2 uM
|
2,5 ul
|
|
Tampon X 10
|
1 X
|
2,5 ul
|
|
Taq DNA polymerase
|
-
|
(0,5/15) ul
|
|
Eau PCR
|
-
|
13 ul
|
|
Volume réactionnel partiel
|
22,5 ul
|
Pour réaliser la PCR, chaque tube PCR
a comporté en plus de ce milieu réactionnel commun, 2,5 à
4 ul d'ADN d'une et unique souche bactérienne. Ainsi, le volume
réactionnel total par souche a été de 25 ul, le volume
d'eau PCR ayant été ajusté en conséquence dans
chaque cas. Au total, il y a eu 169 tubes PCR à 25 ul de réactif
pour autant de souches à amplifier. Pour chaque réaction
d'amplification, un microtube contenant uniquement le milieu réactionnel
commun a été utilisé comme témoin
négatif.
VII.2.3.2.3 - Programme de la PCR
Au cours de notre PCR, le cycle
d'amplification à 3 étapes a été
répété 35 fois. Celui-ci définit avec 2 autres
cycles (une phase de préchauffage à 94 °C pendant 5 minutes
et une phase terminale de réparation des bouts des fragments à
72°C pendant 7 min), le programme total de la PCR qui dure 2 heures 7
minutes. Ce programme se présente comme suit :
Etape 1 : 94°C-------- 5 minutes
Etape 2 : 94°C-------- 30
secondes
Etape 3 : 55°C-------- 30 secondes
35 cycles
Etape 4 : 72°C-------- 1 minute 30
seconde
Etape 5 : 72°C-------- 7 minutes
A la fin de la PCR, les amplifiats peuvent
être conservés dans le thermocycleur à une
température de 4°C. Cette phase de conservation correspond à
une étape dite de pause. Dans le cas contraire, vers la fin du temps
nécessaire à la PCR (2 h 7 min), un gel d'agarose à 0,8%
est préparé pour réaliser l'électrophorèse
des gènes une fois l'amplification terminée.
VII.2.3.2.4 - Electrophorèse sur gel d'agarose
et visualisation des gels
· Préparation du gel d'agarose
Le gel d'agarose permet de suivre la
migration des fragments d'ADN en fonction de leur taille et en milieu non
dénaturant. Pour le préparer, 0,8g d'agarose est pesé et
mélangé à 100 ml de tampon TBE (Tris Borate EDTA) 0,5X.
L'ensemble est fondu au four micro-ondes et refroidi partiellement
jusqu'à environ 60°C. Le gel ainsi obtenu est coloré au BET
(Bromure d'Ethydium) et coulé dans un porte gel où des peignes
sont préalablement placés pour créer les puits de
migration. Après solidification complète du gel, les peignes sont
enlevés et ce dernier est ainsi prêt pour
l'électrophorèse.
· Dépôt des amplifiats
Le gel ainsi solidifié est
placé à l'intérieur d'une cuve
d'électrophorèse et recouvert de tampon TBE 0,5X. Dans chaque
puits, est déposé, cinq à huit microlitres (ul) d'ADN
(amplifiats) préalablement mélangés avec deux microlitres
de tampon de charge (bleu de bromophénol). Parallèlement, 3 ul
d'un marqueur de poids moléculaire 1Kb (100 pb à 12000 bp) est
déposé sur le gel, entre 1 et 2 puits, pour vérifier la
taille attendue de l'ADNr 16S amplifié. Enfin, l'allumage de l'appareil
d'électrophorèse de type Mupid-One permet de mettre en migration
pendant 30 minutes et à 100 V, les amplifiats déposés sur
le gel.
VII.2.3.2.5 - Migration de l'ADN et
visualisation
Après le temps de migration
fixé, le gel est retiré de la cuve
d'électrophorèse, puis déposé sur une plaque de
lumière ultraviolette pour la visualisation. Sur cette plaque, la
visualisation des amplifiats est rendue possible grâce au BET. En effet,
celui-ci, en tant que cation, s'intercale entre les brins de l'ADN et rend
ainsi le complexe ion éthidium-ADN fluoresçant sous
l'éclairage UV. La prise d'images est effectuée à l'aide
du transilluminateur à caméra relié à un
ordinateur.
Après cette visualisation et les prises d'images, les
souches dont le gène ADNr 16S a été correctement
amplifié sont sélectionnées en vue de leur
digestion.
VII.2.3.2.6 - Digestion des amplifiats
La digestion enzymatique du produit de la
PCR a été réalisée sur les souches bien
amplifiées. L'enzyme de restriction utilisée est la Tsp
509I (R0576S). Ses caractéristiques sont données dans le
tableau 6 ci après.
Tableau VI : Caractéristiques de
l'enzyme de restriction Tsp 509I
|
Enzyme de restriction
|
Sites à 4 paires de bases
|
|
Tsp 509I (R0576S)
|
5'...A A T T...3'
3'...T T A A...5'
|
Cette enzyme a été déjà
utilisée pour digérer des amplifiats du gène ADNr 16S dans
des études de diversité.
Le milieu réactionnel de la digestion se
présente comme suit (Tableau 7) :
Tableau VII : Composition du milieu
réactionnel unitaire commun de la digestion
|
Concentration de travail
|
Volume utilisé pour un échantillon
|
|
Tampon X 10
|
1 X
|
1,5 ul
|
|
Enzyme de digestion Tsp 509I
|
-
|
(2/10) ul
|
|
Eau PCR
|
-
|
3,5 ul
|
|
Volume réactionnel partiel
|
5 ul
|
Pour réaliser la digestion, chaque tube a
comporté en plus de ce milieu réactionnel commun, 10 à 15
ul d'amplifiat d'une et unique souche bactérienne. Ainsi, le volume
réactionnel total par souche varie entre 15 et 20 ul.
La digestion a eu lieu pendant 3 à 4 heures dans un
bain marie chauffé à 65°C. Un témoin négatif
constitué uniquement de 15 ul d'un bon amplifiat a accompagné
chaque digestion.
À la fin de la digestion, la migration et la
visualisation des fragments de restriction obtenus ont été
effectuées dans les mêmes conditions que celles des produits PCR.
Cependant, la petite taille des fragments de restriction
générés a nécessité la préparation
d'un gel d'agarose plus résolutif (agarose à 3%) et un temps de
migration de 50 min. Aussi, note-on qu'ici, tout le produit de digestion
de chaque souche (10 à 15 ul) a été entièrement mis
en migration contrairement aux amplifiats.
CHAPITRE VIII : RESULTATS ET DISCUSSION
VIII.1- RESULTATS
Ces résultats concerneront d'une part le comportement
des bactéries observé sur le milieu de culture TY pendant leur
purification et d'autre part le polymorphisme des fragments de restriction de
l'ADNr 16S amplifié de ces isolats.
VIII.1.1- Croissance des bactéries sur milieu TY
solide
La culture des bactéries isolées des nodules du
pois cajan sur milieu TY solide en vue de leur purification a permis de les
subdiviser en deux groupes selon le temps d'apparition des colonies. Le premier
groupe, noté bactéries à croissance rapide ou XR, (65, 1 %
des isolats) développe des colonies denses et bien identifiables en 24
heures d'incubation tandis qu'il faut un temps de 48 heures au deuxième
groupe (bactéries à croissance lente ou XL) pour
développer des colonies ayant les mêmes aspects (figure
15). Ce résultat a été observé pendant
quatre cycles de purification des isolats sur le milieu TY.
 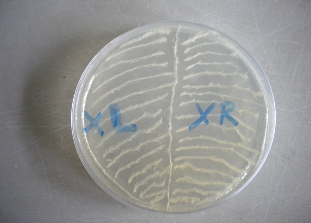
A : Niveau de croissance des deux groupes
de B : Niveau de croissance des deux groupes
de
bactéries 24 h après ensemencement
bactéries 48 h après ensemencement
XR
XR
XL
XL
Figure 15 : Mode de croissance sur milieu
TY en 24 heures et 48 heures des groupes de rhizobia isolés du pois
cajan
VIII.1.2 Amplification par PCR de l'ADNr 16S
La PCR réalisée sur l'ADNr 16S de toutes les
bactéries isolées pour l'étude (169) a permis d'en
amplifier 152, soit un taux d'amplification de 88,4 %. Cette amplification a
généré une bande unique révélée par
électrophorèse chez l'ensemble de ces 152 souches. Evaluée
visuellement par comparaison au marqueur utilisé, la taille de la bande
correspond au poids moléculaire de 1500 pb (figure 16).
M B27.1
B40.3 B35.1 B20.3
B14.4 B39.2 B14.2
B28.2 B23.1 T-
1500 pb
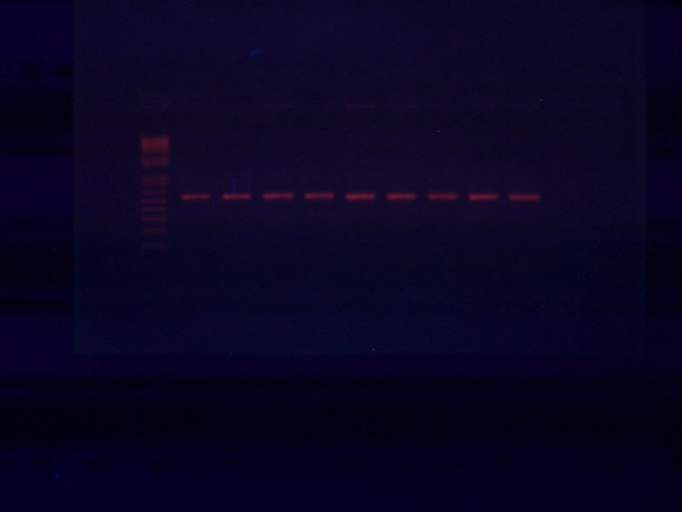
Figure 16 : Résultats d`amplification de
l'ADNr 16S des rhizobia isolés de Cajanus cajan
M : Marqueur de poids moléculaire
1kb ; T- : Témoin négatif (Milieu PCR
sans ADN)
B27.1: Code d'identification (ici,
souche n°1 des quatre cultivées sur la boîte de pétri
n° 27).
VIII.1.3 Produits de digestion
La digestion effectuée sur toutes les souches
amplifiées avec l'enzyme de restriction Tsp 509I a
généré trois profils différents en se basant sur le
nombre de bandes de digestion généré par souche. Il s'agit
d'un profil à deux bandes (A), d'un profil à trois bandes (B) et
d'un dernier profil à quatre bandes (C) (Figure 17).
M B
A A B B B
B B B B B
T-
M C
C C
C C C C
T-
Gel N°2
Gel N°1
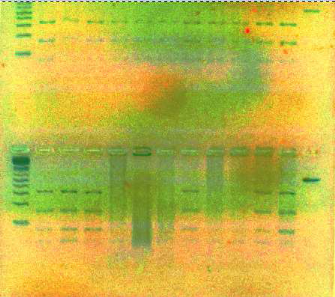
Figure 17 : Différents profils de
digestion obtenus avec l'enzyme de restriction Tsp 509I.
M : Marqueur de poids moléculaire
1kb ; T- : Témoin
négatif (Amplifiat sans Tsp
509I) ;
A : Profil à deux (2)
bandes ; B : profil à trois (3)
bandes ; C : Profil à quatre (4) bandes.
La proportion relative de chaque profil est donnée par
la figure 18:
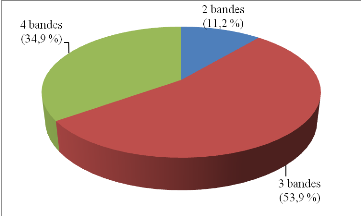
Figure 18 :
Proportions relatives des trois profils obtenus par
PCR-RFLP
La figure 18 montre une abondance relative du profil de
digestion à 3 bandes (53,9 %). Le profil à 2 bandes est le moins
important et représente 11,2 % de l'ensemble des souches
digérées.
Une analyse des profils de digestion en rapport avec la
vitesse de croissance des souches a donné les résultats
présentés par la figure 19:
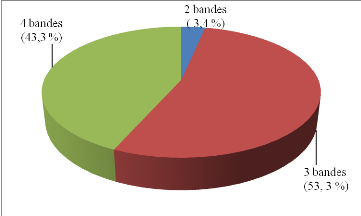
Figure 19 : Proportions des profils de
digestion des souches supposées à croissance lente
Cette figure fait ressortir la présence des trois
types de profils observés chez les
bactéries supposées à croissance lente.
Toutefois, une proportion plus faible (3,4 %) pour le
profil à deux bandes est à souligner.
Ces observations sont complémentaires à celles faites avec
les souches supposées à croissance rapide. Ainsi, l'analyse des
profils de digestion en rapport avec la vitesse de croissance des souches
n'a établi aucun lien particulier entre l'un ou l'autre des groupes
de croissance et les types de profils.
Tous les résultats ci-dessus obtenus feront l'objet d'une
discussion dans le paragraphe suivant.
VIII.2- DISCUSSION DES RESULTATS
La différence de temps de croissance
observée entre les isolats symbiotiques du pois d'Angole, pendant les
cycles de culture et de purification sur milieu TY solide, pourrait être
rapprochée des résultats obtenus en cultivant les rhizobia sur
milieu YMA (Yeast Manitol Agar) (VINCENT, 1970). En
effet, les rhizobia se subdivisent en deux groupes lorsqu'ils sont
cultivés sur le milieu de culture YMA. Ce milieu est celui
utilisé pour l'évaluation de la vitesse de croissance des
bactéries à travers la détermination de leur temps de
génération ainsi que le temps nécessaire pour le
développement de colonies complètes. En l'utilisant, JORDAN
(1982) a pu établir la première classification des
bactéries symbiotiques fixatrices d'azote en deux genres (genres
Rhizobium et Bradyrhizobium). Le premier genre
représente le groupe des bactéries possédant un temps
d'apparition de colonies complètes inférieur ou égal
à cinq jours d'incubation et dites bactéries ``à
croissance rapide''. Quant au second, il représente les bactéries
dites `` à croissance lente '', caractérisées
par le développement de colonies complètes entre cinq et sept
jours d'incubation.
Par ailleurs, en prenant pour critère leur vitesse de
croissance sur le milieu de culture YMA, SY et al. (2000) ont pu
répartir une collection de 126 souches de rhizobia isolées du
Crotalaria spp. en 2 groupes distincts. Un premier groupe de 81
souches à croissance rapide, dont les colonies apparaissent après
48 h de culture, et un second groupe de 45 souches à croissance lente
pour lesquelles les colonies n'apparaissent qu'au bout de 72 h de culture.
Nos résultats sont quasi-identiques à ce
résultat obtenu au Sénégal et révèlent de ce
fait, une diversité physiologique au sein des bactéries
étudiées. Le milieu TY pourrait donc être utilisé
pour évaluer la diversité physiologique des rhizobia en fonction
de leur vitesse de croissance.
Le taux d'amplification de 88,4 % de l'ADNr 16S des isolats
cultivés sur ce milieu est un résultat satisfaisant, tenant
compte du protocole utilisé pour obtenir les fragments d'ADN à
amplifier. En effet, si ce protocole (Préparation des bactéries
par effet thermique) a l'avantage d'être très rapide
comparativement au protocole d'extraction de l'ADN, il ne permet pas par contre
d'éliminer totalement certains inhibiteurs susceptibles d'influencer les
résultats de la PCR (protéines, etc.). Ainsi, un calibrage de
l'ADN dans le milieu réactionnel de la PCR, tenant compte de ces
inhibiteurs, pourrait améliorer le résultat. Par ailleurs, une
variation du volume d'ADN de 2,5 à 4 ul selon les souches a
été nécessaire pour avoir nos résultats.
La taille des amplifiats obtenus (1500 pb) est bien
celle attendue de 1'ADNr 16S parmi les bactéries (WEISBURG
et al., 1991). Ce résultat est aussi identique
à celui obtenu par DUBEY et al. (2010), en amplifiant
cette même région chez des rhizobia isolés également
du pois d'Angole en Inde. Le nombre de profils obtenus après digestion
des amplifiats (3) met en évidence une diversité
génétique au sein des bactéries symbiotes du pois cajan en
Côte d'Ivoire. Ce résultat confirme l'importance de l'ADNr 16S
dans l'évaluation de la diversité des bactéries en
général et des rhizobia en particulier. En effet, grâce
à sa structure très conservée mise en évidence par
WOESE (1987), l'étude du gène de l'ARNr 16S constitue une
excellente approche rapide pour évaluer la variabilité
génétique entre les souches de rhizobia (LAGUERRE et
al., 1994; NOUR et al., 1994b; SYLLA ,1998; NDOYE, 1999). Par
ailleurs, la diversité observée est aussi comparable à
celle obtenue en 2010 par DUBEY et ses collègues en Inde. En
effet, utilisant trois enzymes de restriction différentes (contre une
dans notre cas), leur étude a pu établir une diversité au
sein des symbiotes du pois cajan dans le District de Betul. Les profils
variaient d'un à quatre selon l'enzyme utilisée et quant au
nombre de bandes de digestion généré par profil, il s'est
situé entre deux et trois.
Enfin, l'inégalité constatée entre
les fréquences des trois groupes génétiques
constitués par l'étude peut s'expliquer d'une part par une
capacité compétitive non identique de ces groupes au cours du
processus de nodulation (ZEZE et al., 2001). En effet, les
différentes souches de rhizobia coexistant simultanément dans un
sol présentent des différences de capacité à
rivaliser pour la formation des nodules (POSTMA et al., 1989 ;
HEIJNEN et VAN VEEN, 1991). Ainsi, les deux groupes génétiques
les plus représentés au sein de la population rhizobienne
étudiée pourraient être ceux des souches symbiotiques les
plus performantes dans la nodulation du champ de pois d'Angole
étudié, notamment en termes d'infectivité. D'autre part,
ces résultats peuvent s'expliquer par une densité de population
saprophytique inégale entre les différentes souches rhizobiennes
présentes dans la rhizosphère du pois d'Angole. En effet,
certaines souches maintiennent dans le sol une faible densité de
population en absence de la légumineuse hôte (HIRSCH, 1996 ;
ZEZE et al., 2001), alors que le succès de celles-ci dans la
symbiose est aussi influencé par cette taille initiale (POSTMA et
al., 1989 ; HEIJNEN et VAN VEEN, 1991). Les souches ayant une plus
forte densité de population initiale ont naturellement plus de candidats
potentiels à la nodulation et leur abondance au sein de la population
symbiotique peut par conséquent être plus élevée.
CONCLUSION ET PERSPECTIVES
Notre étude qui s'inscrit dans le cadre de
l'amélioration de la productivité des légumineuses par
l'optimisation de la fixation azotée, a consisté à initier
l'évaluation de la diversité des rhizobia nodulant le pois cajan
en Côte d'Ivoire. L'objectif principal était de déterminer
le niveau de variabilité génétique entre des souches
rhizobiennes isolées d'un champ de ce pois cultivé à
Yamoussoukro. Cette diversité a été évaluée
par l'amplification de l'ADNr 16S des rhizobia suivie d'une digestion des
amplifiats par une enzyme de restriction.
Au terme de cette étude, on retiendra que :
- les souches isolées présentent une
diversité physiologique sur le milieu de culture TY. Deux groupes ont
été identifiés avec une domination des souches
développant des colonies denses et bien identifiables en 24 heures
d'incubation ;
- le nombre de profils (trois) obtenus après digestion
des souches amplifiées met en évidence une diversité
génétique au sein des bactéries symbiotes du pois
cajan ;
- une hétérogénéité dans
les fréquences relatives des trois types de profils a été
aussi observée.
Ces résultats nous donnent une grande satisfaction et
permettent d'affirmer que nos objectifs ont été atteints.
Toutefois, au delà de la variabilité génétique
révélée, la PCR-RFLP de l'ADNr 16S ne permet pas une
connaissance taxonomique de ces isolats symbiotiques. Ainsi, le recourt
à des techniques plus avancées comme le séquençage
de l'ADNr 16S des souches est nécessaire pour révéler
l'identité des groupes constitués par le polymorphisme de la
longueur des fragments de restriction. Par ailleurs, cette approche
moléculaire (séquençage) est souvent utilisée comme
une méthode complémentaire à la PCR-RFLP dans les
études de diversité des rhizobia. Le couplage des deux techniques
permet en effet d'établir des liens phylogénétiques entre
les isolats d'une part et d'autre part entre les isolats et des souches de
références déjà identifiées comme symbiotes
de la légumineuse étudiée (NDOYE, 1998 ;
SYLLA, 1998).
Le renforcement de notre collection initiale des souches
symbiotiques, à travers une étude multilocale, s'avère
également nécessaire pour avoir une vision globale de la
diversité des populations locales de rhizobia symbiotes du pois
d'Angole. Enfin, l'évaluation des caractéristiques symbiotiques
des isolats (infectivité, efficience, aptitude à la
compétition etc.) serait à considérer dans ces
études. Cette évaluation apparaît en effet très
déterminante dans tout processus d'exploitation de la fixation
biologique de l'azote à des fins pratiques.
REFERENCES BIBLIOGRAPHIQUES
ABAIDOO R.C., KEYSER H.H., SINGLETON P.W. &
BORTHAKUR D., 2000. Bradyrhizobium spp. (TGx) isolates
nodulating the new soybean cultivars in Africa are diverse and distinct from
bradyrhizobia that nodulate North American soybeans. International Journal
of Systematic and Evolutionary Microbiology, 50,
225-234.
AHMAD M.H., EAGLESHAM A.R.J. & HASSOUNA S.,
1981 b. Examining serological diversity of cowpea rhizobia by the
ELISA technique. Arch. Microbiol., 130, 281-287.
AHMAD M.H., EAGLESHAM A.R.J., HASSOUNA S., SEAMAN B.,
AYANABA A., MULONGOY K. & PULVER E.L., 1981 a. Examining the
potential for inoculant use of cowpeas in West Africans soils. Tropic.
Agron. (Trin), 58, 325-335.
ALLEN O.N. & ALLEN E.K., 1950.
Biochemical and Symbiotic Properties of the Rhizobia. Bacterial.
Rev., 14, 273-330.
ANONYME, 2002. Mémento de
l'Agronome [CD-ROM]. (2002). France : Ministère de
la coopération et du Développement Paris.
ANONYME., 2008. La plaquette de
présentation du CNRA, 16 p.
ANONYME, 2009. Ecole Supérieure
d'Agronomie, Départements et sections affiliés, 16 p.
ANONYME. Flavonoids
Induce Nodulation, 2010, (page consultée le 24 Avril 2010)
<
http://www.microbiologyprocedure.com/rhizobium-and-legume-root-nodulation/flavonoids-induce-nodualation.html
>.
AVO N.J.D., 2010. Contribution à
l'optimisation de la détection du virus du Swollen Shoot (CSSV)
dans les feuilles de cacaoyer en Côte d'Ivoire. Mémoire de fin
d'études pour l'obtention du diplôme d'Agronomie Approfondie.
Ecole Supérieure d'Agronomie, Yamoussoukro, 50 p.
BALDWIN I. L. & FRED E. B., 1929.
Nomenclature of the root nodule bacteria of the leguminosae. J.
Bacteriol., 17, 141-150.
BARRIOS E., KWESIGA F., BURESH R.J. & SPRENT J.I.,
1997. Light fraction soil organic matter and available nitrogen
following trees and maize. Soil Sci. Soc. Am. J., 61,
826-831.
BARTHOUX, Jean Claude. La famille des
Fabacées ou Légumineuses, 1975, (page consultée le 15
novembre 2009) <
http://www.sologne-nature.org/content/view/158/10/>.
BASHIR J., BURESH R.J. & PLACE F.M. 1998.
Sesbania tree fallows on phosphorus-deficient sites: Maize yield and financial
benefit. Agron. J., 90, 717-726.
BECK D.P., MATERON L.A., AFANDI F., 1993.
Pratical Rhizobium-Legume Technology Manual. ICARDA, Allepo, Syria,
170 p.
BENDER G.L., PLAZINSKI J. & ROLFE B.G.,
1986. A symbiotic acetylene reduction by a
fast-growing cowpea Rhizobium strain with nitrogen structural genes located on
a symbiotic plasmid. Environ. Microbiol., 51,
868-871.
BENTHAM G., 1865. Leguminosae. In Genera
Plantarum. Bentham G. and Hooker J. D. eds., Lovell, Reeve, London, pp.
434-600.
BERINGER J.E., 1974. R factor transfer in
Rhizobium leguminosarum. J. Genet. Microbiol.,
84, 188-198.
BHAGWAT A.A., GROSS K.C., TULLY R.E. & KEISTER D.,
1996. Glucan synthesis in Bradyrhizobium japonicum:
characterization of a new locus (ndvC) influencing â -(1 6)
linkages. J. Bacteriol., 178, 4635-4642.
BOEHRINGER A., & CADWEL R., 1989.
Cajanus cajan (L.) Millsp. as potentiel agroforestry
component in the Eastern Province of Zambia. Agroforestry systems,
9, 127-140.
BORGET M., 1989. Pois cajan (Cajanus
cajan (L.) Millsp). In Les légumineuses vivrières
tropicales. Maisonneuve et Larose, eds., A.C.C.T., Paris, P.
29.
BREEDVELD M. W. & MILLER K. J., 1998.
Cell-surface â-glucans. In The Rhizobiaceae. Spaink
H.P., Kondorosi A., Hooykaas J. J., eds., Kluwer Academic Publishers,
Dordrecht, The Netherlands, pp. 81-96.
BREWIN N.J., DOWNIE J.A. & YOUNG J.P.W.,
1992. Nodule formation legumes. Encyclopedia of
microbiology, M.R Josha Lederberg. Rockefeller University New York,
3, 239-248.
BROUGHTON W. J., JABBOURI S. & PERRET X.,
2000. Keys to symbiotic harmony. J.
Bacteriol., 182, 5641-5652.
CERVANTES E., SHARMA S.B., MAILLET F., VASSE J.,
TRUCHET G. & ROSENBERG C., 1989. The Rhizobium meliloti
host range nod O genes encodes a proteine which shares homology
with translation elongation and initiations factors. M. Microbiol.,
3, 745-755.
CHEN W. X., YAN G. H. & LI J. L., 1988.
Numerical taxonomic study of fast-growing soybean rhizobia and a proposal that
Rhizobium fredii be assigned by Sinorhizobium gen. nov.
Int. J. Syst. Evol. Microbiol., 38, 392-397.
CHEN W.M., JAMES E.K., PRESCOTT A.R., KIERANS M. &
SPRENT J.I., 2003. Nodulation of Mimosa spp. by the
beta-proteobacterium Ralstonia taiwanensis. Mol Plant Microbe
Interact, 16, 1051-1061.
CRONK Q., OJEDA I. & PENNINGTON R.T.,
2006. Legume comparative genomics: progress in phylogenetics and
phylogenomics. Curr Opin Plant Biol, 9, 99-103.
CRUTZEN P.J., MOSIER A.R., SMITH K.A. & WINIWARTER
W., 2007. N2O release from agro-biofuel
production negates global warming reduction by replacing fossil fuels.
Atmos. Chem. Phys. Discuss. 7, 11191-11205.
DAVET P., 1996. Vie microbienne du sol et
production végétale. Institut National de la Recherche
Agronomique. INRA, Paris, 384 p.
DAY D.A., POOLE P.S., TYERMAN S.D. & ROSENDAHL L.,
2001. Ammonia and amino acid transport across symbiotic membranes in
nitrogen-fixing legume nodules. Cell Mol Life Sci, 58,
61-71.
DEBELLE F. & SHARMA S.B., 1986.
Nucleotide sequence of Rhizobium meliloti RCR 2011 genes involved in
host specificity of nodulation. Nucl. Acids Res. H.,
14, 7453-7472.
DEBELLE F., MOULIN L., MANGIN B., DENARIE J. &
BOIVIN C., 2001. Nod Genes and Nod signals and the
evolution of the rhizobium legume symbiosis. Acta Biochimia Polonia
Minireview, 48 (2), 359-365.
DEBELLE F., ROSENBERG C., VASSE J., MAILLET F.,
MARTINEZ E., DENARIE J. & TRUCHET G., 1986. Assignment of
symbiotic developmental phenotypes to cornmon and specifie nodulation (nod)
genetic loci of Rhizobium meliloti. J. Bacteriol.,
l68, 1075- 1086.
DUBEY R.C., MAHESHWARI D.K., KUMAR H. & CHOURE K.,
2010. Assessment of diversity and plant growth
promoting attributes of rhizobia isolated from Cajanus cajan L.
African Journal of Biotechnology, 9(50),
8619-8629.
DUHOUX M. & NICOLE M., 2004. Biologie
Végétale. Associations et Interactions chez les plantes.
Dunod-IRD, Paris, 176 p.
DUKE J.A., 1981a. Manuel de
légumineuses du monde économique. Plenum Press, New York,
pp. 33-37.
EL HILALI I., 2006. La symbiose
rhizobium-lupin : biodiversité des microsymbiotes et mise en
évidence d'une multi-infection nodulaire chez Lupinus luteus.
Thèse de doctorat. Université Mohammed V-AGDAL, Rabat, 189
p.
FAOSTAT. (Page consultée en août
2007) <http://faostat.fao.org/faostat /collections?
Version=ext&hasbulk=0&subset=agriculture>.
FRED E. B., BALDWIN I. L. & MCCOY E.,
1932. Root nodule bacteria and leguminous plants. University of Wisconsin
Press, Madison, Wis. (in: Elkan, 1902).
FREIBERG C., FELLAY R., BAIROCH A., BROUGHTON W.J.,
ROSENTHAL A. & PERRET X., 1997. Molecular basis of symbiosis
between Rhizobium and legumes. Nature, 387,
394-401.
GAGE D.J., 2004. Infection
and invasion of roots by symbiotic, nitrogen-fixing rhizobia during nodulation
of temperate legumes. Microbiol Mol Biol.Rev., 68
(2), 280-300.
GEURTS R. & BISSELING T., 2002.
Rhizobium Nod factor perception and signaling. Plant
Cell, 14, 239-249.
GILLIS M., VANDAMME P., DE VOS P., SWINGS J.Y. &
KERSTERS K., 2001. Polyphasic taxonomy. Pp. 43-48. In Bergey's
Manual of Systematic Bacteriology, 2nd Edition, Volume I. Boone, D.R., R.
W. Castenholz, G. M. Garrity (eds.).
GONZALEZ J.E., REUHS B. L. & WALKER G.C.,
1996. Low molecular weight EPS II of Rhizobium meliloti
allows nodule invasion in Medicago sativa. Proc. Natl. Acad.
Sci. USA, 93, 8636-8641.
GRAHAM P.H., 1991. What are
Rhizobia?. The College of Agricultural Food and Environment Science.
GRAHAM P.H. & VANCE C.P., 2003. Legumes:
importance and constraints to greater use. Plant Physiol, 131,
872-877.
GRAHAM P.H., SADOWSKY M.J., KERSTERS H. H., BARNET
Y.M., BRADLEY R.S., COOPER J.E., DE LEY D.J., JARVIS B.D.W., ROSLYCKY E.B.,
STRIJDOM B.W. & YOUNG J.P.W., 1991. Proposed minimal standards for
the description of new genera and species of root-and
stem-nodulating bacteria. Int. J. Syst. Bacteriol.,
41, 582-587.
GRIMAUD P., 1988. Revue d'Elevage et de
Médecine Vétérinaire de Nouvelle-Calédonie (11),
pp29-36.
GRIMONT F. & GRIMONT P.A.D., 1986.
Ribosomal ribonucleic acid gene restriction patterns as potential taxonomic
tools. An. Inst. Pasteur Microbiol., 137B,
165-175.
GÜRTLER V. & STANISICH V. A., 1996.
New approaches to typing and identification of bacteria using the 16S-23S rDNA
spacer region. Microbiology, 142, 3-16.
HAUKKA K., LINDSTROM K. & YOUNG P.W.,
1998. Three Phylogenetic Groups of nodA and
nifH Genes in Sinorhizobium and Mesorhizobium
Isolates from Leguminous Trees Growing in Africa and Latin America.
Appl Environ Microbiol.Rev., 64 (2), 419-426.
HEIJNEN C. E., VAN VEEN J.A., 1991: A
determination of protective microhabitats for bacteria introduced into soil.
FEMS Microbiol. Ecol., 85, 73-80.
HILLIS D.M. & DIXON M.T., 1991. Ribosomal
DNA: molecular evolution and phylogenetic inference. Q. Rev. Biol.
66, 411-453.
HIRSCH P.R., 1996.
Population dynamics of indigenous and genetically modified rhizobia in
the field. New phyt. 133, 159-171.
HOPKINS W.G., 2003.
Physiologie végétale. Université des Sciences et
Technologie de Lille. Edition de boeck, 514 p.
HORVATH B., KONDORASI E., JOHNSON M., SCHMIDT J.,
TOROK L., GYORGYPAL Z., BARABAS L., WIENEKE V., SCHELL J. & KONDOROSI A.,
1986. Organization structure and syrnbiotic function of Rhizobium
meliloti nodulation genes determining host specilicity for alfalfa.
Cell, 46, 335-343.
HULUGALLE R. N. & LAL R., 1986. Root
growth of maize in compacted gravely tropical alfisoil as affected by rotation
with a woody perennial. Field Crop Research, 13,
33-44.
JAFTHA J.B., STRIJDOM B.W. & STEYN P.L.,
2002. Characterization of pigmented methylotrophic bacteria which
nodulate Lotononis bainesii. Syst Appl Microbiol, 25,
440-449.
JENSEN M. A. & STRAUS N., 1993. Effect of
PCR conditions on the formation of heteroduplex and single-stranded DNA
products in the amplification of bacterial ribosomal DNA spacer regions.
PCR Methods Appl., 3, 186-194.
JORDAN D.C., 1982. Transfer of rhizobium
japonicum Buchanan 1980 to Bradyrhizobium gen. nov., a genus of
slow growing root nodule bacteria from leguminous plants, Int. J.
Syst. Bacteriol., 32, 136-139.
JORDAN D.C., 1984.
Rhizobiaceae. In Bergey's Manual of Systematic Bacteriology.
Krieg N. R. et Holt J. G. eds., Baltimore, pp. 234-245.
KANNAIYAN J., NENE Y.L., REDDY M.V., RYAN J.G., &
RAJU T.N., 1984. Prevalence of pigeonpea diseases and associated crop
losses in Asia, Africa, and America. Trop. Pest Manag.,
30, 62-71.
KNEIP C., LOCKHART P., VOSS C. & MAIER U.G.,
2007. Nitrogen fixation in eukaryotes -new models for symbiosis.
BMC Evol Biol, 7, 55.
KUMAR RAO I.V.D.K., THOMPSON J.A., SASTRY P.U.S.S.,
GILLER K.E. & DAY J.M., 1986. Measurement of N2
fixation in field grown pigeonpea using 15N-labelled fertilizer. Plant
Soil, 101, 102-113.
LAFAY B., BULLIER E. & BURDON J.J., 2006.
Bradyrhizobia isolated from root nodules of Parasponia (Ulmaceae) do not
constitute a separate coherent lineage. Int J Syst Evol Microbiol,
56, 1013-1018.
LAGARES A., CAETANO-ANOLLES G., NIEHAUSS K., LORENZEN
J., LJUNGGREN H.D. & FAVELUKES G., 1992. A Rhizobium meliloti
lipopolysaccharide mutant altered in competitiveness for
nodulation of alfalfa. J. Bacteriol., 174,
5941-5952.
LAGUERRE G., ALLARD M.R., REVOY F. & AMARGER
N., 1994. Rapid identification of rhizobia by restriction fragment
length polymorphism analysis of PCR- amplified 16S rRNA genes. Appl.
Environ. Microbiol., 60, 56-63.
LANCELLE S.A. & TORREY J.G., 1985. Early
development of Rhizobium-induced root nodules of Parasponia rigida. II
Nodule morphogenesis and symbiotic development. Can J Bot,
63, 25-35.
LEPAPE M.C., 1980. Aperçu sur les
fourrages ligneux des îles du Cap-Vert. In `` Les fourrages ligneux
en Afrique. Etat actuel des connaissances''. (Le Houerou, ed.)., CIPEA,
Addis Abeba, pp. 123-125.
LOHNIS F. & HANSEN R., 1921. Nodule
bacteria of leguminous plants. J. Agr. Res., 20,
543-546.
LUDWIG W. & SCHLEIFER K. H., 1999.
Phylogeny of bacteria beyond the 16S RNA Standard. ASM News.,
65, 752-757.
MACCOLL D., 1989. Studies on maize (Zea
mays L) at Bunda, Malawi. II. Yield in short rotation with emphasis on
phosphorus. Soil Sci., 152, 315-320.
MERCADO-BLANCO J. & TORO N., 1996.
Plasmids in rhizobia: The role of nonsymbiotic plasmids. MPMI.,
9, 535-545.
MINJA E.M., 2001. Yield losses due to field
pests and integrated pest management strategies for pigeonpea a synthesis: In,
Silim SN, Mergeai G, Kimani PM (ed). Status and potential of pigeonpea in
East and southern Africa : Proceedings of a regional workshop,
12-15 Sep, 2000, Nairobi, Kenya. B-5030 Gembloux, Belgium: Gembloux
Agricultural University, and Pantecheru, Andhra Pradesh, India: International
Crops Research Institute for the semi-Arid Tropics.
MOULIN L., MUNIVE A., DREYFUS B. & BOIVIN-MASSON
C., 2001. Nodulation of legumes by members of the ß-subclass of
Proteobacteria. Nature, 411, 948-950.
MULLIS K.B. & FALOONA F.A., 1987.
Specific synthesis of DNA in vitro via a polymerase catalysed chain
reaction. Methods Enzymol., 155, 335-350.
NADIMPALLI B.G., JARRET R.L., PATHAK S.C. &
KOCHERT G., 1992. Phylogenetic relationships of pigeonpea (Cajanus
cajan ) based on nuclear restriction fragment length polymorphisms.
Genome, 36, 216-223.
NDABALISHE I., 1995. Agriculture
vivrière ouest africaine à travers le cas de la Côte
d'Ivoire. Monographie. IDESSA, 383 p.
NDOYE I., 1998. Etude taxonomique et
caractérisation des bactéries fixatrices nodulant les
Caesalpinioideae de la sous-Famille des Cassieae (Cassia,
Chamaecrista et Senna). Rapport de stage
séjour scientifique haut niveau. Laboratoire des Symbioses Tropicales et
Méditerranéennes (LSTM), Montpellier, 32 p.
NDOYE I., 1999. Caractérisation
taxonomique des bactéries fixatrices d'azote nodulant Acacia
nilotica var. andansonii et var. tomentosa
(Mimosoideae, sous famille des Acacieae). Rapport de
stage séjour scientifique haut niveau. Laboratoire des Symbioses
Tropicales et Méditerranéennes (LSTM), Montpellier, 35 p.
NENE Y.L. & SHEILA V.K., 1990. Pigeonpea:
geography and importance. In The Pigeonpea. Nene Y.L., Hall S.D. and
Sheila V.K. eds., CAB International, UK, pp. 1-14.
NIYONKURU D. N., 2002. La culture du pois
cajan. SAILD, Cameroun, 23 p.
NOEL K.D., 2009. Bacteria
Rhizobia. Encyclopedia of microbiology, SCHAECHTER M. San Diego. Marquette
University, Milwaukee, WI, USA, 3, 877-893.
NORMAND P., COURNOYER B., NAZARET S. & SIMONET P.,
1992. Analysis of a ribosomal RNA operon in the actinomycete
Frankia. Gene , 3, 119-124.
NORMAND P., PONSOET C., NESME X., NEYRA M. &
SIMONET P., 1996. ITS analysis of prokaryotes. Mol. Microbial.
Ecol. Manual., 3, 1-12.
NOUR S.M., CLEYET-MAREL J. C., BECK D., EFFOSE A.
& FERNANDEZ M. P., 1994b. Genotypic and phenotypic diversity of
Rhizobium isolated from chickpea (Cicer arietinum L.). Can J.
Microbiol., 44, 345-354.
NUTMAN P.S., 1981.
Hereditary host factors affecting nodulation and nitrogen fixation. In
Current Perspectives in Nitrogen Fixation Gibson. A.H. and Newton W.E.
(eds). Elsevier, North Holland, pp. 194- 204.
OLSEN G., WOESE J. C. & OVERBEEK R.,
1994. The winds of (evolutionary) change: breathing new life into microbiology.
J. Bacteriol., 176, 1-6.
OTT T., VAN DONGEN J.T., GUNTHER C., KRUSELL L.,
DESBROSSES G., VIGEOLAS H., BOCK V., CZECHOWSKI T., GEIGENBERGER P. &
UDVARDI M.K., 2005. Symbiotic leghemoglobins are crucial for nitrogen
fixation in legume root nodules but not for general plant
growth and development. Curr Biol, 15, 531-535.
PELMONT J., 1995.
Bactérie et environnement : adaptation physiologique. Vol
2. Office des Publications Universitaires, 897 p.
PEOPLES M.B., HERRIDGE D.F., LADHA J.K.,
1995. Biological nitrogen fixation : An efficient source of Nitrogen
for sustainable agricultural production? Plant and Soil,
174, 3-28.
PERRET X., STAEHELIN C. & BROUGHTON W.J.,
2000. Molecular basis of symbiotic promiscuity. Microbiol Mol
Biol, 64, 180-201.
PETERS N.K. & VERMA D.P.S., 1990.
Phenolic compounds as regulators of gene expression in plant-microbe
interactions. Mol. Plant Microbe Interac., 3,
4-8.
PFEFFER P.E., BECARD G., ROLIN D.B., UKNALIS J., COOK
P. & TU S., 1994. In vivo nuclear magnetic resonance study of the
osmoregulation of phosphocholine-substituted â-1,3;1,6 cyclic glucan and
its associated carbon metabolism in Bradyrhizobium japonicum USDA 110.
Appl. Environ. Microbiol., 60, 2137-2146.
POSTMA J., WALTER S., VAN VEEN J.A., 1989:
Influence of different initial moisture contents on the distribution and
population dynamics of introduced Rhizobium leguminosarum biovar
trifolii. Soil Biol. Biochem., 21,437- 442.
PRIN Y., GALIANA A., DUCOUSSO M., DUPUY N., DE
LAJUDIE P. & NEYRA M., 1993. Les Rhizobiums d'Acacia :
Biodiversité et taxonomie. Bois et Forêts des Tropiques,
238, 5-20.
PUEPPKE S.G. & BROUGHTON W. J., 1999.
Rhizobium sp. NGR234 and R. fredii USDA257 share
exceptionally broad, nested host-ranges. Mol. Plant-Microbe Interact.,
12, 293-318.
RAMSUBHAG A., UMAHARAN P. & DONAWA A.,
2002. Partial 16S rRNA gene sequence diversity and numerical taxonomy of slow
growing pigeonpea (Cajanus cajan L. Millsp) nodulating rhizobia.
FEMS Microbiology Letters, 216, 139-144.
RIVAS R., VELÁZQUEZ E., WILLEMS A.,
VIZCAÍNO N., SUBBA-RAO N. S., MATEOS P. F., GILLIS M., DAZZO F. B. &
MARTÍNEZ-MOLINA E., 2002. A New species of Devosia
that forms a unique Nitrogen-fixing root-nodule symbiosis
with the aquatic legume Neptunia natans (L.f.) Druce.
Appl. Environ. Microbiol., 68, 5217-5222.
ROGER P., DOMMERGUES Y., BALANDREAU J., DREYFUS B.
& SOUGOUFARA B., 1996. Résumé et introduction.
In : ROGER P., DOMMERGUES Y., BALANDREAU J., DREYFUS B. &
SOUGOUFARA B., eds. La fixation biologique de l'azote: quelles
potentialités pour le développement ? Compte rendu de
conférence-débat de l'ORSTOM, 30 Mai, 1996, Paris. 2-7.
SAIKI R.K., SCHARF S., FALOONA F., MULLIS K.B., HOM
G.T., EDITH H.A. & ARUHEIN N., 1985. Enzymatic amplification of
betaglobin genomic sequences and restriction site analysis for diagnostic of
sickle cell anemia. Science, 230, 1350-1354.
SAWADA H., KUYKENDALL L.D. & YOUNG J.M.,
2003. Changing concepts in the systematics of
bacterial nitrogen-fixing legume symbionts. J. Gen. Appl. Microbiol.,
49, 155-179.
SEDGA Z., 1997. Gestion
améliorée de la jachère par l'utilisation des
légumineuses de couverture. In La jachère, lieu de
production. Floret C. eds., CORAF/IRD ex ORSTOM/UE, Dakar, pp.133-139.
SHARMA P.K., KUNDU B.S. & DOGRA R.C.,
1993. Molecular mechanism of host specificity in
legume-rhizobium symbiosis. Biotech. Adv., 11,
741-779.
SIAMBI M., OMANGA P.G.A., SONGA W., 1992.
Status and needs of the national pigeonpea research program in Kenya. Int.
Pigeonpea Newslett., 16, 31-36.
SINGH L., 1991. Overview of
pigeonpea improvement research: objectives, achievements and looking ahead in
the African context. In: Laxman S, Silim SN, Ariyanayagam RP, Reddy MV (eds).
Proceedings of the first East and southern Africa regional legumes
(Pigeonpea) workshop. 25-27 June 1990, Nairobi, Kenya. pp. 5-6. East
Africa Regional Cereals and Legumes (EARCAL) Program, International Crops
Research for the Semi-Arid Tropics.
SKERMAN J.P., 1982. Les
légumineuses fouragères tropicales, FAO, Rome (Italie), 666
p.
SMARTT J., 1990. Grain legumes: evolution
and genetic resources. Cambridge University Press, Cambridge, pp. 278-293.
SOLTIS D.E., SOLTIS P.S., MORGAN D.R., SWENSEN S.M.,
MULLIN B.C., DOWD J.M. & MARTIN P.G., 1995. Chloroplast gene
sequence data suggest a single origin of the predisposition for symbiotic
nitrogen fixation in angiosperms. Proc Natl Acad Sci USA, 92,
2647-2651.
SOMASEGARAN P. & HOBEN H.J.,
1994. Handbook for Rhizobia. Springer
verlag, New York, Inc., p. 450.
SONGOK S., MORAG F., MUIGAI
A.W. & SILIM S., 2010. Genetic diversity in pigeonpea
[Cajanus cajan (L.) Millsp.] Landraces as revealed by simple sequence
repeat markers. African Journal of Biotechnology,
9(22), 3231-3241.
SPAINK H.P., 2000. Root nodulation and
infection factors produced by rhizobial bacteria. Annu. Rev. Microbiol.,
54, 257-288.
SPORE., 2010. Aide à
l'agriculture «des bailleurs en ordre dispersé» N°145,
pp5.
STAHL D.A. & AMMAN R., 1991. Development
and application of nucleic acid probes. In nucleic acid techniques. In
Bacterial systematics. Stackebrandt E. and Goodfellow M. eds., Wiley,
London.
SY A., GIRAUD E., SAMBA R., DE LAJUDIE P., GILLIS M.
& DREYFUS B., 2001. Certaines légumineuses du genre
Crotalaria sont spécifiquement nodulées par une nouvelle
espèce de Methylobacterium. Can. J. Microbiol.,
47, 503-508.
SY A., GIRAUD E., JOURAND P., GARCIA N., WILLEMS A.,
DE LAJUDIE P., PRIN Y., NEYRA M., GILLIS M., BOIVIN-MASSON C. & DREYFUS B.,
2001. Methylotrophic Methylobacterium bacteria nodulate and
fix nitrogen in symbiosis with legumes. J Bacteriol.,
183, 214-220.
SYLLA E.S.N., 1998. Caractérisation
des rhizobiums et l'ontogenèse des nodules chez Pterocarpus
erinaceus et P. lucens. Rapport de stage
séjour scientifique haut niveau. LSTM, CIRAD - IRD,
Montpellier, 16 p.
TA BI D.B., 2010. Influence de la
fertilisation phospho -potassique sur le rendement grainier de Cajanus
cajan L. Mémoire de fin d'études pour l'obtention du
diplôme d'Ingénieur des Techniques Agricoles. Ecole
Supérieure d'Agronomie,Yamoussoukro, 37 p.
TEREFEWORK Z., 2002. Diversity and phylogeny
of Rhizobium galegae, and reflections on molecular evolution of
rhizobium-legume symbiosis. In microbiology. Academic dissertation
eds., University of Helsinki, pp.1239-9469.
USDA. USDA national nutrient database for
standard reference, release 17, 2004, <
http://www.nal.usda.gov/fnic/foodcomp>.
VANCE C.P., GRAHAM P.H. & ALLAN D.L.,
2000. Biological nitrogen fixation. Phosphorus: a critical future
need. In Nitrogen Fixation : From Molecules to Crop Productivity.
Pedrosa F.O., Hungria M. & Yates M.G. eds., Newton WE (Kluwer Academic
Publishers), Dordrecht (The Netherlands), pp. 506-514.
VAN DER MAESEN L.J.G., 1986. Cajanus
DC and Atylosia W&A (Leguminosae). In Agricultural University
Papers 854. Agricole University eds., Wageningen, p. 225.
VAN DER MAESEN L.J.G., 1990.
Pigeonpea: origin, history, evolution, and taxonomy. In The Pigeonpea.
Nene Y.H., Hall S.D., Sheila V.K. eds., CAB International UK, pp15-46.
VAN DER MAESEN, L.J.G. Cajanus cajan
(L.) Millsp., 2006, (page consultée le 16 octobre 2010) <
http://database.prota.org/recherche.htm>.
VANDAMME P., POT B., GILLIS M., DE VOS P., KERSTERS K.
& SWINGS J., 1996. Polyphasic taxonomy, a consensus approach to
bacterial systematics. Microbiol. Rev., 60, 407-
438.
VERINE T., 2008. Analyse fonctionnelle d'EFD,
Un régulateur transcriptionnel de la nodulation au cours de
l'interaction symbiotique entre Medicago truncatula et
Sinorhizobium meliloti. Thèse de doctorat.
Université Toulouse III-Paul Sabatier, Toulouse, 263 p.
VINCENT J.M., 1970. A Manual for the
Practical Study of Root Nodule Bacteria. IBP handbook, no. 15. Blackwell
Scientific Publications, Ltd., Oxford, England.
VISSOH P.V., GBEHOUNOU G., AHANCHEDE A., KUYPER T.W.
& ROLING N.G., 2004. Weeds as agricultural
constraint to farmers in Benin: results of a diagnostic study. NJAS -
Wageningen Journal of Life Sciences, 52, 305-329.
WEISBURG W.G., BARNS S.M., PELLETIER D.A. & LAE D.
J., 1991. 16S ribosomal DNA amplification for phylogenetic study.
J. Bacteriol., 173, 697-703.
WERNER D., 1992. Symbiosis
of plants and microbes. Philipps-University Marburg Germany. Edition Chapman
& Hall.
WILKSON J.K., 1944. Other five hundred
reasons for abandoning the cross inoculation group of legumes. Soil
Sci., 58, 61-69.
WOESE C. R., 1987. Bacterial evolution.
Microbiol Rev., 512, 221-71.
WOLDE-MESKEL E., BERG T., PETERS N.K. & FROSTEGARD
A., 2004a. Nodulation status of native woody legumes, and phenotypic
characteristics of associated rhizobia in soils of southern Ethiopia. Biol
Fertil Soils, 40, 55-66.
YANN P., 2006. Apport de la
génomique dans la compréhension de la symbiose Rhizobium
légumineuses. Master Recherche. Microorganisme du génome aux
interactions avec l'hôte. Université de Paul Sabatier. Toulouse
III.
YOUNG J.P.W. & HAUKKA K. E., 1996.
Diversity and phylogeny of rhizobia. New Phytol., 133,
87-94.
YOUNG N.D., MUDGE J. & ELLIS T.H., 2003.
Legume genomes: more than peas in a pod. Curr Opin Plant Biol,
6, 199-204.
ZAKHIA F. & DE LAJUDIE P., 2001.
Taxonomy of rhizobia. Agronomie, 21,
569-576.
ZEZE A., MUTCH L.A. & YOUNG J.P.W., 2001.
Direct amplification of nodD from community DNA reveals the genetic diversity
of rhizobium leguminosarum in soil. Environ. Microbial.,
3(6), 363-370.
ANNEXES
ANNEXE I : Classification des rhizobia selon les
séquences du gène de l'ARN 16S
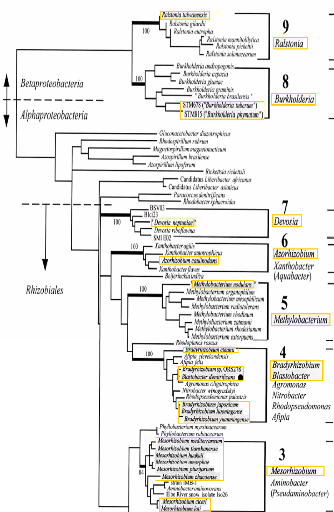
ANNEXE I : (suite)
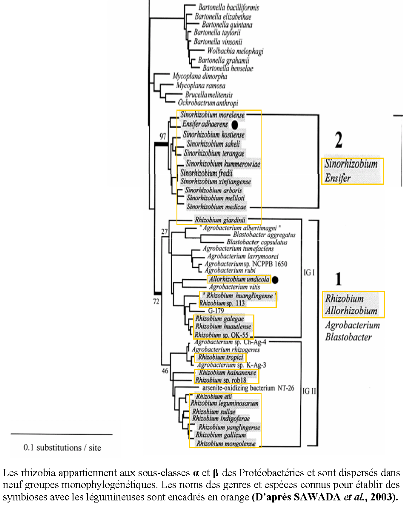
Les rhizobia appartiennent aux sous-classes
á et â des
Protéobactéries et sont dispersés dans neuf groupes
monophylogénétiques. Les noms des genres et espèces connus
pour établir des symbioses avec les légumineuses sont
encadrés en orange (D'après SAWADA et al.,
2003).
ANNEXE II : Présentation des structures et
des laboratoires d'accueil de l'étude
Le principal laboratoire d'accueil de l'étude est
celui d'Agronomie et Productions Végétales de l'Institut National
Polytechnique Félix Houphouët Boigny (INP-HB) de Yamoussoukro.
1- Présentation de l'INP-HB et du Laboratoire
d'Agronomie et Productions Végétales
1.1- Présentation de l'INP-HB
Créé le 4 septembre 1996 par le décret
n° 96-678, l'Institut National Polytechnique Félix Houphouët
Boigny (INP-HB) de Yamoussoukro est un établissement public
d'enseignement supérieur et de recherche. Il est né de la
restructuration et de la fusion de quatre grandes écoles, à la
faveur de la dernière reforme de l'Enseignement Supérieur et de
la Recherche engagée par l'Etat de Côte d'Ivoire. Il reprend,
ainsi, les missions des 4 anciennes grandes écoles de Yamoussoukro que
furent l'Ecole Nationale Supérieure Agronomique (ENSA), l'Ecole
Nationale Supérieure des Travaux Publics (ENSTP), l'Institut Agricole de
Bouaké (IAB) et l'Institut National Supérieur de l'Enseignement
Technique (INSET). L'INP-HB comprend aujourd'hui six grandes écoles
que sont l'Ecole Supérieure d'Agronomie (ESA), l'Ecole de Formation
Continue et de Perfectionnement des Cadres (EFCPC), l'Ecole Supérieure
d'Industrie (ESI), l'Ecole Supérieure des Travaux Publics (ESTP),
l'Ecole des Mines et Géologie (ESMG), l'Ecole Supérieure de
Commerce et d'Administration des Entreprises (ESCAE).
L'Institut compte également des Départements de
Formation et de Recherche (DFR) qui ont pour mission d'effectuer des apports
pédagogiques auprès des 6 écoles, de faire de la
recherche, de la production et de l'expertise. Ils sont au nombre de quatorze
(14) parmi lesquels le DFR Agriculture et Ressources Animales (DFR-ARA) dont
dépend le laboratoire d'Agronomie et Productions
végétales.
1.2- Présentation du Laboratoire
d'Agronomie et Productions végétales de
l'INP-HB
Le Laboratoire d'Agronomie et Productions
végétales est l'un des quatre laboratoires de recherche du
département ARA. Il donne un appui à la formation, par des
travaux pratiques et travaux de fin d'études d'étudiants. Il est
aussi le cadre d'activités des enseignants-chercheurs qui abordent de
nombreux thèmes dont les principaux sont ci-dessous
indiqués :
- Amélioration génétique du riz ;
- Instabilité de la variété de riz NERICA
2 ;
- Rendement du riz et ses composantes en fonction de la
fertilisation azotée ;
- Productivité des principales cultures
maraîchères en zone urbaine et périurbaine ;
- Caractérisation des systèmes
agro-pastoraux : cas de la région des montagnes ;
- Approche régionale des systèmes de culture, de
production et agraires ;
- Efficacité des légumineuses selon
différentes souches de Rhizobium (ANONYME, 2009).
Le thème d'étude s'inscrit dans le champ du
dernier thème de recherche.
La conduite de cette étude a nécessité
une collaboration technique avec le Laboratoire Central de Biotechnologie du
Centre National de Recherche Agronomique (CNRA). Il est important de souligner
que cette collaboration a été possible grâce au partenariat
existant entre l'INP-HB et cette structure.
2- Présentation du CNRA et du Laboratoire
Central de Biotechnologie (LCB)
2.1- Présentation du Centre National de
Recherche Agronomique (CNRA)
Le Centre National de Recherche Agronomique (CNRA) est une
société anonyme à participation financière publique
minoritaire. Le capital social, de 500 millions, est détenu pour 40% par
l'Etat de Côte d'Ivoire et pour 60% par les opérateurs agricoles
et agro-industriels opérant dans le pays.
Créé en 1998, il remplace les trois anciens
instituts de recherche agronomique ivoiriens suivants :
- l'Institut Des Savanes (IDESSA) ;
- l'Institut Des Forêts (IDEFOR) ;
- le Centre Ivoirien de Recherche Technologique (CIRT).
L'objet du Centre National de Recherche Agronomique se
décline en trois points essentiels dont celui de l'accroissement de
façon durable de la production et de la productivité dans les
domaines agricole et agro-industriel. Il s'agit à travers ce point de
faire d'une part, des recherches sur les productions végétales,
animales et forestières, sur les méthodes de conservation et de
transformation etc. D'autre part, il s'agit de transférer les acquis
scientifiques et techniques et de valoriser l'expertise des ressources humaines
auprès des opérateurs publics et privés, locaux et
extérieurs (ANONYME, 2008).
Le CNRA dispose de cinq directions régionales
(Abidjan, Gagnoa, Bouaké, Korhogo et Man) et vingt stations de recherche
correspondant aux différents programmes de recherche du centre. Le
laboratoire central de biotechnologie qui a servi de cadre pour nos travaux
conduit le programme de biotechnologie.
3.2.2- Présentation du Laboratoire Central de
Biotechnologie (LCB) du CNRA
Le LCB est rattaché à la direction
régionale d'Abidjan du CNRA. Il est situé à
Adiopodoumé et composé de six unités
opérationnelles spécialisées qui sont :
- l'unité de génétique
moléculaire ;
- l'unité de génomique fonctionnelle ;
- l'unité de virologie et biologie
moléculaire ;
- l'unité de ressource
génétique ;
- l'unité de microbiologie ;
- l'unité de phytopathologie.
Sa mission est de faire la recherche en exploitant,
transférant et vulgarisant les acquis de la biotechnologie au profit de
la production agricole et de la gestion rationnelle des ressources
génétiques.
L'unité de virologie et de biologie moléculaire
du laboratoire a été particulièrement
sollicitée pour notre étude.
ANNEXE III : Composition du milieu TY (Tryptone
Yeast Agar) en g/l (BERINGER, 1974)
Tryptone 5
Extrait de levure 3
CaCl2 H2O 0.87
Eau distillée 1000 ml
Agar 12
pH 6,8
Autoclavage 120 °C pendant 20 minutes.

* 1 « HELLRIEGEL et
WILFART, 1888 cités par EL HILALI, 2006 »
* 2 « BEYERINCK, 1888
cité par EL HILALI, 2006 »
* 3 « WORONIN, 1866
cité par EL HILALI, 2006 »
* 4 « FRANK, 1879
cité par EL HILALI, 2006 »
* 5 « HELLRIEGEL et
WILFARTH, 1888 cités par EL HILALI, 2006 »
* 6 « BEYERINCK, 1888
cité par EL HILALI, 2006 »
* 7 « FRANK, 1889
cité par EL HILALI, 2006 »
| 


