|
REPUBLIQUE DU CAMEROUN
Paix
-Travail-Patrie
********
UNIVERSITE DE DSCHANG
FACULTE DES SCIENCES
|
|
REPUBLIC OF CAMEROON Peace-work-fatherland
***********
UNIVERSITY OF DSCHANG
FACULTY OF SCIENCE
|
DEPARTEMENT DE BIOCHIMIE
DEPARTMENT OF BIOCHEMISTRY
UNITE DE FORMATION DOCTORALE DES SCIENCES FONDAMENTALES
ET TECHNOLOGIQUES
UNITE DE PARASITOLOGIE ET D'ENTOMOLOGIE MOLECULAIRE
DU
DEPARTEMENT DE BIOCHIMIE
|
CARACTERISATION GENETIQUE DE Glossina pallicera
pallicera CIRCULANT DANS LE FOYER DE LA MALADIE DU SOMMEIL DE CAMPO DU SUD
FORESTIER CAMEROUNAIS
|
THESE
Rédigée et présentée pour
l'obtention du diplôme de Master of Science
(M.Sc) en Biochimie
Option : Biochimie Clinique
Par
GOMSEU DJOUMSIE Emmanuel Boris
Licencié-ès Science en Biochmie
Matricule : CM04-09SCI1597
Sous la Direction de :
Dr SIMO Gustave


Décembre 2015
Chargé de Cours
|
REPUBLIQUE DU CAMEROUN
Paix
-Travail-Patrie
********
UNIVERSITE DE DSCHANG
FACULTE DES SCIENCES
|
|
REPUBLIC OF CAMEROON Peace-work-fatherland
***********
UNIVERSITY OF DSCHANG
FACULTY OF SCIENCE
|
DEPARTEMENT DE BIOCHIMIE
DEPARTMENT OF BIOCHEMISTRY
ATTESTATION DE CORRECTION
THESE DE MASTER OF SCIENCE (M.Sc) EN BIOCHIMIE
DE M. GOMSEU DJOUMSIE Emmanuel Boris
Je soussigné, Pr. TUME Christopher,
Président du jury de thèse de Master de GOMSEU DJOUMSIE
Emmanuel Boris, certifie que la thèse de Master of Science
présentée publiquement le 02 février 2016 à
Université de Dschang, par GOMSEU DJOUMSIE Emmanuel Boris
pour l'obtention du diplôme de Master of Science
(M.Sc) en Biochimie, option Biochimie Clinique intitulé
« Caractérisation génétique de Glossina pallicera
pallicera circulant dans le foyer de la maladie du sommeil de Campo du Sud
forestier camerounais » a été corrigée selon les
recommandations des membranes de jury.
En foi de quoi la présente attestation est
délivrée pour servir et valoir ce que de droit.
Fait à Dschang le .
Le PRESIDENT DU JURY L'EXAMINATEUR
Pr. TUME Christophe Pr. GATSING Donatien
Le RAPPORTEUR
Dr. SIMO Gustave

Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
FICHE DE CERTIFICATION DE L'ORIGINALITE DU
TRAVAIL
Je soussigné, GOMSEU DJOUMSIE Emmanuel
Boris, matricule CM04-09SCI1597, atteste que la présente
thèse est le fruit de mes propres recherches effectuées à
l'Unité de Parasitologie et d'Entomologie Moléculaire du
Département de Biochimie de la Faculté des Sciences de
l'Université de Dschang en collaboration avec le Laboratoire de
Biochimie, d'immunologie et de Biotechnologie de la Faculté de
Médecine et des Science Biomédicales de l'Université de
Yaoundé I et le laboratoire de biologie moléculaire de la
Faculté des Sciences de l'Université de Yaoundé I, sous la
direction de Dr. SIMO Gustave.
Cette thèse est authentique et n'a été
antérieurement présentée pour l'acquisition de quelque
grade universitaire que ce soit.
Visa de l'auteur
Date
GOMSEU DJOUMSIE Emmanuel Boris
Visa du directeur
Date
II
Dr. SIMO Gustave
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
DEDICACE
III
Je dédie ce travail à mes parents papa
Djoumsié Pierre et maman Nzogang Christine
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
REMERCIEMENTS
Avant toute chose, je rends gloire à DIEU
qui m'a aimé et m'a aidé à mener ses travaux
jusqu'à la fin.
Je ne saurais commencer sans remercier « l'European
Foundation Initiative for Negleted Tropical Disease » (EFINTD), «
l'Appui Intégré pour le Renforcement des Equipes Scientifiques du
Sud » (AIRES-SUD) et UMR177 IRD-CIRAD, pour leurs appuis
financiés
et le laboratoire de biologie moléculaire de la
Faculté des Sciences de l'Université de Yaoundé I et le
Laboratoire de Biochimie, d'immunologie et de Biotechnologie de la
Faculté de Médecine et des Science Biomédicales de
l'Université de Yaoundé I, qui m'ont offert un cadre idéal
pour la réalisation de ce travail.
Mes remerciements s'adressent aux:
+ Dr. Simo Gustave, qui m'a donné
l'occasion de réaliser les travaux de recherche dans le domaine de la
biologie moléculaire. Vos critiques non seulement dans la
compréhension des manipulations mais aussi dans la rédaction de
ce mémoire m'ont permis de vaincre plusieurs difficultés. Je
saisis cette occasion pour vous adresser ma profonde reconnaissance ;
+ Pr. Njiokou Flobert qui m'a donné
cette opportunité d'exercer mes travaux de recherche au sein de son
laboratoire ;
+ Pr. Kuiate Jules- Roger, Chef de
département de Biochimie pour m'avoir autorisé à prendre
une inscription en Master II. Je saisis cette occasion pour vous adresser ma
profonde reconnaissance ;
+ Mr. Melachio Tanekou Trésor Tito, le
principal mentor de ce travail, pour sa disponibilité, ses
encouragements et ses apports techniques qui m'ont permis de comprendre
l'utilisation des différents logiciels. Je ne saurais jamais vous
remercier assez ;
+ Enseignants du département de
biochimie pour leur encadrement et pour les énormes sacrifices fait pour
notre formation académique. Il s'agit de : Pr. Gatsing Donatien,
Pr. Kuete Victor, Pr. Telefo Phelix, Pr. Tume Christopher, Pr. Womeni Hilaire,
Pr. Zambou Ngoufack François Dr. Agbor Esther, Dr. Biapa
Prospère, Dr. Kaktcham Pierre Marie, Dr. Kengne Anne Pascale, Dr. Klang
Julie, Dr. Kouitcheu Laure, Dr. Kuate Dieudonné, Dr. Mbaveng Armelle,
Dr. Ngoh Newilah Gérard, Dr. Njateng Guy Sedar Senghor, Mme Goka
Stéphanie, Mr. Innocent Mbulli Ali ;
+ Ainés de laboratoire pour leurs
conseils et leurs soutiens techniques. Il s'agit de : Mme Tchouomene
Labou Judith, Mr. Fogué Soubgwi Pythagore, Mme Kame Ginette, Mr.
Noubouossie Sylvain, Mr. Tiofack Zebaze Arnol, Mr. Kanté Sartrien, et
Mr. Ofon Elvis Amih;
iv
+ Camarades de promotion pour leur encouragement.
+ Amis pour l'organisation et le travail en groupe. Il s'agit
de : Natheu Kamhoua Cerge, Marbou Takougoum Wiliane, Wam Elvis Chongsi
et Temgoua Jules;
+ Mr. Kenfack Michel et Mme Fouafack
Fernande pour leur accueil chaleureux et leur encadrement qui m'ont
aidé a surmonté mes difficultés durant mon séjour
dans la ville de Yaoundé ;
+ Frères et soeurs Tsafack Tsanang Henri,
Kouakam Hugues, Tekoudjou Jule-Fleury, Ngouomo-Guy Aurélien, Mekengmo
Irène, Kengni Darine, Messop Zita et Fomo Dorinette pour leurs
soutiens moraux ;
+ Tantes Noubouossie Thérèse, Kouguem
Clotilde pour leurs soutiens moraux et financiers ;
+ Tous ceux qui de près ou de loin, ont
contribué d'une manière ou d'une autre à la
réalisation de ce travail. Merci infiniment.
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
V
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
TABLE DES MATIERES
ATTESTATION DE CORRECTION i
FICHE DE CERTIFICATION DE L'ORIGINALITE DU TRAVAIL
ii
DEDICACE iii
REMERCIEMENTS iv
TABLE DES MATIERES vi
LISTE DES FIGURES viii
LISTE DES TABLEAUX ix
LISTE DES ABREVIATIONS x
RESUME xi
ABSTRACT xii
INTRODUCTION 1
I. REVUE DE LA LITTERATURE 3
I.1. Les trypanosomoses animales africaines 3
I.2. Historique de la trypanosomose animale africaine (nagana)
3
I.3. La Trypanosomiase humaine africaine 4
I.4. Historique de la THA 4
I.5. Répartition géographique des trypanosomoses
africaines 5
I.6. Les parasites responsables des trypanosomoses africaines
7
I.7. Vecteurs 8
I.7.1. Histoire des glossines 9
I.7.2. Taxonomie 9
I.8. Biologie du vecteur 11
I.8.1. Morphologie 11
I.8.2. Cycle de vie 12
I.8.3. Comportement nutritionnel des glossines 13
I.8.4. Ecologie de la glossine 14
I.8.5. Distribution géographique des mouches
tsétsé 14
I.9. Lutte contre les trypanosomoses 16
I.10. Génétique des populations 18
vi
I.10.1. Principe fondamental de la génétique des
populations 19
I.10.2. Facteurs affectant la génétique des
populations 20
I.10.3. Les types de marqueurs moléculaires
utilisés dans la génétique des populations 21
II. MATERIEL ET METHODES 24
II.1. Site d'étude 24
II.2. Echantillonnage 25
II.3. Extraction de l'ADN 25
II.4. Analyses moléculaires 26
II.4.1. Principe de la PCR 26
II.4.2. Identification moléculaire de G. p. pallicera
27
II.4.2.1. Amplification des loci microsatellites des glossines
28
II.4.2.2. Séparation des produits amplifiés par
électrophorèse sur gel d'agarose 29
II.4.2.3. Séparation des allèles sur gel de
polyacrylamide 30
II.4.3. Analyse génétique des données 31
III. RESULTATS 33
III.1. PCR diagnostic des espèces de glossines 33
III.2. Sensibilité des différents marqueurs
microsatellites 33
III. 3. Polymorphisme des marqueurs utilisés 35
III.4. Analyses génétiques 39
III.4.1. Polymorphisme des marqueurs utilisés 39
III.4.2. L'hétérozygotie et le coefficient de
consanguinité 40
III.4.3. Différenciation génétique entre les
populations 45
IV. DISCUSSION 50
CONCLUSION 54
PERSPECTIVES 55
REFERENCES 56
ANNEXES 67
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
VII
|
LISTE DES FIGURES
|
|
|
Différents foyers de la THA au Cameroun
|
7
|
|
Classification des trypanosomes
|
8
|
|
classification des glossines
|
10
|
|
Représentation schématique d'une glossine
|
11
|
|
Vue ventrale des appareils génitaux mâle et femelle
des glossines
|
12
|
|
Cycle reproductif de la glossine
|
13
|
Figure 1 : Figure 2: Figure 3: Figure 4: Figure 5 :
Figure 6:
Figure 7 : Distribution des espèces de
glossines appartenant au groupe Fusca, Morsitans
et Palpalis en Afrique 15
Figure 8 : Carte du Foyer de la THA de Campo
(Institut National de Cartographie
Yaoundé. 24
Figure 9: Différentes étapes de la
PCR 27
Figure 10: Photo d'un gel d'agarose montrant les
produits d'amplification des ITS1 de
différentes espèces de glossines.
33
Figure 11: Exemple de photo montrant les profils des
bandes obtenues sur gel d'agarose et
illustrant les amplicons issus de l'amplification du locus 55.3.
35
Figure 12: Photo d'un gel de polyacrylamide montrant les
profils des allèles obtenus pour
|
chaque glossine et aux loci C102 et PGP24.
|
36
|
|
Nombre d'allèles en fonction des espèces de
glossines
|
40
|
|
Nombre d'allèles par locus
|
40
|
Figure 13: Figure 14:
Figure 15: Valeurs de la FIS en
fonction des loci et pour les populations de G. p. pallicera42
Figure 16: Les valeurs de la FIS en
fonction des loci et pour les populations de G. p.
palpalis 42
Figure 17 : Courbe de
régression des FIS en fonction des allèles nuls pour
G. p. palpalis
43
Figure 18 : Courbe de régression
des FIS en fonction des allèles nuls pour G. p. pallicera
43 Figure 19: Dendrogramme illustrant les
différences et les similarités génétiques entre
les espèces de glossines 46
Figure 20:
Dendrogramme illustrant les différences et les
similarités entre les populations
de glossines 47
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
VIII
LISTE DES TABLEAUX
Tableau 1: Séquences et
températures d'hybridation des amorces utilisées pour le
génotypage 29
Tableau 2: Nombre de glossines
amplifiées en fonction des espèces et des loci
microsatellites. 34
Tableau 3: Nombre et pourcentage de G. p.
pallicera et G. p. palpalis amplifiés à
chaque locus microsatellite 35
Tableau 4 : Génotypes multilocus
obtenus pour chaque glossine analysée et pour les
cinq loci considérés. 37
Tableau 5: Hétérozygotie et les
valeurs de FIS en fonction des loci chez G. p.
pallicera 41
Tableau 6: Hétérozygotie et les
valeurs de FIS en fonction des loci chez G. p.
palpalis 41
Tableau 7 : Fréquence des
allèles nuls observés et attendus 44
Tableau 8: P-values du
déséquilibre de liaison entre paires de loci 45
Tableau 9: Variation de FST entre
les populations de différentes espèces. 48
Tableau 10 : Variation de FST entre
les sous populations de G. p. pallicera 48
Tableau 11 : Variation de FST entre
les sous populations de G. p. palpalis 49
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
ix
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
LISTE DES ABREVIATIONS
ADN : Acide désoxy-ribonucleique
AFLP: Amplied Fragment Lenght
Polymorphism
AIRES-SUD: Appui Intégré pour le
Renforcement des Equipes Scientifiques du Sud
BET: Bromure d'Ethidium
CIRAD : Centre de coopération
Internationale en Recherche Agronomique pour le
Développement
CTAB: Cetyl Trimethyl Ammonium Bromide
dNTP: Désoxyribonucléotide
5'-TriPhosphate
EDTA: Ethylène Diamine Tétra
Acétate
EFINTD: European Foundation Initiative for
Neglected Tropical Disease
FAO: Food and Agriculture Organisation
IRD: Institut de Recherche pour le
développement
MEGA: Molecular Evolutionnary Genetics
Analysis
MSA : MicroSatellite Analyzer
OMS : Organisation Mondiale de la
Santé
PCR: Polymerase Chain Reaction
RAPD: Randomly Amplified Polymorphic
DNA
SNP: Single-Nucleotide-Polymorphism
SSCP: Single-Stranded Conformational
Polymorphism
TAA: Trypanosomose Animale Africaine
Taq: Thermus aquaticcus
TBE: Tris-Borate-EDTA
TEMED: Tetramethylethylenediamine
THA: Trypanosomiase Humaine Africaine
TIS: Technique des insectes stériles
Tris : Tris
hydroxyméthyl-aminométhane
UV : Ultra-Violet
X
RESUME
Les Trypanosomiases humaines et animales africaines restent un
problème majeur de santé au Cameroun. Ces maladies sont
transmises par un vecteur appelé mouche tsétsé ou
glossine. Glossina pallicera pallicera est une des espèces les
plus répandue et responsable de la trypanosomose animale africaine (TAA)
dans le foyer de Campo. Toutefois, très peu d'informations sont
disponibles sur la biologie et la structure de ces populations de vecteurs.
Pour mieux appréhender la structure génétique des
populations de glossines du sud forestier camerounais, nous avons entrepris une
étude sur la caractérisation moléculaire de Glossina
pallicera pallicera du foyer de la Trypanosomiase Humaine Africaine (THA)
de Campo.
Pour y parvenir, des prospections entomologiques ont
été menées dans trois villages (Akak, Campo Beach et Rio
Campo) du foyer de THA de Campo. Au cours de ces prospections des glossines ont
été capturées à l'aide des pièges
pyramidaux. La méthode du CTAB (Cetyl Trimethyl Ammonium Bromide) a
été utilisée pour extraire l'ADN des pattes des mouches
tsétsé et par la suite, neuf marqueurs microsatellites ont
été utilisés pour caractériser
génétiquement G. p. pallicera.
Dans cette étude, 72 mouches tsétsé
appartenant à 5 espèces différentes ont été
utilisées: 45 Glossina pallicera pallicera, 17 Glossina
palpalis palpalis, 4 Glossina submorsitans, 3 Glossina
caligenea, 2 Glossina nigrofusca et une Glossina
fuscipes. Parmi les neuf marqueurs microsatellites utilisés, quatre
d'entre eux n'ont pas été considérés pour les
études de génétique des populations parce que moins de 50%
des échantillons ont été amplifiés par ces
marqueurs. Pour les cinq marqueurs utilisés pour les analyses
génétiques, le taux d'amplification variait d'un marqueur
à l'autre: 91,11% pour 55.3 ; 97,77% pour PGP13 ; 97,77% pour GPCAG ;
95,55% pour C102 et 84,44% pour PGP24. Les cinq marqueurs microsatellites ont
montré plusieurs génotypes de G. p. pallicera circulant
dans les villages du foyer de la THA de Campo. Ces résultats indiquent
des polymorphismes génétiques au sein des populations de G. p
.pallicera du même village et des différents villages. Nos
résultats ont révélé un déficit en
hétérozygote non significatif (FIS (indice de fixation
des individus dans les sous-populations) = 0,04 ; P = 0,31) entre les
populations de G. p. pallicera. Cependant, un déficit en
hétérozygote significatif (FIS = 0,145 ; P = 0,0001) a
été observé pour les populations de G. p.
palpalis. Ce déficit en hétérozygote serait dû
à la forte variation entre les valeurs de FIS des
différents marqueurs. Entre les sous-populations de G. p.
pallicera, aucune différence significative n'a été
observée pour les valeurs du FST (indice de fixation entre
sous-populations de la population totale) indiquant l'absence de structuration
au sein des sous-populations des différents villages. Néanmoins,
des différences significatives ont été observées
entre G. p. pallicera et d'autres espèces de mouches
tsétsé.
Ces premières investigations portant sur la
génétique des populations de Glossina pallicera pallicera
ont révélé une absence de sous-structuration entre
ces populations de vecteurs et différents génotypes circulant
dans les villages du foyer de la THA de Campo.
Mots clés : marqueurs microsatellites,
Glossina pallicera pallicera, PCR, génétique des
populations
xi
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
ABSTRACT
Human and animal African trypanosomiasis remains a major
health problem in Cameroon. These diseases are transmitted by a vector called
the tsetse fly or Glossina. Glossina pallicera pallicera is one of the
most widespread species and is responsible for animal African trypanosomosis
(AAT) in the focus of Campo. However, very little information about its biology
and population structure is available. In order to better understand the
genetic structure of tsetse fly populations of the Southern Cameroon forest, we
carried out a study on the molecular characterization of Glossina pallicera
pallicera in the human African trypanosomiasis (HAT) focus of Campo.
For this purpose, we carried out entomological studies in
three villages (Akak, Campo Beach and Rio Campo) of the HAT focus of Campo.
Flies were captured using pyramidal traps. The CTAB (Cetyl Trimethyl Ammonium
Bromide) method was used to extract DNA from tsetse thy legs and thereafter,
nine microsatellite markers were used to genetically characterize G. p.
pallicera.
In this study, 72 tsetse flies belonging to 5 different
species were used: 45 Glossina pallicera pallicera, 17 Glossina
palpalis palpalis, 4 Glossina submorsitans, 3 Glossina
caligenea, 2 Glossina nigrofusca and one Glossina
fuscipes. Amongst the nine microsatellite markers used, four of them were
not considered for the population genetics studies because less than 50% of
samples were amplified by these markers. For the five markers considered for
the genetic analyses, the amplification rate varied from one marker to another:
91.11 % for 55.3; 97.77 % for PGP13; 97.77 % for GPCAG; 95.55 % for C102 and
84.44 % for PGP24. The five microsatellite markers showed several
genotypes of G. p. pallicera circulating in villages of the Campo
sleeping sickness focus. These results indicate the genetic polymorphisms
within G. p .pallicera populations of the same village and of
different villages. Our results revealed no significant heterozygote deficiency
(FIS (index of fixing of the individuals in the subpopulations) =
0.04; P = 0.31) between G. p. pallicera populations. However, a
significant heterozygote deficiency (FIS = 0.145; P = 0.0001) was
observed for G p. palpalis populations. This heterozygote deficiency
could be due to strong variations between FIS values at different
loci. Between G. p. pallicera subpopulations, no significant
difference was observed for the FST (index of fixing between
subpopulations of the total population) values; indicating no structuration
within subpopulations of different villages. Nevertheless, significant
differences were observed between G. p. pallicera and other tsetse
flies species.
These first investigations on the population genetics of G
p. pallicera have revealed an absence of sub-structuration between these
vector populations and different genotypes circulating in villages of the Campo
sleeping sickness focus.
Key words: Microsatellite markers, Glossina
pallicera pallicera, PCR, population genetics
XII
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
INTRODUCTION
Les Trypanosomoses Africaines sont des maladies parasitaires
causées par des protozoaires flagellés appelés
trypanosomes. Ces parasites entrainent chez l'homme la trypanosomiase humaine
africaine (THA) encore appelée maladie du sommeil, et chez l'animal la
trypanosomose animale africaine (TAA) encore connue sous le nom de "nagana".
Ces maladies constituent une entrave pour le développement de plusieurs
pays africains. Les trypanosomes sont transmis aux mammifères par des
piqûres d'insectes hématophages appelés mouches
tsétsé ou glossines. Ces glossines responsables de la
transmission des trypanosomoses africaines appartiennent au genre
Glossina. Il existe plusieurs espèces de glossines et leur
distribution est fonction des facteurs éco climatiques. Chaque
espèce de glossine ne transmet qu'un nombre limité
d'espèces de trypanosomes et la distribution des espèces de
trypanosomes peut être liée à celle des glossines. De
manière générale, la distribution des trypanosomoses
africaines suit celle de leur vecteur et couvre environ 10 millions de
Km2, soit un tiers du continent africain (Brunhes et al.,
1994).
La Nagana demeure un obstacle pour le développement de
l'élevage et de l'agriculture en milieu rurale. On estime que 50
millions de bovins et 70 millions de petits ruminants sont exposés aux
risques de la TAA (Trail et al., 1985). La productivité du
bétail et l'utilisation des animaux pour l'agriculture (engrais,
traction) sont également affectées. Alors que la population
humaine augmente à raison de 3,2% par an, les productions de viande et
de lait n'augmentent respectivement que de 1,4 et 2,3%. Ainsi, on estime
qu'à partir du 21ème siècle, une importation de
2 à 5 millions de tonnes de viande et de 10 à 15 millions de
tonnes de lait pour un coût annuel de 15 milliards de dollars US serait
nécessaire pour couvrir les besoins des populations vivant dans les pays
où sévit la TAA (Pangui, 2001). Pour lutter contre les
trypanosomoses africaines, deux principales stratégies sont
déployées notamment l'utilisation des médicaments et la
lutte contre le vecteur. Jusqu'à nos jours, seulement trois trypanocides
sont disponibles et des résistances ont été
signalées contre ces médicaments dans plusieurs pays africains.
Quant à la lutte anti-vectorielle, plusieurs méthodes de lutte
ont été développées: l'utilisation des insectes
stériles (TIS) et des écrans, le piégeage (Cuisance,
1989), l'utilisation des insecticides s'est souvent heurtée aux
écologistes à cause de la rémanence des produits
utilisés et leur action négative sur la biodiversité
(Koeman et Pennings, 1970). L'utilisation de certaines de ces techniques de
lutte n'a donné des résultats positifs que dans des îles
(Salano et al., 2010). Cependant, des tentatives visant
l'éradication des populations de glossines continentales se sont
soldées par des échecs, à cause de la grande
capacité de dispersion des glossines qui ré-envahissent
rapidement les zones traitées (Cuisance, 1985). Pour faire face à
ce problème, des études sur la structure génétique
des populations de vecteurs ont été envisagées. Au cours
des dernières décennies, des outils
1
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par
GOMSEU DJOUMSIE Emmanuel Boris
moléculaires ont été
développés pour caractériser les populations de glossines
afin de mieux comprendre leur écologie et leur structure
génétique. C'est dans ce contexte que des marqueurs
microsatellites ont été identifiés pour affiner la
caractérisation génétique des vecteurs. Les
microsatellites sont des séquences d'ADN répétées
en tandem se caractérisant par une hyper-variabilité et une
abondance dans le génome (Schwenkebecker et al., 2004). Ils
sont largement utilisés dans les études génétiques
et phylogénétiques et ont déjà montré leur
intérêt dans la caractérisation de plusieurs espèces
de glossine (Schwenkebecker et al., 2004). Ces marqueurs ont notamment
permis de confirmer l'isolement génétique des populations
insulaires de glossines (Camara et al., 2006). Les méthodes de
lutte déployées à partir des études sur la
structure génétique des populations de glossines ont
présenté un grand succès dans la région du Niayes
au Sénégal (Solano et al., 2010).
En Afrique centrale et occidentale, des marqueurs
microsatellites ont été utilisés pour analyser la
structure génétique des populations de Glossina palpalis
palpalis et Glossina palpalis gambiensis du Cameroun et du
Burkina Faso respectivement. Ces travaux ont révélé une
diversité génétique importante au sein des populations de
ces glossines, et surtout un niveau de structuration lié à la
distance géographique entre les populations étudiées
(Melachio et al., 2011 ; Solano et al., 2000). La plupart
d'études réalisées sur la structure
génétique des populations de glossines se sont concentrées
sur des glossines péri-domestiques responsables de la transmission des
trypanosomes chez l'homme. Quant aux glossines trouvées essentiellement
en zone forestière comme G. p. pallicera, très peu
d'études génétiques ont été menées
sur ces insectes malgré le fait que ces glossines soient responsables de
la TAA. Il est évident qu'une étude sur la
génétique des populations de G. p. pallicera
améliorerait nos connaissances sur l'épidémiologie et
la lutte contre des trypanosomoses animales.
L'objectif principal de ce travail est de faire une
caractérisation génétique de G. p. pallicera afin
de fournir des données sur la structure génétique des
populations de ces vecteurs avec pour finalité d'obtenir des
informations qui pourraient contribuer à la planification d'une lutte
anti-vectorielle. De manière spécifique, nous avons:
? analysé le polymorphisme génétiquement
au sein des populations de G. p. pallicera en utilisant des marqueurs
microsatellites ;
? évalué le polymorphisme
génétique de G. p. pallicera en comparaison à
d'autres espèces de glossines.
? évalué la structuration
génétique entre les populations de G. p. pallicera.
2
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
I. REVUE DE LA
LITTERATURE
I.1. Les trypanosomoses animales africaines
La trypanosomose animale africaine, encore appelé
«nagana» lorsqu'elle se développe chez les bovins ou des
chevaux, ou "surra" lorsqu'elle se développe chez les porcs domestiques,
est causée par plusieurs espèces de trypanosomes. Cette maladie
réduit le taux de croissance, la productivité de lait et la force
des animaux, conduisant généralement à la mort de ces
derniers. La maladie débute par une phase d'hyperthermie qui correspond
au premier pic de parasitémie. Deux à trois semaines après
la piqûre infectante, le taux d'hémoglobine et
1'hématocrite chutent, reflétant l'anémie qui est le
symptôme majeur des trypanosomoses bovines. Certaines espèces de
bétail sont trypanotolérantes à cause de leurs
capacités à survivre et se développer, même
lorsqu'ils sont infectés par des trypanosomes (Murray et al.,
1979). Ces espèces animales ont cependant des taux de
productivité plus faibles lorsqu'ils sont infectés (Murray et
al., 1979). Trypanosoma congolense et Trypanosoma vivax
sont les deux espèces les plus importantes qui infectent les bovins
en Afrique sub-saharienne. Trypanosoma simiae provoque une maladie
virulente chez les porcs.
I.2. Historique de la trypanosomose animale africaine
(nagana)
Au milieu du XVème siècle, les explorateurs
portugais rapportèrent que les caravanes assurant le commerce de sel et
d'or entre le désert du Sahara et l'Afrique occidentale devaient, au
niveau du Mali, se débarrasser de leurs dromadaires qui étaient
décimés lors de la traversée des zones à glossines
(Cattand, 2001). Les conquêtes des Maures au XVIIème siècle
furent bloquées au-delà du sud du Sahara à cause des
chevaux qui périssaient suite aux attaques des glossines. Dès
lors, toutes les expéditions s'effectuant à chevaux
étaient freinées dès qu'elles devaient traverser les zones
humides.
Les premières espèces de glossines ont
été décrites en 1830 par Wiedemann (G.
longipalpis) et Robineau-Desvoidy (G. p. palpalis) (Rageau et
al., 1953 ). En 1850, le nom «tsétsé» fut
introduit par Gordon Cumming dans son livre «Five years of a Hunter's Life
in the Far Interior of Africa» ; cette onomatopée était
utilisée par les indigènes au Botswana pour désigner les
mouches, Glossina morsitans, qui décimaient les troupeaux de
boeufs. C'est en 1857 que David Livingstone a évoqué le
rôle des glossines dans la transmission de la maladie ;
soupçonnant qu'elles injectaient un venin à leur hôte. En
1875, Louis Figuier écrivait «cette suceuse de sang
sécrète, par une glande située à la base de sa
trompe, un venin si actif que trois ou quatre mouches suffisent pour tuer un
boeuf».
3
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
Les premiers trypanosomes ont été
découverts dans l'intestin de taons en 1680, dans le sang de truite en
1841, puis chez la grenouille, la taupe et le rat (Morlais, 1998). Leur
intérêt médical ou vétérinaire n'a
été démontré que plus tard. En 1880, Griffith Evans
qui était un vétérinaire de l'armée Indienne, mit
en évidence, chez des chevaux et chameaux malades, le parasite qui sera
nommé plus tard Trypanosoma evansi ; montrant ainsi la
pathogénicité de ce parasite. En 1894, Bruce trouva des
trypanosomes dans le sang de bovins souffrant de nagana. Il réussit
ensuite à infecter des chevaux avec des mouches tsétsé,
démontrant alors le rôle vecteur des glossines (Cattand, 2001).
T. b. brucei fut décrit en 1899 par Plimmer et Bradford.
I.3. La Trypanosomiase humaine africaine
La trypanosomiase humaine africaine (THA) commence
après l'inoculation du trypanosome chez le mammifère par la
glossine. Les trypanosomes se multiplient sur le site de l'infection menant
à une réponse immunitaire: un gonflement ou chancre
détectable sur la peau. Le développement de la maladie peut
être séparé en deux étapes sur la base des
symptômes cliniques observés : la phase hémolymphatique qui
est caractérisée par la fièvre, des maux de tête,
des problèmes articulaires et des démangeaisons ; la phase
neurologique commence lorsque le parasite envahit le système nerveux
central en passant à travers la barrière
hémato-encéphalique. La période d'incubation de la THA
varie de quelques jours à plusieurs semaines ou mois pour T. b.
gambienses (forme aiguë), tandis que chez T. b. rhodienses,
la maladie se révèle souvent fatale dans les 9 à 12 mois,
si le malade n'est pas traité.
I.4. Historique de la THA
Le premier cas de la THA a été enregistré
au 14ème siècle en Afrique et a été
décrit par l'historien Ibn Khaldoun. Cet historien a parlé de la
mort de l'empereur malien, sultan Mari Jata après une maladie
caractérisée par une somnolence diurne prolongé (Williams,
1996). En 1734, l'anglais Akins publie le premier rapport médical sur la
maladie du sommeil où il décrit plus précisément
les stades avancés de la maladie, dénommée «sleeping
distemper» (Cox, 2004). Livingstone (1852), missionnaire écossais
soupçonne la relation entre une mouche et la maladie et rapporte
l'existence de la maladie sur les rives du lac Tanganyika où il perdit
tout le bétail après que celui-ci fût piqué par la
mouche tueuse. En 1901, Forde découvre des trypanosomes (nommés
Trypanosoma gambiense par Dutton en 1902) dans le sang d'un
4
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
malade. Puis, Castellani en 1903 rapporte la présence
du protozoaire dans le liquide céphalorachidien (LCR) de la
majorité des malades. La même année, Bruce fournit la
preuve que la maladie du sommeil est transmise par la mouche
tsétsé. En 1910, Stephens et Fantham décrivent chez un
patient de Rhodésie un nouveau trypanosome Trypanosoma rhodesiense.
De morphologie différente, ce dernier s'avère plus virulent
pour l'homme que T. gambiense et est transmis par la glossine
(Mulligan, 1970). C'est en 1917 que la première équipe mobile de
dépistage de la maladie du sommeil mis sur pied par le Docteur Eugene
Jamot a vu le jour au Cameroun.
L'Afrique a connu trois grandes flambées de la maladie
du sommeil (Laveissière et Penchenier, 2000). La première
commence en 1885 au confluent des fleuves Oubangui et Zaïre et
s'étend jusqu'au lac Victoria. Elle fait près de trois cent mille
et cinq cent mille morts respectivement dans le bassin du Congo et les foyers
Busoka. La deuxième commence en Afrique centrale et s'étend
dès 1920 en Afrique de l'ouest. Devant l'ampleur de la situation, les
autorités coloniales de l'époque prirent des mesures permettant
la création d'équipes mobiles de dépistage et de
traitement, l'instigateur fut le célèbre docteur Eugène
Jamot. La pandémie fut alors maîtrisée. La
réémergence des foyers historiques et l'émergence de
nouveaux foyers marquent la troisième épidémie.
I.5. Répartition géographique des
trypanosomoses africaines
La distribution des trypanosomoses africaines suit celle de
leur vecteur (Glossina sp.). Elle se situe en Afrique intertropicale
entre les latitudes 15° Nord et 20° Sud (Brunhes et al.,
1994). La trypanosomose animale touche le tiers de la superficie totale de
l'Afrique. Les glossines sont les vecteurs de la trypanosomose dans 37 pays
africains, couvrant environ 10 millions de kilomètres carrés.
Près de 50 millions de bovins, 230 millions de moutons et 40 millions de
chèvres sont exposés à la trypanosomose animale. Le risque
est plus grave dans les zones subhumides et les parties plus humides des zones
semi-arides qui détiennent le plus grand potentiel d'expansion agricole
du continent. Cette maladie entraîne chaque année le
décès de 3 millions de bétails, la perte de 500 000 tonnes
de viande ainsi qu'un million de tonnes de lait (Duvallet, 2006). La
répartition géographique des trypanosomoses animales
dépend de l'espèce de trypanosome ainsi que de son hôte
spécifique. Selon les données publiées par le PATTEC
(2010), les dix régions du Cameroun sont susceptibles d'être
endémiques. Par ailleurs, les régions de l'Adamaoua, du Nord et
l'extrême Nord constituent les zones les plus atteintes car,
caractérisées par une forte densité de l'élevage
bovin et des
5
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
petits ruminants. Les travaux menés par la mission
d'éradication des glossines dans les régions du Cameroun ont
permis de contrôler le vecteur principal de la nagana dans le plateau de
l'Adamaoua. Les autres zones du Cameroun restent pour la plupart
incontrôlées.
Quant à la THA, 37 pays d'Afrique et environ 60
millions d'hommes sont exposés aux risques de cette maladie et
près de 70 000 personnes portent une infection active. On estime
à moins de 7 000 le nombre de personnes détectées chaque
année (Simarro et al., 2011) et près de 100 le nombre de
morts de suite de la maladie du sommeil par jour (Launois et al.,
2004). La THA se présente sous deux formes : la forme chronique qui
sévit en Afrique Centrale et occidentale et qui est due à T.
b. gambiense et la forme aiguë présente en Afrique orientale
et Australe et qui est causée par T. b. rhodesiense
(Steverding, 2008). Au début du 21ème siècle,
plusieurs foyers actifs de la THA existaient au Cameroun. Ces foyers (Figure 1)
étaient regroupés dans quatre grandes régions
géographiques: les foyers de l'Ouest (Fontem, Mamfé et Plaines
des Mbos), les foyers côtiers (Douala et Campo) les foyers de l'Est
(Bafia et le bassin du Nyong; Doumé, Abong-Mbang et Lomié) et les
foyers du nord (Logone et Chari). Aujourd'hui, plusieurs de ces foyers ont
disparu à cause des modifications du milieu ou de la lutte. C'est ainsi
que l'urbanisation a entraîné la disparition du foyer de Douala et
le climat devenu rude pour les glossines a favorisé l'extinction du
foyer du Logone et Chari. Les foyers de Bafia et du Haut-Nyong ont
été éteints grâce à la lutte anti-vectorielle
et l'assainissement du réservoir animal.
6
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris

Figure 1 : Différents foyers de la THA au
Cameroun (Simo, 2001). I.6. Les parasites responsables des trypanosomoses
africaines
Les trypanosomes sont les parasites responsables des
trypanosomoses africaines. La classification phylogénique des
trypanosomes les situe dans le sous-règne des Protozoaires, le
phylum des Sarcomastigophora, le sous-phylum des
Mastigophora, la classe des Zoomastigophora, l'ordre des
Kinetoplastidae, la famille des Trypanosomidae et le genre
7
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
Trypanosoma (figure 2) (Hoare, 1972). Les
trypanosomes africains pathogènes du bétail appartiennent
à la section des Salivaria, aux sous-genres Nannomonas (T.
congolense), Duttonella (T. vivax), Trypanozoon
(T. b. brucei, T. evansi et T. equiperdum), et
Pycnomonas (T. suis) (Sidibé et Desquesnes, 2003). Les
bovins sont parfois infectés par plusieurs espèces de
trypanosomes pathogènes comme T. congolense, T. b. brucei
et T. simiae.

Figure 2: Classification des trypanosomes (Hoare,
1972)
I.7. Vecteurs
Les glossines sont des insectes hématophages qui
transmettent les trypanosomes chez leurs hôtes spécifiques. Les
vecteurs de la nagana sont retrouvés principalement au bord des cours
d'eau et des lacs, dans des galeries forestières et des vastes
étendues de savane arbustive. Ces glossines ont été
intensivement étudiées en raison de leur importance
médicale, vétérinaire et économique.
8
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
I.7.1. Histoire des glossines
Les glossines ont été décrites pour la
première fois en 1930, mais elles étaient déjà
connues des populations autochtones d'Afrique. Des documents en l'Egypte
Pharaonique, datant plus de 3000 ans avant J.C., mentionnent un insecte
"skaïti" qui provoque la perte du bétail. Des archives de l'Empire
perse (datant depuis 1000 ans avant J.C.) décrivait la glossine comme un
insecte «mortel pour les bêtes et les nouveau-nés»
(Gros, 2004). En outre, les fossiles ont été trouvés dans
les gisements fossilifères florissant dans le Colorado (Nord de
l'Amérique) (Cockerell, 1917). Cela indique que les glossines existent
depuis trente-quatre millions d'années. Le nom mouche
tsétsé a ensuite été adopté par le
scientifique et est maintenant utilisé en référence
à toutes les espèces de mouches et sous-espèces
appartenant au genre Glossina.
I.7.2. Taxonomie
La mouche tsétsé est un insecte diptère
cyclorrhaphe appartenant à la famille des Glossinidae qui ne comporte
qu'un seul genre, Glossina. Les trois sous-genres correspondent
à trois groupes anciennement définis sur les
caractéristiques morphologiques et écologiques. Il existe 33
espèces et sous-espèces de tsétsé, réparties
en trois sous genres ou groupes (figure 3) dont les caractéristiques
écologiques et comportementales sont très différentes
(Leak, 1999).
- Le groupe palpalis Robineau-Desvoidy, 1830,
regroupe les espèces de petite taille vivant près des zones
humides (mouches riveraines). Elles sont généralement moins
mobiles, restant confinées dans leur habitat. Ce groupe réunit
les vecteurs majeurs de la maladie du sommeil chez l'homme comme G.
fuscipes, G. palpalis et leurs sous-espèces. G. p.
pallicera appartient à l'espèce pallicera Bigot,
1891.
- Le groupe morsitans Zumpt, 1935, réunit les
espèces de taille moyenne, très mobile, occupant la savane. A ce
groupe, appartiennent les vecteurs majeurs des trypanosomoses animales comme
G. pallidipes, G. morsitans spp., G. longipennis et
G. austeni.
- Le groupe fusca Townsend, 1921, regroupe des
glossines de grande taille des zones boisées (mouches de forêts)
se nourrissant sur des animaux. Elles sont sans intérêt
médical et sont considérées jusque-là comme des
vecteurs de moindre importance dans la mesure où leur habitat naturel
est moins fréquenté par l'homme et le bétail.
Néanmoins, des espèces comme Glossina brevipalpis sont
des vecteurs non négligeables des trypanosomoses animales.
9
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
Fusca
PHYLUM CLASSE ORDRE
SOUS-ORDRE SERIES SECTION
FAMILLE GENRE GROUPE
ESPECES
Crustacés Myriapodes
G.fusca
G.haningtoni G.nashi G.tabaniformis G.schwetz i G.vanhoofi
G.fuscipleuri
G.palpalis
G.fuscipes
G.tachinoides
G.pallicera
G.caliginea
Arthropodes
Arachnides
Morsitans
G.morsitans
G.swynnertoni
G.longipalpis
G.pallidipes
G.austeni
|
Thysanoures Hemiptères
|
Diptères
|
Lépidoptères
Hyménoptères...
|
|
Nematocères Brachycères
|
Cyclorrhaphes
|
|
Aschiziens
|
Schizophores
|
|
Acalyptères
|
Calyptères
|
Puplparares
|
|
Muscidés
|
Glossinidès
|
OEstridés...
|
Glossina
Palpalis
G.brevipalpis
G.longipennis
G.nigrofusca
G.medicorum G.severini
SOUS-ESPECES
G.f.fusca
G.f.congolensis
G.n.nigrofusca G.n.hopkinsi
G.p.palpalis G.p.gambiense
G.f.fuscipes G.f.martini G.f.quanzensis
G.p.pallicera
G.p.newsteadi
G.m.morsitans G.m.submorsitans G.m.centralis
Figure 3: classification des glossines (OMS,
1986)
10
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
I.8. Biologie du vecteur I.8.1. Morphologie
Les glossines sont des insectes dont la principale
caractéristique est de posséder une seule paire d'ailes. La paire
postérieure étant transformée en deux petites massues
appelées "haltères". Les glossines ont une taille qui varie entre
6 et 16 mm sans le proboscis. On les reconnaît facilement grâce
à leur trompe située horizontalement dans le prolongement de la
tête, leurs ailes qui se recouvrent au repos et qui présentent une
cellule en forme de hache entre la 4ème et la
5ème nervure, leurs antennes qui portent une
soie particulière plumeuse, l'arista (figure 4). Les
mâles sont généralement plus petits que les femelles. Le
poids des adultes est fonction de l'espèce. L'appareil reproducteur
externe du mâle se distingue par la phallique (callosité fortement
convexe) qui est observable sur la face ventrale de l'abdomen alors que chez la
femelle, on observe plutôt des plaques génitales (figure 5).
L'anus et la vulve sont situés à l'extrémité
postérieure et sont entourés par des plaques dont le nombre, la
forme, et la pilosité varient selon les espèces (Launois et
al., 2004). Contrairement aux moustiques et aux tabanidés (taons)
chez qui, seules les femelles piquent et se nourrissent de sang, les
mâles butinant le nectar des fleurs, les mâles et les femelles des
mouches tsétsé sont hématophages (Gouteux, 1995).

Figure 4: Représentation schématique d'une
glossine (Pollock, 1982)
11
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris

a-Appareil a-Apparel
génital male gén b- Appareil génital
femelle
Figure 5 : Vue ventrale des appareils génitaux
mâle et femelle des glossines, (Laveissiere et al.,
2000).
I.8.2. Cycle de vie
Les mouches tsétsé ont un cycle de vie original.
Les femelles ne peuvent fertiliser plus d'un oeuf à la fois. Elles
conservent chaque oeuf dans leur utérus pour avoir une
progéniture développée intérieurement au cours des
premiers stades larvaires, la stratégie est appelée
viviparité adénotrophique. Pendant ce temps, la femelle alimente
sa progéniture avec une substance laiteuse qui est
sécrétée par une glande modifiée dans
l'utérus. Dans le troisième stade larvaire, la larve laisse
l'utérus de la mouche tsétsé et commence une vie
indépendante. Cette étape de la vie est courte et d'une
durée de quelques heures. La larve nouvellement indépendante
rampe dans le sol, forme une coque externe dure appelée pupe dans
laquelle elle achève sa transformation morphologique. Les pupes sont
petites et mesurent moins de 1,0 cm de long (Jordan, 1986). Cette étape
de la vie a une durée variable, généralement trente
à quarante jours selon les espèces de glossines, selon la
température et l'humidité du sol. Dans les pupes, les larves
complètent les deux derniers stades larvaires et le stade de pupaison.
À la fin du stade de pupaison, la jeune mouche utilise des mouvements de
la tête pour sortir de la pupe, après quoi il se déplace
dans le sol jusqu'à la surface (figure 6).
Hors du sol, cette jeune mouche (ténérale)
étend ses ailes, son abdomen se gonfle et sa trompe horizontale se
développe. Elle recherche par la suite un hôte pour prendre son
premier repas de sang. Ce repas fournira les ressources nécessaires au
jeune mouche pour durcir sa cuticule et développer ses muscles. Les
jeunes mâles sont attirés par les phéromones sexuelles
12
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
émises par les jeunes mouches femelles (qui copulent
seulement une fois dans leur vie). Les mâles placent un spermatophore au
fond de l'utérus des femelles. Le sac de spermatophore vide est
éjecté quelques heures après copulation. La
première larviposition aura lieu 18 jours après la copulation
puis dans un intervalle de 10-11 jours en fonction de la température et
la disponibilité de la nourriture (Cuisance, 1989). Techniquement la
mouche tsétsé subit des processus standards de
développement en insectes qui comprend la formation d'ovocytes,
l'ovulation, la fécondation, le développement de l'oeuf, cinq
stades larvaires, un stade de pupaison, l'émergence et la maturation de
l'adulte.

Figure 6: Cycle reproductif de la glossine (Launois
et al., 2004)
I.8.3. Comportement nutritionnel des glossines
Les glossines sont des insectes hématophages dont les
mâles se nourrissent environ une fois tous les quatre jours et dont les
femelles se nourrissent trois fois au cours de la
13
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
gestation: la première fois au cours de la formation de
la paroi intra-utérine entre la deuxième et troisième
phase du stade larvaire, la seconde à un moment donné et la
troisième seulement après larviposition (Itard et Cuissance,
2004). Chaque espèce de mouche tsétsé à une
préférence alimentaire. Certaines espèces du groupe
palpalis sont opportunistes et adaptent leurs régimes aux
conditions locales. Ils se nourrissent volontiers sur les animaux à sang
chaud ainsi que sur les animaux à sang froid. D'autres ont des
préférences très marquées. G. f. fuscipes
préfèrent se nourrir sur les lézards tandis que
G. f. quanzensis préférera se nourrir du sang des porcs
(Omolo et al., 2009). Les mouches ténérales ont une
préférence alimentaire supérieure à celle des
vieilles mouches qui préfèrent se nourrir des hôtes sur
lesquels elles ont pris leur premier repas de sang.
Les mouches tsétsé ont des récepteurs
sensibles au CO2, à certains phénols
(4-méthylphénols, 3-n-propylphénol etc.), à des
corps cétoniques (acétone) et à l'alcool
(1-octèn-3-ol) trouvés dans l'urine, les excréments et
d'autres sécrétions. Ceux-ci sont exploités par les
mouches pour suivre leur hôte. Les glossines se déplacent dans une
série de vols courts vers leur hôte suivants le gradient de
concentration de ces odeurs. Le facteur visuel est important à de
courtes distances. Il est activé par le système olfactif pendant
le repos. Ce facteur est plus développé chez les mouches
riveraines en comparaison avec les mouches de savane. G. p. pallicera,
la glossine d'intérêt dans cette étude se nourrit de
préférence sur les bovidés, les suidés, les
primates, les humains et les animaux à sang froid (Gouteux et
al., 1982).
I.8.4. Ecologie de la glossine
La végétation est l'élément
essentiel des biotopes de la glossine car elle entretient une humidité,
une température et une luminosité qui convient à cette
dernière. Les glossines du groupe palpalis ont pour habitat le
bord des cours d'eau, les lacs et la forêt. La plupart des espèces
du groupe fusca vivent dans la forêt ombrophile des
basses-terres ou dans les galeries forestières. Par contre, les
glossines du groupe morsitans occupent de vastes étendues de
savane arborescente. Plusieurs espèces se sont adaptées à
des conditions écologiques atypiques. C'est ainsi qu'il existe des
espèces du groupe palpalis qui ont des biotopes
péri-domestiques en zone forestière ou
préforestière, particulièrement G. p.
pallicera.
I.8.5. Distribution géographique des mouches
tsétsé
Les mouches tsétsé sont distribuées de
façon discontinue sur 11 millions de kilomètres carrés de
la superficie de l'Afrique sub-saharienne. Cette zone se situe entre 14°
N
14
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
et 29° S. La répartition des différents
groupes de mouches est liée à leurs préférences
d'habitat. Les mouches appartenant au groupe Fusca se trouvent
principalement dans la forêt ou dans la forêt dense riveraine,
tandis que celles du groupe Morsitans sont limitées
principalement aux savanes boisées. Les mouches tsétsé
appartenant au groupe Palpalis habitent la forêt tropicale, et
elles se trouvent principalement dans la végétation riveraine
(Leak, 1999). La distribution de chacun de ces groupes est
représentée dans la figure 7. Cette figure montre que les
différents groupes et espèces de mouches sont inégalement
répartis sur les zones infestées. En général, les
groupes Fusca et Palpalis se trouvent principalement en
Afrique centrale et occidentale tandis que les groupes Morsitans sont
largement distribués dans les zones sèches d'Afrique de l'Ouest
et de l'Est. La répartition géographique des mouches
tsétsé est entravée par des conditions climatiques
extrêmement chaudes ou froides (au-dessus de 2000 m d'altitude), la
mauvaise pluviométrie, la végétation clairsemée, la
nourriture insuffisante et la nature des sols (inadaptés pour la
pénétration des larves).

Figure 7 : Distribution des espèces de
glossines appartenant au groupe Fusca, Morsitans et Palpalis
en Afrique (Leak, 1999).
15
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
En plus de G. p. pallicera, on retrouve aussi au
Cameroun G. p. palpalis, G. caligenea, G.
tabaniformis, G. nigrofusca, G. m. morsitans, G. m.
submorsitans, G. longipalpis, G. nigrofusca, G.
tachinoïdes, G. fuscipes, G. haningtoni, G.
fuscipleuris, G. nashi, G. newsteadi, G. brevipalpis
(Melachio et al., 2011 ; Eouzan et Ferrara , 1978 ; Rageau et
Adam, 1953 ; Pollock, 1982).
G. p. pallicera est une mouche des zones
forestières denses sempervirentes. Elle peut occuper les plantations de
café mais reste en général sous le couvert arboré
moins fréquenté par l'homme. Elle se nourrit essentiellement de
gros mammifères, bovidés, suidés et primates (Gouteux
et al., 1982). Les bovidés fournissent l'essentiel des repas
sanguins (60%) à cette espèce (Gouteux et al., 1982).
L'homme n'est pas un hôte négligé et représente
près de 10% des repas (Laveissière et al.,
1980).
I.9. Lutte contre les trypanosomoses
Deux stratégies alternatives sont
généralement utilisées pour lutter contre les
trypanosomoses africaines. La première stratégie est
principalement médicale ou vétérinaire et vise à
réduire la propagation du parasite par le traitement des
mammifères (Hommes et animaux) infectés; la seconde
stratégie est entomologique et met l'accent sur le contrôle de la
mouche tsétsé.
Le dépistage systématique par des équipes
mobiles permet de détecter les malades au stade précoce. Ce qui
permet de les traiter et d'éviter la progression de la maladie. Il
permet aussi de reduire le réservoir humain. Cependant, des animaux
domestiques et sauvages ont été identifiés comme des
réservoirs potentiels (Simo et al., 2000; Njiokou et al.,
2006, 2010) à partir desquels des trypanosomes peuvent être
réintroduits dans la population humaine. Gérer le
réservoir animal est difficile parce qu'il est constitué d'une
variété d'animaux domestiques (porcs, moutons, chèvres,
chiens...) et sauvages (singes, rats, crocodiles...). Dans un tel contexte,
contrôler le vecteur apparaît comme une méthode alternative
pour lutter contre le réservoir animal. De nombreuses techniques ont
été utilisées dans le but de réduire les
populations de glossines. Ces méthodes comprennent :
? L'abattage des animaux sauvages
Cette technique ancienne consistait à abattre les
animaux sauvages sur lesquels se nourrissaient les mouches tsétsé
(Hocking et al., 1963; Jordan, 1986). Dans l'île de Principe
16
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
par exemple, les porcs sauvages ont été
entièrement éliminés dans les années 1930, ce qui a
entrainé la disparition des mouches. Dans les années 1950, les
mouches ont ré-envahi cette île. Cependant, la nouvelle population
des glossines était indemne de la maladie. L'élimination des
animaux sauvages n'est pas facile à réaliser, surtout quand il
consiste à détruire les petits animaux comme le
phacochère, le potamochère et les petites antilopes qui sont
également des hôtes de nombreuses espèces de trypanosomes.
Ce procédé d'abattage des animaux sauvages avait
été abandonné, car il était moins efficace et
n'assurait pas la protection de la faune sauvage.
? Destruction de la végétation
La destruction de la végétation peut se faire
par usage des bulldozers, des feux de brousse, par défrichage partiel ou
total suivi quelque fois du remplacement de la végétation
naturelle par des cultures. Ces méthodes sont onéreuses,
nécessitent une main d'oeuvre abondante et présentent un
inconvénient majeur car elles perturbent l'équilibre de
l'environnement, augmente des quantités importantes de CO2 dans
l'atmosphère, ce qui conduit au réchauffement climatique. De
plus, elle favorise la mise en place des glossines péri-domestiques plus
dangereuses (Nash, 1940; Morris, 1946; Kernaghan et Davies, 1960; Maillot,
1966; Challier, 1968).
? Les campagnes de pulvérisations des
insecticides
Les insecticides ont été
utilisés pour lutter contre les mouches tsétsé durant le
XXe siècle. Cette technique a été étendue
après la seconde guerre mondiale avec des grandes campagnes
aériennes (aéronefs) et terrestres (pulvérisateurs)
basée sur utilisation des insecticides organiques comme le DDT
(Dichlorodiphényltrichloroéthane). Le problème majeur est
le risque de pollution. Ces insecticides n'étaient pas
spécifiques à une espèce et pouvaient donc affecter
d'autres arthropodes non-pathogènes. De plus les modalités de la
lutte chimique contre les glossines sont complexes et doivent tenir compte des
facteurs climatiques, du relief et de la végétation (Kirkby et
Blasdale, 1960; Macdonald, 1960 ; Mahood, 1960; Maclennan, 1963).
17
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
? Piégeage
L'usage des pièges ou des écrans occupe une
place importante dans les stratégies de lutte contre les glossines. Les
pièges et les écrans sont souvent imprégnés
d'insecticides. Les pièges se sont avérés plus efficaces
avec de nombreux modèles actuellement disponibles: le piège
Vavoua (Lavéissière et Grébaut, 1990), le piège
pyramidal (Gouteux et Lancien, 1986), et le piège biconique (Challier et
Lavéissière, 1973). Ces pièges et écrans sont
composés de couleurs attractives (bleu) et de couleurs sombres (noir).
L'usage des pièges exigent cependant une logistiqu e importante et un
personnel qualifié ayant une bonne connaissance de l'écologie des
mouches.
? Utilisation des mâles stériles
La technique du lâché des insectes
stériles (TIS) repose sur la libération de mâles
irradiés rendus ainsi stériles. Ces insectes en surnombre par
rapport aux mâles sauvages de l'espèce cible s'accouplent avec les
femelles sauvages, entraînant des accouplements infertiles, ce qui est
« catastrophique » pour la dynamique de leur population, qui
décline jusqu'à l'extinction si elle est isolée. Cette
technique a l'avantage de toucher exclusivement l'espèce ciblée.
La TIS s'applique aux glossines qui présentent une faible densité
des populations, un faible taux de reproduction, un accouplement en
général unique (Bouyer ,2006).
Le problème majeur dans cette technique réside sur
l'élevage de masse des glossines, la compétitivité des
mâles de laboratoire et l'effectif des individus à lâcher
(Lavéissière et al., 2000).
Ces méthodes de lutte anti-vectorielle ayant
présenté plusieurs limites, de nouvelles études se sont
axées dans le domaine de la biologie moléculaire et celles-ci a
permis le développement d'autres méthodes de lutte.
I.10. Génétique des populations
La génétique des populations
est un domaine de la biologie qui étudie la composition
génétique d'une population biologique (l'étude de la
distribution, de la fréquence allélique) (Postlethwalt, 2009) et
des modifications dans la composition génétique sous l'influence
de quatre processus d'évolution: la sélection naturelle, la
dérive génétique, les mutations et le flux des
gènes. Elle tient compte également des facteurs de recombinaison,
de la subdivision
18
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
de la population et de la structure de la population. Elle
essaye d'expliquer les phénomènes comme l'adaptation et la
spéciation.La génétique des populations s'est
développée dans les années 1920 et 1930 grâce au
travail de Fisher, Haldane et Wright (BS). Leur réalisation devait
intégrer les principes de la génétique Mendélienne
qui avait été redécouverte à la fin du
siècle avec la sélection naturelle darwinien.
I.10.1. Principe fondamental de la
génétique des populations
La génétique de population
concerne la constitution génétique de la population et
comment cette constitution change avec le temps. Une population
est un groupe localisé d'individus appartenant à la
même espèce. Par exemple, toutes les G. p. pallcera qui
vivent dans une forêt isolée représente une population. Un
gène simple dans cette population peut avoir plusieurs formes
alternatives, qui expliquent des variations entre les phénotypes des
organismes. Un patrimoine héréditaire est
l'ensemble complet d'allèles pour un gène dans une population
simple; la fréquence d'allèle mesure la fraction
du patrimoine héréditaire composé d'allèle simple.
L'évolution se produit quand il y a des changements des
fréquences des allèles dans une population d'organismes se
reproduisant. L'Allèle est l'état
héréditaire dans lequel un locus se présente. Le
déséquilibre de liaison est expression d'une association
non aléatoire entre les loci. Le flux des gènes
est toutes les formes de migration des gamètes, des individus
ou des groupes d'individus d'une sous-population à une autre. Cette
force s'oppose à la dérive génétique et favorise
l'homogénéisation de la population. Le gène
est une portion d'ADN qui code pour une information. Un
génotype est la composition allélique d'un individu
à un locus donné ou à une série de loci
spécifique. L'homozygote est l'état d'un locus
chez un individu diploïde présentant deux fois le même
allèle. L'hétérozygote est l'état
d'un locus chez un individu diploïde présentant deux allèles
différents. Un locus est l'emplacement d'un gène
sur un Chromosome. Ce terme est vulgairement utilisé pour
désigner une portion d'ADN soumise à une étude. Le
polymorphisme est le terme qui désigne l'existence de plusieurs
formes (allèles) à un locus donné. Une population
structurée est une population constituée de plusieurs
sous-populations entre lesquelles il existe très peu ou pas de flux de
gènes. La panmixie décrit un mode de
reproduction sexuée où les zygotes sont formés par
rencontre aléatoire de tous les gamètes de la population.
19
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
I.10.2. Facteurs affectant la génétique
des populations
Les facteurs qui peuvent provoquer un changement de la
composition génétique d'une population sont: la sélection
naturelle, les mutations, la dérive génétique et le flux
des gènes.
? Sélection naturelle
La sélection naturelle est un processus au cours duquel
la survie et/ou le succès reproducteur d'un individu dépend de
son phénotype ou de son génotype d'une manière plus ou
moins directe. Au cours des générations et grâce à
une combinaison de caractères avantageux hérités de leurs
parents, seuls les individus les mieux adaptés participeront à
l'établissement de la prochaine génération.
? Mutation
Les mutations sont des changements dans la séquence de
l'ADN du génome des cellules qui sont provoquées par les
rayonnements, les virus, les transposons, les produits chimiques
mutagéniques, les erreurs qui se produisent pendant la méiose ou
la réplication de l'ADN. Si des dommages se produisent naturellement
dans l'ADN, des mécanismes de réparation peuvent s'effectuer dans
les cellules. Néanmoins, la réparation ne renvoie parfois pas
à l'ADN original. Ces erreurs créent de grands changements
structuraux dans l'ordre de duplication de l'ADN, l'inversion ou la suppression
des régions entières, ou échange accidentel des parties
entières entre différents chromosomes (translocation).
? Dérive génétique
La dérive génétique est une force
évolutive qui traduit l'échantillonnage aléatoire des
allèles de la population parentale qui seront représentés
dans la descendance. Elle est directement liée à la taille finie
des populations naturelles. Plus les populations seront de taille restreinte,
plus l'influence de la dérive génétique sera importante.
Elle agit en faveur de l'augmentation de la différenciation entre
populations.
? Flux de gènes
Le flux génétique est l'échange des
gènes entre les populations de la même espèce. Il peut
également être défini comme le transfert des allèles
d'une population à l'autre. La migration dans ou hors d'une population
peut être responsable d'un changement marqué des fréquences
d'allèle. L'immigration peut avoir comme conséquence l'addition
de nouvelles variantes génétiques pour établir le
patrimoine héréditaire d'une espèce ou d'une population
particulière. Réciproquement, l'émigration peut enlever
des variantes génétiques. Le flux
20
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
génétique est entravé par des chaines de
montagnes, des océans, des déserts ou même des structures
synthétiques. Le flux maintenu de gène entre deux populations
peut mener à une combinaison de deux patrimoines
héréditaires, réduisant la variation
génétique entre les deux groupes. C'est pour cette raison que le
flux génétique agit fortement contre la spécialisation.
I.10.3. Les types de marqueurs moléculaires
utilisés dans la génétique des populations
Un marqueur moléculaire est un fragment d'ADN
(information génétique) ou son expression moléculaire
(ARN, protéines) permettant la caractérisation
génétique d'un individu ou d'une population. Ces marqueurs ont
plusieurs domaines d'utilisation comme les études d'apparentement, les
études de systématique (la définition des espèces
jumelles), les études phylogénétiques (relations entre
espèces) mais surtout les études de structure
génétique des populations (estimation des effets de la
dérive génétique, des flux de gènes) (Raymond,
1996). Il existe plusieurs catégories de marqueurs moléculaires:
la SSCP (Single-Stranded Conformational Polymorphism), l'AFLP (Amplied Fragment
Lenght Polymorphism), la SNP (Single-Nuleotide-Ploymorphism), les
microsatellites. Les microsatellites sont les marqueurs les plus
fréquemment utilisés dans les études sur la
caractérisation génétique des espèces de parasites
ainsi que leur vecteur (Sunnucks, 2001). Les allozymes et les marqueurs
mitochondriaux sont aussi très utilisés dans la
génétique des populations.
? Allozymes
Les allozymes sont en fait des enzymes du métabolisme
de base des cellules. Les allozymes sont moins polymorphes, notamment chez les
organismes parasites, et requièrent de travailler sur du matériel
frais, ce qui s'avère souvent difficile, en particulier dans les pays du
Sud. Le matériel biologique à utiliser doit se trouver en
quantité suffisante, ce qui est souvent difficile pour les organismes
parasites, souvent de taille modeste et difficilement cultivable.
? Marqueurs mitochondriaux
Les marqueurs cytoplasmiques correspondent à des loci
présents dans le génome mitochondrial. L'ADN mitochondrial s'est
montré extrêmement informatif dans des études
phytogéographiques, car il présente des taux d'évolution
relativement rapides et ne subit pas de recombinaisons entre loci (Avise et
al., 1987 ; Avise, 2000). Cependant, pour les études de
génétique des populations, les propriétés de ces
marqueurs sont loin d'être idéales car l'ADN
21
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
mitochondrial présente une
hérédité uniparentale, typiquement maternelle. Ces
marqueurs sont haploïdes et ne peuvent par conséquent pas
renseigner clairement sur le régime de reproduction locale de
l'espèce étudiée (De Meeûs, 2012).
? Marqueurs microsatellites
Ce sont des séquences d'ADN formées par des
répétitions continues (en tandem) de motifs (2, 3 etc.)
composés de 1 à 6 nucléotides. Les microsatellites ont une
taille relativement petite. L'existence des microsatellites dans les
génomes est connue depuis plusieurs décennies mais ils ont
été longtemps considérés comme des séquences
de peu d'intérêt. Ce n'est qu'à la fin des années
1980 qu'on a réalisé qu'il s'agissait probablement d'un des plus
puissants marqueurs mendéliens qu'on ait mis en évidence
jusqu'à présent. Leur fonction biologique reste floue (Epplen
et al., 1993). Certaines études montrent que les variations de
longueur de certains microsatellites ont des effets phénotypiques sur la
physiologie et le développement de certains organismes (King et Soller,
1998).
Les avantages importants des loci microsatellites comme
marqueurs pour les études de génétique de populations sont
les suivants:
- les loci microsatellites ont des taux de mutations et de
polymorphismes (10-3, 10-4) très
élevés dans les populations naturelles (Ellegren, 2000 ; Balloux
et Lugon-Moulin, 2002 ; Ellegren, 2004)
- ils sont relativement denses dans les génomes ;
- sont codominants et relativement aisés à
manipuler ; - ont des séquences relativement courtes ;
- sont amplifiables par PCR à partir des tissus
conservés dans l'alcool pendant une durée assez longue et dans
n'importe quelle condition (De Meeus, 2012).
Grâce à leur ubiquité, leur abondance dans
le génome et leur fort taux de mutation qui leur confère un
niveau de polymorphisme élevé, les microsatellites sont
couramment utilisés dans de nombreux domaines d'études comme
- la génétique des populations (Bossart et
Pashley Prowell, 1998), la cartographie du génome (Bell et Ecker, 1994;
Zheng et al., 1996), l'analyse des traits quantitatifs (Gorman et
al., 1996),
22
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
- la génétique humaine (Sutherlands et Richards,
1995), de plus en plus dans la phylogénie et l'histoire de
l'humanité (Bowcock et al., 1994; Goldstein et al.,
1995).
Apport de la génétique des populations
dans la lutte anti-vectorielle
Avec le développement des outils de la
génétique moléculaire, une nouvelle page s'est ouverte
dans la lutte anti-vectorielle. Aujourd'hui, deux approches sont envisageables
dans la lutte anti-vectorielle: la suppression et l'éradication. Le
choix d'une des approches va dépendre de l'isolement ou non des
populations de glossines. La suppression désigne la baisse des
densités de glossines en deçà d'un seuil de transmission
et qui est adoptée lorsque la population de glossines concernées
n'est pas génétiquement isolée des populations voisines.
L'éradication quant à elle désigne la destruction
complète de la population de glossines concernées. Elle est
adoptée lorsque cette population est génétiquement
isolée des autres (Solano et al., 2010a, 2010b). Pour faire le
choix entre suppression et éradication, il est obligatoire
d'entreprendre des études sur la génétique des populations
de glossines.
23
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
II. MATERIEL ET
METHODES
II.1. Site d'étude
Le foyer de la maladie du sommeil de Campo (2 O 20'N, 9 O
52'E) est situé à 80 Km de la ville de Kribi dans la
région du Sud Cameroun (figure 8). C'est un foyer hypo-endémique
de THA où aucune évolution épidémique de la maladie
n'y a été signalée depuis plusieurs années
(Melachio et al., 2011) . Le foyer de la THA de Campo se trouve le
long de la côte Atlantique et s'étend le long du fleuve Ntem qui
constitue la frontière entre le Cameroun et la Guinée
Equatoriale. Le climat est de type équatorial avec quatre saisons. La
végétation est constituée d'une forêt dense
équatoriale moins dégradé avec des terres agricoles, des
zones marécageuses et des marais le long de la côte (Simo et
al., 2014). Il s'agit d'une zone de forêt équatoriale avec un
réseau hydrographique dense, des zones marécageuses et des
mangroves. Cette zone est riche en faune sauvage et fait partie du parc
national de ?Campo Ma'an? (Njiokou et al., 2004a). L'agriculture, la
pêche et la chasse sont les principales activités menées
dans cette localité.

Figure 8 : Carte du Foyer de la THA de Campo
(Institut National de Cartographie Yaoundé Cameroun,
1976).
24
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
II.2. Echantillonnage
Les glossines utilisées dans le cadre de cette
étude ont été capturées dans deux villages de Campo
(Akak, campo Beach) et deux villages limitrophes de la Guinée
Equatoriale (Edjabé et Rio campo) du 29 mars au 12 avril 2012 en
utilisant des pièges pyramidaux. Quelques glossines provenant de Fontem
(Région du Sud-Ouest, Cameroun) et Dodéo (Région de
l'Adamaoua, Cameroun) ont été utilisées pour des
comparaisons génétiques. Lors de la prospection entomologique,
des pièges pyramidaux (Gouteux et Lancien, 1986) ont été
installés dans des biotopes (les points d'eau, les puits et les points
ombragés, les bords des routes ou des pistes) favorables aux mouches
tsétsé. Les pièges étaient installés pendant
quatre jours consécutifs et les glossines étaient
collectées une fois par jour.
Une fois les mouches tsétsé collectées,
chacune d'elles a subi une identification morphologique pour déterminer
l'espèce et le sexe. Après cette identification, les pattes de
chaque glossine ont été enlevées et introduites dans des
microtubes contenant de l'alcool à 95° et la carasse
conservée dans de l'éthanol 95° pour d'autres études
ultérieures. Les microtubes ont été conservés sur
le terrain à la température ambiante. Une fois au laboratoire,
ces microtubes ont été conservés à -20°C
jusqu'à l'extraction d'ADN.
II.3. Extraction de l'ADN
L'ADN a été extrait de 6 pattes de glossines en
utilisant le CTAB 5% (Cetyl Trimethyl Ammonium Bromide) selon la méthode
décrite par Navajas et al. (1998). Brièvement, les tubes
contenant les pattes des glossines ont été sorties du
congélateur, puis laissés ouverts sur la paillasse toute la nuit
dans le but de faire évaporer l'alcool contenu dans les tubes.
Après l'évaporation complète de l'éthanol, 600 ul
de la préparation du CTAB (composition en annexe 1) ont
été ajoutés, puis les pattes ont été
broyées à l'aide des pistons. Le mélange obtenu a
été incubé pendant une heure au bain marie. Ensuite, 600
ul d'un mélange de chloroforme et d'alcool isoamylique (24/1, v/v) ont
été ajoutés dans chaque tube. Après une
homogénéisation lente pendant 10 minutes, l'ensemble a
été centrifugé à 8000 trs/min pendant 10 minutes.
La phase supérieure (phase aqueuse) contenant les acides
nucléiques a été transférée dans un nouveau
tube où 430 ul d'isopropanol ont été ajoutés afin
de précipiter les acides nucléiques. L'ensemble a
été homogénéisé puis incubé à
-20°C pendant une heure pour faire précipiter l'ADN. Une fois cette
incubation terminée, les tubes ont été centrifugés
à 13000 trs/min pendant 10 minutes et le surnageant a été
vidé en conservant le culot d'acides
25
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
nucléiques (extrait d'ADN). Ce culot a
été lavé avec 900 ul d'éthanol 70°C et
centrifugé à 13000trs/min pendant 10 minutes. L'éthanol a
été entièrement vidé et les tubes contenant le
culot d'acides nucléiques ont été laissés ouverts
sur la paillasse pendant deux heures afin d'éliminer alcool
résiduel. Le culot a été suspendu dans 50 ul d'eau et
conservé à -20 °C pour les analyses moléculaires
ultérieures.
II.4. Analyses moléculaires II.4.1. Principe de la
PCR
La PCR est une technique moléculaire permettant
d'amplifier in vitro une région spécifique d'un acide
nucléique donné afin d'en obtenir une quantité
exponentielle de copies, suffisante pour être détectée et
étudiée. Au cours des réactions PCR, les produits obtenus
à la fin de chaque cycle servent de matrice pour le cycle suivant.
Chaque cycle comprend trois étapes:
? La dénaturation des doubles brins d'ADN à
température élevée (90-98°C).
? L'hybridation des amorces qui se fait à une
température définie selon la nature des amorces (cette
température varie généralement de 40 à 65°C).
Au cours de cette étape, les amorces qui sont des
oligonucléotidiques se lient, par appariement des bases
complémentaires au moyen des liaisons hydrogènes, à la
séquence cible à copier.
? L'élongation à 72°C, qui est la
température d'activité optimale de la Taq ADN polymérase.
Au cours de cette étape, les brins complémentaires d'ADN sont
synthétisés à partir des extrémités 3'OH
libre des amorces hybridées (Borde, 2006). La figure 9 ci-dessous
récapitule les différentes étapes de la PCR.
26
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris

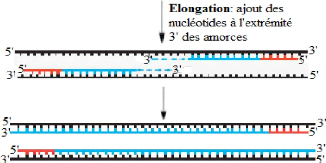
Figure 9: Différentes étapes de la PCR
(Jackson et al., 2006)
II.4.2. Identification moléculaire de G. p.
pallicera
Pour confirmer l'appartenance taxonomique issue des
critères morphologiques, nous avons amplifié l'ITS1 (internal
transcribed spacer 1) de tous les échantillons qui ont
été retenus pour cette étude. En effet, des amorces
consensus ont été mises au point et permettent d'amplifier l'ITS1
de toutes les espèces et sous espèces de mouches
tsétsé. Une fois que l'ITS1 est amplifié, l'identification
des différentes espèces se fait à partir du polymorphisme
de longueur qui s'observe entre les différentes espèces de
glossines.
L'identification moléculaire de G. p. pallicera
a été faite suivant la méthode décrite par
Dyer et al. (2008) en utilisant le couple d'amorces Diag forward
(5'TGGACTTCGGATTAAGTACAACA-3') et Diag
reverse
27
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
(5'TCATTATGCGCTATTAAGGTAAGC-3'). Les réactions
d'amplification ont été faites dans un milieu réactionnel
de 25ul contenant 2,5ul de tampon PCR 10X (10 mM Tris-HCl ; 50 mM KCl; 1,5 mM
de MgCl2), 10 pmoles de chaque amorce, 200 uM de dNTPs; 5U de Taq ADN
polymérase et 2,5 ul d'extrait d'ADN dilué au 10eme.
Les amplifications ont été réalisées dans un
thermocycleur de marque TECHNE selon le programme suivant : une étape de
dénaturation initiale à 950C pendant 3 minutes, suivie
de 30 cycles d'amplification comprenant chacun une étape de
dénaturation à 950C pendant 30 secondes, une
étape d'hybridation des amorces à 560C pendant 30
secondes, et une étape d'élongation à 720C
pendant 1 minute. A la fin, une extension finale a été
réalisée à 720C pendant 5 minutes. Les produits
d'amplification ont été séparés sur gel d'agarose
à 2% en vue de distinguer différentes espèces de
glossines.
II.4.2.1. Amplification des loci microsatellites des
glossines
Pour cette étude, nous avons analysé neuf loci
microsatellites ; B3, B104, C102, B110 et A10 de ROBINSON (FAO/IAEA), Gpg55.3
de Solano et al. (1997), Pgp13 et Pgp24 de Luna et al. (2001)
et GpCAG133 de Baker et Krafsur (2001). Chaque locus microsatellite a
été amplifié en utilisant les amorces dont les
séquences sont contenues dans le tableau 1. Les réactions
d'amplifications ont été faites dans un milieu réactionnel
de 25ul contenant 2,5ul de tampon PCR 10X (10 mM Tris-HCl ; 50 mM KCl; 1,5 mM
de MgCl2), 10 pmoles de chaque amorce, 200 uM de dNTPs, 5U de Taq ADN
polymérase et 5 ul d'extrait d'ADN. Le programme d'amplification
contenait une étape de dénaturation initiale à
950C pendant 5 minutes et 40 cycles d'amplification comprenant
chacun une étape de dénaturation à 950C pendant
30 secondes, une étape d'hybridation des amorces pendant 30 secondes
à des températures variables (voir tableau 1) en fonction du
couple d'amorces utilisées, une étape d'élongation
à 720C pendant 1minute. Une extension finale a
été réalisée à 720C pendant 10
minutes.
28
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
Tableau 1: Séquences et températures
d'hybridation des amorces utilisées pour le
|
génotypage
|
|
|
|
|
Caractéristique
Marqueurs
|
Séquence de l'amorce
5' 3'
|
Motif du
microsatellite
|
Température d'hybridation
|
Références
|
|
55.3
|
F:5'GTACTCAACGTGGTGCTTAAAGTTG3' R: 5'
GTCTGAGATAGGACCATTTATCG3'
|
(GT)GC(GT)
|
51,0
|
Robinson (FAO/IAEA)
|
|
Pgp13
|
F:5'AAAATAACGAACGGGTGCAG3'
R: 5' GACGCACGCACATAGTTACG3'
|
(GT)TT(GT)
|
54,5
|
Luna et al.,
2001
|
|
Pgp24
|
F:5'AATTCCCGCCGGTAAAGTT3'
R: 5' TGTTTGCGTATGCATCTGTG3'
|
(TA)CA(TA) T (GT)
|
53,5
|
Luna et al.,
2001
|
|
C102
|
F:5'CGTTAAATTGTCACGATGAAG3' R:5'AGCACATTTTTGGTACATTTATACC3'
|
(TGA)
|
52,0
|
Robinson (FAO/IAEA)
|
|
GpCAG133
|
F:5'ATTTTTGCGTCAACGTGA 3'
R: 5' ATGAGGATGTTGTCCAGTTT 3'
|
(CAG)
|
50,0
|
Baker et
Krafsur
(2000)
|
|
B104
|
F:5'ACGATTGTTCTTCTTCATTCC3' R:5'AAGAAGCCAAACTTTCAACGCTCGC3'
|
(GA)
|
52,0
|
Robinson (FAO/IAEA)
|
|
B3
|
F:5'TTCGCTTTTGTCAGAAGTG3'
R: 5' TCCCAGACGGTTXATATTAC3'
|
(GA)
|
56,0
|
Robinson (FAO/IAEA)
|
|
B110
|
F:5'ATAGATCGGCTAAATCCATGTAG3'
R:
5'TTTATTGCTTGTAGTGGTGAGAG3'
|
(GA)
|
51,5
|
Robinson (FAO/IAEA)
|
|
A10
|
|
|
56,0
|
Robinson (FAO/IAEA)
|
II.4.2.2. Séparation des produits
amplifiés par électrophorèse sur gel d'agarose
Principe de la technique : Elle est
basée sur la séparation des acides nucléiques
chargés négativement sous l'effet d'un champ électrique.
Cette séparation s'effectue à travers une matrice (agarose dans
ce cas): les molécules de petites tailles se déplaceront plus
rapidement et migreront plus loin que les molécules de tailles
supérieures.
29
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
La séparation des produits amplifiés sur gel
d'agarose a été faite dans le but de vérifier si les
réactions d'amplifications étaient bonnes. Pour cela, nous avons
préparé un gel d'agarose à 2% par un mélange de 2g
d'agarose et 100 ml de tampon (Tris-Borate-EDTA) TBE 0,5X (annexe 1). Le
mélange a été porté à ébullition dans
un four à micro-ondes pendant 3 minutes. Après refroidissement,
3ìl de Bromure d'Ethidium (BET) ont été ajoutés et
l'ensemble a été versé dans un moule. A refroidissement et
solidification du gel, le peigne a été enlevé. Par la
suite, 6ul des produits d'amplification ont été
mélangés à 2ul de bleu de charge. Le mélange a
été déposé dans les puits du gel et la
séparation a été faite à 100 volts pendant 30
minutes dans le tampon TBE 0,5X (Tris-Borate-EDTA). La révélation
a été faite par observation du gel sur un trans-illuminateur
à UV. Les échantillons ayant présenté des bandes
d'ADN ont été sélectionnés dans le but de
déterminer le nombre et la taille des allèles sur un gel de
polyacrylamide.
II.4.2.3. Séparation des allèles sur gel
de polyacrylamide
Les amplicons ou produits de la PCR ont été
séparés sur gel d'acrylamide afin d'observer le polymorphisme
génétique qui se reflète par le polymorphisme de longueur
des séquences microsatellites amplifiées. A cet effet, la
résolution des produits d'amplification s'est faite sur gel de
polyacrylamide à 10% (seuls les échantillons qui ont
présenté une bande sur gel d'agarose ont été
sélectionnés). Pour ce faire, le gel de polyacrylamide a
été préparé en mélangeant : 18,33 ml
d'acrylamide/bis acrylamide (19:1) à 30%, 5,5 ml de Tris Borate EDTA
(TBE) 10X, 30,62 ml d'eau distillée, 481,25 ul d'APS (10%) et 68,75 ul
de TEMED. Le mélange a été introduit dans le moule
vertical d'électrophorèse avec un peigne pour former les puits.
Après polymérisation du gel (environ 30 minutes), le peigne a
été enlevé et les puits ont été lavés
avec le tampon de migration Tris Borate EDTA (TBE) 1X. Par la suite, 19ul de
chaque produit d'amplification ont été mélangés
à 3ul de bleu de charge. Les mélanges ont été
introduits dans les puits du gel préalablement nettoyés. A
côté des échantillons, 7ul de marqueur de poids
moléculaire ont été introduits dans un autre puits pour
l'estimation de la taille des allèles. La migration s'est faite dans du
tampon TBE 1X à 150 Volts pendant environ 17 heures. A la fin de la
migration, le gel a été coloré dans un bain de bromure
d'éthidium (30 ul de bromure d'éthidium pour environ 600 ml de
tampon TBE 1X). L'ensemble a été incubé pendant 20 minutes
à l'abri de la lumière. Par la suite, le gel a été
lavé à l'eau afin d'éliminer l'excès de bromure
d'éthidium. Le gel a été enfin visualisé sous UV,
puis photographié.
30
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
Etant donné que les mouches tsé-tsé sont
des organismes diploïdes, on s'attend après résolution des
produits d'amplification à deux bandes (2 allèles) pour un
individu hétérozygote pour le locus analysé et une bande
pour un individu homozygote. A la fin de chaque électrophorèse,
la taille de chaque allèle a été estimée à
partir des marqueurs de poids moléculaire (PM) en traçant la
courbe du logarithme décimal du poids moléculaire en fonction de
la distance de migration (D) (log PM =f (D)).
II.4.3. Analyse génétique des
données
Après avoir déterminé la taille des
allèles, une base de données contenant pour chaque individu son
génotype multilocus (génotype à différents loci) a
été confectionnée à partir du logiciel
Excel®. A partir de cette base de données, le logiciel
CREATE version 1.0 a permis de créer des fichiers en formats
appropriés pour les autres logiciels d'analyses FSTAT, GENETIX, GENEPOP,
MSA et MICRO CHECKER.
L'hétérozygotie observée (Ho) et celle
attendue (Hs) ont été calculées pour chaque locus sous
l'hypothèse de l'équilibre de Hardy-Weinberg. Chez les
mâles et pour les loci liés au chromosome X (55.3, PGP13), les
tailles des allèles ont été codées en
données manquantes par les chiffres zéro (000/000). Ceci est
dû au fait que ces mâles sont tous des « faux »
homozygotes et contribuent à un déficit en
hétérozygotes.
La structure génétique des populations de
glossines a été étudiée à l'aide des
estimateurs non biaisés de Weir et Cockerham (1986) (f et O)
des statistiques F (FIS et FST) de Wright (Wright,
1965). Le FIS ou coefficient de consanguinité mesure
l'écart à la panmixie (croisements au hasard entre individus)
dans une sous-population. Il exprime la variabilité
génétique au sein des individus par rapport à la
sous-population et permet d'expliquer l'excès ou le déficit
d'hétérozygotes à l'intérieur de chaque
sous-population. Ses valeurs varient de -1 (excès en
hétérozygotes) à 1 (déficit en
hétérozygotes). Le FST (ou indice de fixation) mesure la
différenciation génétique entre deux ou plusieurs
sous-populations. Il mesure, sur la base des différences de
fréquences alléliques entre les sous-populations, le
déficit en hétérozygotes issu de la structuration en
sous-populations. Sa valeur est comprise entre 0 (pas de structuration) et 1
(toutes les sous-populations sont fixées pour un allèle au
moins).
Les statistiques F de Wright ont été
estimées à l'aide du logiciel FSTAT version 2.9.4 (Goudet, 2003).
Etant donné que f mesure l'écart des fréquences
génotypiques par rapport à celles attendues dans une
sous-population sous équilibre de Hardy-Weinberg, la signification
31
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
de cet écart a été testée en
randomisant 10000 fois les allèles entre les individus au sein des
différentes sous-populations et à chaque fois, f
était calculé (procédure automatique dans le logiciel
Fstat). La P-value de ce type de test est la proportion du nombre de
fois que la valeur de f estimée dans les réplicas
formés par randomisations est supérieure ou égale à
la valeur réelle de f obtenue (ou inférieure ou
égale lorsque la valeur réelle est négative). Une
procédure analogue a été utilisée dans le logiciel
Fstat pour tester la significativité de la valeur de O. Le
polymorphisme au niveau de chaque marqueur a été obtenu en
comptant le nombre d'allèles différents mis en évidence
par le marqueur considéré, ceci grâce au logiciel FSTAT.
Les fréquences alléliques et
l'hétérozygotie ont été respectivement
déterminés par les logiciels FSTAT version 2.9.4 et GENETIX
version 4.0.5 Une courbe de régression linéaire de FIS
en fonction du nombre d'allèle nul a été construite afin
de savoir si les allèles nuls étaient à l'origine du
déficit en hétérozygote. Le logiciel Micro-checker version
2.2.3. (Van Oosterhout et al., 2004) a été
utilisé dans le but de savoir si les allèles nuls expliquent la
totalité du déficit en hétérozygote. Ceci a
été évalué grâce au coefficient de
corrélation (R) qui varie de -1 à 1. Si R est proche de -1 ou 1,
il existe une forte corrélation et si R est éloigné de -1
ou 1, il existe une corrélation faible.
Pour apprécier le polymorphisme
génétique, c'est-à-dire les distances
génétiques entre les glossines analysées, le logiciel MEGA
3.1 (Kumar et al., 2004) a été utilisé pour la
construction d'un dendrogramme en utilisant des matrices de distances
harmoniques de Cavalli-Sforza et Edwards (1967). Ces matrices ont
été générées à l'aide du logiciel MSA
(Dieringer et Schlotterer., 2003).
Le logiciel XLSTAT 2015 a permis de comparer le niveau de
sensibilité entre les différents marqueurs microsatellites.
32
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
III. RESULTATS
III.1. PCR diagnostic des espèces de
glossines
Pour cette étude, 72 glossines ont été
analysées dont 45 G. p. pallicera ,17 G. p. palpalis,
4 G. submorsitans, 3 G. caligenea, 2 G. nigrofusca
et une G. fuscipes. La figure 10 montre un exemple de gel obtenu
à la suite de la séparation des produits d'amplifications des
ITS1 de différentes espèces de glossines. Cette figure montre une
différence de taille entre les ITS1 de G. nigrofusca (254
Paires de bases) et G. caligenea (179 Pbs). Une différence a
été aussi observée entre G. p. pallicera (266
Pbs) et G. p. palpalis (241 Pbs). Aucune différence n'a
été observée entre la taille d'ITS1 de G. submorsitans
et G. p. pallicera, ainsi qu'entre G. fuscipes et G.
p. palpalis.

254*
179*
179* 241*
241*
266*
266*
266*
266 *
1000 Pbs
500 Pbs 300 Pbs
200 Pbs
Figure 10: Photo d'un gel d'agarose montrant les
produits d'amplification des ITS1 de différentes espèces de
glossines.
* : pbs (Paires de bases) ; M =
Marqueurs de poids moléculaires (100pbs) ; 1 et 2 =
G. nigrofusca ; 3 et 4 = G. caligenea
; 5 = G. fuscipes ; 6=
G. p. palpalis ; 7 et 8 =
G. p. pallicera ; 9 et 10 =
G. submorsitans
III.2. Sensibilité des différents
marqueurs microsatellites
Lors de l'amplification des loci microsatellites sur les 45
échantillons de G. p. pallicera, les 9 marqueurs n'ont pas
amplifié avec la même efficacité. Les marqueurs 55.3,
GPCAG133, PGP13, PGP24 et C102 ont amplifié avec une sensibilité
très variable. Le tableau 2 présente, en fonction des
espèces, le nombre de glossines amplifiées à chaque locus
microsatellite. Pour les marqueurs A10 et B110, aucune amplification n'a
été observée pour les 72 glossines analysées ;
raison pour laquelle ces deux marqueurs ne figurent pas dans le
33
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
tableau 2. Les marqueurs B3 et B104 ont amplifié moins
de 50% (34 glossines pour B3 et 31 pour B104) des 72 glossines
analysées. Ces deux marqueurs n'ont pas été
considérés dans les analyses de génétiques des
populations. C102 est le marqueur ayant amplifié le plus grand nombre de
glossines (70), suivi de PGP13 (69 glossines amplifiées), CPCAG, 55.3 et
PGP24.
Tableau 2: Nombre de glossines amplifiées en
fonction des espèces et des loci
|
microsatellites.
|
|
|
|
|
|
|
|
|
Espèce
|
Total
|
PGP13 C102
|
55.3
|
GPCAG PGP24
|
B3
|
B104
|
|
G. caligenea
|
3
|
3
|
3
|
3
|
3
|
3
|
0
|
1
|
|
G. fuscipes
|
1
|
1
|
1
|
1
|
1
|
1
|
0
|
1
|
|
G. nigrofusca
|
2
|
1
|
2
|
0
|
1
|
2
|
0
|
2
|
|
G .p. pallicera
|
45
|
44
|
43
|
41
|
44
|
38
|
20
|
13
|
|
G. p. palpalis
|
17
|
16
|
17
|
16
|
15
|
13
|
13
|
13
|
|
G. m. submorsitans
|
4
|
4
|
4
|
3
|
4
|
3
|
1
|
1
|
|
TOTAL
|
72
|
69
|
70
|
64
|
68
|
60
|
34
|
31
|
Pour comparer les taux d'amplification obtenus à chaque
locus, nous avons considéré uniquement les espèces G.
p. pallicera et G. p. palpalis pour lesquelles plus de 10
glossines ont été analysées. La taille de
l'échantillonnage des autres espèces étant très
faible notamment une G. fuscipes, deux G. nigrofusca, trois
G. caligenea et quatre G. submorsitans. En comparant les taux
d'amplification des glossines à chaque locus, une différence
significative (P= 0,024) a été observée entre G. p.
palpalis et G. p. pallicera pour le locus B3.
Concernant le marqueur B104, une différence
significative (P= 0,001) a été observée entre les nombres
de G. p. palpalis et G. p. pallicera amplifiés.
Par contre, pour les marqueurs PGP13, C102, 55.3, GPCAG et PGP24, aucune
différence significative n'a été observée entre les
nombres de G. p. palpalis et G. p. pallicera amplifiés
(tableau 3). Pour ces comparaisons, le seuil de significativité
était égal à 0,05.
34
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par
GOMSEU DJOUMSIE Emmanuel Boris
Tableau 3: Nombre et pourcentage de G. p. pallicera
et G. p. palpalis amplifiés à chaque locus
microsatellite
|
Total
|
PGP13
|
C102
|
55.3
|
GPCAG
|
PGP24
|
B3
|
B104
|
|
G. p. pallicera
|
45
|
44
|
43
|
41
|
44
|
38
|
20
|
13
|
|
G. p. palpalis
|
17
|
16
|
17
|
13
|
15
|
13
|
13
|
13
|
|
P-value
|
/
|
0,466
|
0,054
|
0,125
|
0,118
|
0,463
|
0,024*
|
0,001*
|
* : différence statistiquement significative
III. 3. Polymorphisme des marqueurs utilisés
Pour les analyses portant sur le polymorphisme
génétique, nous avons considéré uniquement les
marqueurs ayant amplifié au moins 80% des glossines analysées.
Sur cette base, seulement 5 marqueurs ont été
considérés : GPCAG, 55.3, C102, PGP13 et PGP24.
La figure 11 montre un exemple de photo illustrant la
séparation, sur gel d'agarose, des produits d'amplifications obtenus au
locus 55.3. Quel que soit le locus analysé, aucune différence de
taille n'a été détectable à la suite de la
séparation des produits d'amplifications sur gel d'agarose. Par
ailleurs, une seule bande d'ADN illustrant l'amplification du locus
microsatellite a été obtenue sur gel d'agarose pour chaque
glossine analysée. Les allèles ayant des tailles très
proches, ne peuvent être bien séparés sur gel d'agarose ;
raison pour laquelle nous avons fait recours au gel de polyacrylamide.

Figure 11: Exemple de photo montrant les profils des
bandes obtenues sur gel d'agarose et illustrant les amplicons issus de
l'amplification du locus 55.3.
C- = Contrôle négatif ; 1= G. caligenea ;
2= G. fuscipes ; 3, 13-15= G. p. palpalis ; 4-12= G. p. pallicera
35
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
La figure 12 présente un exemple de profils de bandes
obtenues après résolution sur gel de polyacrylamide des produits
d'amplification pour les loci C102 et PGP24.
Homozygotes Hétérozygotes

263* 263* 280* 280* 289* 289* 266*
300* 295* 289*
275*
250*
225*
200*
204*
208*
248* 257* 257* 257* 260* 266* 257* 263* 257*
257* 257* 257* 263*
175*
186* 188*
Figure 12: Photo d'un gel de polyacrylamide montrant
les profils des allèles obtenus pour chaque glossine et aux loci C102 et
PGP24.
* : paire de base ; 1, 2 et 11 =
G. p. palpalis ; 2-9 et 17 = G. p.
pallicera ; 12-15 = G. submorsitans.
C102 ; 16= G. caligenea ; 10 =
marqueur de taille 25pbs. Le locus C102
(échantillon 1-15), le locus PGP24
(échantillon 16 et 17).
Le degré de polymorphisme des marqueurs microsatellites
varie d'un locus à l'autre. La taille des allèles varie de 151
à 247 paires de bases pour le marqueur GPCAG133, de 165 à 193 pb
pour 55.3, de 154 à 210 pour PGP13 et de 242 à 296 paires de
bases pour C102.
Le tableau 4 donne, en fonction du sexe, du village, des
espèces et des loci, la taille des allèles obtenus pour chaque
glossine analysée.
36
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
Tableau 4 : Génotypes multilocus obtenus pour
chaque glossine analysée et pour les cinq
|
loci considérés.
|
|
|
|
|
|
|
|
/
|
/
|
/
|
/
|
Génotypes à chaque locus
|
|
|
|
Ind
|
Espèce
|
Village
|
Sexe
|
55.3
|
GPCAG
|
PGP13
|
C102
|
PGP24
|
|
1
|
Gnigr
|
Akak
|
F
|
000/000
|
000/000
|
000/000
|
245/245
|
249/249
|
|
2
|
Gpal
|
Akak
|
F
|
167/167
|
217/217
|
180/166
|
254/242
|
241/227
|
|
3
|
Gpal
|
Akak
|
M
|
167/167
|
220/214
|
166/166
|
260/260
|
241/227
|
|
4
|
Gpal
|
Akak
|
F
|
167/167
|
214/214
|
180/166
|
251/251
|
241/227
|
|
5
|
Gpal
|
Akak
|
M
|
167/167
|
214/211
|
166/166
|
251/251
|
241/227
|
|
6
|
Gpal
|
Akak
|
F
|
167/167
|
220/220
|
180/166
|
254/245
|
241/227
|
|
7
|
Gpal
|
Akak
|
M
|
167/167
|
217/211
|
166/166
|
257/257
|
241/227
|
|
8
|
Gnigr
|
CampoBeach F
|
000/000
|
151/151
|
178/166
|
260/260
|
205/205
|
|
9
|
Gpal
|
Memel
|
M
|
000/000
|
220/214
|
166/166
|
251/251
|
207/201
|
|
10
|
Gcal
|
Campo
|
F
|
167/167
|
202/202
|
186/172
|
263/263
|
261/245
|
|
11
|
Gcal
|
Campo
|
F
|
167/167
|
205/220
|
188/174
|
260/260
|
263/247
|
|
12
|
Gcal
|
Akak
|
F
|
181/167
|
202/202
|
184/176
|
260/260
|
265/251
|
|
13
|
Gpal
|
Campo
|
M
|
167/167
|
202/199
|
166/166
|
260/251
|
265/251
|
|
14
|
Gfus
|
Memel
|
F
|
181/181
|
196/220
|
184/184
|
251/251
|
241/227
|
|
15
|
Gpp
|
Campo
|
F
|
000/000
|
000/000
|
000/000
|
260/260
|
000/000
|
|
16
|
Gpal
|
Memel
|
F
|
167/167
|
202/202
|
000/000
|
266/266
|
000/000
|
|
17
|
Gpal
|
Campo
|
M
|
167/167
|
214/214
|
166/166
|
269/269
|
241/227
|
|
18
|
Gpp
|
Campo
|
F
|
181/171
|
202/202
|
170/154
|
263/251
|
243/243
|
|
19
|
Gpp
|
Campo
|
M
|
177/177
|
211/211
|
192/192
|
263/257
|
209/205
|
|
20
|
Gpal
|
Campo
|
M
|
167/167
|
217/217
|
166/166
|
260/260
|
241/227
|
|
21
|
Gpal
|
Campo
|
F
|
181/167
|
214/214
|
204/190
|
260/260
|
241/227
|
|
22
|
Gpal
|
Campo
|
F
|
181/167
|
238/214
|
204/190
|
254/242
|
241/227
|
|
23
|
Gpal
|
Campo
|
M
|
167/167
|
217/217
|
166/166
|
254/242
|
241/227
|
|
24
|
Gpp
|
Campo
|
F
|
193/181
|
211/211
|
210/198
|
257/257
|
207/193
|
|
25
|
Gpal
|
Akak
|
F
|
000/000
|
220/220
|
180/166
|
000/000
|
000/000
|
|
26
|
Gpal
|
Akak
|
M
|
000/000
|
220/214
|
166/166
|
000/000
|
000/000
|
|
27
|
Gpal
|
Akak
|
M
|
167/167
|
214/214
|
166/166
|
260/260
|
241/227
|
|
28
|
Gpal
|
Akak
|
M
|
167/167
|
214/214
|
166/166
|
260/260
|
241/227
|
|
29
|
Gpal
|
Akak
|
F
|
181/167
|
214/205
|
180/166
|
260/260
|
241/227
|
|
30
|
Gpal
|
Akak
|
F
|
181/167
|
217/217
|
180/166
|
260/260
|
241/227
|
|
31
|
Gpal
|
Akak
|
M
|
167/167
|
217/217
|
166/166
|
260/260
|
241/227
|
|
32
|
Gpal
|
Akak
|
F
|
181/167
|
217/217
|
180/166
|
260/260
|
243/229
|
|
33
|
Gpal
|
Akak
|
M
|
000/000
|
217/217
|
166/166
|
257/257
|
241/227
|
|
34
|
Gpal
|
Akak
|
F
|
181/167
|
217/217
|
180/166
|
260/260
|
241/227
|
37
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
|
35
|
Gpal
|
Akak
|
M
|
167/167
|
217/217
|
166/166
|
260/260
|
241/227
|
|
36
|
Gpal
|
Akak
|
M
|
167/167
|
220/220
|
166/166
|
257/257
|
000/000
|
|
37
|
Gpal
|
Akak
|
M
|
167/167
|
217/217
|
166/166
|
260/260
|
241/227
|
|
38
|
Gpal
|
Akak
|
F
|
177/177
|
160/160
|
180/166
|
260/254
|
000/000
|
|
39
|
Gpal
|
Akak
|
M
|
177/177
|
220/217
|
166/166
|
260/260
|
241/227
|
|
40
|
Gpal
|
Akak
|
M
|
189/189
|
000/000
|
172/172
|
260/260
|
000/000
|
|
41
|
Gpal
|
Akak
|
F
|
181/167
|
217/217
|
180/166
|
260/260
|
241/227
|
|
42
|
Gpal
|
Akak
|
F
|
181/167
|
217/217
|
180/166
|
260/260
|
241/227
|
|
43
|
Gpal
|
Akak
|
F
|
181/167
|
217/217
|
180/166
|
254/254
|
241/227
|
|
44
|
Gpal
|
Akak
|
M
|
167/167
|
223/223
|
166/166
|
263/263
|
241/227
|
|
45
|
Gpal
|
Akak
|
F
|
167/167
|
217/217
|
180/166
|
260/260
|
241/227
|
|
46
|
Gpal
|
Akak
|
M
|
167/167
|
217/217
|
166/166
|
260/260
|
243/229
|
|
47
|
Gpal
|
Akak
|
M
|
167/167
|
220/217
|
166/166
|
254/254
|
241/227
|
|
48
|
Gpal
|
Akak
|
M
|
165/165
|
220/214
|
166/166
|
287/257
|
241/227
|
|
49
|
Gpal
|
Akak
|
F
|
181/171
|
217/202
|
180/166
|
254/254
|
241/227
|
|
50
|
Gpal
|
Akak
|
M
|
167/167
|
217/202
|
166/166
|
257/257
|
241/227
|
|
51
|
Gpal
|
Akak
|
M
|
167/167
|
217/217
|
172/172
|
257/257
|
000/000
|
|
52
|
Gpal
|
Akak
|
F
|
181/167
|
217/202
|
180/166
|
257/257
|
241/227
|
|
53
|
Gpal
|
Akak
|
F
|
181/167
|
217/202
|
180/166
|
257/257
|
241/227
|
|
54
|
Gpal
|
Akak
|
F
|
181/167
|
217/217
|
180/166
|
260/260
|
243/227
|
|
55
|
Gpal
|
Mabiog
|
F
|
167/167
|
217/217
|
180/166
|
257/257
|
225/225
|
|
56
|
Gpp
|
RioCampo
|
F
|
181/167
|
000/000
|
184/170
|
296/266
|
245/231
|
|
57
|
Gpp
|
RioCampo
|
F
|
181/167
|
190/181
|
194/180
|
260/260
|
000/000
|
|
58
|
Gpp
|
Edjabe
|
M
|
167/167
|
247/208
|
190/190
|
266/266
|
219/203
|
|
59
|
Gpp
|
Edjabe
|
M
|
179/179
|
221/202
|
178/178
|
266/251
|
219/203
|
|
60
|
Gpp
|
RioCampo
|
M
|
177/177
|
205/205
|
186/186
|
260/260
|
000/000
|
|
61
|
Gpp
|
RioCampo
|
F
|
165/165
|
202/202
|
200/194
|
260/260
|
000/000
|
|
62
|
Gpp
|
Fontem
|
F
|
183/175
|
211/202
|
186/172
|
266/266
|
225/211
|
|
63
|
Gpp
|
Fontem
|
F
|
187/177
|
211/211
|
198/186
|
260/260
|
225/211
|
|
64
|
Gpp
|
Fontem
|
F
|
193/183
|
208/208
|
186/174
|
263/251
|
225/213
|
|
65
|
Gpp
|
Fontem
|
F
|
193/183
|
208/202
|
188/172
|
263/251
|
225/211
|
|
66
|
Gpp
|
Fontem
|
F
|
183/171
|
208/208
|
186/174
|
257/245
|
227/213
|
|
67
|
Gpp
|
Fontem
|
F
|
183/171
|
205/205
|
190/178
|
251/243
|
227/213
|
|
68
|
Gpp
|
Fontem
|
F
|
181/167
|
208/190
|
188/176
|
269/269
|
215/199
|
|
69
|
Gmp
|
Adamaoua
|
M
|
167/167
|
208/199
|
202/202
|
263/263
|
223/209
|
|
70
|
Gmp
|
Adamaoua
|
M
|
000/000
|
220/220
|
208/208
|
263/263
|
223/209
|
|
71
|
Gmp
|
Adamaoua
|
M
|
167/167
|
217/217
|
194/194
|
263/263
|
000/000
|
|
72
|
Gmp
|
Adamaoua
|
M
|
193/193
|
214/214
|
220/220
|
290/263
|
221/207
|
Ind : Individu ; M : Mâle
; F : Femelle ; Gmp : G. submorsitans
; Geal : G. caligenea ; GPP :
G. p. palpalis ; Gpal : G. p. pallicera
; Gnigr : G. nigrofusca ; Gfus :
G. fuscipes.
38
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
Il découle de ce tableau que 69 allèles (dont 36
allèles pour G. p. pallicera) ont été
identifiés pour les cinq marqueurs microsatellites utilisés,
illustrant ainsi une certaine diversité génétique au sein
des différentes populations de glossines.
III.4. Analyses génétiques
III.4.1. Polymorphisme des marqueurs
utilisés
Les cinq marqueurs considérés se sont
montrés polymorphes avec des nombres d'allèles variables. La
figure 13 donne les fréquences alléliques par locus et pour
chaque espèce de glossines. Nous n'insisterons pas sur les
espèces pour lesquelles peu de glossines ont été
analysées.
Chez G. p. pallicera, 6, 10, 5, 10 et 9
allèles ont été respectivement identifiés aux loci
55.3, GPCAG, PGP13, C102 et PGP24. Au sein des populations de G. p.
pallicera, la comparaison des nombres d'allèles de
différents loci n'a montré aucune différence significative
(P= 0,47). Les loci GPCAG et C102 étaient les plus polymorphes avec
environ dix allèles distincts pour chacun des loci.
Chez G. p. palpalis, 10, 8, 16, 9 et 15
allèles ont été identifiés respectivement pour les
loci 55.3, GPCAG, PGP13, C102 et PGP24. Les loci PGP13 et PGP24 étaient
les plus polymorphes avec respectivement 16 et 15 allèles. Aucune
différence significative (P= 0,20) n'a été observée
en comparant les nombres d'allèles de différents loci au sein des
populations de G. p. palpalis.
Les valeurs de la richesse allélique entre G. p.
pallicera (4,960) et G. p. palpalis (10,785) montrent que la
variation du nombre d'allèle est non-significative entre ces deux
espèces (P= 0,33). Ces valeurs ont été calculées
parce qu'il était impossible de comparer le nombre d'allèle entre
ces deux espèces à cause de la disproportion entre le nombre
d'échantillons de G. p. pallicera et G. p.
palpalis.
39
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris

Figure 13: Nombre d'allèles en fonction des
espèces de glossines
Gnigr : G. nigrofusca ; Gpal
: G. p. pallicera ; Gcal : G. caligenea
; Gfus : G. fuscipes; Gpp :
G. p. palpalis ; Gmp : G. morsitans
La figure 14 donne le nombre total d'allèles par locus
pour l'ensemble des espèces de glossines. Le locus PGP24 a
été le plus polymorphe avec environ vingt-six allèles
distincts


Figure 14: Nombre d'allèles par locus
III.4.2. L'hétérozygotie et le
coefficient de consanguinité
L'analyse du coefficient de consanguinité (FIS)
révèle un déficit en hétérozygote non
significatif (FIS = 0,04 ; P = 0,31) pour G. p. pallicera.
Par contre, chez G. p. palpalis, le déficit en
hétérozygote est significatif (FIS = 0,145 ; P =
0,0001).
Les différentes valeurs de FIS reflètent
la différence entre les valeurs de Ho et Hs de G. p. pallicera
(tableau 5) et G. p. palpalis (tableau 6)
40
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
Tableau 5: Hétérozygotie et les valeurs de
FIS en fonction des loci chez G. p. pallicera
|
Loci
|
55.3
|
GPCAG
|
PGP13
|
C102
|
PGP24
|
Moyennes
|
|
Ho
|
0,167
|
0,294
|
0,451
|
0,154
|
0,750
|
0,363
|
|
Hs
|
0,319
|
0,753
|
0,523
|
0,768
|
0,494
|
0,571
|
|
FIS
|
-0,199
|
0,519
|
-0,681
|
0,778
|
-0,535
|
0,040
|
Ho = Hétérozygotie
observée Hs = Hétérozygotie attendue
Tableau 6: Hétérozygotie et les valeurs de
FIS en fonction des loci chez G. p. palpalis
|
Loci
|
55.3
|
GPCAG
|
PGP13
|
C102
|
PGP24
|
Moyenne
|
|
Ho
|
0,917
|
0,467
|
1,000
|
0,500
|
0,923
|
0,761
|
|
Hs
|
0,886
|
0,857
|
0,955
|
0,879
|
0,952
|
0,906
|
|
FIS
|
-0,034
|
0,456
|
-0,048
|
0,431
|
0,030
|
0,163
|
Ho = Hétérozygotie
observée Hs = Hétérozygotie attendue
Les valeurs des FIS sont variables entre loci et sont
toutes différentes de zéro. Les figures 15 et 16
présentent les valeurs des FIS par locus, respectivement pour
G. p. pallicera et G. p. palpalis. La valeur moyenne de
FIS est proche de zéro pour G. p. pallicera et
éloignée de zéro pour G. p. palpalis.
41
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris

Figure 15: Valeurs de la FIS en fonction des
loci et pour les populations de G. p. pallicera


Figure 16: Les valeurs de la FIS en fonction
des loci et pour les populations de G. p. palpalis
Effet des allèles nuls sur les déficits
en hétérozygote ou les valeurs du FIS pour G. p.
palpalis.
Compte tenu du fait que la présence des allèles
nuls peut avoir un effet sur le déficit en hétérozygote,
nous avons analysé la relation entre les allèles nuls et les
FIS. La figure 17 illustre l'association entre le nombre
d'allèles nuls et les FIS pour G. p. palpalis. Cette
courbe de régression des allèles nuls (figure 17) donne la
relation entre les valeurs de FIS et les allèles
42
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
nuls. Le coefficient de corrélation (R2 =
0,00627) montre qu'il existe une corrélation négative entre les
allèles nuls et les valeurs de FIS.

Figure 17 : Courbe de régression des FIS
en fonction des allèles nuls pour G. p. palpalis
Effet des allèles nuls sur les déficits en
hétérozygote ou les valeurs du FIS pour G. p.
pallicera.
La figure 18 montre la courbe de régression des
FIS en fonction des allèles nuls. Le coefficient de
corrélation (R2 = 0,4681) montre qu'il existe une
corrélation négative entre les allèles nuls et les valeurs
de FIS.

Figure 18 : Courbe de régression des FIS
en fonction des allèles nuls pour G. p. pallicera
43
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
La fréquence des allèles nuls de la sous
population G. p. palpalis a été estimée en
utilisant la deuxième méthode de Broockfield. En comparant les
allèles attendus, il ressort que les loci GPCAG et C102 ont
présenté des allèles nuls qui auraient contribué
à augmenter leurs valeurs de FIS. Tous les autres loci n'ont
pas présenté d'allèles significativement nuls (tableau
7).
Tableau 7 : Fréquence des allèles nuls
observés et attendus
|
Locus
|
Stuttering
|
Méthode de
|
PB22
|
N
|
N'
|
Blancs
|
Blancs
|
|
|
Brookfield 2
|
|
|
|
observés attendus
|
|
553
|
non
|
0
|
0
|
7
|
7
|
0
|
1,142
|
|
GPCAG
|
non
|
0,1047
|
0,0109
|
7
|
10
|
3
|
1,714
|
|
PGP13
|
non
|
0
|
0
|
7
|
7
|
0
|
1,142
|
|
C102
|
non
|
0,1538
|
0,0236
|
7
|
10
|
3
|
0,999
|
|
PGP24
|
non
|
0
|
0
|
7
|
7
|
0
|
1,428
|
PB22 : Fréquence
génotypique attendue des individus homozygotes nuls ;
Méthode de Brookfield 2 : Nous donne la
fréquence attendue des allèles nuls ; N : Nombre
d'individus génotypés ; N' : Correspond
à la somme de N et les blancs (homozygotes nuls) observés.
Déséquilibre de liaison
Pour voir s'il existait des associations entre les loci
microsatellites analysés, nous avons évalué le
déséquilibre de liaison. En effet, Le déséquilibre
de liaison est évalué sous l'hypothèse de l'absence
d'association statistique entre les loci. Globalement, les valeurs (tableau 8)
du test de déséquilibre de liaison montrent globalement une
absence d'association statistiquement significative aussi bien pour les
populations de G. p. palpalis que celles de G. p. pallicera
(tableau 8). Néanmoins, une association significative a
été observée au sein des populations de G. p.
pallicera pour les combinaisons de loci 55.3 et PGP13 (P = 0,002) au seuil
de significativité P= 0,0025 (en fait, en combinant les P-values de
plusieurs tests pour obtenir une P-value générale pour chaque
paire de locus, le logiciel recalcule le nouveau seuil qui est en
général 0,05/nombre de tests combinés). Ces loci sont donc
en déséquilibre de liaison.
44
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
Tableau 8: P-values du déséquilibre de
liaison entre paires de loci
|
(Seuil de significativité : P=
0,0025)
|
|
|
|
|
Permutation
|
Gpal
|
Gpp
|
Tous
|
|
55.3 X GPCAG
|
0,125
|
1,000
|
0,152
|
|
55.3 X PGP13
|
0,002*
|
1,000
|
0,002
|
|
55.3 X C102
|
0,407
|
0,467
|
0,357
|
|
55.3 X PGP24
|
0,550
|
1,000
|
0,572
|
|
GPCAG X PGP13
|
0,035
|
1,000
|
0,035
|
|
GPCAG X C102
|
0,005
|
1,000
|
0,005
|
|
GPCAG X PGP24
|
0,225
|
1,000
|
0,255
|
|
PGP13 X C102
|
0,882
|
1,000
|
0,887
|
|
PGP13 X PGP24
|
0,115
|
1,000
|
0,115
|
|
C102 X PGP24
|
0,217
|
1,000
|
0,252
|
Gpal : G. p. pallicera; Gpp
: G. p. palpalis ; * : association statistiquement
significative
III.4.3. Différenciation génétique
entre les populations
Pour évaluer la similarité entre les
espèces de glossines, un dendrogramme (contenant des bootsraps) a
été construit. Ce dendrogramme regroupe les glossines en fonction
des espèces (figue 19). Un total de 6 espèces a été
analysé dans cette étude. Pour construire ce dendrogramme, nous
avons regroupé les glossines en fonction des espèces. A la suite
de ce regroupement, nous avons évalué les distances
génétiques entre les espèces. Le dendrogramme
résultant montre les distances génétiques entre les
différentes espèces de glossines. Il ressort que les distances
sont faibles entre G. p. palpalis et G. nigrofusca, G. fuscipes et G. p.
pallicera. Par contre, ces distances génétiques sont
élevées entre les autres espèces comme par exemple G.
caligenea et toutes les autres espèces.
45
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris

Figure 19: Dendrogramme illustrant les différences
et les similarités génétiques entre les espèces de
glossines.
Gpp : G. p. palpalis ; Gnigr : G.
nigrofusca ; Gfus : G. fuscipes ; Gpal :
G. p. pallicera ; Gmp : G. submorsitans
; Gcal : G. caligenea
Pour évaluer le niveau de polymorphisme
génétique entre les populations de glossines, nous avons
utilisé les génotypes multilocus pour caractériser chaque
espèce. Par la suite, un dendrogramme de similarité a
été construit à partir des données obtenues. Un
total de 69 génotypes multilocus distincts a été
identifié à partir des 72 échantillons examinés. Le
dendrogramme généré indique un nombre important de
génotypes différents (figure 20). Ce dendrogramme peut être
subdivisé en quatre clusters : le premier cluster contenant 41 glossines
dont la majorité appartenant à l'espèce G. p.
pallicera provenant de Campo, deux G. p. palpalis de Rio Campo et
G. fuscipes provenant de Bipindi. Le deuxième cluster,
(contenant 10 échantillons) contient trois G. p. palpalis dont
deux provenant de Campo et une autre de Fontem, deux G. caligenea de
Campo, une G. nigrofusca provenant de Campo Beach et quatre G. p.
pallicera de Campo. Le cluster 3 contient majoritairement l'espèce
G. p. palpalis dont cinq provenant de Fontem et trois autres de Campo,
une espèce G. submorsitans de l'Adamaoua. Le cluster 4 contient
trois G. submorsitans provenant de l'Adamaoua et une G. caligenea
provenant de Campo. Le Cluster 5 contient deux G. p. palpalis
d'Edjabé et une G. p. pallicera provenant de Bipindi.
D'une manière générale, on constate que les espèces
de glossines sont isolées les unes des autres.
46
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris

Cluster 1
Cluster 2
Cluster 3
Cluster 4
Cluster 5
LEGENDE
Gmp : G. submorsitans Gpp
: G. p. palpalis Gca : G. caligenea
Gpa : G. p. pallicera Gni :
G. nigrofusca Gfu : G. fuscipes
Admw: Adamaoua Cmpo: Campo
Mmel: Memel
CmpoB: Campo Beach Edjb:
Edjabé
Mbgo: Mabiogo
Figure 20: Dendrogramme illustrant les
différences et les similarités entre les populations de
glossines
47
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
Pour évaluer la structuration entre les
différentes espèces de glossines étudiées, les
valeurs de FST ont été déterminées entre
elles (tableau 9). Ces valeurs de FST permettent d'estimer les
différences génétiques entre les différentes
populations de glossines. Lorsque les valeurs de FST sont proches ou
égales à zéro, on conclut à une absence totale de
structuration ; lorsqu'elles sont différentes de zéro, on conclut
à une structuration de la population en sous populations. A la
lumière de ces considérations, nous observons que toutes les
valeurs de FST sont significatives entre G. p. pallicera et
G. caligenea (0,1894), G. p. pallicera et G. p. palpalis
(0,2006) ; G. p. pallicera et G. morsitans, G. p.
palpalis et G. morsitans (0,2396).
Tableau 9: Variation de FST entre les
populations de différentes espèces.
FST Gcal Gpp Gmp
Gpal 0,1894* 0,2006* 0,2396*
Gcal 0,0356 0,0769
Gpp 0,0967
* : valeur statistique ("P-value") Significative au seuil 0,05
Gpal : G. p. pallicera ; Gcal
: G. caligenea ; Gpp : G. p. palpalis
; Gmp : G. submorsitans.
Pour évaluer la structuration entre les sous
populations (ensemble des glossines du même village) de G. p.
pallicera et de G. p. palpalis en fonction des villages (Campo,
Edjabe, Fontem, Rio Campo), les valeurs de FST ont
été calculées (tableaux 10 et 11). Ces valeurs sont
faibles et non significatives entre les sous populations aussi bien pour G.
p. pallicera que G. p. palpalis (tableaux 9 et 10).
Tableau 10 : Variation de FST entre les sous
populations de G. p. pallicera
Akak Campo Mabiogo Memel
Akak 0.0000 0.0127
NA 0.1939
Campo 0.0000
NA 0.0559
Mabiog 0.0000
NA
Memel
0.0000
Seuil de significativité à 0,005
48
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
Tableau 11 : Variation de FST entre les sous
populations de G. p. palpalis
|
Campo
|
Edjabe
|
Fontem
|
Rio Campo
|
|
Campo
|
0,0000
|
0,1195
|
0,0459
|
0,0277
|
|
Edjabe
|
|
0,0000
|
0,1009
|
0,0866
|
|
Fontem
|
|
|
0,0000
|
0,0464
|
|
Rio Campo
|
|
|
|
0,0000
|
Seuil de significativité à 0,005
49
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
IV. DISCUSSION
Au cours de cette étude, neuf marqueurs microsatellites
ont été testés pour la caractérisation
génétique de G. p. pallicera du Sud forestier
camerounais. Les marqueurs microsatellites utilisés dans la
présente étude avaient déjà été
utilisés dans de nombreux travaux notamment sur G. p. palpalis
des foyers de la THA du Cameroun et de la République
Démocratique du Congo (RDC) (Melachio et al., 2011, 2015), et
même sur G. p. gambiensis d'Afrique occidentale (Solano et
al., 2009).
De ces marqueurs, deux (A10 et B110) n'ont amplifié
aucune des 72 glossines analysées dans cette étude. Or, les
travaux menés avec ces marqueurs sur G. p. gambiensis (Solano
et al., 2009) et G. p. palpalis (Melachio et al.,
2011, 2015) avaient révélé un taux d'amplification
important pour ces espèces de glossines. Cette absence d'amplification
pourrait s'expliquer par le fait que ces glossines n'appartiennent pas aux
mêmes espèces. De plus, la plupart des marqueurs microsatellites
utilisés dans cette étude ont été
synthétisés à partir du génome de G. p.
gambiensis. Ces deux marqueurs ne semblent donc pas appropriés pour
la caractérisation génétique de G. p. pallicera
du sud forestier Camerounais. A côté de ces deux marqueurs,
deux autres (B3 et B104) ont amplifié moins de 50% de glossines
analysées. Ces résultats pourraient s'expliquer par un
polymorphisme au niveau du site de fixation de ces amorces. Le nombre faible
d'échantillons de G. p. pallicera amplifiés avec B3 et
B104 suggère aussi que ces deux marqueurs ne sont pas appropriés
pour la caractérisation génétique de G. p. pallicera
du sud forestier Camerounais. Des neuf marqueurs utilisés dans
cette étude, seulement cinq (55.3, GPCAG, C102, PGP24 et PGP13) d'entre
eux ont donné des résultats d'amplification permettant de faire
des analyses de génétique des populations. Les marqueurs C102,
PGP13, PGP24, 55.3 et GPCAG se sont montrés polymorphes pour G. p.
pallicera et toutes les espèces de glossines ; corroborant ainsi
les résultats obtenus chez G. p. palpalis (Melachio et
al., 2011, 2015), G. p. gambiensis (Solano et al., 2009,
2010), G. tachinoïdes (Adam et al., 2014) . La
variabilité observée au niveau de la sensibilité de ces
cinq marqueurs microsatellites corrobore avec les résultats
antérieurs (Melachio et al., 2011 ; Solano et al.,
2009). Des cinq espèces de glossines analysées dans cette
étude, les marqueurs GPCAG et C102 se sont montrés plus
polymorphes (10 allèles chacun) chez G. p. pallicera et les
marqueurs 55.3, PGP24 et PGP13 (9, 15, 16 allèles respectivement) chez
G. p. palpalis. Ces résultats montrent que le taux
d'amplification obtenu pour chaque marqueur dépendrait de
l'espèce de glossine et du lieu d'échantillonnage.
Les niveaux de polymorphisme obtenus pour G. p. palpalis
ont été supérieurs à ceux de G. p.
pallicera dans cette étude. L'hétérozygotie moyenne
attendue a été de 0,906 pour G.
50
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
p. palpalis alors qu'elle a été de
0,571 pour G. p. pallicera. De même,
l'hétérozygotie moyenne observée était de 0,761
alors qu'elle a été de 0,363 pour G. p. pallicera. Cette
supériorité s'explique par le fait que les marqueurs
microsatellites utilisés ont été désignés
pour G. p. palpalis et sont donc plus adaptés chez ces
derniers.
Les cinq marqueurs microsatellites retenus pour les analyses
génétiques ont montré un polymorphisme
génétique aussi bien au sein des populations de G. p.
pallicera que celles de G. p. palpalis. La combinaison des
résultats obtenus au niveau de chaque locus a permis d'établir
des génotypes multilocus pour chaque glossine analysée. C'est
ainsi que le dendrogramme de la figure 18 illustre les similarités
génétiques obtenues à partir de 5 loci. Ce dendrogramme
confirme le polymorphisme allélique observé au niveau de chaque
locus. Il montre un grand polymorphisme génétique non seulement
au sein des populations de G. p. pallicera du même village, mais
aussi des villages distincts. Ce dendrogramme ne montre aucun regroupement des
échantillons de G. p. pallicera en fonction des villages ;
confirmant ainsi les résultats des FST qui ont montré
une absence de structuration entre les populations de G. p. pallicera.
Le dendrogramme montre que presque tous les échantillons de G. p.
pallicera se concentrent sur une partie du dendrogramme. Ces
résultats indiquent que les populations de G. p. pallicera sont
génétiquement différentes des autres espèces de
glossines. Ces différences s'illustrent par les distances
génétiques entres les populations de G. p. pallicera et
celles d'autres espèces, mais aussi par les valeurs de FST
statistiquement significatives entre G. p. pallicera et les autres
espèces de glossines analysées dans cette étude. Pour les
populations de G. p. palpalis, le dendrogramme montre un grand
polymorphisme génétique non seulement au sein des populations de
G. p. palplis du même village, mais aussi des villages
distincts. Il ne montre aucun regroupement des échantillons en fonction
des villages; confirmant ainsi les résultats des FST qui ont
montré une absence de structuration entre les populations de G. p.
palpalis.
En considérant uniquement l'espèce G. p.
pallicera, il ressort que la valeur globale de FIS est
significativement proche de zéro (FIS = 0,04). Cette valeur
serait due à la grande variation des FIS entre les
différents loci (FIS = -0,199; -0,681; -0,534; 0,519; 0,778
respectivement pour 55.3; PGP13 ; PGP24 ; GPCAG et C102). Ces variations ont
été aussi observées au sein des populations de G. p.
palpalis. Les variations observées entre les valeurs des
FIS ne permettent pas à présent de tirer une conclusion
fiable sur la reproduction de G. p. pallicera c'est-à-dire si
la reproduction se fait de manière panmictique ou pas. Malgré la
grande variation observée entre les valeurs des FIS entre les
loci, les résultats de cette étude
51
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
montrent néanmoins et de manière
générale des déficits en hétérozygotes aussi
bien chez G. p. palpalis (FIS = 0,145) que chez G. p.
pallicera. Ces déficits observés au sein des populations de
G. p. pallicera et G. p. palpalis corroborent les
résultats obtenus pour la plupart des populations de glossines. Comme
dans la plupart des études, ce déficit en
hétérozygote aurait pour origine la présence
d'allèles nuls, l'effet Wahlund dû au mélange des
populations génétiquement différentes et le stuttering qui
se produit lorsque les amorces ne s'hybrident pas très bien provoquant
un bégaiement de la Taq polymérase qui amplifie deux fois un
allèle de même taille pour un individu hétérozygote.
(Solano et al., 2010 ; Melachio et al., 2011). Pour voir si
le déficit en hétérozygote observé dans cette
étude était dû aux allèles nuls, nous avons
estimé le coefficient de corrélation entre les allèles
nuls et les valeurs du FIS. Le coefficient de corrélation
(R2 = 0,0627) montre une corrélation négative entre
les allèles nuls et les valeurs de FIS pour l'espèce
G. p. palpalis. Pour G. p. pallicera, le coefficient de
corrélation (R2 = 0,4681) montre aussi une corrélation
négative entre les allèles nuls et les valeurs de FIS.
Ces deux analyses suggèrent que les allèles nuls ne seraient pas
à l'origine du déficit en hétérozygote
observé. Ce déficit résulterait très probablement
de l'effet Wahlund.
Le test de déséquilibre de liaison a
montré globalement une absence d'association statistiquement
significative aussi bien pour les populations de G. p. palpalis que
celles de G. p. pallicera. Néanmoins, une association
significative a été observée au sein des populations de
G. p. pallicera pour la paire de 55.3 et PGP13 (P = 0,002). Cette
association s'expliquerait par le fait que ces deux loci (55.3 et PGP13) sont
situés sur le chromosome X et contribueraient à une
déviation des proportions génotypiques.
Pour déterminer si les populations de glossines peuvent
être structurées, des valeurs de FST ont
été évaluées. Nos résultats ont
montré une structuration entre les différentes espèces de
glossines (P= 0,0003). La valeur de FST entre G. p. pallicera
et G. caligenea est statistiquement très significative (P=
0,0002), suggérant ainsi que ces deux espèces sont
génétiquement différentes. Entre les autres espèces
de glossines notamment G. p. pallicera et G. p. palpalis, G. p.
pallicera et G. morsitans, les valeurs de FST sont
également significatives et suggèrent également des
différences génétiques entre ces espèces. Ces
résultats des FST sont en accord avec les distances
génétiques observées sur le dendrogramme illustrant les
différences et les similarités génétiques entre les
espèces de glossines. Ces résultats confirment donc les
différences taxonomiques entre ces espèces. Par contre, les
valeurs des FST entre les sous populations de G. p. pallicera
et G. p. palpalis sont faibles et non significatives entre les
villages où ces glossines ont été
échantillonnées ; indiquant ainsi une
52
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
absence de structuration au sein des populations de G. p.
pallicera et G. p. palpalis des villages du foyer de la THA de
Campo. Ces résultats indiquent aussi une absence de populations de
glossines génétiquement isolées et suggèrent des
échanges entre les populations des différents villages. Les
résultats générés dans cette étude
suggèrent une approche de lutte visant la suppression et non
l'éradication des populations de glossines du foyer de la THA de
Campo.
53
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
CONCLUSION
Cette étude sur la caractérisation
génétique de G. p. pallicera a fourni des données
sur la structure génétique de ces populations de vecteurs. Les
marqueurs microsatellites utilisés nous ont permis d'identifier
plusieurs génotypes circulant au sein des populations de G. p.
pallicera, illustrant un polymorphisme génétique au sein des
populations du même village et des villages distincts. Cette
étude a montré que les populations de G. p. palpalis des
villages du foyer de la THA de Campo sont plus polymorphes que les populations
de G. p. pallicera. Elle a montré des valeurs de FST
non significatives entre les sous populations de G. p. pallicera ainsi
que celles de G. p. palpalis. Il n'existe pas de sous structuration
entre les sous populations de G. p. pallicera. Les populations de
G. p. pallicera des villages du foyer de la THA de Campo ne sont pas
génétiquement isolées. Par contre, les valeurs des
FST sont significatives entre les différentes espèces de
glossines ; confirmant ainsi la classification taxonomique de ces
différentes espèces de glossines. Les résultats
générés dans cette étude suggèrent une
approche de lutte par suppression.
54
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
PERSPECTIVES
Pour mieux comprendre la génétique des
populations de glossines, nous comptons dans l'avenir :
? tester les marqueurs disponibles sur d'autres populations de
G. p. pallicera d'autres
régions afin d'évaluer le niveau de polymorphisme
réel au sein de ces vecteurs ;
? mettre au point d'autres marqueurs microsatellites
adaptés à G. p. pallicera afin de générer
des données qui permettront de mieux appréhender la
génétique des populations de G. p. pallicera qui sont
des vecteurs très peu étudiés.
55
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
REFERENCES
Adam Y., Bouyer J., Dayo G.K., Mahama C.I., Vreysen
M.J.B., Cecchi G., Abd-Alla A.M.M., Solano P., Ravel S. & De Meeûs T.
(2014). Genetic comparison of Glossina tachinoides
populations in three river basins of the Upper West Region of Ghana and
implications for tsetse control. Infection, Genetics and Evolution,
28: 588-595.
Akol G.W.O. & Murray M. (1986). Parasite
kinetics and immune responses in efferent prefemoral Iymph draining skin
reactions induced by tsetse transmitted Trypanosoma Congolense.
Veterinary Parasitology, 19: 281-293.
Authié E., Bringaud F., Bakalara N., Tetaud E.
& Baltz T. (1999). Trypanosomoses humaines et animales: maladie du
sommeil et Nagana. Annales de l'Institut Pasteur, 10:
27-50
Avise J.C. (2000). Phylogeography: the
history and formation of species. Harvay University Press ambridge,
Massachusers.
Avise J.C., Arnold J., Ball R.M., Bermingham E., Lamb
T., Neigel J.E., Reeeb C.A. & Saunders N.C. (1987). Intraspecific
phylogeography: the mitochondrial DNA bridge between population genetics and
systematic. Annual Review of Ecology and Systematics,
18: 489-522.
Baker M.D. & Krafsur E.S. (2001).
Identification and properties of micro-satellite markers in tsetse
flies Glossina morsitans sensu lato (Diptera: Glossinidae).
Molecular Ecology Notes, 1: 234-236.
Balloux F. & Lugon-Moulin N. (2002). The
estimation of population differentiation with microsatellite markers.
Molecular Ecology, 11: 155-65.
Bell C.J. & Ecker J.R. (1994). Assignment
of 30 microsatellite loci to the linkage map of Arabidopsis.
Genomics, 19:137-144.
Borde I. (2006). LA PCR: Polymerase
chain reaction, ou réaction de polymérisation en
chaîne. Biologie et Multimédia. Université Pierre
et Marie Curie. Disponible sur
http://www.snv.jussieu.fr/bmedia/PCR/.
Consulté le 12/05/2015.
Bossart J.L. & Pashley-Prowell D. (1998).
Genetic estimates of population structure and gene flow: limitations,
lessons and new directions. Trends in Ecology and Evolution,
13: 202-206.
Bouteille B. & Jannin J. (2004). La
trypanosomose humaine Africaine : épidémiologie, historique,
situation actuelle. Développement et Santé,
171: 2-4.
56
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
Bouyer J. (2006). Ecologie des glossines du
Mouhoun au Burkina Faso : intérêt pour
l'épidémiologie et le contrôle des trypanosomoses
africaines. Sciences Chimiques et Biologiques pour la Santé,
190 p.
Bouyer J., Guerrini L., César J., De la Rocque
S. & Cuisance D. (2005). A phytosociological analysis of the
distribution of riverine tsetse flies in Burkina Faso. Medical and
Veterinary Entomology, 19: 372-378.
Bouyer J., Ravel S., Dujardin J. P., DE Meeüs T.,
Vial L., Thevenon S., Guerrrini L., Sidibe I. & Solano P. (2007).
Population structuring of Glossina palpalis gambiensis
(Diptera: Glossinidae) according to landscape fragmentation in the Mouhoun
river, Burkina Faso. Journal of Medical Entomology,
44: 788-795.
Bowcock A.M., Ruiz L.A., Tomfohrde J., Minch E., Kidd
J.R. & Cavalli-Sforza L. L. (1994). High resolution of human
evolutionary trees with polymorphic microsatellites. Nature,
368: 455-457.
Brunhes J., Cuisance D., Geoffroy B., Hervy J-P. &
Lebbe J. (1994). Les glossines ou mouches tsétsé Paris.
Logiciel d'identification Glossine Expert. Manuel illustré
d'utilisation. Collection «Didactiques » ORSTOM Paris, 160
p.
Camara M., Caro-riaño H., Ravel S., Dujardin
J.P., Hervouet J.P., De Meeüs T., Kagbadounou M.S., Bouyer J. & Solano
P. (2006). Genetic and Morphometric Evidence for Population Isolation
of Glossina palpalis gambiensis (Diptera: Glossinidae) on the Loos
Island, Guinea. Journal of Medical Entomology, 43:
853-860.
Carrié J., Challier A., Durand B., Duvallet G.
& Laveissière C. (1980). La trypanosomiase humaine à
Trypanosoma (Trypanozoon) gambiense Dutton et ses
vecteurs en Afrique occidentale et centrale. Etudes médicales,
1: 1-59.
Cattand P. (2001).
L'épidémiologie de la trypanosomiase humaine africaine:
Une histoire multifactorielle complexe. Médecine tropicale,
61: 313-322.
Cavalli-sforza L.L. & Edwards A.W.F. (1967).
Phylogenetic analysis: models and estimation procedures. American
Journal of Human Genetics, 19: 233-257.
Challier A. (1968). La lutte contre les
vecteurs de la maladie du sommeil en Afrique Occidentale. Cahier ORSTOM,
Série Entomologie Médicale, 4: 257-264.
57
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
Challier A. (1986). La lutte contre les
glossines dans les foyers de la maladie du sommeil : tendances actuelles.
IV ième congrès sur la protection de la santé humaine
et des cultures en milieu tropical, Marseille, 593-598 p.
Challier A. & Laveissière C. (1973).
Un nouveau piège pour la capture des glossines (Glossina:
Diptera, Muscidae): description et essais sur le terrain. Cahier
O.R.S.T.O.M., série Entomologie médicale et Parasitologie,
10: 251-262.
Clausen P.H., Sidibé L, Bassinga A., Richard
X., Bauer B. & Pohlit H. (1993). Pathogenesis and pathology of
African trypanosomosis in Baoulé, N'DamalBaoulé, cross bred and
zebu cattle in Burkina Faso. Clinical performance under high natural tsetse
challenge. Tropical Medicine and Parasitology, 44:
99-107.
Cockerell T.D.A. (1917). A fossil tsetse fly
and other Diptera from Florissant, Colorado. Proceedings of the Biological
Society of Washington, 30: 19-22.
Cox G. (2004). History of sleeping sickness
(African trypanosomiasis). Infectious Disease Clinics of Norht
America, 18: 231-245.
Cuisance D. (1989). Le piégeage des
tsé-tsé. Etudes et synthèses de l'Institut d'Elevage
et de Médecine Vétérinaire des Pays tropicaux. Centre de
coopération internationale en recherche agronomique pour le
développement (Maisons Alfort), 72 p.
Cuisance D., Fevrier J., Dejardi J. & Fillidier J.
(1985). Linear dispersion of Glossina palpalis gambiensis and
Glossina tachinoïdes in a forest gallery in the Sudan Guinean
Area (Burkina Faso). Revue d'Elevage et de Médecine
Vétérinaire des Pays Tropicaux, 38:
153-172
De La Rocque C. (2003). Glossine et
trypanosomoses. CIRAD-EMVT campus International de Baillarguet, Montpellier, 79
p.
Davis C.S. & Strobeck C. (1998).
Isolation, variability, and cross-species amplification of polymorphic
microsatellite loci in the family Mustelidae. Molecular Ecology,
7: 17761778.
Dyer N.A., Lawton S.P., Ravel S., Choi K.S., Lehane M.J.,
Robinson A.S., Okedi L.A., Hall M., Solano P. & Donnelly M.J. (2008).
Molecular phylogenetics of tsetse flies (Diptera:
Glossinidae) based on mitochondrial (CO1, 16S, ND2) and nuclear ribosomal
DNA sequences, with an emphasis on the palpalis group. Molecular
Phylogenetics and Evolution, 49: 227-239.
58
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
De meeûs T. (2012).
Initiation à la génétique des populations
naturelles: application aux parasites et à leurs vecteurs. IRD
Edition, 338 p.
Dieringer D. & Schlötterer C. (2003).
Microsatellite analyzer (MSA): a platform independent analysis tool
for large microsatellite data sets. Molecular Ecology Notes,
3: 167169.
Domergue D. (1981). La Lutte contre la
Trypanosomiase en Côte d'Ivoire, 1900-1945. The Journal of African
History, 22: 63-72.
Duvallet G. (2006). Maladie du sommeil:
terrible fléau pour l'Afrique. Futura-Sciences,
25 p.
Ellegren H. (2000). Microsatellite mutations
in the germline: implications for evolutionary inference. Trends in
Genetics, 16: 551-557.
Ellegren H. (2004). Microsatellites: simple
sequences with complex evolution. Nature Reviews Genetics,
5: 435-455.
Emery D.L., Wells P.W. & Tenywa T. (1980).
Trypanosoma congolense: specifies transformation in vitro
of leukocytes from infected or immunized cattle. Experimental
Parasitology, 50: 358-368.
Eouzan J-P. & Ferrara L. (1978).
Comportement péri-domestique de Glossina palpalis palpalis
(R.-D.) dans un foyer de maladie du sommeil au Cameroun. Cahier
O.R.S.T.O.M., série Entomologie médicale et Parasitologie,
26: 237-242.
Epplen C., Melmer G., Siedlaczck I., Scwaiger F.W.,
Mäueler W. & Epplen J.T. (1993). On the essence of
«meaningless» simple repetitive DNA in eukaryote genomes.
In: DNA Fingerprint: State of the Science. Molecular Human
Genetic, 29-45 p.
Evans D.A. (1979). Cyclical transmission of
Trypanosoma brucei rhodesiense and Trypanosoma congolense by
tsetse flies infected with culture-form procyclic trypanosomes. Journal of
Protozoology, 26: 425-427.
FAO (1992). Biologie, systématique et
répartition des mouches tsétsé. Manuel de lutte contre
la mouche tsé-tsé, 273p.
FAO (1990). Fao Agristat, Rome, 140
p.
Garside L., Bailey M. & Gibson W. (1994).
DNA content and molecular karyotype of trypanosomes of the subgenus
Nannomonas. Acta Tropica, 57: 21-28.
59
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
Gibson W.C., Osinga K.A., Michels P. A. M. & Borst
P. (1985). Trypanosomes of subgenus Trypanozoon are diploid
for housekeeping genes. Molecular and Biochemical Parasilology,
16: 231-242.
Goldstein D.B., Linares A.R., Cavalli-Sforza L.L.
& Feldman M.W. (1995). Genetic absolute dating based on
microsatellites and the origin of modern humans. Proceeding of National
Academy of Science USA, 92: 6723-6727.
Gorman M.J., Cornel A.J., Collins F.H. & Paskewitz
S.M. (1996). A shared mechanism for melanotic encapsulation of
CM-Spehadex beads and a malaria parasite, Plasmodium cynomolgi B, in
the mosquito, Anopheles gambiae. Experimental Parsitology,
84: 380-386.
Goudet, J. (2003). Fstat (ver. 2.9.4), a
program to estimate and test population genetics parameters. Available from:
http://www.unil.ch/izea/softwares/fstat.html
website. Updated from Goudet (1995).
Gouteux J-P., Laveissiere C. & Boreham P.F.L.
(1982). Écologie des glossines en secteur pré-forestier
de Côte d'ivoire. Les préférences trophiques de
Glossina pallicera et G. nigrofusca. Comparaison avec G. palpalis
et implications épidémiologiques. Cahier
O.R.S.T.O.M., série Entomologie médicale et
Parasitologie, 20: 109-124.
Gouteux J-P. & Lancien J. (1986). The
pyramidal trap for collecting and controlling tsetse flies (Diptera:
Glossinidae). Comparative trials and description of new collecting
technics. Tropical Medicine and Parasitology, 37:
61-66.
Gouteux J-P. (1995). La mouche
tsétsé: une mouche pas comme les autres. Insectes et
santé, 99: 2-4.
Hoare C.A. (1972). The trypanosomes of
mammals. A Zoological Monograph.-Oxford: BlackwelI Scientific
Publications, 749 p.
Holmes P.H. (1997). New approaches to the
integrated control of trypanosomiasis. Veterinary Parasitology,
71: 121-135.
Hard J. (2000). Trypanosomoses animales
africaines. In Précis de parasitologie vétérinaire
tropicale. Londres, New York, Paris: Ed. TEC & DOC.-Ed. med. Int,
773 p.
Hocking K.S., Lamerton J.F. & Lewis E.A. (1963).
Tsetse-Fly Control and Eradication. Bulletin of the World Health
Organization 1963, 28: 811-823.
60
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
Itard J. & Cuisance D. (2003). Vecteurs
cycliques des trypanosomoses. In Principales Maladies Infectieuses et
Parasitaires du Bétail. Europe et Régions Chaudes, 139-165 p.
Jackson D.S., Abbey C.S. & Nugent D.
(2006). DNA profiling of the D1S80 locus: a forensic analysis for
undergraduate biochemistry laboratory. Journal of Chemical. Education,
83: 774-776.
Jordan A.M. (1986). Trypanosomiasis Control
and African Rural Development. Longman Inc., New York, 78 p.
Kernaghan R.H. & Davies J.B. (1960).
Field trials of the control of Glossina palpalis by
obstructive clearing. Bulletin of entomological Research,
50: 449-466.
King D.G. & Soller M. (1998). Variation
and fidelity: The evolution of simple sequence repeats as functionnal elements
in adjustable genes. In Evolutionary Theory and Processes: modern
perspectives, 65-82 p.
Kirkby W.W. & Blasdale P. (1960). The
eradication of Glossina morsistans submorsitans and Tachinoides
in part of a river flood plain Nigeria by chemical means. Part II.
Bulletin of entomological Research, 51:
253-264.
Koeman J.H. & Pennings J.H. (1976). An
Orientational Survey on the Side-effects and Environmental Distribution of
Insecticides Used in Tsetse-Control in Africa. Bulletin of Environmental
Contamination & Toxicology, Vol. 5, No. 2, published
by Springer-Verlag New York, Inc.
Kumar S., Tamura K. & Nei M. (2004).
MEGA3: integrated software for molecular evolutionary genetics
analysis and sequence alignment. Brief Bioinformatic,
5: 150-163.
La Rocque S. & Cuisance D. (2005). Une
mouche singulière et dangereuse! Insectes,
136: 27-31.
Launois M., Charbonnier G., Garcia-Laveissière
G., Cuisance D. & Duvallet G. (2004). La mouche
tsétsé pédagogique. CIRAD, 151 p.
Laveissière C., Gouteux J.P. & Couret D.
(1980). Essais de méthodes de lutte contre les glossines en
zone pré-forestière de Côte d'ivoire. Cah.
O.R.S.T.O.M., série Entomologie médicale et Parasitologie,
4: 315-322.
Lavéissière C. & Grébaut P.
(1990). Recherche sur les pièges à glossine (Diptera :
Glossinidae). Mise au point d'un modèle économique : le
piège Vavoua. Tropical Medicine and Parasitology,
41: 185-192.
61
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
Laveissière C., Grébaut P., Herder S.
& Penchenier L. (2000). Les glossines vectrices de la
Trypanosomiase Humaine Africaine. Institut de Recherches pour le
Développement, OCEAC Yaoundé Cameroun, 240 p.
Laveissiere C. & Penchenier L. (2000).
Manuel de la lutte contre la maladie du sommeil en Afrique Centrale et
Occidentale: généralités. OCEAC, 66p.
Leak S.G.A. (1999). Tsetse biology and
ecology: their role in the epidemiology and control of trypanosomosis. CAB
International Publishing, Wallingford, 568 p.
Levine R.A., Corliss J.O., Cox F.E., Deroux G. &
Honigberg B. (1980). A newly revised classification of the protozoa.
Journal of Protozoology, 27: 37-58.
Luna C., Bonizzoni M., Cheng Q., Robinson S., Aksoy S.
& Zheng L. (2001). Microsatellite polymorphism in tsetse flies
(Diptera: Glossinidae) Journal of Medical Entomology,
38: 376 381.
Macdonald W.A. (1960). Insecticidal spraying
against Glossina palpalis in Nigeria based on a study of its nocturnal
resting sites with UV light. Commisson de Coopération Technique en
Afrique au Sud du Sahara, 62: 243-245.
Maclennan K.J.R. (1963). Simultaneous control
of three species of Glossina by the selective application of the
insecticide. Bulletin of entomological Research, 54:
199-212.
Mahood A. (1960). An experiment of the
control of Glossina palpalis in Southern Nigeria, by chemical means.
Commission de Coopération Technique en Afrique au Sud du
Sahara, 62: 269-276.
Maillot L. (1966). Glossine et
végétation. Indications de l'éclaircissement
sélectif et de l'utilisation des insecticides. Environnement et
santé publique, 19: 21-415.
Melachio T.T.T., Gustave S., Sophie R., De Meeûs
T., Causse S., Solano P., Lutumba P., Asonganyi T. & Njiokou F. (2011).
Population genetics of Glossina palpalis palpalis from
central African sleeping sickness foci. Parasites and Vectors,
4: 140-148.
Morlais I. (1998). Identification des
trypanosomes chez les glossines en zones endémiques de trypanosomose
humaine au Cameroun. Thèse en Parasitologie sur la Biologie des
Populations et Ecologie: Biologie des Systèmes Intégrés,
Agronomie, Environnement, Montpellier, 208p.
Morlais I., Grebaut P., Bodo J.M., Djoha S., Cuny G.
& Herder S. (1998). Detection and identification of trypanosomes
by polymerase chain reaction in wild tsetse flies in Cameroon. Acta
Tropica, 70: 109-117.
62
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par
GOMSEU DJOUMSIE Emmanuel Boris
Morris K.R.S. (1946). The control of
trypanosomiasis (of man and animals) by entomological mean. (Eradication of
Glossina palpalis and Glossina tachinoïdes by selective
clearing in the Gold Coast). Bulletin of entomological Research,
37: 201-250.
Mulligan H.W. (1970). The african
trypanosomiasis. Eds allen and Union LTD, London, 159 p.
Murray M., Morrison W.I., Murray P.K., Clifford D.J.
& Trail J.C.M. (1979). Productivity of trypanotolerant livestock.
A quarterly journal on animal health, production and products, 31
p.
Nash T.A.M. (1940). The effect upon
Glossina of changing of climate of the true habitat by partial
clearing of vegetation. Bulletin of entomological Research,
31: 69-84.
Navajas M., Lagnel J., Gutierrez J. & Bourset P.
(1998). Species wide homogeneity of nuclear ribosomal ITS2 sequences
in the spider mite Tetranychus urticae contrasts with extensive
mitochondrial COI polymorphism. Heredity, 80:
742-752.
Njiokou F., Simo G., Mbida Mbida J. A., Truc P., Cuny
G. & Herder S. (2004a). A study of host preference in tsetse flies
using a modified heteroduplex PCR-based method. Acta Tropica,
91: 117-120.
Njiokou F., Laveissiere C., Simo G., Nkinin S.,
Grebaut P., Cuny G. & Herder S. (2006). Wild fauna as a probable
animal reservoir for Trypanosoma brucei gambiense in Cameroon.
Infection, Genetic and Evolution, 6: 147-153.
Njiokou F., Nimpaye H., Simo G., Njitchouang G.R.,
Asonganyi T., Cuny G. & Herder S. (2010). Domestic animals as
potential reservoir hosts of Trypanosoma brucei gambiense in sleeping
sickness foci in Cameroon. Parasite and Vector, 17:
61-66
Omolo M., Hassanali A., Mpiana S., Esterhuizen J.,
Lindh J., Mike J.L., Solano P., Rayaisse J.B., Glyn A. V., Torr S.J. &
Tirados I. (2009). Prospects for Developing Odour Baits to Control
Glossina fuscipes spp., the Major Vector of Human African
Trypanosomiasis. Neglected Tropical Diseases, 3:
e435.
Organisation Mondiale de la Santé (1986).
La Trypanosomiase Africaine: épidémiologie et lutte.
Report of a WHO Expert Committee. WHO Technical report Series, 739
p.
63
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
Ouma J.O., Marquez J.G. & Krafsur E.S. (2005).
Macrogeographic population structure of tsetse fly Glossina
pallidipes (Diptera: Glossinidae). Bulletin of Entomological
Research, 95: 437-447
Pangui L.J. (2001). La trypanosomose, une
contrainte majeure de l'élevage en Afrique Sub-saharienne (30-33). In
Utilisation des trypanocides en Afrique Sub-saharienne, Actes du
séminaire sous régional tenu du 06 au 09 février 2001,
Dakar, Sénégal-Dakar : EISMV, 170
p.
PATTEC (2010). Plan stratégie national
intégré pour l'élimination des trypanosomoses humaines et
animales au Cameroun 2008-2015, 4-50 p.
Peacock L., Cook S., Ferris V., Bailey M. & Gibson W.
(2012). The life cycle of Trypanosoma (Nannomonas)
congolense in the tsetse fly. Parasites and vectors,
5:109-122.
Pollock J.N. 1982. Manuel de lutte contre la
mouche tsétsé. Biologie, systématique et
répartition des tsétsé. Rome, FAO. Vol 1. 310 p.
Pollock J.N. (1982). Biologie,
systématique et répartition des tsé-tsé. Manuel
de lutte contre la mouche tsétsé, 310 p.
Postlethwalt J. (2009). Modern Biology.
Holt, Rinehart and Winston, 317 p.
Rageau J. & Adam P. (1953).
Répartition des glossines au Cameroun français.
Revue d'élevage et de médecine vétérinaire des
pays tropicaux, 2 :73-76.
Schofield S. & Torr S.J. (2002). A
comparison of the feeding behaviour of tsetse and stable flies. Medical and
Veterinary Entomology, 16: 177-85
Schwenkenbecker J.M., Frohlich C., Gehre F., Schnur
L.F. & Schonian G. (2004). Evolution and conservation of
microsatellite marker for Leishmania tropical. Infection, Genetics
and Evolution, 4: 99-105.
Simarro P. P., Diarra A., Postigo J. A., Franco J. R.
& Jannin J. G. (2011). The Human African Trypanosomiasis Control
and Surveillance Programme of the World Health Organization 2000-2009: The Way
Forward. Plos Negleted Tropical Diseases, 5:
e1007.
Simo G., Mbida Mbida J. A., Ebo'o Eyenga V., Asonganyi
T., Njiokou F. & Grebaut P. (2014). Challenges towards the
elimination of Human African Trypanosomiasis in the sleeping sickness focus of
Campo in southern Cameroon. Parasit and Vectors, 7:
374381.
64
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
Simo G., Njiokou F., Nkinin S., Mgbedié M.,
Laveissière C. & Herder S. (2000). Etude de la
prévalence des infections à trypanosomes chez les animaux
sauvages du foyer de la maladie du sommeil de Bipindi, Cameroun. Bulletin
de Liaison et de Documentation de l'OCEAC, 33: 8-15.
Simo G., Njitchouang G.R., Njiokou F., Cuny G. &
Asongannyi T. (2011). Trypanosoma brucei s.l.: microsatellite
makers revealed high level of multiple genotypes in the mid-guts of wild tsetse
flies of Fotem sleeping sickness focus of Cameroon.
Experimental Parasitology, 128: 272-278.
Solano P., Cuny G., Cuisance D., Duvallet G., Ravel
S., Sidibé I. & Touré S.M. (1997). Les techniques de
génétique moléculaire au service de
l'épidémiologie des trypanosomoses. Intérêt de
l'étude du polymorphisme des microsatellites des glossines.
Pathologie parasitaire, 50: 297-301.
Solano P., De La Rocque S., De Meeus T., Cuny G.,
Duvallet G. & Cuisance D. (2000). Microsatellite DNA markers
reveal genetic differentiation among populations of Glossina palpalis
gambiensis collected in the agro-pastoral zone of Sideradougou, Burkina Faso
Insect. Molecular Biology, 9: 433-439.
Solano P, Kaba D., Ravel S., Dyer N.A., Sall B.,
Vreysen M.V., Seck M.T., Darbyshir H., Gardes L., Donnelly M.J., De Meeüs
T. & Bouyer J. (2010a). Population Genetics as a Tool to Select
Tsetse Control Strategies: Suppression or Eradication of Glossina palpalis
gambiensis in the Niayes of Senegal. PLoS Neglected Tropical
Diseases, 4: e692.
Solano P., Ravel S. & De Meeüs T. (2010b).
How can tsetse population genetics contribute to African
trypanosomiasis control? Trends in Parasitology, Vol. 26
No.5.
Sunnucks, P. (2001). Efficient genetic
markers for population biology. Trends in Ecology and
Evolution, 15: 199-203.
Sutherlands G.R. & Richards R.I. (1995).
Simple tandem DNA repeats and human genetic disease. Proceedings
of National Academy of Science USA, 92: 3636-3641.
Torr S.J. & Hargrove J.W. (1998). Factors
affecting the landing and feeding responses of the tsetse fly Glossina
pallidipes to a stationary ox. Medical and Veterinary Entomology,
12: 196-207.
65
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
Trail J.C.M., Murray M., Sones K., Jibbo J.M.C., Darkin
J. & Light D. (1985). Boran cattle maintained by chemoprophylaxis
under Trypanosomiasis risk. Journal of Agricultural Science,
105: 147-166.
Van-Oosterhout C., Hutchinson W.F., Wills D.P.M. &
Shipley P. (2004). Micro-checker: software for identifying and
correcting genotyping errors in microsatellite data. Molecular Ecology
Notes, 4: 535-538.
Vickerman K. (1985). Developmental cycles and
biology of pathogenic trypanosomes. British Medecine Bulletin,
41: 105-114.
Weir B.S. & Cocker ham C.C. (1984).
Estimating F-Statistics for the Analysis of Population Structure.
Evolution, 38: 1358-1370.
Williams J.G.K., Kubelik A. R., Livak K. J., Rafalski
J.A. & Tingey S. V. (1990). DNA polymorphisms amplified by
arbitrary primers are useful as genetic markers. Nucleic Acids
Research, 18: 6531-6535.
Wright S. (1965). The Interpretation of
Population Structure by F-Statistics with Special Regard to Systems of Mating.
Evolution, 19: 395-420.
Zheng L., Benedict M.Q., Cornel A.J., Collins F.H.
& Kafatos F.C. (1996). An integrated genetic map of the african
human malaria vector mosquito, Anopheles gambiae. Genetics,
143: 941-952.
66
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
ANNEXES
Annexe 1 : Préparation de quelques solutions
SOLUTION DE MIGRATION
1.1. TBE
? Tampon de stockage TBE 10X pH 8,2
- Tris base : 109 g
- Acide borique : 55,6 g
- Na2EDTA 2H2O : 7,44 g
Ajouter progressivement de l'eau distillée tout en agitant
(agitateur magnétique)
jusqu'au trait de jauge : 1000 mL
? Tampon TBE 1X
Prélever 100 mL de TBE 10X et ajouter 900 mL d'eau
distillée. Homogénéiser et
conserver à température ambiante.
? Tampon 0,5X
Prélever 50 mL de TBE 10X et ajouter 950 mL d'eau
distillée. Homogénéiser et
conserver à température ambiante.
1.2. Gel d'agarose à 2 %
- Peser 2g de gel en poudre
- Ajouter 100 ml de TBE 0,5X
Homogénéiser jusqu'à obtenir un liquide
blanc laiteux. Chauffer jusqu'à clarification et ajouter 3 ul de BET.
Maintenir à température ambiante dans une cuve à
fabrication des puits jusqu'à solidification.
1.3. Gel de polyacrylamide 10%
-Acrylamide/Bisacrylamide : 18,33 ml
-TBE 10X: 5,5 ml
-H2O sterile: 30,62 ml
-APS: 481,25 ul
-TEMED: 68,75 ul
67
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
1.4. Mix d'extraction
-CTAB 5% : 16 ml
-Tris 1M pH8 : 4 ml -EDTA 0,5%: 1,6 ml
-NaCl 5M: 11,2 mL
-H2O D : 7,2 mL
- â mercaptoethanol : 160 uL
68
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
|
Annexe 2 : Fiche de donnée brute
|
|
|
|
|
|
|
|
|
|
|
|
ind Espèce Village
|
Codage
|
Sexe
|
553
|
|
GPCAG PGP13
|
C102
|
|
PGP24
|
|
1
|
Gnigr
|
Akak
|
Gni_Akak
|
F
|
0
|
0
|
0
|
0
|
0
|
0
|
245
|
245
|
249
|
249
|
|
2
|
Gpal
|
Akak
|
Gpa_Akak
|
F
|
167
|
167
|
217
|
217
|
180
|
166
|
254
|
242
|
241
|
227
|
|
3
|
Gpal
|
Akak
|
Gpa_Akak
|
M
|
167
|
167
|
220
|
214
|
166
|
166
|
260
|
260
|
241
|
227
|
|
4
|
Gpal
|
Akak
|
Gpa_Akak
|
F
|
167
|
167
|
214
|
214
|
180
|
166
|
251
|
251
|
241
|
227
|
|
5
|
Gpal
|
Akak
|
Gpa_Akak
|
M
|
167
|
167
|
214
|
211
|
166
|
166
|
251
|
251
|
241
|
227
|
|
6
|
Gpal
|
Akak
|
Gpa_Akak
|
F
|
167
|
167
|
220
|
220
|
180
|
166
|
254
|
245
|
241
|
227
|
|
7
|
Gpal
|
Akak
|
Gpa_Akak
|
M
|
167
|
167
|
217
|
211
|
166
|
166
|
257
|
257
|
241
|
227
|
|
8
|
Gnigr
|
CmpoB Gni_CmpB
|
F
|
0
|
0
|
151
|
151
|
178
|
166
|
260
|
260
|
205
|
205
|
|
9
|
Gpal
|
Memel
|
Gpa_Mmel
|
M
|
0
|
0
|
220
|
214
|
166
|
166
|
251
|
251
|
207
|
201
|
|
10
|
Gcal
|
Campo
|
Gca_Cmpo
|
F
|
167
|
167
|
202
|
202
|
186
|
172
|
263
|
263
|
261
|
245
|
|
11
|
Gcal
|
Campo
|
Gca_Cmpo
|
F
|
167
|
167
|
205
|
220
|
188
|
174
|
260
|
260
|
263
|
247
|
|
12
|
Gcal
|
Akak
|
Gca_Akak
|
F
|
181
|
167
|
202
|
202
|
184
|
176
|
260
|
260
|
265
|
251
|
|
13
|
Gpal
|
Campo
|
Gpa_Cmpo
|
M
|
167
|
167
|
202
|
199
|
166
|
166
|
260
|
251
|
265
|
251
|
|
14
|
Gfus
|
Memel
|
Gfu_Mmel
|
F
|
181
|
167
|
196
|
220
|
184
|
184
|
251
|
251
|
241
|
227
|
|
15
|
Gpp
|
Campo
|
Gpp_Cmpo
|
F
|
0
|
0
|
0
|
0
|
0
|
0
|
260
|
260
|
0
|
0
|
|
16
|
Gpal
|
Memel
|
Gpa_Mmel
|
F
|
167
|
167
|
202
|
202
|
0
|
0
|
266
|
266
|
0
|
0
|
|
17
|
Gpal
|
Campo
|
Gpa_Cmpo
|
M
|
167
|
167
|
214
|
214
|
166
|
166
|
269
|
269
|
241
|
227
|
|
18
|
Gpp
|
Campo
|
Gpp_Cmpo
|
F
|
181
|
171
|
202
|
202
|
170
|
154
|
263
|
251
|
243
|
243
|
|
19
|
Gpp
|
Campo
|
Gpp_Cmpo
|
M
|
177
|
177
|
211
|
211
|
192
|
192
|
263
|
257
|
209
|
205
|
|
20
|
Gpal
|
Campo
|
Gpa_Cmpo
|
M
|
167
|
167
|
217
|
217
|
166
|
166
|
260
|
260
|
241
|
227
|
|
21
|
Gpal
|
Campo
|
Gpa_Cmpo
|
F
|
181
|
167
|
214
|
214
|
204
|
190
|
260
|
260
|
241
|
227
|
|
22
|
Gpal
|
Campo
|
Gpa_Cmpo
|
F
|
181
|
167
|
238
|
214
|
204
|
190
|
254
|
242
|
241
|
227
|
|
23
|
Gpal
|
Campo
|
Gpa_Cmpo
|
M
|
167
|
167
|
217
|
217
|
166
|
166
|
254
|
242
|
241
|
227
|
|
24
|
Gpp
|
Campo
|
Gpp_Cmpo
|
F
|
193
|
181
|
211
|
211
|
210
|
198
|
257
|
257
|
207
|
193
|
|
25
|
Gpal
|
Akak
|
Gpa_Akak
|
F
|
0
|
0
|
220
|
220
|
180
|
166
|
0
|
0
|
0
|
0
|
|
26
|
Gpal
|
Akak
|
Gpa_Akak
|
M
|
0
|
0
|
220
|
214
|
166
|
166
|
0
|
0
|
0
|
0
|
|
27
|
Gpal
|
Akak
|
Gpa_Akak
|
M
|
167
|
167
|
214
|
214
|
166
|
166
|
260
|
260
|
241
|
227
|
|
28
|
Gpal
|
Akak
|
Gpa_Akak
|
M
|
167
|
167
|
214
|
214
|
166
|
166
|
260
|
260
|
241
|
227
|
|
29
|
Gpal
|
Akak
|
Gpa_Akak
|
F
|
181
|
167
|
214
|
205
|
180
|
166
|
260
|
260
|
241
|
227
|
|
30
|
Gpal
|
Akak
|
Gpa_Akak
|
F
|
181
|
167
|
217
|
217
|
180
|
166
|
260
|
260
|
241
|
227
|
|
31
|
Gpal
|
Akak
|
Gpa_Akak
|
M
|
167
|
167
|
217
|
217
|
166
|
166
|
260
|
260
|
241
|
227
|
|
32
|
Gpal
|
Akak
|
Gpa_Akak
|
F
|
181
|
167
|
217
|
217
|
180
|
166
|
260
|
260
|
243
|
229
|
|
33
|
Gpal
|
Akak
|
Gpa_Akak
|
M
|
0
|
0
|
217
|
217
|
166
|
166
|
257
|
257
|
241
|
227
|
|
34
|
Gpal
|
Akak
|
Gpa_Akak
|
F
|
181
|
167
|
217
|
217
|
180
|
166
|
260
|
260
|
241
|
227
|
|
35
|
Gpal
|
Akak
|
Gpa_Akak
|
M
|
167
|
167
|
217
|
217
|
166
|
166
|
260
|
260
|
241
|
227
|
|
36
|
Gpal
|
Akak
|
Gpa_Akak
|
M
|
167
|
167
|
220
|
220
|
166
|
166
|
257
|
257
|
0
|
0
|
|
37
|
Gpal
|
Akak
|
Gpa_Akak
|
M
|
167
|
167
|
217
|
217
|
166
|
166
|
260
|
260
|
241
|
227
|
|
38
|
Gpal
|
Akak
|
Gpa_Akak
|
F
|
177
|
177
|
160
|
160
|
180
|
166
|
260
|
254
|
0
|
0
|
69
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris
|
39
|
Gpal
|
Akak Gpa_Akak M
|
177
|
177
|
220
|
217
|
166
|
166
|
260
|
260
|
241
|
227
|
|
40
|
Gpal
|
Akak Gpa_Akak M
|
189
|
189
|
0
|
0
|
172
|
172
|
260
|
260
|
0
|
0
|
|
41
|
Gpal
|
Akak Gpa_Akak F
|
181
|
167
|
217
|
217
|
180
|
166
|
260
|
260
|
241
|
227
|
|
42
|
Gpal
|
Akak Gpa_Akak F
|
181
|
167
|
217
|
217
|
180
|
166
|
260
|
260
|
241
|
227
|
|
43
|
Gpal
|
Akak Gpa_Akak F
|
181
|
167
|
217
|
217
|
180
|
166
|
254
|
254
|
241
|
227
|
|
44
|
Gpal
|
Akak Gpa_Akak M
|
167
|
167
|
223
|
223
|
166
|
166
|
263
|
263
|
241
|
227
|
|
45
|
Gpal
|
Akak Gpa_Akak F
|
167
|
167
|
217
|
217
|
180
|
166
|
260
|
260
|
241
|
227
|
|
46
|
Gpal
|
Akak Gpa_Akak M
|
167
|
167
|
217
|
217
|
166
|
166
|
260
|
260
|
243
|
229
|
|
47
|
Gpal
|
Akak Gpa_Akak M
|
167
|
167
|
220
|
217
|
166
|
166
|
254
|
254
|
241
|
227
|
|
48
|
Gpal
|
Akak Gpa_Akak M
|
165
|
165
|
220
|
214
|
166
|
166
|
287
|
257
|
241
|
227
|
|
49
|
Gpal
|
Akak Gpa_Akak F
|
181
|
171
|
217
|
202
|
180
|
166
|
254
|
254
|
241
|
227
|
|
50
|
Gpal
|
Akak Gpa_Akak M
|
167
|
167
|
217
|
202
|
166
|
166
|
257
|
257
|
241
|
227
|
|
51
|
Gpal
|
Akak Gpa_Akak M
|
167
|
167
|
217
|
217
|
172
|
172
|
257
|
257
|
0
|
0
|
|
52
|
Gpal
|
Akak Gpa_Akak F
|
181
|
167
|
217
|
202
|
180
|
166
|
257
|
257
|
241
|
227
|
|
53
|
Gpal
|
Akak Gpa_Akak F
|
181
|
167
|
217
|
202
|
180
|
166
|
257
|
257
|
241
|
227
|
|
54
|
Gpal
|
Akak Gpa_Akak F
|
181
|
167
|
217
|
217
|
180
|
166
|
260
|
260
|
243
|
227
|
|
55
|
Gpal
|
Mabiog Gpa_Mbgo F
|
167
|
167
|
217
|
217
|
180
|
166
|
257
|
257
|
225
|
225
|
|
56
|
Gpp
|
RioCpo Gpp_Rcpo F
|
181
|
167
|
0
|
0
|
184
|
170
|
296
|
266
|
245
|
231
|
|
57
|
Gpp
|
RioCpo Gpp_Rcpo F
|
181
|
167
|
190
|
181
|
194
|
180
|
260
|
260
|
0
|
0
|
|
58
|
Gpp
|
Edjabe Gpp_Edjb M
|
167
|
167
|
247
|
208
|
190
|
190
|
266
|
266
|
219
|
203
|
|
59
|
Gpp
|
Edjabe Gpp_Edjb M
|
179
|
179
|
221
|
202
|
178
|
178
|
266
|
251
|
219
|
203
|
|
60
|
Gpp
|
RioCpo Gpp_Rcpo M
|
177
|
177
|
205
|
205
|
186
|
186
|
260
|
260
|
0
|
0
|
|
61
|
Gpp
|
RioCpo Gpp_Rcpo F
|
165
|
165
|
202
|
202
|
200
|
194
|
260
|
260
|
0
|
0
|
|
62
|
Gpp
|
Fontem Gpp_Fntm F
|
183
|
175
|
211
|
202
|
186
|
172
|
266
|
266
|
225
|
211
|
|
63
|
Gpp
|
Fontem Gpp_Fntm F
|
187
|
177
|
211
|
211
|
198
|
186
|
260
|
260
|
225
|
211
|
|
64
|
Gpp
|
Fontem Gpp_Fntm F
|
193
|
183
|
208
|
208
|
186
|
174
|
263
|
251
|
225
|
213
|
|
65
|
Gpp
|
Fontem Gpp_Fntm F
|
193
|
183
|
208
|
202
|
188
|
172
|
263
|
251
|
225
|
211
|
|
66
|
Gpp
|
Fontem Gpp_Fntm F
|
183
|
171
|
208
|
205
|
186
|
174
|
257
|
245
|
227
|
213
|
|
67
|
Gpp
|
Fontem Gpp_Fntm F
|
181
|
171
|
205
|
205
|
190
|
178
|
251
|
242
|
227
|
213
|
|
68
|
Gpp
|
Fontem Gpp_Fntm F
|
181
|
167
|
208
|
190
|
188
|
176
|
269
|
269
|
215
|
199
|
|
69
|
Gmp
|
Adama Gmp_Admw M
|
167
|
167
|
208
|
199
|
202
|
202
|
263
|
257
|
223
|
209
|
|
70
|
Gmp
|
Adama Gmp_Admw M
|
0
|
0
|
220
|
220
|
208
|
208
|
263
|
263
|
223
|
209
|
|
71
|
Gmp
|
Adama Gmp_Admw M
|
167
|
167
|
217
|
217
|
194
|
194
|
263
|
263
|
0
|
0
|
|
72
|
Gmp
|
Adama Gmp_Admw M
|
193
|
193
|
214
|
214
|
220
|
220
|
290
|
263
|
221
|
207
|
70
Caractérisation génétique de Glossina
pallicera pallicera circulant dans le foyer de la maladie du sommeil de
Campo du Sud forestier Camerounais rédigé par GOMSEU DJOUMSIE
Emmanuel Boris



