|
Université Montpellier 2 Faculté des Sciences et
Techniques Département Informatique
Bât 16, RC Place Eugène Bataillon 34095 Montpellier
cedex 05
|
|
MASTER 2 INFORMATIQUE
Spécialité-MOCA : Modélisation,
Optimisation, Combinatoire, Algorithme Année universitaire
2013-2014
RAPPORT DE STAGE
Effectué à :
|
INRA
UR 1115 Plantes et systèmes de culture Horticoles Domaine
St Paul, 84914 Montfavet cedex 9
|
|
Du 10 Mars 2014 au 31 août 2014
Sous le
thème :
Conception d'idéotypes de tomate
adaptés au stress hydrique
Par:
Ould Mohamed Abdellahi Cheikh Mehdi
|
Devant le jury :
Maître de stage : Mme. Bertin NADIA
Tuteur universitaire : M. Rodolphe GIROUDEAU
Rapporteur: M. Philippe JANSSEN
|
Soutenu à Montpellier le 05/09/2014
|
1
Table des matières
|
1
|
Introduction
1.1 Problématique .................................
1.2 Présentation de l'entreprise
..........................
1.3 Contexte général du stage
..........................
1.3.1 Les données observées
........................
1.3.2 Présentation du modèle
.......................
1.3.3 L'objectif: ajustement des paramètres
génotype-dépendants . . .
|
10
10
11
12 12 12 14
|
|
2
|
Méthodologie
|
17
|
|
2.1
|
L'optimisation mono-objectif .........................
|
17
|
|
|
2.1.1 Construction de la fonction objectif .................
|
17
|
|
2.2
|
L'optimisation multi-objectif .........................
|
18
|
|
|
2.2.1 Construction des fonctions multi-objectif à
minimiser .......
|
19
|
|
2.3
|
NSGA-II ....................................
|
19
|
|
|
2.3.1 Notion de domination et le front de Pareto ............
|
19
|
|
|
2.3.2 Historique ...............................
|
20
|
|
|
2.3.3 Le principe de la distance de Crowding ...............
|
20
|
|
|
2.3.4 Les différentes étapes de NSGA-II
..................
|
21
|
|
2.4
|
La somme pondérée
..............................
|
23
|
|
|
2.4.1 Utilisation de la somme pondérée
..................
|
25
|
|
2.5
|
Utilisation de NSGA-II ............................
|
25
|
|
|
2.5.1 La recherche du meilleur compromis sur le front de Pareto
. . .
|
26
|
|
|
2.5.1.1 Tracer le front de Pareto pour les meilleurs compromis
.
|
26
|
|
|
2.5.1.2 Critère de sélection par la distance
minimale .......
|
27
|
|
|
2.5.1.3 Critère de sélection par la distance
maximale ......
|
27
|
|
|
2.5.1.4 Critère de sélection par seuil
................
|
29
|
|
3
|
Les résultats sur les génotypes
|
30
|
|
3.1
|
Les résultats d'ajustement indépendant
...................
|
31
|
|
3.2
|
Les résultats d'ajustement parallèle
.....................
|
41
|
|
3.3
|
Analyse de la variabilité des paramètres
...................
|
46
|
|
|
3.3.1 L'analyse avec la fonction pairs ...................
|
46
|
|
|
3.3.2 L'analyse avec ACP ..........................
|
46
|
|
|
3.3.3 Le critère de décision sur une
corrélation forte ...........
|
47
|
|
|
3.3.4 Analyser sur six paramètres
.....................
|
47
|
|
3.4
|
Les résultats finaux ..............................
|
48
|
2
4
|
5
|
Synthèse de la solution apportée
Conclusions
|
62
65
|
|
5.1
|
Résultats obtenus ...............................
|
65
|
|
5.2
|
Difficultés rencontrées
............................
|
65
|
|
5.3
|
Apports ....................................
|
65
|
|
|
5.3.1 Pour l'entreprise ...........................
|
65
|
|
|
5.3.2 Apport personnel ...........................
|
66
|
|
6
|
Bibliographie
|
67
|
|
6.1
|
Références bibliographiques
.........................
|
67
|
|
6.2
|
Webographies .................................
|
68
|
3
Table des figures
1.1 L'ensemble des nuages de points qui présentent
l'évolution de MFobs et de
MSobs. Chaque point représente un fruit
prélevé à un stade donné. . . . 13
1.2 Le
fonctionnement du modèle croissance de fruit..............14
1.3 L'évolution de MFpred et de MSpred pour le
génotype SSD45. ...... 15
1.4 Illustration du problème sur un génotype dans
une condition donnée. . . 16
2.1 Le principe de regroupement par
cluster...................18 2.2 La notion de domination et le front de
Pareto................20 2.3 Le principe de distance de
crowding.....................21
2.4 Comparaison basée sur la dominance pour la
population R(t). ...... 23
2.5 Le scénario général de
NSGA-II....................... 24
2.6 Les compromis construisant le front de Pareto se trouvent sur
la courbe
rouge, comment choisir le meilleur ?. . . . . . . . . . . . . .
. . . . . . . 28
2.7 Critère de sélection par la distance maximale
................ 28
3.1 La teneur en matière sèche prédite par
le modèle au cours de 24H..... 31
3.2 L'ajustement indépendant des paramètres du
génotype Levovil. ..... 33
3.3 L'ajustement indépendant des paramètres du
génotype Cervil. ...... 34
3.4 L'ajustement indépendant des paramètres du
génotype CervXLev..... 35
3.5 L'ajustement indépendant des paramètres du
génotype SSD106. ..... 36
3.6 L'ajustement indépendant des paramètres du
génotype SSD133. ..... 37
3.7 L'ajustement indépendant des paramètres du
génotype SSD45....... 38
3.8 L'ajustement indépendant des paramètres du
génotype SSD18....... 39
3.9 L'ajustement indépendant des paramètres du
génotype SSD173. ..... 40
3.10 L'ajustement parallèle des paramètres des
génotypes Cervil et Levovil. . . 42
3.11 L'ajustement
parallèle des paramètres des génotypes CervXLev et SSD106.
43 3.12 L'ajustement parallèle des paramètres des
génotypes SSD133 et SSD45. . 44 3.13 L'ajustement parallèle des
paramètres des génotypes SSD18 et SSD173. . 45
3.14 Les graphes des corrélations entre les huit
paramètres par génotype. . . . 49
3.15 Les graphes des
corrélations entre les huit paramètres par génotype et
sur
l'ensemble des génotypes............................50
3.16 Les graphes d'analyse en composantes principales entre les huit
paramètres
par génotype et sur l'ensemble des
génotypes................51
3.17 Les histogrammes obtenus pour num et pi_f0. ...............
52
3.18 La convergence des num et pi_f0.......................
53
3.19 Les graphes des corrélations entre les six paramètres
par génotype et sur l'ensemble des
génotypes............................54
4
3.20 Les graphes d'analyse en composantes principales entre les
six paramètres par génotype et sur l'ensemble des
génotypes................55
3.21 La convergence des ph_max, Y_param, el et tstar.
............ 56
3.22 Les 20 meilleurs compromis pour SSD45. ..................
58
3.23 Le meilleur des 20 meilleurs compromis pour SSD45.
........... 59
3.24 Les graphes des corrélations et d'analyse en
composantes principales entre les six paramètres sur 37
génotypes......................60 3.25 La convergence des ph_max,
Y_param, el et tstar dans les résultats finaux. 61
4.1 Les schémas d'illustrations sur les objectifs atteints
dans ce travail. . . . 64
5
List of Algorithms
|
1
|
La fonction multi-objectifs à minimiser . . . . . . . .
. . . . . . . . . . .
|
19
|
|
2
|
Calcul de la distance de Crowding d'un individu i sur un front
F .....
|
21
|
|
3
|
Solution-non-dominées(P) [Deb et al, 2002]
.................
|
22
|
|
4
|
NSGA-II ....................................
|
25
|
|
5
|
Tracer le front de Pareto à partir des nuages des points
des compromis .
|
27
|
|
6
|
Sélection par seuil ...............................
|
29
|
6
Liste des tableaux
2.1 Les compromis à traiter par le critère de
seuil................29
3.1 Tableau des valeurs de paramètres de NSGA-II pour
analyser sur 8 para-
mètres......................................32 3.2 Les
valeurs des RRMSE(s) trouvées salon les critères de
sélection pour le
génotype SSD173 en condition
témoin....................32 3.3 Les 50 estimations trouvées par
génotype pour faire une analyse sur huit
paramètres...................................48 3.4
Tableau des valeurs de paramètres de NSGA-II pour analyser sur six
pa-
ramètres.....................................48
3.5 Tableau des valeurs de paramètres de NSGA-II pour les
résultats finaux. 57
7
Résumé:
Dans ce rapport de stage, nous allons aborder les chapitres
suivants :
> Une introduction qui aboutira à l'identification du
problème posé.
> Une méthodologie où nous étudierons en
détail la solution adaptée pour ce problème.
> Des résultats sur les génotypes où nous
présenterons et analyserons les résultats obtenus selon la
solution adaptée.
> Une synthèse de la solution apportée où
nous donnerons une description synthétique de la solution
proposée.
> Et une conclusion sur les résultats obtenus.
Mots clés : génotype, ajustement, NSGA-II, front de
Pareto, compromis, corrélation.
8
Remerciements :
Durant mon stage, j'ai bénéficié du soutient
moral et technique de plusieurs personnes ce qui a rendu mon environnement de
travail très agréable.
J'adresse tout particulièrement mes plus chaleureux
remerciements à mon responsable de stage Madame Nadia Bertin,
pour m'avoir permis d'intégrer l'équipe de recherche
dans le cadre de ce stage, pour son écoute, sa disponibilité et
ses conseils précieux.
Mes remerciements les plus chaleureux vont également aux
membres de mon équipe pour m'avoir bien accueilli, intégré
et de leurs aides illimités. Chacun d'eux a permis de son niveau, le bon
déroulement de ce stage, je tiens à les remercier
sincèrement :
> Gilles Vercambre > Michel
Génard
> Pierre Valsesia
> Valentina Baldazzi
J'adresse une attention particulière à Monsieur
Gilles Vercambre grâce à lui, j'ai pu
dépasser tous les défis techniques rencontrés durant ce
travail.
Mes plus chaleureux remerciements s'adressent également
à mon tuteur universitaire et responsable de ma formation Monsieur
Rdlphe GIROUDEAU, pour avoir accepté de diriger ce
travail.
Mes remerciements vont également à Monsieur
Philippe JANSSEN pour avoir accepté être le
rapporteur de ce travail.
Je tiens aussi à remercier sincèrement les
membres du jury qui me font le grand honneur d'évaluer ce travail.
Enfin, je tiens à remercier mes parents qui m'ont
soutenu et aidé tout au long de mes études, en particulier mes
frères Bah et Elhusseine, qui
veillaient sur mes études, je vous remercie infiniment.
9
Lexique :
MF : Matière fraiche (poids total du fruit)
MS : Matière sèche (poids du fruit après
séchage)
MFobs : Matière fraiche observée
expérimentalement
MSobs : Matière sèche observée
expérimentalement
MFpred : Matière fraiche prédite par le
modèle
MSpred : Matière sèche prédite par le
modèle
Condition : Soit témoin, soit stress
Anthèse (la floraison) : la période fonctionnelle
pour la pollinisation de la fleur
RMSE : Root-Mean-Square Error
RRMSE : Relative Root Mean-Squared Error
Les huit paramètres à ajuster :
> phi_max : utilisé
pour le calcul de l'extensibilité de la paroi cellulaire
> Y _param : correspond au seuil de la
pression de turgescence pour la croissance > pi_f0 :
utilisé pour le calcul de la pression osmotique du fruit
> Lp : conductivité de la membrane composite
pour le transport de l'eau
> nu_m : vitesse maximale
de transport du carbone par unité de masse sèche utilisé
dans le calcul de Ua (absorption active de
sucre).
> tstar : utilisé dans le calcul de
Ua (absorption active de sucre) >
tau_a : utilisé dans le calcul de
Ua (absorption active de sucre) >
el : utilisé dans le calcul de
Ua (absorption active de sucre)
10
Chapitre 1
Introduction
1.1 Problématique
Depuis plusieurs années, les
météorologistes constatent des modifications des conditions
climatiques à l'échelle mondiale, et surtout régionale,
allant vers un réchauffement de la planète. Ce changement
climatique entraîne une fréquence accrue
d'évènements climatiques extrêmes, parmi lesquels des
périodes de sècheresse provoquant un stress hydrique important
sur les plantes cultivées avec pour conséquence une diminution
des rendements et une modification de la qualité des produits (Seki et
col. , 2003; Farooq et col. ;2009a,b et 20011). En effet, l'eau est l'un des
principaux facteurs qui influent sur la croissance et le développement
des plantes, car elle constitue la force motrice de la multiplication et de
l'élongation cellulaire qui sont les mécanismes clés de la
croissance des plantes. Pour croître, les plantes ont besoin d'un apport
régulier d'eau. Un manque d'eau au niveau du sol peut affecter le
contenu en eau des feuilles, le transport et l'accumulation des
éléments nutritifs et par là même la croissance des
plantes cultivées. Le déficit hydrique peut donc être
définit comme un manque d'eau par rapport aux besoins de la plante pour
une croissance et un développement optimal durant son cycle de vie
(Manivannan et al., 2008).
La tomate est l'une des plus importantes plantes
cultivées à travers le monde ainsi qu'une source importante de
vitamines et d'antioxydants (H.K Klee et col., 2011). Comme toute plante, la
tomate est aussi sensible au déficit hydrique, qui provoque une perte de
rendement et une modification de la qualité des fruits. La
qualité du fruit est un objectif de recherche majeur chez la tomate et
un caractère complexe car de nombreux critères doivent être
pris en compte. Pour l'industrie, la taille des fruits, le contenu en
matière sèche, la couleur et la viscosité sont des
critères importants. Plus récemment, des critères de
qualité pour la valeur santé et le plaisir des consommateurs sont
aussi pris en compte. Pour le marché de frais, la taille des fruits,
leur texture et leur composition en sucres et acides sont des critères
très importants.
Aujourd'hui, les agriculteurs doivent se conformer à un
nombre croissant de réglementations afin de réduire l'impact des
productions sur l'environnement et la santé humaine. Dans ce contexte,
des exigences de qualité des aliments et de régularité de
la production les obligent à envisager toutes les alternatives possibles
pour pallier à ces questions. Pour cela les systèmes de
production doivent s'adapter au contexte climatique, mais aussi faire
11
face à la demande sociale croissante en termes de
qualité organoleptique, nutritionnelle et environnementale des produits
alimentaires. Par conséquent, une question cruciale pour l'avenir est de
savoir comment mieux exploiter la variabilité génétique
pour concevoir de nouvelles variétés plus adaptées au
nouveau contexte climatique et à des pratiques culturales respectueuses
de l'environnement notamment la réduction des apports d'eau. Une
approche intégrée est nécessaire pour gérer les
liens et les antagonismes entre les processus biologiques sous l'influence du
génotype et des pratiques culturelles. Dans cet objectif, un
modèle de simulation décrivant les interactions entre les
principaux processus de croissance du fruit pourrait éventuellement
être utilisé pour analyser la variabilité
génétique et aider à la conception de génotypes
adaptés aux contraintes environnementales (on parle d'idéotype,
c'est-à-dire de génotype ideal dans des conditions
données). Les modèles peuvent également permettre la
construction de systèmes de production innovants, en appliquant une
méthodologie d'optimisation.
Durant ces dernières années, des approches de
modélisation écophysiologique ont été
développées pour comprendre les interactions entre les processus
clés impliqués dans le contrôle génétique de
caractères complexes et prédire les interactions
génotypeXenvironnement Xpratiques
culturales et pour la conception de nouveaux idéotypes adaptés
aux environnements cibles (Génard et col., 2007). Pour la tomate,
plusieurs modèles ont été développés pour
prédire le développement et la croissance du fruit, sa
concentration en sucre ainsi que l'endoréduplication ou endomitose de
l'ADN (Bertin et col., 2003; Bertin et col., 2007; Bussières, 2002; Liu
et col. 2007). Ces modèles sont des outils potentiels d'analyse de la
variabilité génétique et des interactions
génotypeXenvironnement ainsi que des outils de
phénotypage (Genard et col., 2010).
La conception de systèmes de production durable
combinant idéotypes est un problème multicritères, car il
y a beaucoup des critères antagonistes à remplir et de nombreuses
contraintes à respecter. Lorsque les critères sont choisis et les
contraintes définies, la sélec-tion/conception à base de
modèles de systèmes intégrés de production
peut-être formulée comme un problème d'optimisation
multi-objectifs. Par conséquent, nous devons recourir à des
méthodes d'évaluation multicritères "efficaces", des
algorithmes d'optimisation multi-objectifs pour la conception elle-même
et des techniques multicritères d'évaluation afin
d'évaluer la pertinence des systèmes de culture conçus.
1.2 Présentation de l'entreprise
Le centre Inra Provence-Alpes-Côte
d'Azur est un centre de recherche tourné vers l'agroécologie des
systèmes de culture sous serres et en vergers et la modélisation
de l'impact régionalisé du changement climatique à
l'échelle du paysage. Parmi ses unités des recherches,
l'unité PSH (Plantes et Systèmes de culture Horticoles) travaille
sur les productions horticoles alimentaires, et principalement les fruits et
légumes consommés en frais. Ses objectifs finalisés sont
de contribuer à la mise au point de scénarios techniques et
paysagers permettant de promouvoir la qualité des produits
récoltés et le respect de l'environnement. La réalisation
de ces objectifs s'appuie sur des études au niveau de la plante, de ses
fruits, et de populations de bioagresseurs, destinées à mieux
comprendre
12
leurs réponses à l'environnement. Elle s'appuie
également sur des études au niveau des systèmes de culture
permettant de représenter le fonctionnement des plantes en interaction
avec l'environnement physique et biotique et les interventions culturales.
1.3 Contexte général du stage
L'unité PSH est structurée en 2 équipes
de recherches dont l'équipe (Ecophysiologie de la Plante Horticole). Ce
stage se déroule au sein de cette équipe et s'inscrit dans un
projet plus vaste sur l'adaptation de la tomate au déficit hydrique,
financé par le CTPS (Comité Technique Permanent de la
Sélection).
1.3.1 Les données observées
Une expérimentation a été menée en
2013 sur une population de 120 lignées cultivées en condition
témoin et en condition de stress hydrique. Cette population est issue du
croisement entre une lignée (le génotype Cervil) à petits
fruits mais aux qualités orga-noleptiques remarquables et une
lignée (le génotype Levovil) à gros fruits (Causse et al.
2002). On traite tout génotype (élément de la population
précédente) sous deux conditions (témoin et stress), sous
chaque condition donnée, on s'intéresse aux variables MF (la
matière fraiche) et MS (la matière sèche) qui varient en
fonction de l'âge (des jours après anthèse de la fleur) de
ce génotype.
Deux types de données : les premières issues de
l'expérimentation, ce qui a permis de mesurer la MF et la MS, ce qu'on
appelle dans ce cas, la MFobs (matière fraiche observée) et la
MSobs (matière sèche observée) pour tout génotype
et dans chaque condition. Le deuxième type de données est issu de
la simulation qu'on fait grâce au modèle (la sous section
suivante). Au niveau des axes, l'axe X =
»des_jours» (il n'est considéré
qu'à partir du jour=8) et sur l'axe Y =(MF ou MS). La
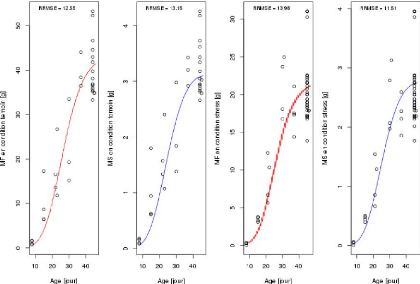
Figure1.1 illustre pour l'un des génotypes
(appelé SSD45) les mesures de matière fraiche
observée et matière sèche observée obtenues pour
les 2 conditions de culture (témoin et stress). On voit que les stress
hydrique a réduit la croissance du fruit.
1.3.2 Présentation du modèle
Le modèle développé pour la tomate permet
de simuler un ensemble des variables [Lui et al.2007]. On se
concentrera dans ce travail uniquement sur l'un des modules du modèle
VirtualFruit, appelée "modèle croissance du fruit",
liée aux flux d'eau et de carbone et à l'expansion
cellulaire. Ce modèle nous permet d'obtenir le deuxième type de
données, en simulant à un pas de temps horaire, la croissance du
fruit, en matière fraîche et sèche qu'on appelle dans ce
cas : MFpred (matière fraiche prédite) et MSpred (matière
sèche prédite) . Pour faire une simulation avec le modèle
de croissance du fruit, il faut donner le jour de début, le jour de fin
de la simulation et deux fichiers sont nécessaires : > Un fichier
des entrées : il contient les informations suivantes
renseignées à un pas de temps horaire :
c des données climatiques : température,
humidité de l'air dans la serre.
c des données plantes : potentiel hydrique de tige et
concentration en sucre dans le phloème.
En condition témoin
I

|
MFobs [g]
e? -
N
0
4 -
|
a
(c) o
a a
CO
o o
a
o
O
O
IP
|
MSobs [g]
c~ -
N --
4 -
|
o o
O
0
a
o
o
O
|
I I I I I I I I
10 20 30 40 10 20 30 40
Agejours Agejours
|
|
i
|
|
En condition stressée
|
i
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
0
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
o
ca
|
|
|
|
|
|
|
ua --
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
a O
O
o
D o O
0.1
MFobs [g]
Q _ N
U)
MSobs [g] L _

o
o
o
o
13
p O
O --
O
I
I I I I f
10 20 30 40 10 20 30 40
Agejours Agejours
FIGURE 1.1 L'ensemble des nuages de points qui
présentent l'évolution de MFobs et de MSobs. Chaque point
représente un fruit prélevé à un stade
donné.
14
~ une donnée fruit : la conductance de la cuticule du
fruit à la vapeur d'eau (rho) .
> Un fichier des paramètres : il
contient tous les paramètres nécessaires au modèle
croissance avec :
~ des paramètres correspondant à des constantes
physiques (constantes des gaz parfait, masse molaire des sucres et de l'eau . .
.)
~ des paramètres plantes fixés quel que soit le
génotype (taux de respiration, effet de la température sur la
respiration . . .)
~ des paramètres plantes
génotype-dépendants
A la fin de la simulation, le modèle
génère un fichier de sorties, contenant toutes les informations
calculées par le modèle au cours de la simulation, et en
particulier la MF-pred et la MSpred qui nous concerneront dans ce qui suit. La
Figure 1.2 illustre l'idée générale du
fonctionnement du modèle de croissance du fruit.
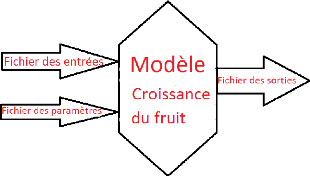
FIGURE 1.2 Le fonctionnement du modèle croissance de
fruit.
1.3.3 L'objectif: ajustement des paramètres
génotype-dépendants
Pour que le modèle simule correctement
l'évolution au cours du temps des deux variables MF et MS, il est
nécessaire d'ajuster huit paramètres ayant un fort impact sur ces
variables. On considère que ces paramètres sont génotype
dépendants, mais ne dépendent pas de l'environnement. Pour un
même génotype, les plants en condition témoin et
stressé partagent les mêmes valeurs de paramètres (voir la
Figure1.3). Le travail du stage va donc consister dans un
premier temps à ajuster ces paramètres pour un génotype
quelconque dans chacune des conditions (témoin ou stress hydrique) pour
simuler le mieux possible l'évolution mesurée au cours du temps
de la matière fraîche et de la matière sèche. La
Figure1.4 montre un exemple où la valeur des
paramètres ne permet pas une simulation correcte de la dynamique de
croissance du fruit. Cette question sera appelée "ajustement
indépendant", dans le sens où pour un même
génotype, les paramètres peuvent être différents
dans les 2 conditions de culture. Dans un deuxième temps, on cherchera
à ajuster les paramètres d'un génotype dans les deux
conditions simultanément,
c'est-à-dire que la valeur des paramètres sera
identique dans les 2 conditions de culture. On parlera dans cette étape
"d'ajustement parallèle".
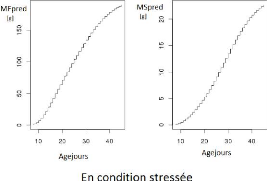
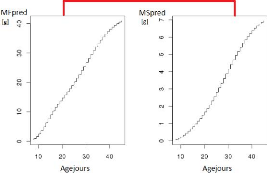
15
FIGURE 1.3 L'évolution de MFpred et de MSpred pour le
génotype SSD45.
Si on résume, les objectifs du stage sont les trois points
suivants :
i) Recalibrer le modèle de croissance du fruit existant
(Liu et al. 2007) sur la population de lignées recombinantes et
éventuellement le modifier pour qu'il soit capable de prédire les
effets du stress hydrique :
- Faire l'ajustement indépendant sur huit
génotypes de la population.
- Faire l'ajustement parallèle sur huit génotypes
de la population.
ii) Analyser la variabilité génétique des
paramètres du modèle.
iii) Proposer au final la meilleur combinaison de
paramètres permettant d'avoir le meilleur ajustement parallèle
par génotype pour toute la population.
16
FIGURE 1.4 Illustration du problème sur un
génotype dans une condition donnée.
17
Chapitre 2
Méthodologie
Résumé : Nous proposons dans ce chapitre de
présenter les méthodes (algorithmes) principalement
utilisées pour arriver aux objectifs proposés. Dans les deux
premières sections, nous allons montrer comment l'ajustement des
paramètres du modèle peut se transformer en un objectif
d'optimisation mono-objectif et éventuellement multi-objectifs, en
profitant de méthodes (algorithme) existant dans le domaine de
l'optimisation. Et dans les trois sections suivantes, nous présentons un
algorithme évolutionnaire multi-objectif et une méthode classique
choisis pour résoudre notre problème et des critères pour
traiter l'ensemble des solutions trouvées.
2.1 L'optimisation mono-objectif
En optimisation mono-objectif, on cherche à
minimiser/maximiser une fonction objectif unique. Cette fonction objectif varie
en fonction d'un ou plusieurs paramètres chacun de ces paramètres
étant borné (contraintes). On va présenter dans la sous
section suivante comment construire nos fonctions objectifs à partir des
données observées et prédites par le modèle de la
croissance du fruit.
2.1.1 Construction de la fonction objectif
Pour toute condition donnée, le modèle permet la
simulation de la matière fraîche et de la matière
sèche pendant la croissance du fruit à un pas de temps horaire.
Les mesures ont été réalisées au cours du temps.
Comme ces mesures sont destructives, plusieurs fruits ont été
récoltés à des âges après anthèse
variable. En général, le nombre de mesures à la
récolte est très important alors qu'à contrario, peu de
fruit ont été récoltés au cours de la croissance
pouvant aller jusqu'à 1 seule mesure à certaine date. De plus,
les fruits n'ont pas été cueillis toujours au même
âge, mais plutôt sur certaines plages d'âges. Au vu du jeu de
donnée (âge variable, nombre de mesures très variable entre
les différents âges), nous avons, donc construit des clusters
permettant à la fois de regrouper différentes mesures
réalisés dans des fenêtres d'âge proche et permettant
de donner un poids identique à ces différentes dates en prenant
pour chaque cluster la moyenne des différences entre les données
observées et prédites (illustration avec la Figure2.1
: chaque cluster est illustré par une couleur). Dans un
deuxième temps, il a fallu définir une fonction objectif pour
chacune des variables MF et MS. Pour chacune de ces variables, il est
nécessaire de
(2.1)
(2.2)
18
construire une fonction objectif qui nous permettra
d'optimiser la valeur des paramètres. L'objectif de cette fonction doit
être de nous permettre de minimiser les écarts entre les
données simulées et les données observées.
|
nClusterP Pn
1 1
RMSE = (Dobsi,j -
Dpredi,j)2
nCluster n
i=1 j=1
|
Les variables de l'équation
(équatin2.1) sont :
Dobs : (MF ou MS).
Dpred :(MFpred ou MSpred).
nCluster : nombre de culster.
n : nombre de points des données
observées au niveau de culster i.
La fonction à minimiser est le RRMSE
(équatin2.2), aussi bien pour la matière
fraîche que pour la matière sèche. L'objectif est bien de
trouver la meilleure combinaison de paramètre permettant de simuler le
mieux possible la matière fraîche et la matière
sèche, donc de minimiser simultanément RRMSEMF et RRMSEMS. Nous
nous trouvons donc dans la situation d'avoir à optimiser sur deux
critères, et nous nous plaçons dans le cadre
général de l'optimisation multi-objectifs (voir section
suivante).
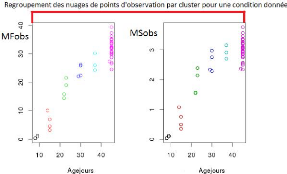
FIGURE 2.1 Le principe de regroupement par cluster.
2.2 L'optimisation multi-objectif
La plupart des problèmes du monde réel
nécessitent l'optimisation selon plusieurs objectifs, ces objectifs
pouvant potentiellement être contradictoires et/ou dépendants
les
19
Algorithm 1 La fonction multi-objectifs à
minimiser
1: k : le nombre des fonctions objectifs et défini selon
la procédure d'ajustement.
2: Initialisation :
~ Initialiser x=(Les_paramètres_à_ajuster) // un
vecteur contenant ces paramètres.
3: Calcul des fonctions objectifs
for (i=1; i<=k; i++) {Calculer la fonction objectif
fi(x)}
4: Remplir le vecteur de fonctions objectifs f :
f(x) = (f1(x),
f2(x), ...,
fk(x))
5: Retourner f(x).
uns des autres. Pour l'optimisation à objectif unique
(ou à critère unique ou encore mono-objectif), la solution
optimale est généralement clairement définie. Pour les
problèmes à objectifs multiples (ou à critères
multiples ou encore multi-objectifs), il n'y a généralement pas
une solution optimale, mais plutôt un ensemble de solutions qui sont des
compromis. Dans la sous-section suivante, on va créer une fonction
multi-objectifs qui regroupera nos fonctions objectifs à minimiser.
2.2.1 Construction des fonctions multi-objectif à
minimiser
Notre problème se présente donc comme un
problème d'optimisation multi-objectif, avec deux fonctions à
minimiser RRMSEMF et
RRMSEMS, qui seront notées fMF
et fMS. Ces fonctions
dépendent des valeurs des paramètres à ajuster,
x=(Les_paramètres_à_ajuster). La minimisation de ces fonctions
doit être réalisée sous contrainte, les valeurs des
paramètres étant bornées. L'objectif est de minimiser ces
fonctions en respectant les contraintes sur les paramètres.
L'algorithme1 résume la création des fonctions
multi-objectifs à minimiser. Un algorithme d'optimisation multi-
objectifs évolutionnaire NSGA-II et une méthode
classique la somme pondérée sont utilisés
à ces fins (les sections suivantes).
2.3 NSGA-II
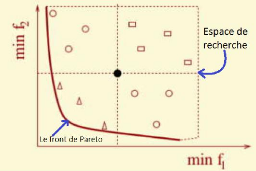
2.3.1 Notion de domination et le front de Pareto
Sur la Figure2.2, on illustre la notion de
domination et le front de Pareto tel que, le point noir :
> domine chacun des carrés.
> est dominé par chacun des
triangles.
> n'est pas comparables avec les cercles.
Le front de Pareto représente l'ensemble des points de
l'espace de recherche tels qu'il n'existe aucun point qui est strictement
meilleur qu'eux (les domine) sur tous les objectifs simultanément. Il
s'agit de l'ensemble des meilleurs compromis réalisables entre les
objectifs contradictoires, et l'objectif de NSGA-II va
être d'identifier cet ensemble de compromis optimaux.
20
FIGURE 2.2 La notion de domination et le front de Pareto.
2.3.2 Historique
NSGA-II (Non-dominated Sorting Genetic Algorithm) a
été proposé par [Deb et al, 2002] et est classé
comme l'un des algorithmes phares dans le domaine de l'optimisation
évolutionnaire multi-objectif. Il tient son appellation de l'algorithme
NSGA qui a été proposé auparavant par [Srinvas et Deb,
1994]. L'algorithme NSGA reprend l'idée proposée par Goldberg sur
l'utilisation du concept de classement par dominance (sous-section
précédente) dans les algorithmes
génétiques [Goldberg 1989]. Dans plupart des aspects NSGA-II est
très différent de NSGA, cependant le nom a été
gardé pour indiquer les origines de cette approche. NSGA-II
intègre un opérateur de sélection d'un individu
(solution), basé sur un calcul de la distance de "Crowding"
(voir sous-section suivante). Comparativement à NSGA,
NSGA-II obtient de meilleurs résultats sur toutes les instances
présentées dans les travaux de [Deb et al, 2002], ce qui fait que
cet algorithme est l'un des algorithmes les plus efficaces pour trouver
l'ensemble optimal de Pareto avec une excellente variété des
solutions.
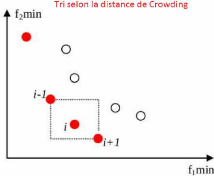
2.3.3 Le principe de la distance de Crowding
La distance de Crowding (ou
surpeuplement) est un opérateur de sélection,
utilisé pour estimer la densité au voisinage d'un individu
(solution) j dans l'espace de recherche. Il calcule la distance moyenne sur
chaque objectif, entre les deux points les plus proches situés de part
et d'autre de la solution j. Cette distance notée di sert
d'estimateur de taille du plus large hypercube incluant le point j sans inclure
un autre point de la population et formé par les solutions du même
front de Pareto les plus proches de j (voir la Figure2.3, les points rouges
appartiennent du même front de Pareto). L'algorithme2 montre les
étapes nécessaires pour calculer cette distance (di)
pour un individu j.
21
FIGURE 2.3 Le principe de distance de crowding.
Algorithm 2 Calcul de la distance de Crowding
d'un individu j sur un front F
1: Initialiser :
l : le nombre d'individus de front F.
di = 0 : pour tout individu j
se trouve sur F.
in = 1 : un compteur sur les objectifs.
2: Réordonner l'ensemble F de façon que
les valeurs de l'objectif fm sur ses
éléments diminuent. Notons Im
=
sort[fm>](F) le vecteur des
indices, c'est à dire Imi
dénote l'indice de la solution j dans la
liste ordonnée selon l'objectif
fm
3: Mettre à jour la valeur de
di
Pour chaque solution j telle que 2
Imi (l
- 1) :
~fm[Im ~
i +1]-fm[Im i -1]
di = di +
max(fm)-min(fm)
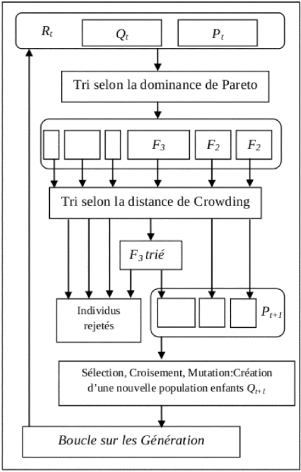
2.3.4 Les différentes étapes de NSGA-II
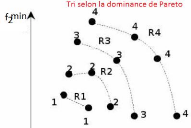
Soient deux populations de même taille N,
P(t) (des parents) générée
aléatoirement et Q(t) (des enfants)
créée à partir de la population P(t), en
utilisant les opérateurs génétiques
(sélection-croisement-mutation) (voir le projet TER).
Ensuite, elles sont réunies en ensemble pour former la population mixte
R(t) = P(t) U Q(t), qui
est triée selon le principe de dominance en un nombre des fronts de
Pareto distincts Rj de la façon suivante :
tous les individus non-dominés de R appartiennent à
l'ensemble R1 (premier front) ; ensuite, tous les
éléments non-dominés de R\R1
sont placés dans l'ensemble R2
(deuxième front) et ainsi de suite jusqu'à ce que
tous les individus (solutions) de R(t) soient
attribués à un front (voir la Figure2.4,
L'algorithme3 montre les étapes nécessaires pour
faire un tri de dominance pour une population donnée). Notons que entre
deux solutions de même front, aucune ne peut être
considérée meilleure que l'autre.
Quand toute la population R(t) est
triée, la population suivante P(t + 1) est remplie
22
Algorithm 3 Solution-non-dominées(P) [Deb et al, 2002]
1: for each p E P // P : une population
à trier selon le principe de dominance
Sp = O // Sp :
une liste ayant contenu tous les individus(solutions) dominés par
p
np = 0 // np : le nombre
d'individus(solutions) qui dominent p
for each q E P
if (p -< q) then // si p domine
q
Sp = Sp U {q} // mettre
q dans Sp
else if (q -< p) then // si q domine
p
np = np + 1
if (np = 0) then //p appartient au
premier front
pfront = 1
F1 = F1 U {p}
2: i = 1 // Initialize the front counter
while (Fi =6 O)
Q = O // Used to store the members of the next
front
for each p E Fi
for each q E Sp
nq = nq - 1
if (nq = 0) then // q appartient au
front suivant
qfront = i + 1
Q=QU{q}
i = i + 1
Fi = Q
par les solutions des sous-ensembles non-dominé de
R(t) l'un après l'autre en ajoutant les fronts, en
commençant par le premier front R1, second front R2,
... etc. Pour choisir les solutions du front qui vont survivre dont seulement
une partie peut être placée dans la population suivante,
l'opérateur de sélection utilise la distance de Crowding
(l'algo-rithme2). Tant que |P(t + 1)| ne
dépassent pas N, un tri selon la distance de Crowding est
appliqué sur les individus du premier front suivant, non inclus dans
P(t+1). Ce tri a pour objectif d'insérer les
(N-|P(t+1)|) meilleurs individus (solutions) qui manquent dans la
population P(t + 1).
Une fois que les individus appartenant à la population
P(t + 1) sont identifiés, une nouvelle population
enfant Q(t + 1) est créée. La sélection
des individus pour la création de Q(t + 1) à
partir de P(t + 1) se fait en utilisant un opérateur
sélection appelé de tournoi qui est appliqué entre n
individus tirés au hasard dans P(t + 1). Un
individu i gagne un tournoi contre un individu j si et
seulement si une des deux conditions suivantes est vraie :
> Front(i) <
Front(j)
> Ou Front(i) =
Front(j) et di > dj
Étant donné que ce tournoi permet à
NSGA-II d'éviter le problème de non élitisme,
c'est-à-dire perdre le meilleur individu de la population P(t) dans la
population P(t + 1).
23
FIGURE 2.4 Comparaison basée sur la dominance pour la
population R(t).
Le processus continue, d'une génération à
la suivante, jusqu'à la dernière. Le scénario
général de NSGA-II est illustré dans la
Figure2.5 et résumé dans
l'algrithme4.
Remarque 2.1. [Deb et al, 2002] ont
montré que la complexité du NSGA-II est de l'ordre de
O(MN2) où M est le nombre d'objectifs (critères) et N
la taille de la population. Cette complexité est induite par la
procédure du tri (l'algrithme3) des solutions non
dominées de l'algorithme.
Remarque 2.2. NSGA-II a un
problème pour résoudre les problèmes ayant un grand nombre
des critères, ses performances se détériorent au
delà de 3 critères, la plupart des individus (solutions) de la
population sont des solutions non dominées, ils ne peuvent pas se
déplacer vers la région optimale au sens de Pareto. La taille de
la population peut être augmentée pour surmonter ce
problème, mais ce qui rend le travail de l'algorithme très lent.
Donc une méthode classique "la somme pondérée" a
été utilisée pour limiter le nombre de nos critères
(les sorties de l'algrithme1) à 2. Elle sera
traité dans la section suivante.
2.4 La somme pondérée
Cette méthode de résolution est la plus
évidente et la plus largement utilisée en pratique parmi toutes
les méthodes classiques. Elle consiste à ramener le
problème multi-objectif à un problème d'optimisation
mono-objectif, il s'agit d'associer à chaque fonction objectif un
coefficient de pondération et à faire la somme des fonctions
objectifs pondérées pour obtenir une nouvelle et unique fonction
objectif (l'équatin2.3). Ces coefficients sont
généralement choisis en fonction de l'importance relative que le
décideur attribue à l'objectif.
F = min k i=1 wifi(x), k = 2
(2.3)
Où les poids 0 < wi < 1 sont tels
que k i=1 wi = 1
24
FIGURE 2.5 Le scénario général de
NSGA-II.
25
Algorithm 4 NSGA-II
1: Initialiser les populations P(0), Q(0) de
taille N, L : nombre de génération, t
= 0.
2: while t < L
do
3: R(t) = P(t) ?
Q(t) ;//Création de R(t), P(t) et
Q(t) désignent respectivement la population et la
progéniture à la génération t.
4:
F=Algorithme3(R(t)) //Trier R(t)
selon le principe de dominance, F = (F1, F2, F3, ...).
5: P(t + 1) = Ø, i =
1 ;// La nouvelle population
6: while |P(t + 1)| +
|Fi| < N do
7: P(t + 1) = P(t + 1) ?
Fi ;
8:
Algorithme2(Fi);
9: i + +;
10: end while
11: P(t+1) =
P(t+1)?Fi[1 :
(N-|P(t+1)|)]//Ordonner l'ensemble Fi
selon la distance de Crowding et inclure les N
-|P(t+1)| solutions ayant les valeurs di
les plus grandes.
12: Q(t + 1) =
mak_new_pop(P(t +
1))//Créer la population des enfants Q(t + 1) à
partir de la population des parents P(t + 1).
13: t + +;
14: end while
2.4.1 Utilisation de la somme pondérée
Il est à noter que la variable k (le
nombre des fonctions objectifs dans l'algorithme1) ne prend
que deux valeurs dans ce travail :
> 2 si on fait un ajustement
indépendant (ajuster sous une seule condition (témoin ou stress)
la MFobs et la MSobs d'où deux fonctions objectifs seulement).
> 4 si on fait un ajustement
parallèle (les deux conditions témoin et stress d'où deux
fonctions objectifs par condition). Pour cet ajustement la somme
pondérée est utilisée pour fusionner les 2 fonctions
objectifs par condition à une seule par condition
(l'équation2.4).
fCondition = w ×
fMF + (1 - w) × fMS
(2.4)
Avec w=0.50 : l'importance relative
attribuée aux fonctions objectifs fMF et
fMS est identique, chacune de ces fonction
ayant importance identique.
2.5 Utilisation de NSGA-II
L'algorithme NSGA-II est déjà
implémenté sous R (l'outil utilisé pour
la programmation) et appeler de la façon suivante :
nsga2(fn, idim, odim,
generations, popsize,lower.bounds, upper.bounds, cprob , cdist,mprob , mdist )
avec,
fn : la fonction à minimiser
(l'algorithme1 (section2.2.1) pour nous).
idim : la taille des entrées de la
fonction fn (le nombre de nos paramètres à ajuster)
odim : la taille des sorties de la fonction fn (la
taille de vecteur des sorties de l'algorithme1, qui est 2 pour
nous et quelque soit la procédure d'ajustement).
26
generations : le nombre de
générations souhaitées et aussi le critère
d'arrêt à respecter.
popsize : la taille de la population, il
présente aussi le nombre des compromis qu'on
pourra avoir.
lower.bounds : les bornes inférieures des
entrées de la fonction fm (les bornes inférieures
de nos paramètres )
upper.bounds : les bornes supérieures des
entrées de la fonction fm (les bornes supé-
rieures de nos paramètres )
cprob : Crossover probability
cdist : Crossover distribution index
mprob : Mutation probability
mdist : Mutation distribution index
Lorsqu'on fait un appel de l'algorithme NSGA-II sur un
génotype pour un ajustement (indépendant ou parallèle),
les sorties sont stockées dans un fichier contenant des colonnes pour
les valeurs obtenues des nos paramètres à ajuster et deux
colonnes particulières représentant les valeurs des fonctions
objectifs à minimiser f1 et f2 (les sorties de
l'algorithme1 (section2.2.1)). Cet algorithme va donc donner
en sortie les "popsize : nombre de compromis" meilleurs compromis obtenus. Et
la question posée est comment choisir le compromis ayant des valeurs
minimales à la fois de f1 et puis f2 ?, cette question
sera traitée dans la sous section suivante.
2.5.1 La recherche du meilleur compromis sur le front de
Pareto
Comme toute méthode d'optimisation multi-objectif,
l'algorithme NSGA-II vise à déterminer le front de Pareto ayant
contenu l'ensemble des meilleurs compromis optimaux. NSGA-II nous propose donc
les valeurs de fonctions objectifs (f1, f2), pour les
meilleurs compromis trouvés. Pour lesquels, il faut tracer le front de
Pareto.
2.5.1.1 Tracer le front de Pareto pour les meilleurs
compromis
Pour qu'un compromis (une solution) A, ayant les
coordonnées (xA, yA)
soit sur le front de Pareto, il ne faut pas qu'il existe un autre compromis
B ayant les coordonnées (xB,
yB), qui vérifie la contrainte suivante :
xB < xA et yB
< yA (2.5)
L'algorithme5 résume les étapes
nécessaires pour sélectionner les compromis formant le front de
Pareto, tel qu'à l'étape2 (while), on fait les tests sur les
compromis, s'il existe un compromis B qui vérifie la
contrainte2.5 contre un compromis A, cela veut dire que A est
rejeté sinon retenu et stocké dans le tableau Pareto[k,2]. Nous
obtenons donc un tableau qui récapitule l'ensemble des solutions
minimisant les fonctions objectifs (f1, f2). La
Figure2.6 donne un exemple sur l'application du
critère.
Maintenant la question devient comment sélectionner le
meilleur compromis se trouvant sur le front de Pareto?. Trois critères
de sélection ont été développés pour
répondre à cette question.
27
Algorithm 5 Tracer le front de Pareto à
partir des nuages des points des compromis
1: Initialiser :
k = 0;
Pareto[k, 2]
: Il prendra les compromis ayant construit le front de Pareto. i =
1;
2: while(i<= f1 )) // Notons que, f1 =
f2
{
for(j = 1; j <= f1 ; j++)
{
if((f1[j]<f1[i]) and
(f2[j]<f2[i]))
{
boule=False;
break;
}
else{boule=True ;}
}
if(boule==True)
{
k=k+1;
Pareto[k,] =(f1[i],f2[i])
}
i=i+1;
}
3: Tracer les éléments de Pareto.
Remarque 2.3. La cntrainte2.5 traduit juste
le principe de dominance entre deux individus (solutions) dans l'espace de
recherche (vu en sectin2.3.1 et algrithme3).
2.5.1.2 Critère de sélection par la
distance minimale
On calcule la distance de chaque compromis se trouvant sur le
front de Pareto par rapport au point d'origine du repère (f1,
f2) et on sélectionne comme choix final le compromis ayant une
distance minimale (l'équation2.6).
\/dmin = f2 1 + f2 2
(2.6)
2.5.1.3 Critère de sélection par la
distance maximale
L'idée de ce critère est de partir des deux points
A(min(f1), max(f2)) et
B(max(f1),
min(f2)),
les extrêmes du front de Pareto et de calculer la distance
de chaque compromis (solution)
se trouvant sur le front de Pareto par rapport à la droite
D(A, B) passant par les points
A et B. Le compromis (solution) retenu est
celui présentant la distance maximale à la
droite D (voir la Figure2.7).
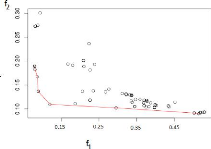
FIGURE 2.6 Les compromis construisant le front de Pareto se
trouvent sur la courbe rouge, comment choisir le meilleur?.
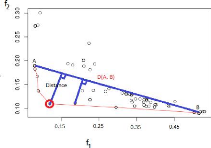
28
FIGURE 2.7 Critère de sélection par la distance
maximale
29
f1
|
a1
a2
a3
..
ai
.. an
|
f2
b1
b2
b3
..
bi
.. bn
|
TABLE 2.1 Les compromis à traiter par le critère
de seuil.
Algorithm 6 Sélection par seuil
1: Initialiser :
Lmax = {Ø}
: // La liste ayant contenu les maximums des couples
(ai, bi)
2: for(i=1; i<=|f1|
; i++)
{
Lmax =
{max(ai, bi)}
}
3: Lk =
min(Lmax)
4: Sélectionner (ak,
bk)
2.5.1.4 Critère de sélection par
seuil
Les critères de sélection par la distance
minimale/maximale peuvent amener à choisir une solution (compromis)
très bonne pour une des fonctions objectif mais beaucoup moins bonne
pour la seconde fonction objectif. Ce critère a été
développé de façon à trouver la solution
étant le meilleur compromis sur les 2 fonctions simultanément.
Sur les deux colonnes associées aux f1 et
f2 dans le fichier des sorties (voir la
Table2.1), on sélectionne le maximum de chaque couple
(ai, bi), on le met dans
une liste Lmax, puis on choisit le compromis
associé à la valeur minimale de cette liste (voir
l'algorithme6).
30
Chapitre 3
Les résultats sur les génotypes
Résumé : Il est à noter
que les résultats de ce travail seront illustrés dans ce rapport
sur huit génotypes, qui ont été choisis parmi les 120
génotypes à traiter. Les 112 génotypes restants seront
traités dans un autre document. Ceci est du au fait que pour chaque
génotype, il faudrait 4 à 5 figures (soit au moins deux pages par
génotype pour rendre les figures visibles), ce qui prendrait plus de 200
pages dans le rapport. Donc pour illustrer la qualité du travail et
diminuer le volume du rapport, nous nous bornerons à présenter
seulement les résultats pour ces 8 génotypes. Rappelons aussi
qu'on applique le même traitement sur les 120 génotypes. Trois
documents seront préparés pour contenir les résultats sur
les 120 génotypes :
-Le rapport (document actuel), contiendra les 8
génotypes choisis.
-Un grand document, contiendra les résultats finaux sur
toute la population (les 112 Génotypes restants + aussi
les 8 génotypes choisis).
-Un fichier des valeurs des paramètres des 120
génotypes.
Comme dit à la sectin1.3.3, on va
ajuster selon deux procédures, la première consiste à
ajuster indépendamment sur les deux conditions (témoin et
stress). Cette procédure aboutit donc à deux vecteurs de
paramètres différents pour les deux conditions et la
deuxième procédure consiste à ajuster les
paramètres simultanément pour les deux conditions. On aboutit
alors à une combinaison de paramètres communes aux deux
conditions. Dans les deux premières sections, on traitera ces deux
procédures d'ajustement, puis en troisième section on fera une
analyse sur la variabilité des paramètres ajustés et
à la dernière section on présentera les résultats
finaux.
Il est à noter aussi que dans toute procédure
d'ajustement, l'heure de récolte prise en compte au niveau de
modèle de croissance du fruit est 10H qui correspond
à l'heure moyenne des prélèvements de fruits pour les
valeurs observées (sur par exemple la Fi-gure3.1, on
voit bien que la teneur en matière sèche pour un génotype
dans une condition donnée commence à croitre à partir de
l'heure 10H) et les huit paramètres seront ajustés sur les bornes
suivantes :
> phi_max E [2.0e - 03, 3.0e - 01] et
Y_param E [0.1, 20.0]
> Lp E [2.0e - 04, 1.0] et tstar E
[1.0, 1500.0]
> tau_a E [100.0, 400.0] et el E
[0.0, 0.30]
> pi_f0 E [0.0, 30.0] et nu_m E [1.0e
- 03, 0.050]
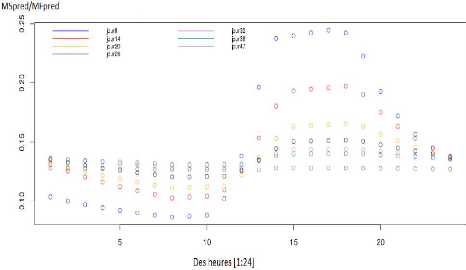
31
FIGURE 3.1 La teneur en matière sèche
prédite par le modèle au cours de 24H.
3.1 Les résultats d'ajustement
indépendant
Dans cette section, on illustre les résultats selon la
première procédure d'ajustement sur les 8 génotypes
choisis représentatifs de la population (120 génotypes). Pour
avoir ces résultats, l'algorithme NSGA-II a été
appelé sur les valeurs de la Table3.1 (revoir la
sectin2.5) par génotype. Les paramètres cprob et
mprob sur cette table, représentent respectivement la probabilité
d'appliquer les opérateurs de croisement et de mutation sur l'ensemble
des individus de la population dans l'espace de recherche. L'opérateur
de croisement a pour but d'enrichir la diversité de la population en
manipulant la structure des individus. L'opérateur de mutation a pour
but de garantir l'exploration de l'espace de recherche, c'est-à- dire le
fait que tout point de l'espace de recherche peut être atteint en un
nombre fini de mutations.
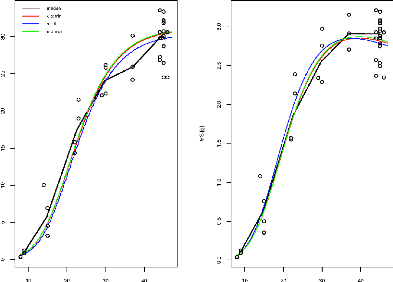
Sur l'ensemble des figures (Figure3.2, ...,
Figure3.9) de ces résultats, l'axe X représente
l'age des fruits (jours) et l'axe Y représente soit
MF(observée-point ou prédite-ligne) soit MS(observée-point
ou prédite-ligne). Et comme indiqué sur les graphiques chaque
courbe représente le meilleur compromis choisi par l'un des trois
critères de choix :
-dist min : Critère de sélection par la distance
minimale.
-dist max : Critère de sélection par la distance
maximale
-seuil :Critère de sélection par seuil
Il apparaît aussi que certains compromis sont identiques
ou très proches ne permettant pas de les distinguer sur les courbes.
32
|
Les paramètres de NSGA-II
|
Les valeurs des paramètres de NSGA-II
|
|
idim
|
8
|
|
odim
|
2
|
|
generations
|
50
|
|
popsize
|
100
|
|
cprob
|
0.7
|
|
cdist
|
5
|
|
mprob
|
0.1
|
|
mdist
|
10
|
TABLE 3.1 Tableau des valeurs de paramètres de NSGA-II
pour analyser sur 8 paramètres.
|
Critère de sélection
|
Le RRMSE de MF
|
Le RRMSE de MS
|
|
Distance minimale
|
12.54
|
12.80
|
|
Seuil
|
12.54
|
12.80
|
|
Distance maximale
|
28.86
|
12.25
|
TABLE 3.2 Les valeurs des RRMSE(s) trouvées salon les
critères de sélection pour le génotype SSD173 en condition
témoin.
Sur l'ensemble des génotypes, on voit bien que chaque
compromis trouvé par chacun des critères de sélection
simule correctement les données observées pour le génotype
désigné. Pour choisir un seul compromis parmi ces trois
trouvés par génotype, on choisit celui ayant des valeurs
minimales à la fois de RRMSE de MF et puis de RRMSE de MS (revoir la
sectin2.5). Par exemple sur la Table3.2, on
choisit pour le génotype SSD173 en condition témoin le compromis
trouvé par les critères de sélection par distance minimale
et seuil (même compromis). Nous allons donc voir dans ce qui suit qu'un
seul critère de sélection sera adapté. Donc d'une
manière globale, le modèle simule correctement l'évolution
au cours temps des deux variables MF et MS sur l'ensemble des génotype.
Nous allons maintenant voir les résultats selon la deuxième
procédure (section suivante).
Remarque 3.1. La courbe
médiane est la courbe passant par les médianes
des clusters et tracer pour voir la qualité d'ajustement pour le
meilleurs compromis choisi au final par génotype.
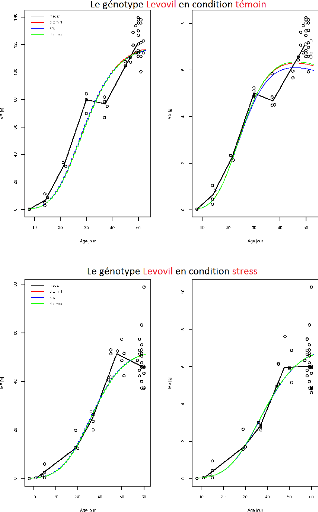
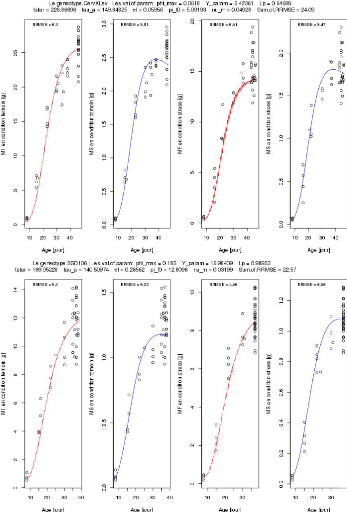
FIGURE 3.2 L'ajustement indépendant des paramètres
du génotype Levovil.
33
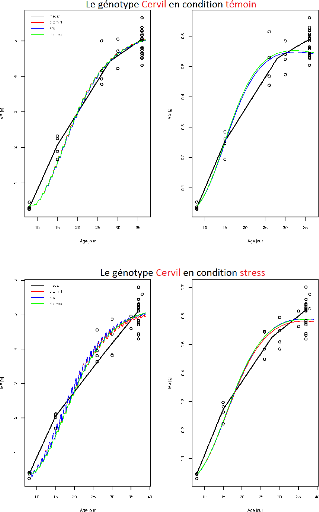
FIGURE 3.3 L'ajustement indépendant des paramètres
du génotype Cervil.
34
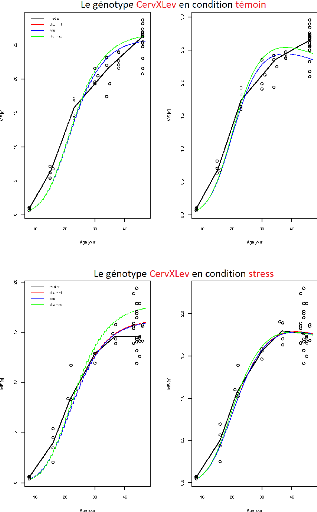
FIGURE 3.4 L'ajustement indépendant des paramètres
du génotype CervXLev.
35
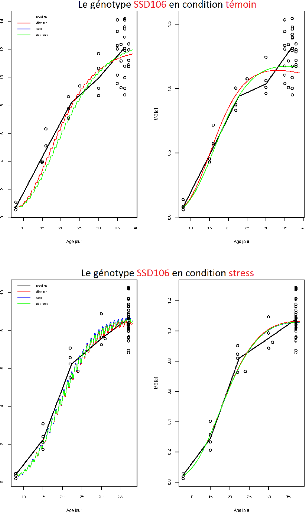
FIGURE 3.5 L'ajustement indépendant des
paramètres du génotype SSD106.
36
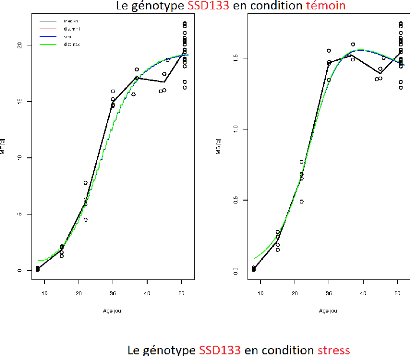
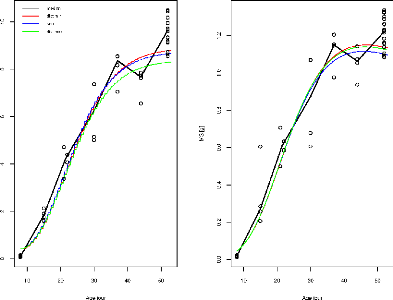
FIGURE 3.6 L'ajustement indépendant des paramètres
du génotype SSD133.
37
Le génotype SSD45 en condition témoin
Age jour Age jour
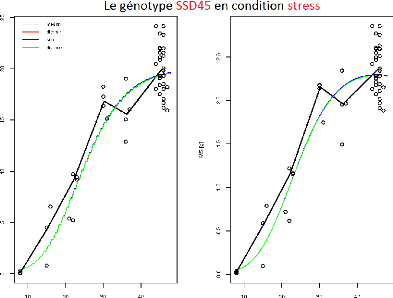
Le génotype SSD45 en condition stress
N(V
FIGURE 3.7 L'ajustement indépendant des paramètres
du génotype SSD45.
Age jour Age jour
38
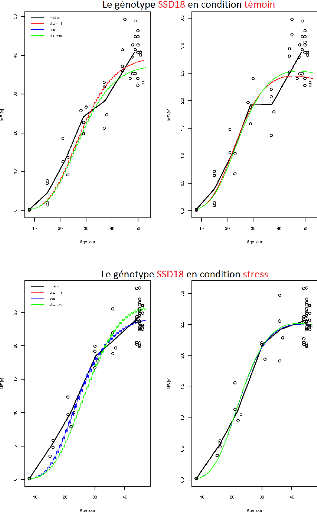
FIGURE 3.8 L'ajustement indépendant des paramètres
du génotype SSD18.
39
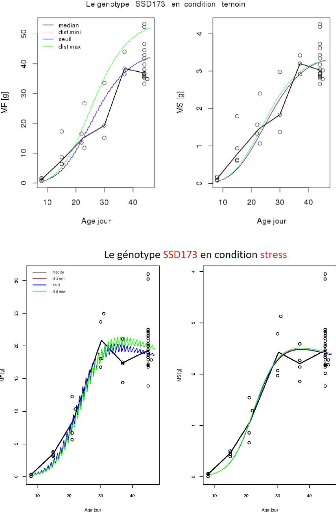
FIGURE 3.9 L'ajustement indépendant des paramètres
du génotype SSD173.
40
41
3.2 Les résultats d'ajustement
parallèle
Dans la section précédente, pour chaque
génotype, 8 paramètres ont été ajustés
indépendamment suivant les conditions donnant donc 8 valeurs de
paramètres en conditions témoin et 8 en conditions stress, soit
16 valeurs par génotype. Avec la procédure d'ajustement
parallèle, nous allons ajuster tous les génotypes avec un seul
jeu de paramètre, quelques soient les conditions (témoin ou
stress). Avec toujours les mêmes huit génotypes, on illustre les
résultats selon cette procédure. Ces résultats ont
été obtenus après avoir appelé l'algorithme
NSGA-II sur encore la même Table3.1 par
génotype. Sur l'ensemble des figures (Figure3.10, ...,
Figure3.13) de ces résultats deux génotypes par
figures, les informations indiquées sont :
> Le nom de génotype.
> Les.val.of.param : Les valeurs des paramètres
trouvées pour ce génotype.
> sum.of.RRMSE : le somme des valeurs des RRMSE(s) de
MF(respectivement, MS) en condition témoin (respectivement, stress).
La valeur de RRMSE est relative avec la qualité
d'ajustement de la courbe associée, c'est-à-dire que si cette
valeur est petite, alors la courbe va beaucoup mieux ajuster les données
observées sinon elle sous-ajuste. D'une manière
générale, on trouve que le modèle simule
l'évolution au cours temps des deux variables MF et MS sur l'ensemble
des génotype selon cette procédure, ce qui nous permet d'aller
faire une analyse sur la variabilité des paramètres (section
suivante).
Vu que cette procédure est celle choisie pour cette
analyse et éventuellement les résultats finaux, il a fallu faire
un seul choix sur les critères de sélections et comme dit
à la sectin2.5.1.4, c'est le critère de seuil
qui est le plus performant par rapport aux critères de sélection
par distance suivi du critère de la distance minimale. En effet le
critère de sélection par la distance minimale est forte pour deux
critères quand on minimise les RRMSE(s) seulement mais vu que cette
procédure a ajouté la somme pondérée
(sectin2.4), ce critère se détériore
parfois en amenant à choisir un compromis très bonne pour une des
fonctions objectif mais beaucoup moins bonne pour la seconde fonction objectif.
Ainsi que avec la minimisation de quatre critères avant découvrir
la Remarque2.2 de NSGA-II. Donc dans tout ce qui suit, seule
la procédure d'ajustement parallèle couplée au
critère de sélection par seuil sera utilisée.
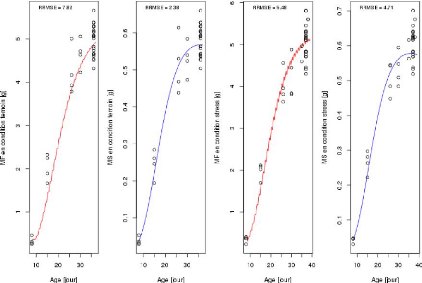
Le gneotype Cervil ; Les.val.of.param: phimax = 0.1609 Y_param
= 10.64766 Lp = 0.81023
tstar = 29.31592 tau a = 1 E0.66633 el = 0.28917 pi f0 =
6.87033 nu_m = 0.04017 Sum.of.RRMSE = 20.39
40
I I I I 1
10 20 30
Age Dour]
Age Dour]
10 20 30
Age []our]
10 20 30 40
Age []our]
MS en condition stress [g]
a
MF en condition temoin [g]
N
O
o
MS en condition temoin [g]
C)
nd tion stress [g]
m LL
Cd
O
ô
ô
o
N
ô
RRMSE -4.71
o
O
6 a
a 0
a
Age [Our]
Age [Our]
Age []our]
Age []our]
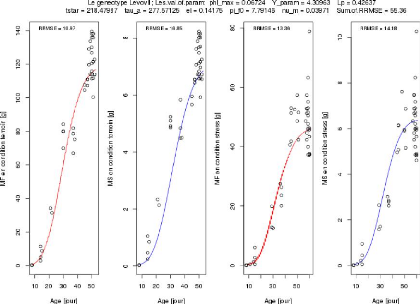
Le genotype Levovil ; Les.val.of.param: phi_max = 0.06724 Y_
cram = 4.30963 Lp = 0.42637
tatar = 218.47987 tau a = 277.57125 el = 0.14175 pi 0= 7.79146
nu_m = 0.03971 Sum.of.RRMSE = 55.36
1 1 I I I
10 30 50
1 1
10 30 50
MFen condition temoin [g]
ME en condition stress [g]
RRMSE- 10137
0
0
RRMSE - 1933
O
N
O
o -
10
a
MS en condition stress [g]
o a
O N
10 20 30 40 50
10 20 30 40 50
42
FIGURE 3.10 L'ajustement parallèle des paramètres
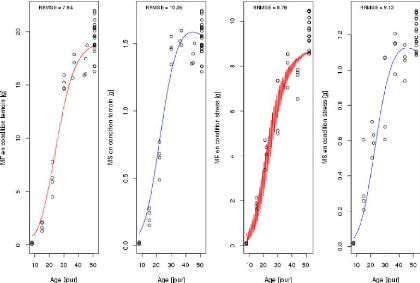
des génotypes Cervil et Levovil.
43
FIGURE 3.11 L'ajustement parallèle des paramètres
des génotypes CervXLev et SSD106.
MS en co nd do n te mo in [g]
ME en condition stress [g]
MS en condition stress [g]
MFen condition temoin [g]


Le wmotype SSD133 Les.val.of.param: phimax = 0.07864 Y_ram =
5.82745 Lp = 0.99707
tstar = 401.77526 tau a = 186.68099 el = 0.14069 pi f0 = 6.60399
nu_m = 0.01725 Sum.of.RRMSE = 35.87
Age [Our]
Age [Our]
Age [pur]
Age [pur]
MF en condition temoin [g]
10 20 30 40 50
MS en condition temoin [g]
10 20 30 40 50
MF en cond fion stress [g]
MS en condition stress [g]
N
N
O
10 20 30 40 50
10 20 30 40 50
Le gneotype SSO45: Les.val.of.param: phi_max = 0.04307 Y_ram =
10.83638 Lp = 0.71487
tstar = 203.84713 tau a = 178.54754 el = 0.06858 pi f0=
10.93544 nu_m = 0.04633 Sum.of.RRMSE = 31.67

LP N
N
I 1 I
10 20 30 40
Age [pur]
I I I
10 20 30 40
Aga [pur]
I
10 20 30 40
Age [Our]
I 1 I
10 20 30 40
Age [pur]
44
FIGURE 3.12 L'ajustement parallèle des paramètres
des génotypes SSD133 et SSD45.
50
I 1 I
10 20 30 40
RRMSE- 12.1 00
0
,
b b°
o
4 00
O
O
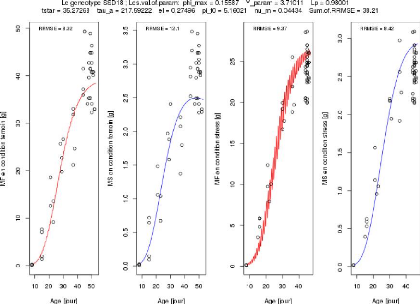
Le geneotype 55018 ; Les.val.of.param: phi_max = 0.15587
Y_param = 3.71011 Lp = 0.9E031
tstar = 35.27268 tau a = 217.59222 el = 0.27496 pi f0 = 5.19321
nu_m = 0.04434 Sum.of.RRMSE = 38.21
o -
Age []our]
I I I
10 20 30 40 50
Age []our]
10 20 30 40
Age []our]
10 20 30 40
Age []our]
MF-i7 condition stress [g]
MS en condition stress [g]
o
o
In
o
rI
In
o
MS en condition temoin [g]
o
o
N
MF en coud tion temoin [g]
RRMSE-222
4
0
o
a
00
00
O
°
O
b
Le geneotype 550173: Les.val.of.param: phi_rnax = 0.04503 Y_ram
= 4.74944 Lp = 0.35584
tstar = 231.03342 tau a = 207.55825 el = 0.16539 pi f0 =
6.33521 nu_m = 0.03257 Sum.of.RRMSE = 51.19
I I
10 20 30 40
Age []our]
MS en condition stress [g]
-r
MFen condition temoin [g]
ms en .:o nd tio n te mo in
o
I 1 I
10 20 30 40
I I I
10 20 30 40
10 20 30 40
Age [Our]
Age [pur]
Age [pur]
RRMSE-1255
a
a °
°
45
FIGURE 3.13 L'ajustement parallèle des paramètres
des génotypes SSD18 et SSD173.
46
3.3 Analyse de la variabilité des
paramètres
Après avoir obtenu les différentes combinaisons
pour les valeurs des paramètres, il était nécessaire de
faire une analyse graphique et statistique des relations entre ces
paramètres. L'objectif est éventuellement de pouvoir
réduire le nombre de paramètres à ajuster, si certains
paramètres sont très fortement corrélés. Pour faire
cette analyse, nous avons fait 50 estimations selon l'ajustement
parallèle (section précédente) sur chacun
de nos 8 génotypes, ce qui a donné 50 valeurs par
paramètre sur tous les génotypes. Chaque estimation a
été trouvée après avoir appelé NSGA-II sur
toujours la même Table3.1 par génotype. Sur la Table3.3, les
paramètres sont les colonnes et les estimations (compromis) sont les
lignes. Deux méthodes d'analyses ont été utilisées
à savoir, la fonction "pairs" et la méthode ACP (analyse
composant principale), elle seront traitées dans les sous sections
suivantes.
3.3.1 L
|
'analyse avec la fonction pairs
|
pairs est une fonction graphique sous R de
haut niveau qui utilise un ensemble de fonctions et paramètres par
défaut pour faire des graphes de corrélations de variables 2
à 2. Elle peut être appelée par génotype sur la
Table3.3 (voir les Figure3.14 et
Figure3.15) et pour l'ensemble des génotypes (voir la
sous-figure "l'ensemble des génotypes" en bas sur la
Figure3.15).
Sur ces graphiques, le nom des paramètres sont
indiqués sur la diagonale, les coefficients de corrélations
au-dessus de la diagonale. Trois couples de paramètres sont
corrélés entre eux 2 à 2,
(tstar et tau_a),
(tstar et nu_m) et
(Y _param et
pi_f0). Ces deux derniers couples sont les
plus fortement corrélés aussi bien au niveau de l'analyse par
génotype que dans l'analyse globale. Les valeurs des paramètres
nu_m et pi_f0 seront fixés dans ce qui
suit et seuls les paramètres tstar et Y_param
peuvent être ajustés parmi ces deux couples.
3.3.2 L
|
'analyse avec ACP
|
La méthode ACP (analyse en composantes principales),
consiste à transformer des variables liées entre elles (dites
"corrélées" en statistique) en nouvelles variables
décorrélées les unes des autres. Ces nouvelles variables
sont nommées "composantes principales", ou axes principaux. Elle permet
au praticien de réduire le nombre de variables et de rendre
l'information moins redondante [wikipedia]. Elle peut être appelée
par génotype sur la Table3.3 (voir la
Figure3.16) ou pour l'ensemble des génotypes (voir la
sous-figure "l'ensemble des génotypes" en bas sur la
Figure3.16).
Sur les sous figures, ACP sélectionne les deux axes qui
explique en pourcentage le plus de variance. Dans les cercles si deux
paramètres forment :
> Un angle inférieur à 90°,
ils sont en corrélation positive. Plus l'angle est fermé (l'angle
converge vers 0°) plus la corrélation positive est
forte,
> Un angle supérieur à 90°,
ils sont en corrélation négative. Plus l'angle est ouvert
(l'angle converge vers 180°), plus la corrélation
négative est forte,
47
> Un angle proche de 90?, ils
ne sont pas corrélés,
La partie à gauche des sous figures représente
la distribution des estimations sur les axes choisis. On trouve encore les
mêmes couples (tstar et n_m), en très
fortement corrélation négative et (Y _param et
pi_f0), très fortement corrélés
positivement, aussi bien sur l'analyse par génotype et que pour
l'ensemble des génotypes. Donc les résultats de cette
méthode confirment bien ceux de la fonction pairs. La question
maintenant, est de savoir à quelles valeurs fixer les paramètres
n_m et pi_f0?
A partir des 400 valeurs trouvées (50 valeurs pour
chacun des huit génotypes) pour les paramètres n_m
et pi_f0, on dessine les histogrammes associés
(la Figure3.17). Chaque histogramme présente la
distribution des valeurs prises par le paramètre désigné
et la médiane "median" associée se trouve dans
l'intervalle le plus fréquenté dans cette distribution. On va
fixer les paramètres aux valeurs de leurs médianes, soient donc
0.030 pour n_m et 7.36 pour
pi_f0. Cependant le fait de fixer un paramètre peut
dégrader la qualité d'ajustement et très probablement
certains génotypes deviennent quasiment non ajustables sur une des
courbes (MF ou MS) en condition témoin ou stressée. Pour
éviter ce problème, il a fallu construire un critère
permettant de prendre une décision sur une corrélation forte. La
décision soit oui (la corrélation sera prise en compte) soit non
(la corrélation ne sera pas prise en compte), (voir la sous section
suivante).
3.3.3 Le critère de décision sur une
corrélation forte
Pour ce faire, on trie les n (50 ici) compromis
trouvés par génotype du meilleur jusqu'au moins bon. Dès
que ce tri est fait par le critère de sélection par seuil
(sectin2.5.1.4), on dessine les valeurs des paramètres
à fixer en fonction des compromis triés et une droite horizontale
représentant la valeur de la médiane pour laquelle, il faut fixer
le paramètre désigné (la Figure3.18).
Rappelons que la valeur de la médiane est bien celle trouvée sur
400 valeurs (pour les huit génotypes), donc c'est une valeur globale
pour les histogrammes de paramètre associé et non locale. La
décision est oui, si la droite (la valeur de la médiane) simule
au moins une partie de l'ensemble des nuages des points de son paramètre
sur tous les huit génotypes et non en cas d'existence d'un
génotype qui ne vérifie pas cette contrainte. Donc d'après
la Figure3.18, la décision est oui pour les couples de
corrélations (tstar et n_m) et (Y
_param et pi_f0).
Remarque 3.2. La variable
(Val_de_critère_de_seuil) sur la Figure3.18 représente les
valeurs de la somme pondérée (car on applique l'ajustement
parallèle) dans la liste L_max (revoir encore la sectin2.5.1.4).
Maintenant, on fixe les paramètres n_m
et pi_f0 à leurs valeurs indiquées
à la section précédente, puis on ajuste les six
paramètres restants avec éventuellement la recherche des
nouvelles corrélations.
3.3.4 Analyser sur six paramètres
Cette analyse a été faite de la même
manière que celle sur huit paramètres, nous avons fait 50
estimations selon l'ajustement parallèle sur chacun de nos 8
génotypes. Chaque estimation a été trouvée
après avoir appelé NSGA-II sur la
Table3.4 (juste
48
??????????????? Paramètres Estimation
|
phi_max
|
Y_param
|
Lp
|
tstar
|
tau_a
|
el
|
pi_f0
|
n_m
|
|
Estimation1
|
a1,1
|
a1,2
|
..
|
..
|
..
|
...
|
...
|
a1,8
|
|
Estimation2
|
a2,1
|
a2,2
|
..
|
..
|
..
|
..
|
..
|
a2,8
|
|
..
|
..
|
..
|
..
|
..
|
..
|
..
|
..
|
..
|
|
Estimationi
|
ai,1
|
ai,2
|
..
|
..
|
..
|
..
|
..
|
ai,8
|
|
..
|
..
|
..
|
..
|
..
|
..
|
..
|
..
|
..
|
|
Estimation50
|
a50,1
|
a50,2
|
..
|
..
|
..
|
..
|
..
|
a50,8
|
TABLE 3.3 Les 50 estimations trouvées par génotype
pour faire une analyse sur huit paramètres.
|
Les paramètres de NSGA-II
|
Les valeurs des paramètres de NSGA-II
|
|
idim
|
6
|
|
odim
|
2
|
|
generations
|
50
|
|
popsize
|
100
|
|
cprob
|
0.7
|
|
cdist
|
5
|
|
mprob
|
0.1
|
|
mdist
|
10
|
TABLE 3.4 Tableau des valeurs de paramètres de NSGA-II
pour analyser sur six paramètres.
le paramètre idim qui a changé) par
génotype. D'après l'appel de la fonction pairs
(la Figure3.19) et la méthode ACP
(la Figure3.20), deux nouveaux couples fortement
corrélés aussi bien au niveau de l'analyse par génotype
que dans l'analyse globale sont apparus, (phi_max et Y
_param) et (tstar et el). Pour ces deux couples
de corrélations, il n'apparait pas possible de fixer l'un des
paramètres à la valeur de sa médiane sans prendre en
compte la qualité d'ajustement (voir la Figure3.21, la
décision du critère est non). Sur cette figure, deux
génotypes ne vérifient pas la contrainte du critère de
décision sur les quatre paramètres des ces deux couples des
corrélations fortes. Rappelons encore que la valeur de la médiane
est globale (la médiane sur les 400 valeurs des huit
génotypes).
Pour résumer, aucun paramètre n'a alors
été fixé dans cette analyse et les résultats finaux
(section suivante) sont obtenus en ajustant les six paramètres
restants.
3.4 Les résultats finaux
La tâche principale de ce travail est de proposer au
final la meilleure combinaison de paramètres permettant d'avoir le
meilleur ajustement parallèle par génotype. Pour trouver la
meilleure combinaison paramétrique par génotype, il faut donner
des tailles de

49
FIGURE 3.14 Les graphes des corrélations entre les huit
paramètres par génotype.

FIGURE 3.15 Les graphes des corrélations entre les
huit paramètres par génotype et sur l'ensemble des
génotypes.
50
Varlablea factor map (PCA)
|
Y -
|
individuels
factor map (PCA)
|
|
|
|
39
0 42
· GB -1
·I- ~8-_
. 23
·
P
|
1.0 -0.5 0.0 0.5
|
|
|
-4 -2 0 2 4
Dim 1 (32.37%)
Dim1 (3022%)
CeNI I Variables factor map (PCA)
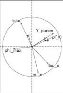
-1.0 -0.5 0.0 0.5 1.0
Dim 1 (30.22%)
Celle-ci est une sous figure
Individuals factor map (PCA) Levovil

Dim 1 (32.37%)
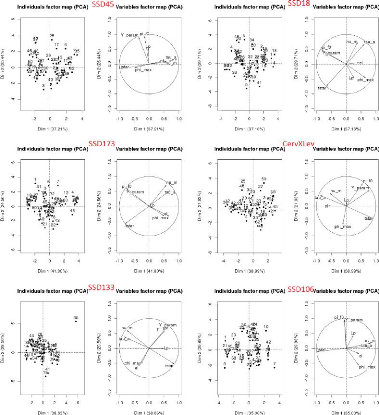
-2 0 2 4
1,0 -0.5 0.0 0.5 1.0
-1,0 -0.5 0.0 0.5 1.0 4 -2 0 2 4
Dim 1 (37.21%)
Dim 1 (37.21%)
Dim, (37.18%)
Dim 1 (37.18%)
48
1 8 7
31 3:
a2 12 4
· as'2 ).6° âti
5073
.2 0 2 4
Dim, (41.80%)
1.0 -2 5 0.0 0.5
Dim 1 (38.99%)
1.0
1.0 -0.5 OD 0.s 1.0 4 -2 0 2
Dim, (41.80%) Dim1 (38.99%)
Individuals factor map (PCA)
SSD45 Variables factor map (PCA)
Individual, factor map (PCA)
SSD18 Variables factor map (PCA)
SSD173
Individual, factor map (PCA) Variables factor map
(PCA)
CervXLev
Individual, factor map (PCA) Variables factor map
(PCA)
Individuals factor map (PCA)SSD133 Variables
factor map (PCA)
Individuels factor map (PCA)
SSD1Oâyariablea factor map (PCA)
Dim 1 (38.83%)
21994 10 23 4.0.t
9~~ 42
2145 3
· 33 u.
I
2 0 2 4
Dim, (35 00%)
1.0 -05 0.0 0.5 1'0
Dim 1 (35.00%)
24
É
E
~0 iT843
21 22
·
·
25 50
45 27
· ~°
· 33
qqaa
1 zz y
3fi2 2
·reeïT
·9 14b2i~ 7s ~
·
· 4 353
·
·
·
·
· 34 2, 2
Al2 6 32 a§
,,p4,~~ L+tA9
5
· 41
L'ensemble des génotypes
Individual, factor map (PCA) Variables factor map
(PCA)
p\(2
tau_ Y.peram
51
FIGURE 3.16 - Les graphes d'analyse en composantes principales
entre les huit paramètres par génotype et sur l'ensemble des
génotypes.
I l I l
4 -2 0 2 4
Dire 1 (27.87%)
1.0 -0.5 0.0 0.5 1.0
Dim 1 (27.87%)
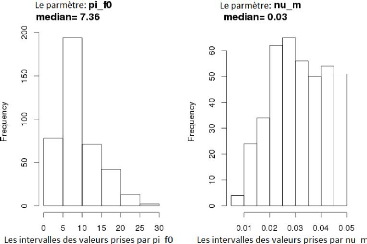
52
FIGURE 3.17 Les histogrammes obtenus pour num et pi_f0.
génération/population permettant à
l'algorithme NSGA-II de bien explorer l'espace de recherche vers le meilleur
front de Pareto possible. Après plusieurs tests, qui ont
été faits durant ce travail sur l'ensemble des génotypes,
le choix le plus judicieux qui a été défini est d'appeler
NSGA-II sur une génération de taille 250 et une population de
taille 400 par génotype. Avec ces tailles (voir la Table3.5),
on a fait 20 estimations par génotype (la Figure3.22
pour le génotype SSD45).
Pour faire le graphique associé à ces 20
meilleurs compromis, on a appliqué le principe de cluster (vu en
sectin2.1.1) sur les nuages de points de données
observées (MFobs, MSobs) en remplaçant chaque cluster par sa
moyenne et son écart-type (le point segment) puis on trace les 20
courbes associées aux MFpred et MSpred. Même chose pour les sous
graphiques associés aux teneurs en matière sèche (MS/MF)
en condition témoin et stress.
Sur le graphique associé à ces 20 meilleurs
compromis, on trouve que les courbes simulent presque les points-segment des
données observées de la même manière, ce qui prouve
la convergence de ces compromis vers la même zone du meilleur compromis
possible.
Au niveau des courbes des teneurs, on voit qu'elles n'ajustent
pas bien les données dans un premier temps et cela vient du fait que les
poids du fruit (MF, MS) sont très petits au début (assez
proches). Mais dès que ces poids commencent à croître, les
teneurs prédites simulent relativement bien les teneurs
observées.
Sur chacun des sous graphiques associés à la
convergence des paramètres (toujours la Figure3.22), on
voit bien que chaque paramètre converge presque vers la même
valeur (ou disons reste restreint dans le même petit intervalle), ce qui
nous permet de
|
ol
|
Levovll
|
|
Levovll
|
|
Coral!
|
ô
|
Cervll
|
o -- median
so
o o °o 0
° ô 0
0
tô
|
° --maan
0 0
0 00
°°° ° 0
0 0 °
°o a o0
|
-- median
ô
0 0 0
0 0 0
° 0 0 ° o
0 0
0
|
oe -- median
oao 0
0 0 °
o 0
o$
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
o
|
|
8 10 12 14 18
|
8 10 12 14 18
|
11 12 13 14
|
11 12 13 14
|
|
Val de critere de seuil Val de critere de seuil
|
Val_de_critere_de_seu I I
|
Vol_de_critere_de_seu I I
|
SSD173 SSD173 CervXLev CervXLev

median
median
0 0° o 0
o° 0
8
°°
p0 0 0
00 0
° o
0 0
08 ° ZP°0 o g2/0 0
00
0 0
median
o
00 8 0
o o A
°° O
0
0
0 00ô0ô
~o
° 0
.0.4%
GO ° ° o0 0
° ° o
13 14 15 16 17 18 19
Vol_de_critare_de_seu i l
13 14 15 18 17 18 19
V al_de_critere_de_seuil
9 8 10 12 14 8 8 10 12 14
Val_de_critere_de_seu i l Vol_de_critere_de_seu i l
E o
SSD133
-- madian
oYd g~o
moo®
° 0 0
9 10 11 12 13
Val_de_critere_de_seu I I
SSD133

00 0
ô °
0 0
o °9 0 0o 0ô 0 0
o 0
0 o
o
median
9 10 11 12 13
V al_de_critere_de_sauil
SSD106
median
04 o° °
° ° o
0
10 12
Val_de_critere_de_seu I I
SSD106

median
0 °
8 10 12
Val_de_critere_de_seu il
53
FIGURE 3.18 La convergence des num et pi f0.





Levavll
Cervll
50 150 250
0 05 0 15
0 05 0 20
0.83
phi_max
phi_max
0.62
0.34
:fl
Y_param
0.70
Y_param
0.51
KIM
Lp
Lp
0.99
0.93
0.87
0.86
tatar
tatar
0.81
tau_a
tau_a
0.62
el
el
0 05 0 15
SSD45
SSD18
0.87
phi_max
0.81
phi_max
0.48
0.50
com
0.40
0 35
Y_param
Y_param
0.61
0.50
0.52
0.54
044
0.44
0.49
8 ° m8
;bEAe a Wi-rr~-rr
Lp
Lp
0.98
0.91
0.98
0.77
tatar
tatar
0.83
tau_a
tau_a
0.63
el
el
SSD173
CervXLev
150 250 950
005 0 15 025
0.81
phi_max
phi_max
0.71
054
0.43
0.40
0.43
0.40
Y_param
Y_param
0.49
0.59
0.55
0.57
v ldI
°ôd
oo 8
o. o. o oo t,o o
Lp
Lp
054
0.46
ria
0.99
0.98
0.88
0.83
tatar
tatar
0.81
tau_a
tau_a
0.72
el
el
SSD133
SSD106
100 200

0 15 0 25
0.88
phi_max
phi_max
0.44
0,42
0.49
0.40
Y_param
0.71
0.70
Y_param
0.61
0.64
052
0.47
044
Lp
Lp
0.52
0.46
â
· e
002 001 014 04 Od 01 10
0.93
0.84
0.83
0.80
tatar
tatar
tau_a
tau_a
r
el
el
200 240
120 150 200
L'ensemble des genotypes
phi_max
0.64
Y_param
0.58
0 07
!>
i
Lp
0.80
tatar
rr 711
tau_a
el
·
00 04 08
54
FIGURE 3.19 Les graphes des corrélations entre les six
paramètres par génotype et sur l'ensemble des
génotypes.
Individuals factor map (PCA) CONS Variables factor map
(PCA)
Individual. factor map (PCA) sols Variable. factor map
(PCA)
|
|
|
Dim 1 (80.78%)
|
|
Dim 1 (83.]5%)
|
|

Individuals factor map (PCA) Levovil
Variables factor map (PCA)
38 31
.43
·
24 10
·
· 512 54),
4,21
331.- 1,! -2 - 3.
·zé
·
1 ..138
48
· 4 08 .0
· ,Ar5
·
r
.25
-4 -2 0 2
Dlm 1 (48.94%)
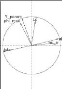
1.0 -05 00 0.5 1.0
Dim 1 (48. 94%)
Indlvlduala factor map (PCA) 555 ° Variables factor
map (PCA)
1.0 -0.5 00 0.5 1.0
Dim 1 (5792%) Dim 1 (5792%)
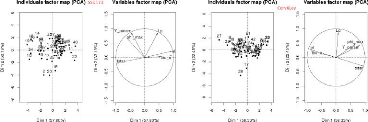
1.0 -0.9 00 0.5 110
Dim 1 (58.33%)
8 -0 -2 0 2 4 Dim 1 (58.33%)
Indlvlduala factor map (PCA) 55 c^ Variables factor map
(PCA)
Individual. factor map (PCA) Variable. factor map
(PCA)
EervxLev
Dim 1 (57.80%)
Dim 1 (5].80%)
10 -5 5 00 0.5 1.8
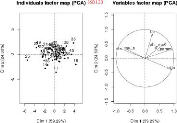
Indlvlduala factor map (PCA) 554133
Variables factor map (PCA)
Individuals factor map (PCA) OSE100
Variable. factor map (PCA)
E ô 9
-8 -4 -2 0 2 4 Dim 1 (59.29%)
1.0 -0.5 0.0 0.s 10
Dim 1 (59.29%)

-4 -2 0 2 4
Dim 1 (52.83%)
Yyera
E
1.0 -0.s 00 5.s 10
Dim 1 (52 83%)
N
5-
L'ensemble des génotypes
Indlvlduala factor map (PCA) Variables factor map
(PCA)
FIGURE 3.20 - Les graphes d'analyse en composantes principales
entre les six paramètres par génotype et sur l'ensemble des
génotypes.
-4 -2 0 2 4
|
Dim 1 (3829%)
|
55
|
1.0 -0.5 0.0 0.5 1.0
Dlm l (38.29%)
|
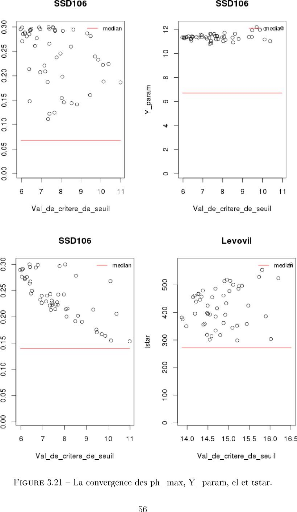
SSD106
I I I I
6 7 8 9 10 11
Val de critere de seuil Val de critere de seuil
SSD106 Levovil
a
0 D -- median
o
C}
in
C1--
6 e 00D o0
Q 00 O
N - 0 0
d 0 O
o o
0
a -
d Î I I I I I
6 7 8 9 10 11
14.0 14.5 15.0 15.5 16.0 16.5
Val de critere de seuil Val de critere de seuil
FIGURE 3.21 -- La convergence des ph max, Yparam, el et tstar.
56
1
9
6 7 8
SSD106
Q
median
·
0o
o
CV
Q
a
{V --
m 0
D 00
0 p0
Q
0 0 00
o
00
a
d
o
a
d
114-7
d
LU
a
0
d
edia0
Vie: Opt
0 O OD
ç
·
2 POP
a,
cv
0
CO -
a --
06, 0
o
c
o
o
o
0 a -
mediar
o
0
m 0 0 0
· 0 ki0
o 0 co
o
CV
a
a -
0 a -
57
|
Les paramètres de NSGA-II
|
Les valeurs des paramètres de NSGA-II
|
|
idim
|
6
|
|
odim
|
2
|
|
generations
|
250
|
|
popsize
|
400
|
|
cprob
|
0.9
|
|
cdist
|
20
|
|
mprob
|
0.1
|
|
mdist
|
20
|
TABLE 3.5 Tableau des valeurs de paramètres de NSGA-II
pour les résultats finaux.
sélectionner le meilleur de ces 20 compromis ayant la
meilleure combinaison possible de paramètres, c'est-à-dire qui
permet d'avoir le meilleur ajustement parallèle
(Figure3.23).
Pour résumer, on appliquera le même traitement
(20 estimations par génotype, puis la sélection du meilleur des
20 meilleurs) sur l'ensemble des génotypes, qui seront traités
dans le grand document.
Remarque 3.3. Sur les résultats
finaux de 37 génotypes qui ont été déjà
traités, on a refait encore l'analyse de la variabilité des
paramètres et la Figure3.25 confirme
l'invalidité des corrélations trouvées dans la
Figure3.24. Ce qui nous confirme encore ce qui a
été trouvé en sectin3.3 pour ces
corrélations.
Le graphique associe aux 20 estimations pour le genotype
SSD45
10 20 30 40 50
10 20 30 40
50
O
o
N
O
Teneur en MS [gig]
O
o
o O
If1
0 N
o
Teneur en MS [gr'g]
o
|
|
|
|
|
|
|
|
|
|
|
|
|
o
N
LL
· -
0
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
u-
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
O
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
I I I I
|
|
|
|
10 20 30 40 50
|
o ri
N
o
_ N co 0
T2 r
o
o
O
o
N
o
N
~
· r
o
o
o
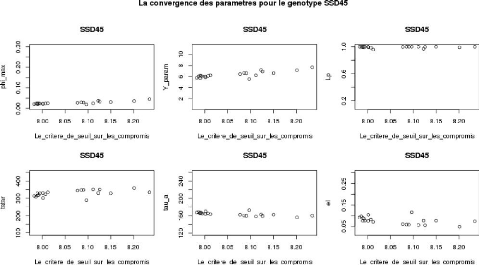
o
aari D 6 O O 4
La convergence des pa ra metres pour le genotype
SSD45
o
o
O
o
E m
N -
0% ô°%
o
0 N
o
o
I
E
E
SSD45
SSD45
6.00 8.05 8.10 8.15 8.20
Le_crite re_de_se u il_su r_les_co m pro m s
SSD45
D
o
o -
6.00 8.05 2.10 8.15 8.20
Le_crite re_de_se u il_su r_les_co m pro m s
O
N -
8.00 6.05 6.10 6.15 8.20
Le_crite re_de_se u il_su r_les_ca m prom s
8.00 6.05 6.10 6.15 8.20
Le_crite re_de_se u il_su r_les_ca m prom s
SSD45
N
O
o
40 0on 0
pdD 0
o
ow O 4 O 0 o
cktEtop
SSD45
8.00 2.05 8.10 8.15 8.20
Le_crite re_de_se u il_su r_les_ca m prom s
SSD45
8.00 8.05 8.10 8.15 8.20
.131
Le_crite re_de_se u il_su r_les_ca m prom s
O
6
N -
0
0
0 -
o
m
0 4 O a
m
0
N _ ô
58
FIGURE 3.22 Les 20 meilleurs compromis pour SSD45.
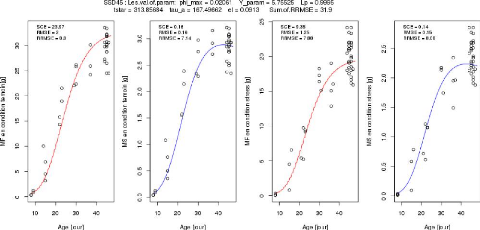
00
4 d
SCE-L35RMSE-125 RRMSE-71!
55045 : Les.val.of.param: phi_rrax = 0.02061 Yjram =
5.75525 Lp = 0.9995
tstar = 313.85684 tau a = 16749662 el = 0.0913 Sum.of.RRMSE =
31.9
10 20 30 40
Age [our]
10 20 30 40 10 20 30 40
Auge []our] Age Dur]
MF en condition temoin[g]
MS en condition temoin [g]
MF en condition stress [g]
Age [pur]
MS en condition stress [g]
10 20 30 40
D
u_
u_ en-
teneur simule
Le genotype 991345 en condition temoin
Le genotype SSD45 en condition stress
teneur simule
tn
o _
a I I I I
59
10 20 30 40 50 60 10 20 30 40 50 60
Temps [jour] Temps [jour]
FIGURE 3.23 Le meilleur des 20 meilleurs compromis pour SSD45.
Sur 37 genotypes

phi_max
0 4 e
I I L
0.62
Y_param
0.00 0.15 0.30
0.89
_o
0.0 0.4 0.8
150 250 350
tstar
0.27
el
0.00 0.15 0.30
I
0.46
0.49
O
O
0 200 400
11 I I I 1
0.37
0.47
Lp
5 15
aiG
Individuals factor map (PCA) Variables factor map
(PCA)
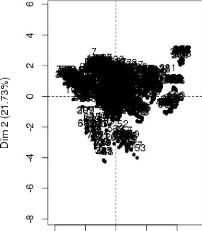
I I I I
Dim 2 (21.73%)
0
cv
00 --
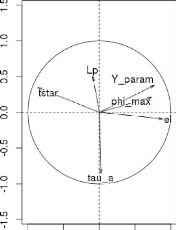
L(
rn
L(.
Lp
Y_param
tstar
ph.iimax
ta __a
I I
-4 -2 Cl 2 4 -1.0 -0.5 0.0 0.5 1.0
OH 1 (44,47%) Dim 1 (44.47%)
60
FIGURE 3.24 -- Les graphes des corrélations et
d'analyse en composantes principales entre les six paramètres sur 37
génotypes.
61
SSD1O6
|
|
SSD1O6
|
|
|
|
|
-
ô
u,
N --
O O
N --
O
ce
E _
ç O
Q
O O
u,
4 --O
O
O 0
|
medi
|
E
a
Q
}
|
0
03
m
N --
O -
|
(=MO ® n media
|
|
|
|
I I I 1 I I
|
|
5.94 5.98 6.02
|
5.94 5.98 6.02
|
|
Val de critere de seuil
|
Val de critere de seuil
|
|
SSD1O6
|
Levovil
|
|
0 en --ô
u,
|
|
|
0
O --Ln
|
|
q 0 median
q 0 c7
|
median
|
|
N --
|
|
|
|
|
|
O
|
|
|
|
|
|
|
|
O -
|
D
|
|
0
|
|
|
|
0
|
|
--
O
|
|
|
|
|
|
|
|
O
|
|
|
|
|
O -
|
|
|
Ln
|
|
ce
|
|
|
|
O
|
|
.N.
|
|
|
|
|
|
O
|
|
|
O
|
|
|
O --
N
|
|
|
4
u,
O --
|
|
|
O
O -
|
|
|
O
|
|
|
|
|
|
4
|
|
|
|
|
|
O -
|
|
|
O -
|
|
|
O
|
|
|
|
I I I I I
|
5.94 5.98 6.02 13.80 13.90 14.00
Val de critere de seuil Val de critere de seuil
FIGURE 3.25 -- La convergence des ph max, Yparam, el et tstar
dans les résultats finaux.
62
Chapitre 4
Synthèse de la solution apportée
Dans le cadre de ce projet, nous avons vu que le modèle
de croissance du fruit ne permettait pas de simuler correctement
l'évolution au cours du temps des deux variables MF et MS de
manière simultanée en condition témoin et stress. Par
conséquent, le premier objectif du stage était de réaliser
la procédure d'ajustement indépendant (sectin3.1)
sur les huit paramètres ayant un fort impact sur les variables
à prédites pour l'ensemble des génotypes.
Pour cela, nous avons défini une fonction objectif
(sectin2.1.1) pour chacune des variables MF et MS. L'objectif
de cette fonction était de nous permettre de minimiser les écarts
entre les données simulées et les données
observées. Par la suite, nous avons construit une fonction
multi-objectif (sectin2.2.1) ayant comme sortie ces deux
fonctions objectifs. Pour minimiser cette dernière, nous avons
utilisé l'algorithme NSGA-II (sectin2.3) qui nous
permettait de trouver le front de Pareto contenant les meilleurs compromis
entre les deux fonctions objectifs de MF et MS. Enfin, nous avons
développé trois critères (sectin2.5.1)
permettant de sélectionner le meilleur des meilleurs compromis
se trouvant sur le front de Pareto et ajustant les données
observées.
Dès que cet objectif était atteint, le
deuxième objectif a consisté à développer une
procédure d'ajustement parallèle (sectin3.2).
Pour cela, on a appliqué le même principe que pour la
procédure précédente mais il a fallu régler
auparavant trois problèmes majeurs : > Faire que le
modèle puisse être appelé parallèlement sur les deux
conditions en même temps. En effet le modèle pour l'instant ne
peut être appelé que pour une seule condition et la solution la
mieux adaptée à ce problème est de faire deux appels
successifs pour les deux traitements témoin et stress dans le corps de
la fonction multi-objectif, ce qui a augmenté fortement le temps de
calcul.
> Un problème au niveau d'une
équation dans le corps du modèle liée à
l'évolution de la variable MSpred, qui a été
réglé par l'équipe et nous a fait changer la version du
modèle. > Et le problème de NSGA-II
(Remarque2.2), qu'on a résolut grâce à la somme
pondérée (sectin2.4.1).
Le schéma1 sur la Figure4.1
illustre les étapes nécessaires pour arriver à
ces deux objectifs (les deux procédures d'ajustements).
63
Ensuite un troisième objectif est l'analyse de la
variabilité des paramètres (sectins3.3).
Étant donné que cette analyse visait à
réduire le nombre de paramètres à ajuster mais aussi
à déterminer les paramètres génétiques du
modèle. Pour ce faire, on a travaillé sur la recherche de
l'existence de corrélations fortes entre ces paramètres en
utilisant la fonction pairs (sectin3.3.1) et la méthode
ACP (sectin3.3.2). On vérifie tout d'abord si les deux
méthodes indiquent une corrélation forte entre les
paramètres. Si la corrélation est forte, un critère de
décision (sectin3.3.3) a été construit
pour voir si la fixation d'un des paramètres n'engendrait pas une forte
dégradation de la qualité de l'ajustement. La corrélation
forte est rejetée dans deux cas, un cas de désaccord entre les
méthodes (pairs et ACP) ou un cas d'une réponse négative
au test du critère de décision. Le schéma2
sur la Figure4.1 résume les étapes
nécessaires pour faire cette analyse.
Enfin ces trois objectifs se résument à un seul
objectif principal, trouver la meilleure combinaisons de paramètres, qui
permet d'avoir le meilleur ajustement parallèle par génotype
(sectin3.4). En effet, cet objectif nous a permis d'aboutir
aux résultats finaux sur l'ensemble des génotypes de la
manière suivante, appliquant sur tout génotype un ajustement
parallèle couplé au critère de sélection par seuil
(sectin2.5.1.4), qui fait l'appel à NSGA-II
sur une génération de taille 250 et une population de
taille 400 (revoir la Table3.5). Ensuite le critère de
sélection par seuil, nous permet de sélectionner le meilleur des
meilleurs compromis, après avoir atteint 20 estimations (une seule
estimation donne un meilleur compromis) par génotype. Le
schéma3 sur la Figure4.1 illustre les
processus permettant d'atteindre ce dernier objectif.
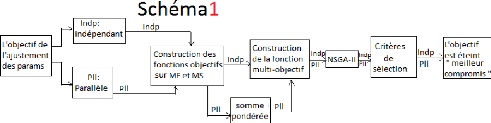
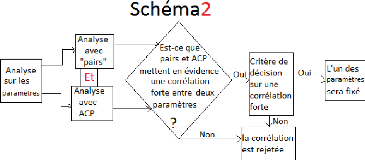
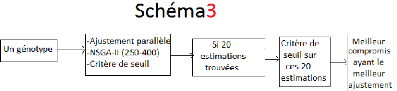
64
FIGURE 4.1 Les schémas d'illustrations sur les
objectifs atteints dans ce travail.
65
Chapitre 5
Conclusions
5.1 Résultats obtenus
L'objectif principal du stage a été suivi dans
un premier temps via l'ajustement indépendant (sectin3.1)
de 8 paramètres en obtenant 16 valeurs par génotype.
Dans un deuxième temps un ajustement parallèle
(sectin3.2) a permis de réduire le nombre de
paramètres de 16 à 8 par génotype. Ensuite grâce
à l'analyse de la variabilité des paramètres
(sectin3.3), on a pu réduire encore le nombre des
paramètres à ajuster de 8 à 6. Et au final l'objectif
principal du stage à savoir, estimer les paramètres du
modèle de croissance du fruit pour simuler correctement
l'évolution au cours du temps des deux variables MF et MS pour
l'ensemble des génotypes a été atteint via l'ajustement
parallèle de six paramètres.
5.2 Difficultés rencontrées
Les principales difficultés du stage étaient de
pouvoir s'adapter aux situations de la vie en laboratoire de recherche,
découvrir et exploiter les travaux précédents sur le
modèle dans un court laps de temps, comprendre le vocabulaire de
biologie et travailler avec l'outil de programmation R (exigé par
l'équipe) que je n'aurais jamais utilisé auparavant. Du
coté technique les trois problèmes évoqués en
chapitre4, m'ont pris la majorité du temps. Il a fallu
découvrir chacun d'eux après un long temps et
éventuellement chercher la solution la mieux adaptée.
5.3 Apports
5.3.1 Pour l'entreprise
Le bilan de ce stage est positif, les principaux objectifs
(sectin1.3.3) du projet sont accomplis. Désormais, le
modèle de croissance du fruit permet de simuler correctement
l'évolution au cours du temps des deux variables MF et MS pour
l'ensemble des génotypes via l'ajustement parallèle
(sectin3.2) de six paramètres. En conséquence,
nous pouvons dire, sans aucune prétention, que le modèle
développé est un outil efficace pour remplir plusieurs objectifs
envisagés dans le cadre du projet TOMSEC (conception
d'idéotypes
66
culturaux adaptés à la contrainte hydrique et
améliorés sur des critères de qualité gustative et
nutritionnelle), notamment les deux objectifs suivants :
> Utiliser le modèle pour prédire le
comportement de différentes combinaisons
allé-liques/paramètres génétiques dans des
conditions d'alimentation en eau variables. On suppose que les valeurs des
paramètres du modèle dépendent des allèles
présents chez les génotypes.
> Proposer des combinaisons de paramètres
génétiques permettant d'optimiser le rendement (MS/MF) et la
qualité des fruits dans des conditions variables d'environnement. Il
s'agit de trouver par l'optimisation multi-objectif les combinaisons de
paramètres qui permettent un calibre et une teneur en MS
répondant aux exigences du consommateur et ce, dans des conditions
respectueuses de l'environnement.
5.3.2 Apport personnel
Ce stage était l'occasion de me plonger dans un milieu
professionnel où j'ai eu la chance de travailler au sein d'une
équipe de recherche très accueillante et chaleureuse, ce qui me
permettait de rencontrer des chercheurs et de partager leurs
expériences. Ce stage m'a ainsi permis de mettre en pratique les
connaissances théoriques et pratiques acquises lors de ma formation, et
d'acquérir un nouvel outil de programmation R qui est très
utilisé dans le monde professionnel et surtout les centres de recherche
et de calcul là où ma formation est orientée. Il m'a
permis également d'approfondir mes connaissances dans le domaine de
l'optimisation multi-objectif qui est aussi très demandée
aujourd'hui dans la plupart des secteurs, soit il peut s'agir de minimiser un
coût de production, d'optimiser le parcours d'un véhicule,
d'améliorer les performances d'un circuit électronique, d'affiner
un modèle de calcul, de fournir une aide à la décision
à des managers, etc. Enfin ce stage est une expérience
enrichissante, qui me donne un aperçu du travail dans le milieu de la
recherche.
67
Chapitre 6
Bibliographie
6.1 Références bibliographiques
[R1] : J.BONNEFOI (2013). Phénotypage d'une population
de tomate dans deux conditions d'alimentation en eau pour le calibrage d'un
modèle de qualité du fruit et l'analyse QTL de paramètres
de modèles: Mémoire fin d'étude. UR 1115 PSH, INRA
d'Avignon, France.
[R2] : M-M.Ould-Sidi, F.Lescourret (2010). Model-based design
of integrated production systems : a review. PSH, INRA d'Avignon, France.
[R3] : B.Quilot-Turnion, M-M.Ould-Sidi, A.Kadrani, N.Hilgert,
M.Génard, F.Lescourrt (2012). Optimization of parameters of the "Virtual
Fruit" model ton design peach genotype of sustainable production systems.
INRA-Avignon-Montpellier, France.
[R4] : K. Deb and A. Pratap and S. Agarwal and T. Meyarivan,
A fast and elitist mul-tiobjective genetic algorithm : NSGA-II, IEEE T. Evolut.
Computat. 6, 182197, 2002.
[R5] : X.Li, L.Amodeo, F.Yalaoui, H.Chehade
(210).METAHEURISTIQUES MULTIOB-JECTIF POUR UN PROBLEME D'ORDONNANCEMENT DE
MACHINES PARAL-LELES. Institut Charles Delaunay, LOSI, STMR. Page5.
[R6] : M.MÈndez, D.Greiner, B.Galvan (2010).
Algorithme Èvolutionnaire multi-objectif qui surélève la
région d'intérêt. Toulouse.
[R7] : C.Chatelain, Y.Oufella, S.Adam, Y.Lecourtier, L.Heutte
(2008). Optimisation multi-objectif pour la sélection de modèles
SVM. Laboratoire LITIS, Université de Rouen, Avenue de
l'université, France.
[R8] : Y.OUAZENE, H.CHEHADE, A.YALAOUI. Approches mono et
multi-objectives pour la conception d'un système de production à
deux machines et un stock-tampon. Université de Technologie de Troyes,
10010 Troyes cedex, FRANCE. Page19.
[R9] : S.Amédée, R.Francois-Gérard
(2004). ALGORITHMES GENETIQUES : TE de
68
fin d'année.
[R10] : T.Gräbener, A.Berro (2008). Optimisation
multiobjectif discrète par propagation de contraintes.
IRIT-Université de Toulouse
[R11] : M.Yagoubi (2012). Optimisation évolutionnaire
multi-objectif parallèle : application à la combustion Diesel.
Université de Paris Sud XI
[R12] : M.Schoenauer (2009). Techniques avancées. Equipe
TAO -INRIA Futurs et LRI . Page48.
[R13] : A.Berro (2008). Algorithmeévolutionnaire
Algorithme évolutionnaire pour l'optimisation multiobjectif. Toulouse,
France.
[R14] : M-M.Ould-Sidi, B.Quilot-Turion, A.Rolland.
Méthodes multicritères pour le tri de fruits virtuels.
INRA-Avignon, Bron Cedex, France.
[R15] : P-L.GONZALEZ. L'analyse en composantes principales
(A.C.P).
[R16] : D.Puthier (2009). Introduction au logiciel d'analyse
statistique R. MARSEILLE cedex 09, FRANCE. Les pages 34-35.
6.2 Webgraphies
[W1] : http ://
fr.wikipedia.org/wiki/
[W2] : http ://
pinville.com/algorithmes-evolutionnistes.php
[W3] : http ://
fr.slideshare.net/paskorn/rnsgaii-presentation.
Le point3.
[W4] : http ://
www.recherche.enac.fr/opti/papers/thesis/HABIT/main002.html
[W5] : http ://
www.r-project.org/other-docs.html
[W6] : http ://
www.unige.ch/ses/sococ/cl/r/scatmat.f.html
[W7] : http ://www.lirmm.fr/
rgirou/enseignement/pageenseignement.html
[W8] : http ://
www.inside-r.org/packages/cran/mco/docs/nsga2
| 


