Généralités :
Etudier le polymorphisme génétique des virus de
TYLCV sur la tomate par la technique PCR.
Identifier les virus TYLCV de la tomate par la technique de
PCR.
Déterminer les fréquences des différentes
souches de TYLCV présentes dans la zone d'étude.
Evaluer le polymorphisme génétique des virus
TYLCV dans la zone d'étude.
Etablir la relation entre les symptômes et le type de
virus.
Matériels et Méthodes
Echantillonnage : La collecte des
feuilles sera faite dans toutes les zones d'étude et les feuilles sont
stockée à -20°C avant l'extraction de l'ADN.
Extraction : l'ADN sera extrait
à partir des feuilles infectées de tomate selon la méthode
CTAB 3%).
A chaque tube, contenant 1 ou 2 centimètres de feuille
de tomate infectée, 500ul du tampon de CTAB (100mM de Tris PH 8.0, 50mM
d ; EDTA, 500mM de Na Cl) sera ajoutée puis incubée à
65 ° C pendant 10 minutes. Ajouté 400ul de chloroforme pour centrifuger
à 10000rpm durant 10 mn. Après centrifugation
récupérer le surnageant dans un autre tube contenant de
l'isopropanol pour précipiter l'ADN. Apres lavage avec l'éthanol
70%, 200ul de TE est ajouté pour la PCR et sa conservation.
Amplification par PCR des feuilles collectées.
Les conditions du thermocycleur sont optimisées pour la
détection des souches Israélite et Sardingue avec les amorces
spécifiques aux trois isolats (TYLCV, X15656, TYLCSV-ES [2], L27708
etTYLCV-M1d, X76319). Les paramètres pour la réaction de PCR sont
optimisés pour un volume de 25 ul. La concentration finale des
composantes de réaction : 2ul dNTPs, 2.5ul Buffer 10X PCR, 0.25ul
de Taq Polymerase, 1ul de l'amorce sens et 1ul de l'anti-sens et 1ul
d'ADN. Les paramètres de cycle sont comme suit : un cycle à
94° C pendant 10 minutes; 30 cycles à 94°C pendant pour1 minute, 58°C
pour 1minute, 72°C pour 1minute, 72°C pendant 10 minutes et enfin 4°C pour
toujours .
Résultats Sur un total de
quarante cinq échantillons, vingt quatre ont été
traités par la technique de multiplex PCR. Chaque échantillon a
été amplifié par les trois paires d'amorces, quinze se
sont révélés positifs et neufs négatifs.
Tableau IV : distribution de la taille des bandes aux
isolats et la répartition des virus
|
TYLCV
|
TYLCSV-ES [2]
|
TYLCV Mld
|
|
N
|
Présence
|
6
|
15
|
6
|
|
Absence
|
18
|
9
|
18
|
|
Moyenne de la taille des bandes
|
629.1667
|
386.4667
|
284.5000
|
|
Minimum de la taille des bandes
|
618.00
|
360.00
|
276.00
|
|
Maximum de la taille des bandes
|
638.00
|
401.00
|
290.00
|
Le tableau IV montre la répartition des
échantillons et la taille des fragments observés aux trois
isolats ainsi que les variations de la taille des bandes par les
différentes PCR.
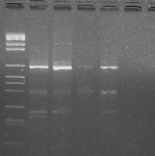
MVI 1 2 3 4
629pb
TYLCV
Figure 2: gel d'agarose présentant les bandes
amplifiées par les marqueurs de TYLCV
La figure2 présente les bandes de TYLCV sur gel
d'agarose dont la taille est estimée à 629 pb. Chacun des
échantillons au niveau des puits 1, 2, 3 et 4 présentent une
bande unique. Le premier puits contient le marqueur de poids moléculaire
VI (Roche Diagnostics Corporation, Mannheim, RFA)
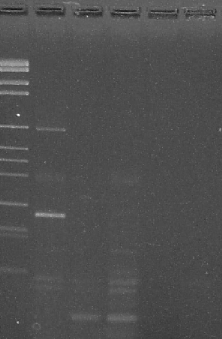
MVI 1 2 3
284pb
TYLCVMld
Figure 3: gel d'agarose montrant la bande de
l'amplification attendue par les marqueurs de TYLCVM1d
La figure3 présente une bande de TYLCVMld sur gel
d'agarose dont la taille est estimée à 284 pb. Le premier puits
contient le marqueur de poids moléculaire VI (Roche Diagnostics
Corporation, Mannheim, RFA)
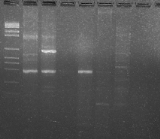
MVI 1 2 3
4 5 6
386pb
TYLCSV-ES [2]
Figure 4: gel d'agarose montrant l'amplification de
trois échantillons avec les marqueurs TYLCSV-ES [2]
La figure4 présentes les bandes de TYLCSV-ES [2] sur
gel d'agarose dont la taille moyenne est estimée à 386 pb. Les
échantillons 1, 2, 4 et 5 ont produit des bandes qui correspondent au
génotype TYLCSV-ES [2]. Le premier puits contient le marqueur de poids
moléculaire VI (Roche Diagnostics Corporation, Mannheim, RFA)
Tableau V : La fréquence relative des
génotypes obtenus par PCR
|
souches
|
Isolats
|
Positivité des échantillons par PCR
|
Fréquences
|
|
Souche Israélite
|
TYLCV
|
9
|
37.5%
|
|
TYLCVMld
|
|
Souche Sardinia virus
|
TYLCSV-ES [2]
|
15
|
62.5%
|
Le tableau V présente la fréquence des
génotypes obtenus par amplification par PCR par les amorces TYLCV ;
TYLCVMld (Souche israélite) et TYLCSV-ES [2] (souche Sardingue). Nous
avons observé que la souche Sardingue était la plus
fréquemment retrouvée (62,5) soit 15/24.
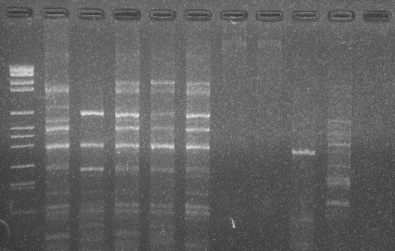
MVI 1 2 3 4 5
6 7 8 9 10
TYLCV
Non Spécifique
TYLCSV-ES [2]
TYLCVMld
Figure 5: Polymorphismes du TYLCV sur gel d'agarose. Cette
photo présente les bandes des trois isolats sur gel d'agarose
amplifiées simultanément avec les 3 paires d'amorce dans le
même test tube. Il faut noter que bandes non spécifiques sont
visibles sur le gel d'agarose. Le premier puits contient le marqueur de poids
moléculaire VI (Roche Diagnostics Corporation, Mannheim).
Tableau VI : fréquence et pourcentage des types
d'infections
|
Type d'infection
|
Fréquence
|
Pourcentage
|
|
Mono infection
|
9
|
37,5
|
|
PCR négative
|
9
|
37.5
|
|
Poly infection
|
6
|
25,00
|
|
Total
|
24
|
100,00
|
Dans le tableau VI, nous avons reparti les échantillons
selon qu'une seule (mono - infection) ou plusieurs bandes (poly-infection) sont
observées avec les trois amorce données.
Nous n'avons pas pu examiner des échantillons à
cause de la négativité des PCR.
Un quart de l'infection est multiple avec la présence
de plus d'un génotype alors que la mono infection était de 37,5%.
Nous notons que neuf échantillons sont restés
indéterminés parce que leur amplification a été
négative.
Tableau VII : Distribution des mono et poly infections
aux différentes souches.
|
souches
|
Isolâtes
|
Mono infection
|
Poly infection
|
fréquence
|
Pourcentage
|
|
Israélite
|
TYLCV
|
0
|
6
|
6
|
40%
|
|
TYLCV Mld
|
0
|
6
|
|
Sardingue
|
TYLCSV-ES [2]
|
9
|
6
|
9
|
60%
|
|
Total
|
|
9
|
6
|
15
|
100%
|
Le tableau VII montre qu'avec l'amorce TYLCV, plusieurs
génotypes ont été observés simultanément sur
les pieds de tomate avec une fréquence de 40%, aucun cas d'infection
mono spécifique n'est observer. Par contre, l'amorce TYLCV-ES [2]
présente des infections monoclonales (60%) et poly clonales (40%).
Tableau VIII : Relation entre les symptômes et les
types de virus
|
Souches
|
Isolats
|
Symptômes
|
|
Enroulement de la feuille
|
Rabougrissement
|
Variation de couleur des feuilles
|
|
Israélite
|
TYLCV
|
-
|
+
|
+
|
|
TYLCVMld
|
-
|
+
|
+
|
|
Sardingue
|
TYLCSV-ES [2]
|
+
|
+
|
-
|
Le tableau VIII donne la relation entre les symptômes et
les souches virales.
L'examen de la relation entre les symptômes et les
génotype montre que les génotypes Sardingue ne présentent
pas de variation de couleur mais des feuilles enroulées et rabougries et
cela de façon statistiquement significative (X2
= 13,333 et p = 0.001) tandis que les génotypes
israélites ne présentent pas de feuilles enroulées mais
des plants rabougris dont les couleurs varient. Cette relation n'était
pas statistiquement significative (X2 = 4,126 et p =
0,078).
| 


