CHAPITRE 3
Etude bio-informatique :
sélection de 3 domaines PRODH
Lorsque nous avons débuté l'étude
structurale de PRODH, il n'existait aucune
structure connue de proline déshydrogénase
d'un quelconque organisme. La publication en février 2003 de la
première structure d'un domaine PRODH chez E. coli (PutA669)
nous a permis d'aborder une stratégie structurale alternative qui
consiste à sélectionner des domaines PRODH humains à
partir de cette structure et de prédictions bio-informatiques.
La démarche consiste dans un premier temps
à prédire la délimitation du domaine catalytique
humain en reportant les éléments de structure secondaire
de PutA669 sur l'alignement de séquences qui a été
présenté dans le chapitre 1. Ceci permet de caractériser
l'organisation des domaines de la protéine humaine de
manière prédictive, et ainsi de restreindre notre champ
d'investigation à un ou plusieurs domaines supposés
structuralement indépendants. Un certain nombre d'outils
bio-informatiques de prédiction de structure secondaire, et de
visualisation des résidus hydrophobes, sont alors utilisés afin
de délimiter de manière précise les domaines
structuraux sélectionnés. Avant de présenter les
résultats de cette étude bio-informatique, qui a
été menée en étroite collaboration avec le
Dr. Sophie Zinn-Justin du DIEP, la structure du domaine proline
déshydrogénase de E. coli sera brièvement
décrite.
1) Description de la structure du domaine PRODH de E.
coli
Comme je l'ai évoqué dans le premier chapitre, la
protéine de liaison à l'ADN PutA
est l'orthologue procaryote de PRODH humaine chez E.
coli. La structure des 669 résidus
N-terminaux de cette protéine (PutA669), qui
correspondent au domaine proline déshydrogénase de E.
coli, a été résolue par cristallographie des rayons X
(Lee et al., 2003). Dans cette structure, PutA669 est un homodimère
composé de 3 domaines : un domaine N- terminal de dimérisation
(87-139), un second domaine N-terminal de fonction inconnue (140-
260), et un domaine catalytique C-terminal de près
de 350 résidus qui assure la fonction proline oxydase
(261-612).
Le domaine de dimérisation est constitué d'un bras
de 3 hélices á qui embrasse les 3
hélices homologues de l'autre sous-unité. Le second
domaine N-terminal comporte 6 hélices
á qui n'établissent aucun contact significatif avec
le domaine catalytique ou la seconde sous- unité. Lee et al.,
ont dans un premier temps suggéré que ce domaine, dont le
repliement est
proche des motifs caractéristiques de liaison
à l'ADN « hélice-coude-hélice », puisse
être
responsable de la fixation aux acides nucléiques. Plus
récemment, Gu et al., ont montré que le domaine de
liaison à l'ADN est en réalité localisé dans
l'extrémité N-terminale 1-90 de PutA
qui est déstructurée dans la structure
cristalline de PutA669 (Gu et al., 2004). Au vu de l'alignement de
séquences initial qui a été réalisé
dans le chapitre 1, il apparaît que les 2 domaines N-terminaux de
PutA669 ne sont présents ni chez l'Homme, ni chez aucune espèce
eucaryote. En effet, la longueur des séquences PRODH est
très différente d'une espèce à l'autre (de
475 résidus chez la plante Oryza à 669
résidus chez la mouche) et elles ne s'alignent que dans la
moitié C-terminale des séquences eucaryotes qui correspond
au domaine catalytique. De plus, contrairement à PutA qui est capable de
réguler la transcription
de son gène dans le cytoplasme, il n'a jamais
été montré qu'une proline oxydase eucaryote,
localisée dans la mitochondrie, soit capable de lier l'ADN.
La superstructure du domaine catalytique de PutA669 est
un tonnelet de type á8â8 dans lequel 8 feuillets â
forment un coeur hydrophobe autour d'un cofacteur FAD et d'un
inhibiteur lactate (Figure 3.1). Cette poche hydrophobe est
protégée du solvant par 10 hélices
á situées en périphérie du domaine.
Les molécules de FAD et de lactate sont fortement liées
à
ce domaine catalytique par de nombreuses interactions non
covalentes avec l'hélice C- terminale á8 et les
extrémités C-terminales des feuillets â.
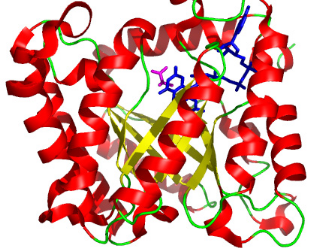
Figure 3.1 : Structure du
tonnelet á8â8 du domaine catalytique de PutA669. Les
hélices apparaissent en rouge, les feuillets en jaune, et les
boucles en vert. Le cofacteur FAD est coloré en bleu, et
l'inhibiteur lactate en violet.
La comparaison de séquence de la région C-terminale
conservée de PRODH humaine
(340-600) avec PutA669 met en évidence une faible
identité de séquence d'à peine 15 %. De manière
intéressante, ce pourcentage atteint 47 % lorsqu'on restreint la
comparaison aux résidus situés à moins de 5 Å du
cofacteur FAD ou de l'inhibiteur lactate. Ainsi, sur les 40 résidus
à proximité de ces 2 molécules, 19 sont rigoureusement
identiques et 6 sont similaires. Sur la base de cette forte conservation
au niveau de ces résidus critiques, il apparaît que
PutA669 et PRODH humaine comportent le même site actif et par
conséquent, un domaine catalytique qui pourrait être de repliement
similaire. La présence d'une molécule de lactate dans le coeur
hydrophobe de PutA669 conforte cette hypothèse. En effet, le lactate est
connu pour sa capacité à inhiber l'activité de la
proline déshydrogénase chez les organismes eucaryotes
(Kowaloff et al., 1977), ce qui n'avait jamais été
constaté chez les PRODH procaryotes.
| 


