2) Caractérisation de l'organisation de PRODH
humaine
2.1) Report de la structure secondaire de PutA669 sur
l'alignement initial
La délimitation du domaine catalytique humain
peut être prédite en reportant les éléments de
structure secondaire de PutA669 sur l'alignement initial (cf. chapitre
1) mené avec 9 séquences eucaryotes et la séquence
de PutA. Sur la base de ce report de structure secondaire, il
apparaît que le domaine catalytique humain est situé entre les
résidus 270 et 600 (non montré). Cette délimitation
est en accord avec les prédictions réalisées
par les programmes de threading de type 3D-PSSM, qui
confirment l'alignement initial en construisant un modèle par
pseudo homologie du domaine catalytique humain qui s'étend des
résidus 267 à 570.
2.2) Organisation de PRODH humaine
Dans l'optique de caractériser l'organisation des
domaines de PRODH humaine, nous avons voulu tester la
véracité de l'alignement initial en vérifiant
l'état de conservation des résidus impliqués dans
l'interaction avec le cofacteur ou l'inhibiteur qui sont supposés
être fortement conservés. De fortes identités
sont effectivement observables au niveau de l'ensemble des
feuillets â du tonnelet à l'exception des 2 brins
N-terminaux â1 et â2 qui apparaissent non conservés. Ce
résultat semble surprenant dans la mesure où 2 résidus du
brin
â2 établissent des contacts avec le cofacteur FAD
dans la structure de PutA669. Nous avons
soumis les séquences correspondant aux brins
â1 et â2 de PutA669 au programme PSI- BLAST afin de
déterminer les homologues séquentiels de ces
régions. De manière intéressante, les résultats
de cette recherche montrent des homologies significatives avec des segments de
la région 110-240 de PRODH humaine qui ne sont pas alignés avec
la séquence
de PutA sur l'alignement initial (cf. chapitre 1). Le
programme PSI-BLAST met également en évidence des homologies avec
des fragments N-terminaux de plusieurs séquences de proline
déshydrogénase eucaryote qui sont également non
alignés avec PutA sur l'alignement initial.
Sur la base de cette analyse, nous avons
corrigé l'alignement de séquences qui s'avérait
inexact dans la région N-terminale du domaine catalytique. La
totalité de ce nouvel alignement de 10 séquences eucaryotes avec
PutA669 qui s'étend sur près de 850 résidus est
présenté dans la figure 3.2. Cet alignement corrigé
fait apparaître 2 insertions au sein du domaine catalytique chez
les eucaryotes supérieurs comme le ver Caenorhabditis elegans,
la mouche Drosophila Melanogaster, et l'Homme. La
première de ces insertions, composée d'une cinquantaine de
résidus, est située entre le feuillet â1 et l'hélice
á1 du tonnelet á8â8. La seconde, d'une taille deux fois
plus importante, est localisée entre le feuillet â2 et
l'hélice á2.
Figure 3.2 :
Alignement corrigé de 10 séquences de
proline déshydrogénase réalisé avec l'aide du
logiciel clustalw. Les éléments de structure secondaire
reportés sont relatifs à la structure
tridimensionnelle du domaine catalytique de PutA669, résolue par
diffraction des rayons X (Lee et al., 2003). Les feuillets â sont en
vert, et les hélices á en rouge. Les 2 insertions dans
la région N-terminale du domaine catalytique
sont mises en évidence. Les résidus sont colorés en
rouge lorsqu'ils sont conservés (sigle *), en vert lorsqu'il sont
fortement similaires (sigle :), et en bleu lorsqu'ils sont faiblement
similaires (sigle .).
Les 10 séquences alignées correspondent aux
espèces suivantes :
Human : proline oxydase humaine (600
résidus)
CAEEL : proline oxydase de Caenorhabditis
elegans (564 résidus) Drosophila : proline oxydase
de Drosophila Melanogaster (669 résidus) POMBE
: proline oxydase de Schizosaccharomyces pombe (492
résidus) Arabidopsis : proline oxydase de
Arabidopsis thaliana (499 résidus)
|
Tabacum
|
:
|
proline oxydase de Nicotiana tabacum (493
résidus)
|
|
Oryza
|
:
|
proline oxydase de Oryza sativa (475 résidus)
|
|
Cerevisiae
|
:
|
proline oxydase de Saccharomyces cerevisiae (476
résidus)
|
|
Emericella
|
:
|
proline oxydase de Emericella nidulans (478
résidus)
|
|
PutA
|
:
|
proline dehydrogenase de E.coli (648 premiers
résidus)
|
Chapitre 3 : Etude bio-informatique : sélection de
3 domaines PRODH
Human
1-----------------MALRRALPALRPCIPRFVQLSTAPASR----------EQPAAG
CAEEL
1--------------------------MKIPVALVL-------------------------
Drosophila
1-----------------MALLRSLSAQRTAISLVYGRNSSKSSNSVAVAACRSFHQRGNG
POMBE
1----------------MRAFRLASGVLRNRKVILGIGAGSLITAG---------NIKIRN
Arabidopsis
1----------------MATRLLRTNFIRRSYRLPAFSPVGPPTVTAS-------TAVVPE
Tabacum
1----------------MANKVVCPKAFRDLRSFVRCLNTAPTVPPMN-------FTGAYD
Oryza
1-------------------MAIASRIQKRVLASFAAAAAAKLPEAA--------VAAAGG
Cerevisiae
1----------------MIASKSSLLVTKSRIPSLCFPLIKRSYVSKT-------PTHSNT
Emericella
1--------------MKAATPRPSVRALSSGRSYRTARFVSRTSNAR--------SSLAAD
PutA
1-MGTTTMGVKLDDATRERIKSAATRIDRTPHWLIKQAIFSYLEQLENSDTLPELPALLSG
51
Human
34-PAAVPGGGSAT------------A---------------------VRP-----------PVPA---------VD-FGNAQ--------------------
CAEEL
-T-------------------------------------------III-------------------------E-F------------------------
Drosophila
STSIAGEGAASESTRGVNGARFLHSGDRPLQASTLVQPEVVSSETVKRSMKQESSQEKNPSPAGSPQRDPLDVS-FNDPI--------------------
POMBE
DSKFDAFFAKG----------------------------------------------------------------FPD----------------------
Arabidopsis
ILSFGQQAPEPP---------------------------------LHHP---------KPTEQSHDG-----LD-LSDQA--------------------
Tabacum
ATTVTTPALIP-----------------------------------TD----------QVITADKKV-----IN-FEDVK--------------------
Oryza
AAEAVEEVASS----------------------------------VQEQ-----------VQAQG----AQVLE-FGDTE--------------------
Cerevisiae
AANLMVETPAAN----------------------------------------------------------------ANGN--------------------
Emericella
TNSLLQQAPPS--------------------------------------------------------------P-KKQLA--------------------
PutA
60-AANESDEAPTPAEEPHQPFLDFAEQILPQSVSRAAITAAYRRPETEAVSMLLEQARLPQPVAEQAHKLAYQLADKLRNQKNASGRAGMVQGLLQEFSLSS
Human
60---------------EAYRSRRTWELAR---------------SLLVLRLCAWPALLARHEQLLY--V-----SRKLLGQRLFNKLMK------MTFYGH
CAEEL
----------------FQSKSNTELVR---------------ALVVLRLCGIQTLVNQNQIILN--T-----MRRVLGKNLFKKTLK------NTFFGH
Drosophila
--------------AAFKSKTTGELIR---------------AYLVYMICSSENLVEHNMTLMK--W-----SKNVLGQRLFTLLMK------ATFYGH
POMBE
---------------ELQHRSLFSVLR---------------SAFVYEICSRAWLVKLSLGAMS--L-----CDVFHLSFLYNPFCR------YTFYKH
Arabidopsis
--------------RLFSSIPTSDLLR---------------STAVLHAAAIGPMVDLGTWVMSSKL-----MDASVTRGMVLGLVK------STFYDH
Tabacum
--------------ELFTGVSTLKLIR---------------STLTLQMAATEPMVDVGIWVMNSKL-----MHMPIVKEVILGFVK------GTFYEH
Oryza
--------------RLFAGERSTSLVR---------------TLAVLQALSVGPLVDVATAALR--------SPAVAGSAAGRAAAR------ATAYQH
Cerevisiae
--------------SVMAPPNSINFLQT-------------LPKKELFQLGFIGIATLNSFFLN--------TIIKLFPYIPIPVIK------FFVSSL
Emericella
--------------SPLAKLPLSSVLR---------------SLLILSVSSSSILLKPCIYTLSALAHPKTALLDVAKNPLLNLLVK------HTIYKQ
PutA
160-QEGVALMCLAEALLRIPDKATRDALIRDKISNGNWQSHIGRSPSLFVNAATWGLLFTGKLVSTHNEASLSRSLNRIIGKSGEPLIRKGVDMAMRLMGEQ
51
. : . : :
á0 â1
Chapitre 3 : Etude bio-informatique : sélection de
3 domaines PRODH
Human
117-FVAGEDQ-ESIQPLLRHYRAFGVSA-ILDYGVEEDLSPEEAEHKEMES----------CTSAAERDGSGTNKRDKQYQAHRAFGDRRNGVISARTYFYAN
CAEEL
FVAGETE-EEVRHVVEKLRNYGVKS-ILDYSVEADITSQEATDKTVKGTSVATVKPAAMTPVVDAKTLETTR--ERYTVHEEFGDRRQGVSSARTYFYEG
Drosophila
FVAGEDQ-IKIIPTLERLRSFGVKP-ILDYSVEEDITQEEAEKREVESS---------VSSAGDKKEEGSMP---QYHVDKSFADRRYKVSSARTYFYLN
POMBE
FCGGETP-QAVMATMDTLQAAGITS-CLNYSREVDLDGDMDVNKIAS--------------------QG-------------------VVPPQVPVPSEK
Arabidopsis
FCAGEDADAAAERVRSVYEATGLKG-MLVYGVEHAD---------------------------------------------------------------D
Tabacum
FCAGKDL-IEVRRTVTKLSDVGLKG-MLDYGVEHAT---------------------------------------------------------------E
Oryza
FCAGETAEEAAAAVRRLWRG-GMGG-ILDYGIEDAE---------------------------------------------------------------D
Cerevisiae
YCGGENF-KEVIECGKRLQKRGISNMMLSLTIENSEGTKSLSSTP------------------------------------------------VDQIVKE
Emericella
FNAGENK-LEVQRSINAIKELGYRGVLLGYAREVLVGESKTDPR-------------------------------------------------------D
PutA
259-FVTGETI-AEALANARKLEEKGFRY-SYDMLGEAAL---------------------------------------------------------------T
: *: * *
1ère insertion
á1 â2
52
Human
205-EAKCDSHME--TFLRCIEA--SGRVSDDGF----IAIKLTALGRPQFLLQFSEVLAKWRCFFHQMAVEQGQAGLAAMDTKLEVAVLQESVAKLG-IASRA
CAEEL
EEQCDKNRD--IFKDSINAV-ASATKNEGF----VAVKITALGRPQLLLKLSEAIVQTQNFFKALTGGMS-----LQEGRLTSQEFYKRLGELGVKTDTE
Drosophila
EATCERNME--IFIKCLEAV-SGATFGTGI----TAIKLTALGRPQLLLQLSEVIMRTRKYMEDMVGGQGN----VLTHHKTIKDLEKYYATLG---DNK
POMBE
NQKVLRQIADKAFESNMHIIDMATYKPGTV----CAVKLTPFINPLVLQRYNSILN--------------------------------------------
Arabidopsis
AVSCDDNMQ--QFIRTIEAAKSLPTSHFSS----VVVKITAICPISLLKRVSDLL---------------------------------------------
Tabacum
NESCDQSMK--VFLQTAESTKSLPSSSVSF----VVVKITAICTPKLLKRMSDLL---------------------------------------------
Oryza
GPACDRNAA--GFLAAIDVAAALPPGSAS-----VCIKITALCPVALLEKASDLL---------------------------------------------
Cerevisiae
TISSVHNILLPNIIGQLESK-PINDIAPGY----IALKPSALVDNP-----HEVLY--------------------------------------------
Emericella
EQASRQEIQ--TWLDGTLQT-VDMAQEGDF----VALKFTGMGI--------QAL---------------------------------------------
PutA
294-AADAQAYMV--SYQQAIHAIGKASNGRGIYEGPGISIKLSALHP--------------------------------------------------------
52
:* : :
2ème insertion
á2 â3 á3
Human
296-EIEDWFTAETLGVSGTMDLLDWSSLIDSRTKLSKHLVVPNAQTGQLEPLLSRFTEEEELQMTRMLQRMDVLAKKATEMGVRLMV---DAEQTYFQP-AIS
CAEEL
SVKKFFDEVDFDSDGIVDLHGWNHILDDHVKLGQLFQVLNIKTGSLEPLIQNLSNEEEQEFRNMVRRTLDVAEYAIEKGVRIMV---DAEQTYLQP-AIS
Drosophila
DVKEFLNNVTSDKEGILHLFPWSGIVDEDSQLSDTFRVPDPQTGQMRRLISQIPPKEEEMFRNMIRRLNTIVKAAADLDVRIMV---DAEQTYFQP-AIS
POMBE
---QYP-------------------VESACNYLEHLK---------SP--E-LSTYEVSELKKFWEYADKLCQFAKEKQIPLFI---DAEQTYFQD-CMH
Arabidopsis
---RWEYKSPNFK------LSWK--LKSFPVFSESSPLYHTNS---EP--EPLTAEEERELEAAHGRIQEICRKCQESNVPLLI---DAEDTILQP-AID
Tabacum
---RWEHKNPSFN------LPWK--QKSLPLFSDSSPFYHTPQ---KP--EPLTVEEEHDLQLAHERLMTICKKCLELDVDLLI---DAEDTAIQP-AID
Oryza
---RWQQKHPATK------LPWK--VHGFPVLCVSSPLYLTAA---EP--PALEAEEERELEMAHGRLLAIGERCAEYDIPLLV---DAEYATVQP-AID
Cerevisiae
---NFSN----------------------PAYKAQ------------R--DQLIENCSKITKEIFELNQSLLKKYPERKAPFMVSTIDAEKYDLQENGVY
Emericella
---EYL------------------------------------------------QNQAPPSPFMDEAIKQVCDLAISRNVRLLV---DAEEQAVQP-GIE
PutA
336----RYS-------------R----------------------------------AQYDRVMEELYPRLKSLTLLARQYDIGINI---DAEEADRLEISLD
: : . : : *** :
á3 â4 á4 â5
á5A á5B
Chapitre 3 : Etude bio-informatique : sélection de
3 domaines PRODH
Human
392-RLTLEMQRKFNVEKP---LIFNTYQCYLKDAYDNVTLDVELARREGWCFGAKLVRGAYLAQERAR-AAEIGYEDPINPTYEATNAMYHRCLDYVLEELKH
CAEEL
KITIEMMKKYNKGRG---NIFNTYQAYLKGTLQNMEADMQVARREGWHFGAKLVRGAYMEQERAR-AKAIGYEDPINDNFEATSKMYESCLTRIADEVHR
Drosophila
RITLEMMRKYNKDKA---IVFNTYQCYLRETFREVNTDLEQAKRQNFYFGAKLVRGAYMDQERDR-AKSLGYPDPVNPTFEATTDMYHRTLSECLRRIKL
POMBE
AVTVDLMRKYNKEVA---IVHNTYQLYLKKSRKIMDDHIKKCVAEGWLMGAKLVRGAYLNSEPRF-LIHDTKAETDKDFDSAVEAIIAAAAKFAPGDPAS
Arabidopsis
YMAYSSAIMFNADKDR-PIVYNTIQAYLRDAGERLHLAVQNAEKENVPMGFKLVRGAYMSSEASL-ADSLGCKSPVHDTIQDTHSCYNDCMTFLMEKASN
Tabacum
YFAYSAAIKYHKDDD--PMIFGTIQAYLKDSKERMVIAKKAAEKMGVPMGFKLVRGAYMSSEREL-ASRLGVQSPIHDSIEQTHDCFNSCAEFMLDEISN
Oryza
YFTFAGALAFNGGGR--PIVHGTVQAYLRDARDRLEAMARAAQGERVCLALKLVRGAYLAREARL-AASLGVPSPVHRSIQDTHDCYNGCAAFLLDRVRR
Cerevisiae
ELQRILFQKFNPTSSKLISCVGTWQLYLRDSGDHILHELKLAQENGYKLGLKLVRGAYIHSEKNRNQIIFGDKTGTDENYDRIITQVVNDLIINGEDSYF
Emericella
EWATMYQKYCNSRTPGRAIFYNTYQAYLCSTPATLARHLEISRKEGYTLGVKLVRGAYLKTEPRH-LIWAKKEQTDECYDGIVEALLTRRYNHMLKPASA
PutA
383-LLEKLCFEPELAGWN---GIGFVIQAYQKRCPLVIDYLIDLATRSRRRLMIRLVKGAYWDSEIKRAQMDGLEGYPVYTRKVYTDVSYLACAKKLLAVPNL
. * * : . : :**:*** *
â6 á6 â7 á7 â8
á8
53
Human
488-N--------AKAKVMVASHNEDTVRFALRRMEELG-LHPADHR-VYFGQLLGMCDQISFPLGQ-------------AGYPVYKYVPYGPVMEVLPYLSRR
CAEEL
R------GKTNVSVMVASHNEDTVRFALNLMKEKC-ISPSERV-MCMAQLYGMCDQVSFSLGQ-------------AGFSVYKYLPYGPVEEVLPYLSRR
Drosophila
MKDCDD-DARKIGIMVASHNEDTVRFAIQQMKEIG-ISPEDKV-ICFGQLLGMCDYITFPLGQ-------------AGYSAYKYIPYGPVEEVLPYLSRR
POMBE
ASDPIASRKGKWGIMVASHNKKTMFESVNLAETKK--VDFTKTSFYLAQLLGMADDITYALAYS-------QRNQQPNFCIVKYVSCGPISEVLPYLVRR
Arabidopsis
GSGF--------GVVLATHNADSGRLASRKASDLG-IDKQNGK-IEFAQLYGMSDALSFGLKR-------------AGFNVSKYMPFGPVATAIPYLLRR
Tabacum
GSG---------AVVLATHNIDSGKLAASKAIDLG-IRKDSQK-LQFAQLYGMAEGLSFGLRN-------------AGFQVSKYLPFGPVEQVMPYLIRR
Oryza
GAA---------AVTLATHNVESGQLAAARALELG-IGGGGDRGLQFAQLMGMADGLSLGLRN-------------AGFQVSKYLPYGPVEQIIPYLIRR
Cerevisiae
G-----------HLVVASHNYQSQMLVTNLLKSTQ-DNSYAKSNIVLGQLLGMADNVTYDLITN-----------HGAKNIIKYVPWGPPLETKDYLLRR
Emericella
EHTT---ELPPVSVIVATHNRDSVRKAHALRLEQASRGEKSDVELTYAQLQGMADEISCELLQGFQTAGPENTKVAESPNVYKLLTWGSVKECMGFLLRR
PutA
480-IYP-----------QFATHNAHTLAAIYQLAGQNY----YP-GQYEFQCLHGMGEPLYEQVTGKVADG-------KLNRPCRIYAPVGTHETLLAYLVRR
.*:** .: * ** : : : . *. :* **
á8
Human
565-ALENSSLMKGT--HRERQLLWLELLRRLRTGNLFHRPA-600
// CAEEL ALENGSVLKKA--NKERDLLWKELKRRISSGEFKARSSSSS-564
//
Drosophila
AQENKGVLKKI--KKEKRLLLSEIRRRLMRGQLFYKPKGNYVPI-669 // POMBE
ARENIDALDRC--KEERAYYRQALRRRIF-492 //
Arabidopsis AYENRGMMATG--AHDRQLMRMELKRRLIAGIA-499
// Tabacum AEENRGLLSTS--AFDRQLMRKELTRRFKVATS-493 //
Oryza AEENRGLLSSS--SFDRQLLR-475 //
53
Cerevisiae LQENGDAVR----SDNGWPLIKAIAKSIPKRVGL-476
// Emericella AVENTEAVGRT--KQSQEAMFSELRRRARRAFGLRY-478
//
PutA
557-LLENGANTSFVNRIADTSLPLDELVADPVTAVEKLAQQEGQTGLPHPKIPLPRDLYGHGRDNSAGLDLANEHRLASLSSALLNSALQKWQAL-648
...............
** .
Notre alignement corrigé met ainsi en évidence la
présence de domaines entremêlés
chez PRODH humaine, et permet de prédire que les
segments qui constituent le domaine catalytique humain sont situés
dans les régions 113-150, 204-240, et 346-571. Les prédictions
de structure secondaire réalisées sur la
séquence humaine sont globalement en accord avec le report des
éléments de structure secondaire de PutA669 sur le nouvel
alignement, et notamment au niveau des brins supposés â1
(139-144) et â2 (231-233) de PRODH humaine
qui sont effectivement prédits en feuillet. Cette analyse
confirme ainsi l'exactitude de notre alignement comparé à
l'alignement initial.
Les prédictions de structure secondaire
suggèrent une structuration des 2 insertions,
qui apparaissent dans le domaine catalytique, en hélice
á. Sur notre alignement corrigé, ces 2 insertions sont
respectivement localisées sur la séquence de PutA autour
des résidus L292 pour la première, et R336 pour la seconde
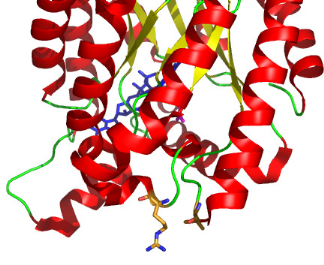
(Figure 3.2). De manière intéressante, ces 2 résidus
appartiennent à des boucles situées sur une même surface
exposée au solvant de la structure tridimensionnelle de PutA669
(Figure 3.3). Cette caractéristique structurale laisse supposer
que ces 2 insertions pourraient former un domaine unique
entremêlé à la périphérie du tonnelet
á8â8 chez les eucaryotes supérieurs.
1ère
insertion
R336
L292
2éme
insertion
Figure 3.3 : Mise en
évidence des 2 sites d'insertion sur la structure du tonnelet
á8â8 de PutA669. Les éléments de structure
secondaire et les molécules de FAD et lactate sont
colorés comme dans la figure précédente. Les
résidus L292 et R336 sont représentés par des sticks. Les
atomes d'azote sont colorés en bleu, et d'oxygène en
rose.
Outre les 2 insertions, l'alignement de la Figure 3.2
fait également apparaître une région N-terminale non
conservée de la bactérie à l'homme dans
l'extrémité 1-112 de PRODH humaine. Cette région, de
fonction inconnue et uniquement conservée chez les
eucaryotes supérieurs, semble structurée au vu des
prédictions de structure secondaire, et du
nombre de résidus hydrophobes qu'elle comporte. Par
conséquent, elle pourrait contenir un domaine de repliement autonome
indépendant du domaine catalytique et des 2 insertions.
| 


