1.2) Stratégie de construction des vecteurs
d'expression
La production de protéines en fusion avec
de multiples partenaires nécessite la réalisation de
nombreux plasmides. Aussi, il est indispensable de recourir à une
méthodologie permettant de minimiser l'effort de clonage. Dans cette
optique, l'utilisation d'un système de clonage par recombinaison
homologue a été choisie : il s'agit de la technologie
Gateway. La stratégie consiste dans un premier temps
à cloner le gène d'intérêt à partir
d'amplicons de PCR dans un plasmide appelé vecteur
d'entrée (Figure 4.1). Le système Gateway
permet alors, à partir de ce seul plasmide, d'obtenir in vitro
et en parallèle, une multitude de vecteurs d'expression
portants chacun le gène d'intérêt et un
partenaire de fusion différent. Tout l'intérêt de cette
technique de recombinaison homologue repose sur la rapidité et
l'efficacité
avec lesquelles sont construits les vecteurs
d'expression.
produit de PCR
Vecteur
d'entrée
Fusion
MBP
Figure 4.1 : Stratégie de
clonage par recombinaison homologue du programme 3PM.
1.3) Criblage des conditions d'expression en
microplaques
Le programme 3PM doit permettre l'analyse de l'influence de
conditions expérimentales sur l'expression et la solubilité de
protéines de fusion recombinantes. Au vu
du nombre de conditions choisies, les techniques
d'expression habituelles ne sont pas adaptées, et constituent un
facteur limitant. Ainsi, à l'exception de l'analyse sur gel
SDS- PAGE, toutes les étapes relatives au criblage des
conditions d'expression, à savoir la transformation de E.
coli, la culture d'expression, la lyse des bactéries, et la
séparation des fractions soluble et insoluble, sont traitées
sur microplaques 96 puits via l'utilisation de pipettes
multicanaux. Outre le traitement plus facile d'échantillons en
parallèle, l'utilisation
de microplaques 96 puits permet de diminuer les volumes
de culture (250uL), de réactifs utilisés, et de rendre
possible une automatisation partielle du processus.
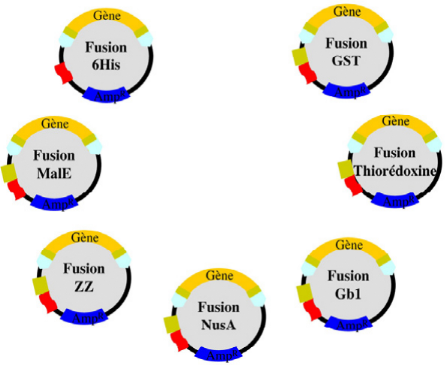
1.3.1) Les partenaires de fusion
Sur la base de l'étude comparative récente des 7
partenaires décrits comme les plus efficaces (Hammarström et
al., 2002), il a été décidé que les
séquences protéiques de l'étiquette 6xHis et des six
partenaires suivants seront systématiquement utilisées pour
être fusionnées en N-terminal avec les protéines
d'intérêt. Il s'agit de :
- GST (26 kDa) :
glutathion-S-transférase de Schistosoma japonicum
- Gb1 (7,5 kDa) : domaine de liaison aux
anticorps de la protéine G de Streptococcus
- ZZ (17 kDa) : double domaine de liaison aux
anticorps de la protéine A de Staphylococcus
- MBP (43 kDa) : protéine de fixation
au maltose de E. coli
- Trx (13 kDa) : thiorédoxine de E.
coli
- NusA (55 kDa) : protéine de E.
coli
1.3.2) Les souches bactériennes
Les souches d'expression utilisées dans le cadre du
programme 3PM dérivent de la souche BL21 déficiente en
protéases lon et ompT. Elles possèdent une
copie chromosomique
du gène codant pour l'ARN polymérase T7 sous le
contrôle d'un promoteur lac inductible par l'IPTG
(désignation DE3). Elles sont destinées à la surexpression
des plasmides d'expression dont la transcription est
contrôlée par un promoteur de type T7 inductible par
cette
polymérase. La production des protéines de fusion
est testée dans les 3 souches suivantes :
· BL21 STAR (DE3) : cette souche
possède un gène rne mutant qui code pour une
RNase E tronquée incapable de dégrader les
ARNm, d'où une augmentation de stabilité de ces derniers.
· ROSETTA pLysS (DE3) : la
souche Rosetta est désignée pour augmenter l'expression
de protéines recombinantes d'origine eucaryote dont les
gènes correspondant contiennent des codons rarement utilisés chez
E. coli. Cette souche contient ainsi un plasmide fournissant les
ARNt correspondant à 6 codons rares (AUA, AGG, AGA, CUA, CCC, GGA).
La souche pLysS exprime le lysozyme T7, qui inhibe l'activité basale
de l'ARN polymérase T7, optimisant de ce fait la
régulation du niveau d'expression. La sélection
spécifique de cette souche est possible en ajoutant du
chloramphénicol dans les milieux de culture.
· C41 (DE3) : recommandée
pour l'étude de protéines toxiques, cette souche
comporte une ou plusieurs mutation(s) de gène(s) inconnue(s)
conduisant à la surproduction de membranes.
1.3.3) Les températures d'expression
D'autres paramètres, comme la température
de la culture d'expression, peuvent influencer et améliorer la
solubilité des protéines recombinantes. Ainsi, une diminution de
la température conduit à une réduction des taux
d'expression. La diminution de ces niveaux d'expression permet d'une part,
de limiter les interactions hydrophobes intermoléculaires qui provoquent
l'agrégation, et d'autre part, d'éviter de saturer les
systèmes d'assistance au repliement formés par les
protéines chaperonnes de E. coli. Pour des raisons pratiques,
et afin
de limiter le nombre de conditions, il a
été décidé de tester l'expression des
protéines de
fusion aux 2 températures suivantes :
37°C et 20°C
| 


