CHAPITRE 2
Production et analyses préliminaires
du domaine K2 de la protéine humaine KIN17
1) Préparation des échantillons pour
l'analyse RMN
1.1) Sélection et optimisation du système
d'expression
Le Laboratoire de Marquage des Protéines du CEA
de Saclay (LMP) a récemment élaboré un Programme de
Production et Marquage des Protéines (3PM) qui a pour objectif de
produire en grande quantité, chez la bactérie E.
coli, des protéines solubles, pures, et sous forme native en
vue de leur caractérisation structurale (Braud et al., 2005). La
stratégie mise
en oeuvre pour obtenir de tels résultats a
été présentée en détail dans le
chapitre 4 de la première partie de ce manuscrit,
consacrée à l'étude structurale de la
protéine PRODH humaine. Elle repose notamment sur un criblage
systématique de plusieurs conditions expérimentales. Toutes
les étapes relatives au clonage, au criblage des
conditions d'expression, à la production à grande échelle
en milieu non marqué, et à la purification du domaine K2 ont
été menées avec succès par les chercheurs du LMP
dans le cadre de cette plate-forme de production. Ainsi, les
quantités de protéine K2 soluble et pure obtenues
à l'issue du processus 3PM étaient suffisantes pour
envisager la préparation des échantillons pour l'analyse
structurale.
La stratégie d'attribution de l'ensemble des raies de
résonance RMN d'une protéine de
111 acides aminés comme K2 repose sur
l'enregistrement d'expériences hétéronucléaires
triple résonance (1H, 15N, 13C),
et nécessite la préparation d'échantillons
uniformément marqués 15N et 15N /
13C. En contrôlant de manière stricte les sources
d'azote et de carbone d'un milieu de culture, il est possible de produire des
protéines simplement ou doublement marquées de façon
uniforme avec des rendements de marquage(s) isotopique(s) proches de
100%. Le contrôle total du milieu impose
généralement des milieux pauvres (milieu minimum), dont le
plus fréquent est le M9 (Maniatis et al., 1982). Par comparaison avec le
LB, ce type de milieu est appauvri en métabolite, nécessite
l'adaptation de l'hôte bactérien, et entraîne par
conséquent une diminution de la croissance bactérienne, ainsi
qu'une baisse du rendement de production. Pour pallier ces
inconvénients, l'une des solutions consiste à enrichir le
milieu M9 avec des vitamines et des oligo-éléments qui
contiennent peu d'atomes
de carbone et d'azote (Jansson et al., 1996). D'autre
part, il existe des milieux riches contenant une réserve de
métabolites bio-disponibles et adaptés au(x)
marquage(s) isotopique(s) uniforme(s) (Reilly & Fairbrother, 1994). Ces
milieux sont pour le plus souvent
issus d'un hydrolysat de microorganismes
photosynthétiques, ce qui est le cas du milieu
Algone préparé au LMP à partir de cultures
de cyanobactéries Spirulina maxima doublement
marquées 15N / 13C de manière
uniforme.
La modification d'une condition expérimentale telle que le
milieu de culture peut nuire
à la qualité du profil d'expression
d'une protéine. Par conséquent, il est
nécessaire d'entreprendre une seconde étape d'optimisation de
l'expression de K2 en milieux marqués avant de préparer les
échantillons RMN. Celle-ci a été réalisée en
milieu minimum sur la base des meilleurs résultats obtenus
à l'issue du criblage des conditions d'expression en
microplaques, puis en milieu Algone.
1.1.1) Résultats du criblage des conditions
d'expression en microplaques
Le domaine K2 de la protéine KIN17 a été
exprimé dans 30 conditions expérimentales différentes : 5
partenaires de fusion (His, Gb1, ZZ, GST, et Trx), 3 souches
d'expression (BL21 Star, BL21 AI, et Rosetta DE3), et 2
températures (37°C et 20°C). Bien que les 3 souches
testées conduisent globalement à de bons taux d'expression
et de solubilité, les meilleurs résultats ont
été obtenus en souche Rosetta (DE3). Le tableau 2.1
présente les taux
de protéines soluble et insoluble obtenus dans cette
souche après analyse sur gel SDS-PAGE.
|
K2
|
His
|
Gb1
|
ZZ
|
GST
|
Trx
|
|
S
|
I
|
S
|
I
|
S
|
I
|
S
|
I
|
S
|
I
|
|
37°C
|
+
|
+
|
++(+)
|
++
|
++
|
+++
|
+
|
+++
|
+
|
+(+)
|
|
20°C
|
++
|
+++
|
+
|
+
|
+++
|
+
|
+++
|
++
|
++++
|
+(+)
|
Tableau 2.1 : Analyse de
l'expression et de la solubilité des protéines de fusion K2 en
souche
Rosetta. Après dépôt des fractions
soluble (S) et insoluble (I) sur gel SDS-PAGE, chaque bande de
surexpression a été analysée, et une valeur
semi-quantitative a été associée à son
intensité, de faible (+) à très forte (++++). Les
meilleures conditions sont mises en évidence par une coloration
orange.
Au vu de ces résultats, on constate que la nature du
partenaire de fusion combinée à la température
d'expression a un effet notable sur la solubilité des protéines
hétérologues K2. Ainsi, les meilleurs taux d'expression de
protéine soluble ont été obtenus à 20°C et
lorsque
K2 est en fusion avec les partenaires ZZ, GST, ou Trx. Au final,
l'association partenaire Trx,
souche Rosetta, et température de 20°C constitue la
meilleure condition d'expression à l'issue
du criblage à moyen débit proposé par le
programme 3PM.
1.1.2) Optimisation de l'expression en milieu minimum et
milieu riche
a) Optimisation en milieu minimum non
marqué
L'expression de K2 en milieu minimum M9 non-marqué a
été menée en volume de
100 mL dans des erlenmeyers de 1 L. Les 3 meilleures conditions
déterminées précédemment
ont été testées ; la protéine K2 a
été exprimée en fusion avec respectivement, GST, Trx, et
ZZ,
en souche Rosetta (DE3) cultivée à 20°C. Pour
chaque construction, les étapes principales du protocole d'expression
sont les suivantes :
La souche Rosetta (DE3) est transformée par le plasmide
d'expression d'intérêt, puis mise en culture en milieu LB
à 37°C. Après quelques heures, une préculture
en milieu minimum est ensemencée puis incubée à 37°C
sous agitation. La culture d'expression de 100 mL est inoculée avec
un volume de préculture, et l'expression de K2 est induite
à 20°C pendant 14 heures par ajout de 1mM d'IPTG lorsque
la DO600 atteint 1.2. Les culots bactériens sont repris et
lysés dans un tampon phosphate, et après centrifugation, les
fractions soluble et insoluble sont analysées sur gel SDS-PAGE (Figure
2.1).
GST
S I
Trx ZZ
S I S I
(kDa)
75
50

37
·
25
·
·
20
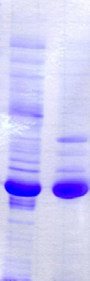
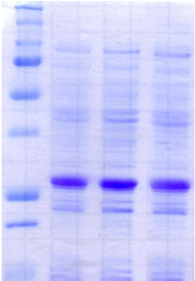
Figure 2.1 : Expression des
protéines de fusion GST-K2 (43.2 kDa), Trx-K2 (28.7 kDa), et
ZZ-K2 (33.3 kDa) en souche Rosetta cultivée
à 20°C en milieu minimum non marqué. Analyse SDS-PAGE des
fractions soluble (S) et insoluble (I) pour les 3 partenaires
testés. Les protéines de fusion sont mises en évidence
par des points rouges.
De manière intéressante, les profils
d'expression obtenus en milieu minimum
divergent significativement de ceux obtenus en milieu LB. La
différence la plus spectaculaire concerne la fusion GST qui conduit
à une expression de protéine très majoritairement
insoluble en milieu minimum. L'expression de K2 en fusion avec Trx
est tout à fait remarquable en milieu minimum, aussi bien dans
la fraction soluble, que dans la fraction insoluble. On retrouve ce
résultat pour le partenaire ZZ, bien que l'expression sous
forme soluble soit plus importante que sous forme insoluble.
Les fusions ZZ et Trx conduisant à des taux
d'expression de protéine soluble comparables, le choix du partenaire
a été guidé par d'autres considérations. Ainsi,
l'apparition d'un trouble persistant dans la fraction soluble de K2
fusionnée à Trx a révélé une propension
lente à l'agrégation de cette construction. De plus, en
anticipant sur la purification de la protéine
hétérologue, les résines d'affinité
spécifique à ZZ s'avèrent moins coûteuses que
celles spécifique au partenaire Trx. Par conséquent, nous
avons choisi de préparer les échantillons marqués
pour l'analyse RMN en produisant le domaine K2 en fusion avec le
partenaire ZZ en souche Rosetta (DE3) cultivée à 20°C.
b) Optimisation en milieu Algone doublement
marqué 15N / 13C
Bien que préparé au LMP, l'Algone est un milieu
riche très onéreux. Afin de réduire
les coûts de production de l'échantillon
doublement marqué 15N / 13C, ce milieu peut
être dilué avec de l'eau MilliQ. Toutefois et à l'image
des milieux pauvres, une diminution de la réserve métabolique
du milieu de culture, entraînée par une dilution, peut
avoir pour conséquence une modification du profil
d'expression de la protéine recombinante. L'optimisation de
l'expression de K2 en milieu Algone a ainsi pour objectif de déterminer
le milieu le plus dilué permettant d'obtenir le meilleur taux de
protéine soluble.
La production de K2 fusionnée à ZZ en souche
Rosetta (DE3) cultivée à 20°C a été
menée en volume de 30 mL dans des erlenmeyers de 300 mL. Trois dilutions
d'Algone 1/3,
1/2, et 2/3 ont été testées, ainsi
que le milieu Algone 100 %. Les résultats de cette optimisation
mettent en évidence que le profil d'expression de la protéine de
fusion ZZ-K2 en milieu Algone est quasiment identique à celui obtenu en
milieu minimum non marqué, quelle
que soit la dilution (Figure 2.2).
1/3
1/2
2/3
100%
S I S I
S I S I
ZZ-K2
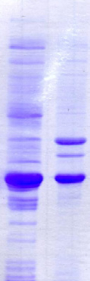
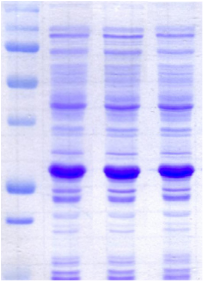
Figure 2.2 : Optimisation de
l'expression de K2 en milieu Algone (fusion ZZ, souche Rosetta
cultivée à 20°C). Analyse SDS-PAGE des
fractions soluble (S) et insoluble (I) correspondant
à différentes dilutions du milieu Algone avec
de l'eau milliQ (1/3, 1/2, 2/3, et 100 % Algone).
Au vu des résultats de l'optimisation de
l'expression de K2 en milieu minimum et milieu riche, il a
été décidé de produire la protéine K2
doublement marquée 15N / 13C en milieu Algone
dilué au 1/3, et K2 simplement marqué 15N, en milieu
minimum.
1.2) Obtention de K2 simplement marquée
15N et doublement marquée 15N /
13C
1.2.1) Stratégie
générale
Cette stratégie a été
présentée en détail dans le chapitre 4 de la
première partie de ce manuscrit. Par conséquent, je me
contenterai de rappeler ses points essentiels.
Le plasmide d'expression de ZZ-K2 encode une protéine qui
contient, une étiquette de
6 résidus histidine (6xHis), le partenaire de fusion ZZ,
le site de reconnaissance TEV (sTEV),
et le domaine K2. La construction obtenue est de la forme
6xHis-ZZ-sTEV-K2 pour un poids moléculaire de 33 kDa. La
préparation de l'échantillon débute par la production de
plusieurs litres de protéine de fusion. Après lyse des
membranes bactériennes et extraction des protéines
cytosolubles, les surnageants sont déposés sur colonne
d'affinité spécifique au fragment protéique ZZ. Cette
étape permet, dans un premier temps, d'éliminer les
protéines endogènes de E. coli, puis de cliver le
partenaire ZZ par une coupure sur colonne avec la protéase
recombinante 6xHis-TEV (produite au laboratoire). Lors de l'élution de
la protéine
K2 clivée, le partenaire ZZ est retenu par la
résine d'affinité. Une seconde étape de
purification est alors réalisée sur
résine de nickel. Contrairement aux protéines
résiduelles
6xHis-ZZ-sTEV-K2 et 6xHis-ZZ, et à la
protéase 6xHis-TEV, la protéine K2 ne comporte
plus d'étiquette 6xHis après clivage de son
partenaire. Elle ne doit donc pas être retenue par la résine de
nickel et peut être séparée de la protéase
6XHis-TEV, ainsi que des 2 protéines résiduelles. Au
final, les échantillons RMN sont obtenus après
concentration sur cellule Amicon.
1.2.2) Production de la protéine de fusion en
milieu minimum et milieu Algone
La première étape de préparation des
échantillons de protéine K2, enrichie en isotopes
15N et 15N / 13C, a
consisté à produire la protéine de fusion dans 3
L de chaque milieu de culture marqué (milieu minimum marqué
15N, et milieu Algone doublement marqué 15N /
13C dilué au 1/3). Le protocole d'expression
résumé en Figure 2.3 a été
préalablement mis au
point en petit volume.
transformants
ensemencement avec plusieurs colonies
Préculture LB (37°C)
50 mL en erlenmeyer
+ Ampi 100 ug/mL
+ CAM 20 ug/mL
+ glucose 0.5 %
ensemencement pour
obtenir DO595 = 0.1
Culture milieu
marqué (20°C)
3 L en fernbach
+ Ampi 50 ug/mL
+ CAM 20 ug/mL
+ IPTG 1mM à DO595 = 1.2
ensemencement pour obtenir DO595 = 0.05
Préculture milieu
marqué (37°C)
100 mL en erlenmeyer
+ Ampi 50 ug/mL
+ CAM 20 ug/mL
Figure 2.3 : Protocole de
surexpression de K2 en milieu marqué (milieu minimum
marqué
15N, ou milieu Algone doublement
marqué 15N / 13C dilué au 1/3).
La souche Rosetta transformée par le vecteur
d'expression encodant la protéine de fusion est étalée
sur boîte LB-agar contenant 100 ug/mL d'ampicilline (Ampi) et 20 ug/mL de
chloramphénicol (CAM), puis incubée à 37°C
pendant 14 heures. Plusieurs colonies sont mises en préculture
dans un erlenmeyer de 500 mL contenant 50 mL de LB, 100 ug/mL
d'Ampi, 20 ug/mL de CAM, et 0.5 % de glucose. L'ensemble est incubé
à 37°C pendant 3 heures sous une agitation de 250 rpm. 100 mL de
milieu marqué contenant 50 ug/mL d'Ampi
et 20 ug/mL de CAM sont introduits dans un erlenmeyer de 1 L,
puis ensemencés avec un
volume de préculture LB de manière à obtenir
une DO initiale à 600 nm égale à 0.1. Cette
deuxième préculture est ensuite incubée à 37°C
sous agitation (250 rpm) pendant 6 heures.
3 L de milieu marqué contenant 50 ug/mL d'Ampi et 20 ug/mL
de CAM sont alors répartis dans 3 fernbachs de 1 L,
préchauffés à 37°C, puis ensemencés pour
obtenir une DO initiale à
595 nm égale à 0.05. Les cultures d'expression sont
ensuite placées à 37°C sous une agitation
de 250 rpm. L'expression de la protéine de fusion K2
est induite par ajout de 1mM d'IPTG dans les milieux de culture lorsque la DO
mesurée à 595 nm atteint 1.2. Les fernbachs sont ensuite
incubés à 20°C sous une agitation de 250 rpm pendant 14
heures. A l'issu de ce délai,
la croissance bactérienne est stoppée en
introduisant les fernbachs de culture dans la glace.
La lyse des cellules procaryotes et l'extraction des
protéines ont été menées de la manière
suivante. Les bactéries sont récoltées par centrifugation
à 2830xg pendant 20 minutes
à 4°C. Après élimination des
surnageants, les culots bactériens sont soumis à un cycle
de congélation-décongélation dans l'azote liquide. Ils
sont ensuite resuspendus dans un tampon Tris contenant, 100 mM de Tris-HCl
(pH=8.0), 150 mM de NaCl, 5 % de glycérol, 1 mM d'EDTA, et 1 mM de
PMSF. La rupture des membranes bactériennes est
réalisée par lyse mécanique en utilisant une presse
d'Eaton. 10 mM de MgCl2, 10 mM de MgSO4, et
0.5 uL/mL de benzonase sont ensuite introduits dans les
lysats. Les fractions soluble et
insoluble sont finalement séparées par
centrifugation à 40000xg pendant 30 minutes à 4°C.
L'expression soluble de K2 en fernbach a
été contrôlée sur gel SDS-PAGE pour chacune
des 3 cultures contenant 1 L de milieu marqué, et pour chaque
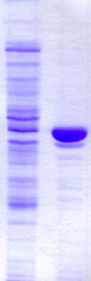
milieu de culture. Comme le montre la Figure 2.4, les niveaux d'expression
obtenus sont très satisfaisants en milieu minimum 15N et en
milieu Algone 15N / 13C. Ces profils d'expression sont
tout à fait comparables à ceux obtenus lors des tests
d'optimisation.
(kDa)
75
50
37
A B C
(kDa)
75
50
37
A B C
ZZ-K2 25
25
20 20
K2 15N Milieu Minimum
ZZ-K2
K2 15N / 13C Milieu
Algone
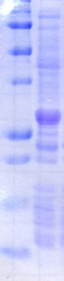
Figure 2.4 : Contrôle de
la production de la protéine de fusion K2 sous forme soluble
en
milieu minimum marqué 15N et en milieu
Algone 15N / 13C pour chaque litre de culture en
fernbach A, B, et C (analyse SDS-PAGE).
1.2.3) Purification de la protéine de fusion et
clivage de son partenaire
La purification de la construction 6xHis-ZZ-sTEV-K2
(ZZ-K2) a été entreprise par chromatographie
d'affinité sur résine IgG Sepharose 6 Fast Flow
(Amersham). Cette résine d'anticorps humains IgG greffés lie
de manière spécifique les fragments protéiques de type ZZ.
La colonne est préalablement équilibrée avec la solution
tampon Tris Saline Tween (TST) contenant, 50 mM de Tris (pH=7.5), 150 mM de
NaCl, 0.05 % de Tween 20, 1 mM de PMSF,
et 0.2 mM d'EDTA. Après injection des surnageants de lyse
bactérienne, la colonne est rincée plusieurs fois avec du tampon
d'équilibration TST jusqu'à retour de l'absorbance mesurée
à
280 nm à la ligne de base. La résine est alors
resuspendue dans du tampon TST et 2.6 mL de protéase recombinante TEV
à 1.8 mg/mL sont introduits dans la colonne. Après une nuit sous
agitation à 4°C, la solution de clivage contenue dans la
colonne est éluée avec du TST en fractions de 30 mL.
L'élution des protéines immobilisées à la
résine est menée par introduction de plusieurs volumes
d'acide acétique 0.5 M (pH=3.4). Toutes les fractions
d'élution sont déposées sur gel
d'électrophorèse SDS-PAGE et quantifiées par mesure
de l'absorbance à 280 nm sur un spectromètre UV. Le pH
des fractions d'acide acétique est préalablement
neutralisé avant analyses.
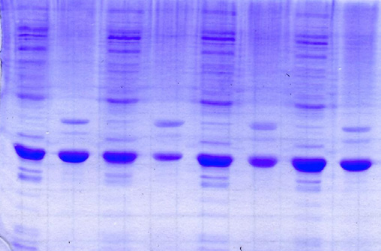
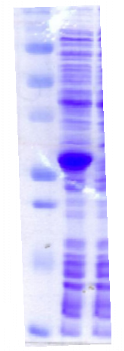
L'analyse SDS-PAGE des fractions de clivage fait
apparaître une bande de forte intensité à une masse
apparente de 13 kDa (Figure 2.5). Cette masse correspond au poids
moléculaire du domaine K2 (13.6 kDa), ce qui indique que
la protéine de fusion a été clivée
par la protéase TEV. Le tampon acide acétique
n'étant pas très adapté à la migration sur gel
d'électrophorèse, les bandes protéiques des fractions
d'élution apparaissent floues et peu intenses. Il est donc
difficile de réaliser une observation fine des protéines
présentes dans ces fractions et de calculer un rendement de coupure. On
peut toutefois observer des nuances de coloration à des masses
apparentes qui correspondent au partenaire ZZ clivé (20 kDa), et
à la protéine de fusion (33 kDa), ce qui démontre que la
coupure sur colonne n'est pas totale.
(kDa)
SN A B EL
(kDa)
SN A B EL
50
50
37
37
ZZ-K2



25 25
20 20
ZZ
15 15
K2
10
10
ZZ-K2
ZZ
K2
K2 15N Milieu Minimum
K2 15N / 13C Milieu
Algone
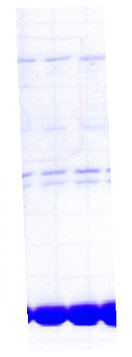
Figure 2.5 : Purification de la
protéine de fusion K2 sur IgG Sepharose et coupure sur
colonne du partenaire ZZ. Analyse SDS-PAGE du surnageant de
lyse bactérienne (SN), des fractions de clivage (A et B), et des
fractions d'élution à l'acide acétique (EL).
Au final, les rendements obtenus sont très
satisfaisants et atteignent, 30 mg/L en milieu minimum, et 25 mg/L en
milieu Algone. A l'issu de cette première étape, le degré
de pureté de K2 est estimé à 90% bien que
quelques bandes contaminantes de faible intensité soient observables
dans les fractions de clivage.
1.2.4) Purification du domaine K2
La seconde étape de purification de la protéine K2
a été réalisée par chromatographie
de pseudo-affinité sur colonne Ni-NTA Agarose
chargée en ions Ni2+ (Qiagen). La colonne est
équilibrée avec un tampon phosphate (50 mM, pH=7.2) contenant,
300 mM de NaCl, 4mM de
â-mercapto-éthanol, et 1mM de PMSF. Les
échantillons de K2 en solution dans du TST sont
directement chargés dans la colonne, et plusieurs
volumes de tampon phosphate sont
introduits jusqu'à retour de l'absorbance mesurée
à 280 nm à la ligne de base. L'élution est
menée par gradient linéaire d'imidazole avec du
tampon phosphate contenant de 10 à 500 mM d'imidazole. Les fractions
d'élution sont analysées sur gel SDS-PAGE (Figure 2.6) et
quantifiées par mesure de l'absorbance à 280 nm.
concentration en
imidazole (mM)
30
0
100
200
50
37



100
40
20
0
200


50
37
25 25
20 20
15
15
10 10
K2 15N Milieu Minimum
K2 15N / 13C Milieu
Algone
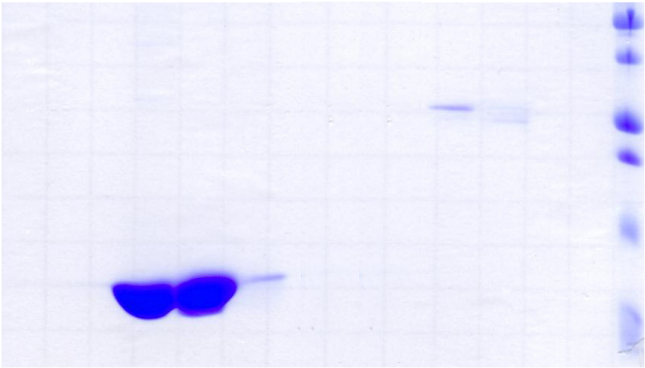
Figure 2.6 : Purification de K2
sur Ni-NTA Agarose. Analyse SDS-PAGE des fractions
d'élution par gradient linéaire d'imidazole (de
0 à 200 mM). La présence de « traînées »
en dessous de la bande correspondant à la protéine K2 sur le gel
de droite est due à un problème
de migration.
L'analyse SDS-PAGE des fractions d'élution fait
apparaître que la protéine K2 est retenue par la
résine de nickel jusqu'à des concentrations d'imidazole de
l'ordre de 30 mM. Sur les 4 histidines que compte K2, les 3
résidus H52, H55, et H57 sont proches dans la séquence
primaire. Par conséquent, ce type de fixation non spécifique,
à des concentrations d'imidazole relativement faibles, s'explique
probablement par la présence d'un amas, contenant ces 3
histidines, situé en surface de K2. Deux bandes
protéiques, de masse apparente autour de 30 kDa, sont observables dans
les fractions contenant une concentration d'imidazole d'environ 150 mM, et
correspondent à la protéase 6xHis-TEV (27kDa), et à la
protéine de fusion 6xHis-ZZ-sTEV-K2 (33 kDa).
Les rendements de l'expression du domaine K2 obtenus à
l'issu de cette seconde étape
de purification sont très satisfaisants car peu de
protéine a été perdue (Tableau 2.2). De plus,
aucune bande contaminante n'est clairement discernable sur
l'analyse SDS-PAGE des
fractions de K2. Par conséquent, nous pouvons
estimer avoir préparé des échantillons simplement et
doublement marqués avec un degré de pureté
supérieur à 95%.
|
Milieu
|
Milieu Minimum 15N
|
Milieu Algone 15N / 13C
|
|
A) Rendement IgG
|
30 mg/L
|
25 mg/L
|
|
B) Rendement final
|
26 mg/L
|
22 mg/L
|
Tableau 2.2 : Rendements de
l'expression de K2, après purification sur IgG Sepharose
et
coupure sur colonne du partenaire (A), et à l'issu de
la 2nde purification sur résine Ni2+ (B).
1.2.5) Préparation du tube RMN
La dernière étape de préparation de
l'échantillon pour l'analyse RMN a consisté à
concentrer la protéine à une valeur proche de 1 mM.
L'ultrafiltration de K2 a été menée sur cellule Amicon de
10 mL équipée d'une membrane YM3 avec un seuil de coupure de 3
kDa. Afin de réduire la concentration en NaCl et d'éliminer
l'imidazole, plusieurs étapes de lavage- concentration ont
précédé la concentration finale de K2. A l'issue
de l'ultrafiltration, la protéine est en solution dans un tampon
phosphate (50 mM, pH=7.0) contenant, 150mM de NaCl, 1 mM d'EDTA, 1 mM de
TCEP, et 1 mM de PMSF. L'échantillon RMN est alors obtenu en
ajoutant 10% de D2O, 2 mM de TSP, 2 mM de cocktail d'inhibiteurs (SIGMA), et
0.01% de NaN3. 600 uL de cette préparation
ont été introduits dans un tube RMN de 5 mm de
diamètre placé ensuite sous atmosphère
d'argon.
| 


