ANNEXE
Criblage des conditions d'expression des
protéines PROentier, PROcatal, PROter, et
PROinser
1) Matériels et Méthodes
1.1) Construction des plasmides d'expression par
recombinaison homologue
1.1.1) Stratégie employée
Commercialisée par la société
Invitrogen, la technologie Gateway s`appuie sur les
propriétés de recombinaison homologue du
bactériophage lambda au niveau des sites spécifiques
att, qu'il utilise naturellement pour s'intégrer dans le
génome d'E. coli (Landy,
1989). Ce mécanisme naturel a été
détourné pour en faire un système de recombinaison
in vitro simple et efficace. Le système de clonage Gateway
présente de nombreux avantages et permet notamment de
s'affranchir des étapes de restriction-ligation obligatoires lors
d'un clonage classique. L'insertion d'un insert d'ADN est spécifique et
directionnelle (conservation du cadre de lecture), et le moyen de
sélection est rapide. Il est alors facile d'obtenir
différents plasmides d'expression à partir d'un seul plasmide
d'entrée.
Le principe de la méthodologie de construction des
différents vecteurs d'expression à partir de la technique
Gateway est représenté dans la Figure A1. La
première étape consiste à adjoindre aux gènes
encodant les domaines PRODH les 2 sites de recombinaison spécifique,
attB1 en amont, et attB2 en aval du gène, par PCR
d'assemblage. Un site de clivage reconnu
par la protéase TEV (Tobacco Etch Virus)
est également incorporé en amont du gène
d'intérêt. Les amplicons de PCR, flanqués des sites
attB, sont alors clonés dans le vecteur donneur pDONR221
(Invitrogen) possédant les sites attP1 et attP2
par réaction de BP clonase. Ce vecteur donneur contient
également un gène de résistance à la kanamycine
(KmR), ainsi qu'un second au chloramphénicol (CmR)
inséré entre les 2 sites attP. Les enzymes de la
BP clonase reconnaissent de manière
spécifique les sites attB et attP, ce qui
conduit à l'insertion du gène d'intérêt dans le
vecteur donneur. Il se forme alors par cette réaction un vecteur
d'entrée possédant le gène de résistance
à la kanamycine (KmR), les sites de recombinaison
homologue AttL1 et AttL2, et le gène
d'intérêt.
Le vecteur d'entrée est utilisé pour
obtenir l'ensemble des vecteurs d'expression à partir de
vecteurs pDEST, qui contiennent un gène encodant un des 6 partenaires de
fusion,
un gène de résistance à l'ampicilline
(AmpR), et les sites de recombinaison attR1 et
attR2. Le
gène d'intérêt est «
transféré » du vecteur d'entrée au
vecteur d'expression de manière unidirectionnelle
grâce aux enzymes de la LR clonase qui reconnaissent les sites attL
et attR
de manière spécifique. Au final, ces
plasmides d'expression comportent, un gène de
résistance à l'ampicilline (AmpR), une
étiquette 6xHis, un gène encodant un des 6 partenaires
de fusion, le gène d'intérêt, et un site de
clivage par la TEV inséré entre le partenaire et le gène
d'intérêt.
Gène
PCR
TEV Gène
CmR
+
attP1 attP2
pDONR
221
attB1 attB2
KmR
BP clonase
Gène
attL1 attL2
Vecteur d'entrée
KmR
LR clonase




CmR
attR1 attR2
pDEST
Gène
attB1 attB2
Vecteur d'expression
AmpR
AmpR
Figure A1 : Résumé
des principales étapes de la construction des vecteurs
d'expression à
partir de la technique de clonage par recombinaison homologue
Gateway.
1.1.2) PCR d'assemblage
Outre l'amplification des gènes
d'intérêt, la PCR d'assemblage a pour objectif d'adjoindre,
un site de reconnaissance AttB1 et un site de clivage
reconnu par la TEV en amont du gène, et un site de reconnaissance
AttB2 et une étiquette Tag-S en aval du gène. Le Tag-S
encode un fragment protéique qui permet de détecter
l'expression des protéines de fusion par un test de
fluorescence. Dans le cas des domaines PRODH, ce test, qui
était en cours de mise au point, n'a pas été
utilisé et l'expression des protéines
hétérologues a été uniquement analysée sur
gel SDS-PAGE.
L'introduction de longues séquences de nucléotides
non spécifiques du gène en amont
et en aval des gènes d'intérêt
(respectivement 57 et 60 bp) nécessite de réaliser 2
réactions de PCR successives. La première PCR (PCR1) fait
intervenir une amorce sens spécifique du gène contenant le
site de clivage par la TEV et une partie du site attB1, ainsi qu'une
amorce anti-sens complémentaire à la fin du gène
comportant l'étiquette Tag-S. L'ensemble des amorces
utilisées pour amplifier les gènes encodant les domaines PRODH
est reporté dans le Tableau A1.
|
PCR
|
Amorces
|
Séquences
|
|
PCR1
|
S
PROentier AS
|
5'-GGCTTCGAGAATCTTTATTTTCAGGGCGGACATTTTGTAGCCGGGGAG-3'
5'-TTAGCTGTCCATGTGTTGGCGTTCGAATTTAGCAGCAGCGGTTTCTTTCTAGGC
AGGGCGATGGAAGAGGTT-3'
|
|
S
PROcatal AS
|
5'-GGCTTCGAGAATCTTTATTTTCAGGGCGGACATTTTGTAGCCGGGGAG-3'
5'-TTAGCTGTCCATGTGTTGGCGTTCGAATTTAGCAGCAGCGGTTTCTTTCTAGGC
AGGGCGATGGAAGAGGTT-3'
|
|
S
Proter AS
|
5'-GGCTTCGAGAATCTTTATTTTCAGGGCGGAGGAGGGTCGGCAACGGCAGTG-3'
5'-TTAGCTGTCCATGTGTTGGCGTTCGAATTTAGCAGCAGCGGTTTCTTTCTAGGA
CTCGTGGTCTTCCCCGGC-3'
|
|
S
Proinser AS
|
5'-GGCTTCGAGAATCTTTATTTTCAGGGCGGGAGACCACAGTTTCTGCTGCAG-3'
5'-TTAGCTGTCCATGTGTTGGCGTTCGAATTTAGCAGCAGCGGTTTCTTTCTACTC
CTCCTCCTCAGTGAACCCGGA-3'
|
|
PCR2
|
AttB1-TEV-S
|
5'-GGGGGGGGGACAAGTTTGTACAAAAAAGCAGGCTTCGAGAATCTTTATTTTCAG
GGC-3'
|
|
AttB2-TagS-AS
|
5'-GGGGGGGGGACCACTTTGTACAAGAAAGCTGGGTCCTTAACTGTCCATGTGCTG
GCGTTCGAA-3'
|
Tableau A1 : Amorces sens (S) et
anti-sens (AS) utilisées pour les PCR d'assemblage. Les
oligonucléotides qui apparaissent en orange
correspondent au site de recombinaison AttB1,
en bleu au site de clivage par la TEV, en violet
au Tag-S, et en rouge au site de reconnaissance AttB2.
Les mélanges réactionnels se composent de
12 ng de vecteur de départ contenant le gène
d'intérêt, 0.1 mM de dNTP, 0.4 uM d'amorce sens, 0.4 uM d'amorce
anti-sens, 4.5 unités de polymérase, et 5uL de tampon Pfu
10X dans un volume final de 50 uL complété avec de l'eau
milliQ. L'enzyme utilisée est la Pfu DNA polymerase
commercialisée par Promega. Ces mélanges sont introduits
dans des barrettes de 16 tubes PCR de 250 uL (Promega) et traités en
parallèle avec des pipettes multicanaux. Les conditions de PCR
utilisées sont : un cycle de dénaturation thermique (2 min
à 94°C), suivi de 30 cycles de PCR (45 sec à 94°C, 45
sec à
50°C, et 4 min 30 à 72°C), puis 10 min
d'élongation finale à 72°C. Les produits de PCR1 sont
purifiés sur gel avec le kit QIAquick purification (Qiagen), et
quantifiés sur gel d'agarose.
La seconde PCR (PCR2) utilise une amorce sens non
spécifique du gène qui adjoint la région
N-terminale du site attB1 en amont du site de
clivage par la TEV. L'amorce anti-sens est également non
spécifique du gène et permet d'insérer le site de
reconnaissance attB2 en aval
du Tag-S. La composition des mélanges réactionnels
et les conditions de PCR sont identiques
à celles utilisées pour la PCR1. Environ 5 ng de
produit de PCR1 purifié servent de matrice.
La purification et la quantification des produits de PCR2 se font
de la même manière que pour
la PCR1.
1.1.3) Construction des vecteurs
d'entrée
Les produits de PCR2, contenant les gènes
d'intérêt flanqués des sites AttB, sont
clonés dans le vecteur donneur pDONR 221 par réaction de BP
clonase. Les réactifs utilisés, ainsi que le protocole
original, sont fournis par Invitrogen. Le mélange
réactionnel est composé de 1 uL de Clonase Reaction
Buffer 5X, 150 ng de vecteur pDONR 221 (1 uL),
1 uL de tampon Tris-EDTA (Tris 10mM à pH 8.0 et EDTA 1mM),
1 uL de produit de PCR2,
et 1 uL de mélange enzymatique BP clonase. L'ensemble est
incubé 3 heures à 25°C, puis traité à la
protéinase K (0.4 ug/uL) pendant 10 min à 37°C afin de
stopper la réaction. 50 uL
de bactéries thermocompétentes DH5á
sont transformées par le produit de réaction BP clonase
par choc thermique à 42°C pendant 45 secondes. Le produit
de transformation est étalé sur boîte LB/agar contenant
50 ug/mL de kanamycine, puis incubé à 37°C pendant toute une
nuit. Le lendemain, une dizaine de colonies sont repiquées sur milieu
LB/agar/kanamycine, puis une PCR sur colonie est réalisée sur
chacun des clones en utilisant une amorce sens (M13) s'hybridant sur le
plasmide, et une amorce anti-sens s'hybridant sur le gène
d'intérêt. Les colonies, conduisant après PCR à une
bande unique dont la taille apparente correspond à la taille
moléculaire attendue, sont mises en culture dans du LB à
37°C. Les vecteurs d'entrée sont alors extraits et
purifiés avec le kit Wizard Plus Minipreps DNA Purification
System (Promega). Les produits de ces minipréparations
sont finalement séquencés sur appareil 310 Genetic
Analyser (ABI Prism, Applied Biosystems).
1.1.4) Construction des vecteurs
d'expression
Les 7 plasmides de type pDEST possèdent un gène
encodant un partenaire de fusion
en aval de l'étiquette 6xHis, à l'exception du
vecteur pDEST-17 (Invitrogen) qui ne contient pas de partenaire. La formation
des vecteurs d'expression est réalisée en mélangeant 1.8
uL
de vecteur d'entrée (300 ng), 1.2 uL de chacun
des vecteurs pDEST (100 à 300 ng), 1 uL de
Clonase Reaction Buffer 5X, et 1 uL de mélange
enzymatique LR clonase. L'incubation dure
5 heures à température ambiante, puis
la protéinase K est ajoutée (0.4 ug/uL). La
transformation des bactéries DH5á thermocompétentes est
alors menée comme
précédemment par choc thermique à 42°C
pendant 45 secondes. L'analyse des transformants
se fait d'une part, par PCR sur colonie en utilisant une
amorce sens spécifique du promoteur T7, et une amorce anti-sens
spécifique du site AttB2. D'autre part, un repiquage sur milieu
LB/agarose-kanamycine, -chloramphénicol, et -ampicilline, permet de
vérifier le profil de résistance des vecteurs d'expression.
Seules les colonies qui présentent une résistance unique
à l'ampicilline sont sélectionnées puis
stockées à -80°C avec du glycérol.
1.2) Criblage des conditions d'expression en
microplaque
Le criblage des conditions d'expression des domaines
PRODH sur microplaques 96 puits (Falcon) a été
réalisé en suivant les protocoles mis au point par les chercheurs
du LMP dans le cadre de la plate-forme 3PM (une microplaque par souche et par
température).
1.2.1) Transformation des souches d'E. coli et
préparation des précultures
Pour chaque puits, 50 uL de bactéries
thermocompétentes sont transformées par 20 ng
de vecteur d'expression par choc thermique
à 42°C pendant 45 secondes. Après ajout de
200 uL de milieu SOC, les microplaques sont incubées
pendant 1 heure à 37°C. Chaque puits
est alors doté des antibiotiques appropriés
(Ampicilline à 100 ug/mL pour la sélection des plasmides
d'expression, et chloramphénicol à 35 ug/mL uniquement pour la
souche Rosetta pLysS), et les plaques sont incubées sur la nuit à
37°C sous une agitation de 250 rpm. On peut ainsi noter que les cultures
de transformation servent également de préculture.
1.2.2) Cultures d'expression
Les cultures d'expression, contenant 240 uL de LB et les
antibiotiques appropriés, sont ensemencées avec un volume de
préculture de manière à obtenir une DO600 initiale de
0.05 (utilisation du lecteur de microplaques ELX 800
Universal Microplate Reader de la
société Bio-Teck Instruments). La croissance est
maintenue sous agitation (1000 rpm) à 37°C jusqu'à une DO600
de 1.2 pour une induction à 20°C, et de 0.4 à 0.6 pour une
induction à 37°C (0.4 pour les BL21 Star, 0.5 pour les C41, et 0.6
pour les Rosetta). Les souches C41 et Rosetta sont induites avec 1mM d'IPTG, et
la souche BL21 Star avec 0.5 mM d'IPTG. L'induction
est alors prolongée pendant 3 heures à 37°C,
ou sur la nuit à 20°C sous une agitation de 1000
rpm. La croissance bactérienne est alors stoppée
par centrifugation à 2830xg et à 4°C pendant
20 minutes.
1.2.3) Lyse des bactéries et séparation des
fractions soluble et insoluble
Les culots bactériens sont lysés par reprise dans
50 uL de tampon de lyse contenant,
100 mM de Tris-HCl (pH 8.0), 150 mM de NaCl, 0.1 % de
Triton X-100, 1 uM de phosphoramidon, et 1 mM de PMSF. 0.04 ug/mL de
lysozyme (Sigma) sont ensuite ajoutés,
et les microplaques sont mises à incuber sous agitation
modérée à 4°C pendant 1 heure. La lyse de l'ADN est
initiée en ajoutant 0.125 U/uL de benzonase (Merck) et 10 mM de MgCl2.
Les microplaques sont alors incubées à température
ambiante pendant 10 minutes sous légère agitation. Les fractions
soluble et insoluble sont séparées par centrifugation à
2830xg pendant
1 heure 30 à 4°C. L'expression des
protéines de fusion est analysée sur gel SDS-PAGE.
10 uL de chaque échantillon sont dilués dans du
« bleu de charge » réducteur 2X [Tris-Hcl
62.5 mM pH 6.8, bleu de bromophénol 0.02 % (w/v), SDS 2
% (v/v), â-mercaptoéthanol 280 mM, glycérol 20 %
(v/v)], avant de subir une dénaturation thermique à
100°C pendant 5 minutes. 10 uL sont finalement déposés sur
gel de polyacrylamide (8 à 12 %).
2) Résultats
Comme je l'ai exposé dans le chapitre 4 de la
première partie de ce manuscrit (cf. § 2.2.2), les domaines
PROcatal et PROentier ont été exprimés dans 28
conditions expérimentales différentes (7 partenaires de
fusion, 2 souches d'expression, et 2 températures), contre 42
conditions pour les domaines PROter et PROinser (7 partenaires, 3 souches, et 2
températures). Pour chaque condition testée, les fractions
contenant les protéines solubles et insolubles ont été
déposées sur gel SDS-PAGE. L'analyse de l'expression des
protéines de fusion à partir de l'interprétation
des profils électrophorétiques des gels SDS- PAGE est
reportée, domaine par domaine, dans un tableau récapitulatif
(Figures A2 à A5).
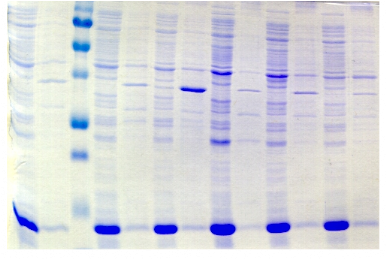
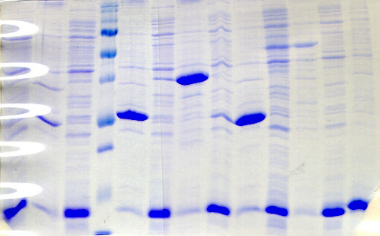
2.1) Le domaine PROcatal
Que ce soit en souche BL21 Star ou Rosetta, les
protéines de fusion PROcatal sont globalement bien exprimées, et
ceci avec l'ensemble des partenaires de fusion (Figure A2). Seule la
construction avec Gb1 conduit à une expression faible, voire nulle en
souche Rosetta
et à 20°C. Cependant, ces bons résultats en
terme d'expression ne sont pas associés à de bons profils de
solubilité. En effet, on constate que la majorité des
protéines hétérologues PROcatal sont exprimées dans
la fraction insoluble. C'est le cas notamment pour les constructions avec
ZZ et la GST, ainsi que pour les fusions Gb1, Trx, et
His, qui conduisent toutefois dans
certaines conditions à une expression soluble très
minoritaire.
PM
(kDa)
250
Gb1
S I
ZZ GST
S I S I
Trx
S I
MBP
S I
His
S I
NusA
S I
PM
(kDa)
250
Gb1
S I
ZZ GST
S I S I
Trx
S I
MBP
S I
His
S I
NusA
S I
150
100
75
50
37
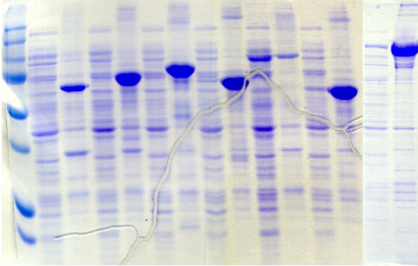
·
·
·
·
·
·
· 150
100
75
50
37
·
·
·
·
·
·
·
25
20
BL21 Star (37°C)
25
20
BL21 star (20°C)
PM
(kDa)
250
150
100
75
Gb1
S I
ZZ GST
S I S I
·
Trx
S I
MBP
S I
·
His
S I
NusA
S I
·
(kDa)
250
150
100
PM Gb1
S I
ZZ GST
S I S I
·
Trx
S I
MBP
S I
·
His
S I
NusA
S I
·
·
·
·
·
50
75
·
·
·
·
50
37
Rosetta (37°C)
37
Rosetta (20°C)
PROcatal
Gb1 ZZ GST Trx MBP His NusA
S I S I S I S I S I S I S I
BL21 Star
37°C - ++(+) - +++(+) - +++(+) +
+++(+) +(+) + - +++(+) +(+) ++++
BL21 Star
20°C + ++ - ++++ - ++++ + ++++ +(+) +
- ++++ +(+) ++++
Rosetta
37°C - ++ - +++ +/- ++(+) + +++(+) +
++ + +++ +(+) +++(+)
Rosetta
20°C +/- +/- - ++++ - +++ - +++(+)
++(+) ++ - ++(+) ++ ++++
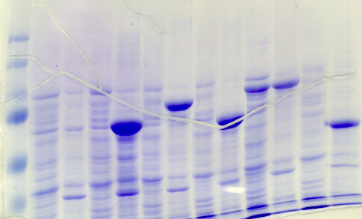
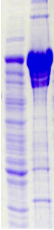
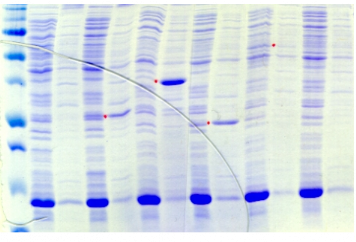
Figure A2 : Analyse de l'expression
et de la solubilité des protéines de fusion PROcatal dans
28 conditions expérimentales
différentes.
Les points rouges sur les gels SDS-PAGE
correspondent aux masses attendues des protéines de fusion.
Après dépôt des fractions soluble (S) et insoluble
(I), chaque bande de surexpression est analysée, et une valeur
semi-quantitative est associée à son intensité, de
faible (+) à très forte (++++). Les fractions non
lisibles sont annotées du sigle NL, et les fractions se
confondant aux protéines intrinsèques à E. coli et
constituant un doute, sont associées au sigle +/-. Les
notations surlignées en couleur mettent en évidence les
meilleures conditions d'expression et de solubilité : couleur
orange pour la meilleure condition, et couleur jaune pour la deuxième
meilleure condition.
De manière plus intéressante, on retrouve ce profil
d'expression pour la construction
PROcatal fusionnée avec le partenaire NusA.
L'expression dans la fraction insoluble y est très intense, mais
contrairement aux autres protéines de fusion, le taux d'expression de
protéines solubles est significatif, et notamment en souche Rosetta
cultivée à 20°C.
Le meilleur résultat en terme d'expression de
protéines solubles PROcatal est obtenu avec le partenaire MBP en
souche Rosetta cultivée à 20°C. La combinaison de
ces trois conditions permet en effet d'obtenir une bonne expression avec un
taux de protéines solubles tout à fait satisfaisant.
Au final, il apparaît que la nature du partenaire de
fusion joue un rôle primordial sur la solubilité des
protéines hétérologues PROcatal. L'influence de la souche
bactérienne et de la température d'expression semble à
première vue moins prononcée, mais elle est flagrante
lorsque PROcatal est exprimée en fusion avec NusA et MBP.
Ainsi, l'association souche Rosetta-température d'expression de
20°C se démarque nettement des autres conditions dans
la mesure où elle permet d'améliorer
sensiblement le profil de solubilité des constructions avec NusA
et MBP.
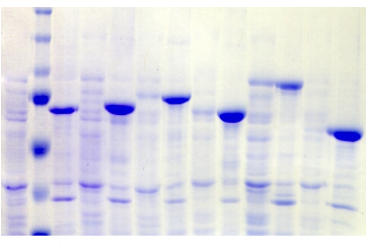
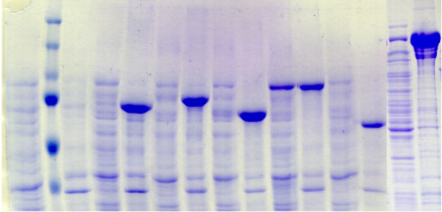
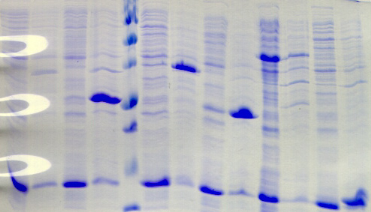
2.2) La protéine PROentier
PROentier est constitué à 86 % des acides
aminés du domaine PROcatal et de manière intéressante, on
constate que le profil d'expression des protéines de fusion
PROentier est similaire à celui des protéines
hétérologues PROcatal, voire identique dans certaines
conditions. Ainsi, le meilleur résultat, en termes d'expression et de
solubilité, est obtenu avec
le partenaire MBP en souche Rosetta cultivée à
20°C.
Comme le met en évidence la Figure A3, la nature
du partenaire de fusion semble jouer un rôle essentiel sur
l'expression et la solubilité des protéines
hétérologues PROentier :
les constructions avec His, Gb1, ZZ, Trx, et GST conduisent
à une expression quasi exclusive
de protéines insolubles ; et même si pour Trx, on
retrouve systématiquement une expression très faible dans la
fraction soluble, seules les constructions avec MBP et NusA permettent, dans
certains cas, d'observer une expression suffisante de protéines
solubles.
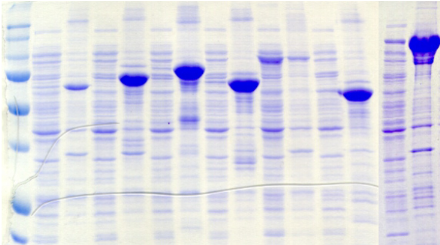
(kDa)
250
150
100
Gb1
S I
ZZ GST
S I S I
Trx
S I
MBP
S I
·
His
S I
NusA
S I
·
(kDa)
250
150
100
Gb1
S I
ZZ GST
S I S I
Trx
S I
MBP
S I
·
His
S I
NusA
S I
·
75
·
·
·
·
·
50
37
75
·
·
·
·
·
50
37
25
20
BL21 Star (37°C)
25
20
BL21 Star (20°C)

(kDa)
250
150
100
Gb1
S I
ZZ GST
S I S I
Trx
Rosetta (37°C)
S I
MBP
S I
·
His
S I
NusA
S I
·
(kDa)
250
150
100
Gb1
S I
ZZ GST
S I S I
Trx
S I
MBP
S I
His
S I
NusA
S I
·
75
·
·
·
·
·
50
·
·
75
·
·
·
·
50
37
37
25
Rosetta (20°C)
PROentier
Gb1 ZZ GST Trx MBP His NusA
S I S I S I S I S I S I S I
BL21 Star
37°C - ++(+) - +++(+) + +++(+) +
+++(+) + + - +++(+) + +++
BL21 Star
20°C - +(+) + ++(+) +/- ++++ + ++++ +
+ - +++(+) + +++(+)
Rosetta
37°C - ++ +/- +++ - ++(+) + +++(+) +
++ + +++ + +++(+)
Rosetta
20°C +/- +/- - +++(+) - +++ + +++ ++
++ - ++ +(+) ++++
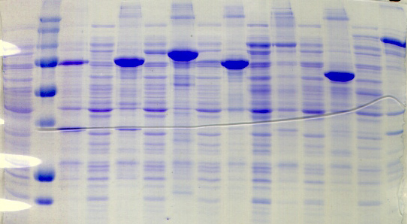
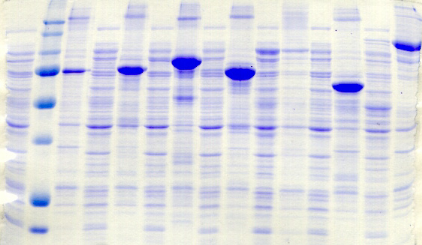
Figure A3 : Analyse de
l'expression et de la solubilité des protéines de fusion
PROentier
dans 28 conditions expérimentales
différentes.
Comme précédemment, les résultats du
criblage des conditions d'expression des
protéines de fusion PROentier ne mettent en
évidence aucun effet direct de la souche bactérienne ou de
la température d'expression sur l'expression et la solubilité des
protéines de fusion recombinantes. Cependant, on retrouve pour les
constructions avec MBP et NusA, un effet souche couplé à un effet
température qui met une nouvelle fois en valeur le pouvoir de
solubilisation de la combinaison souche Rosetta-température d'expression
de 20°C.
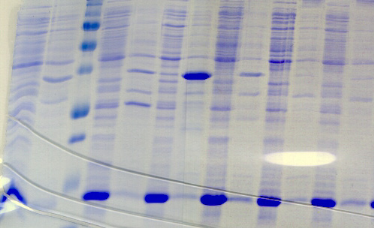
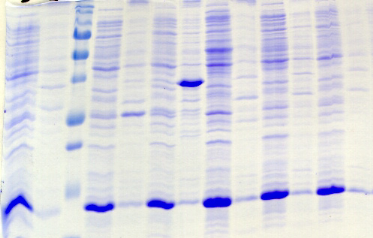
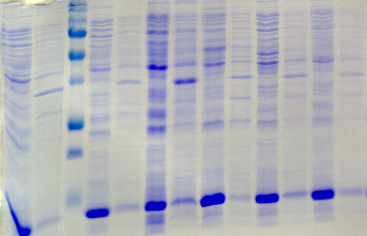
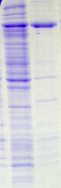
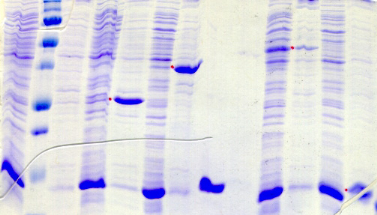
2.3) Le domaine PROter
L'expression des protéines de fusion PROter n'a
été initialement testée qu'en souches BL21 Star et
Rosetta. Comme le montrent les gels SDS-PAGE et le tableau d'analyse de la
Figure A4, la majorité de ces protéines
hétérologues ne s'expriment pas dans ces deux souches ; ou
l'expression est si faible qu'elle n'est pas décelable sur gel SDS-PAGE.
C'est le
cas notamment des constructions avec Gb1, Trx, MBP, et His.
Pour PROter en fusion avec le partenaire ZZ, une expression faible
est observable dans la fraction insoluble, mais uniquement en
souche BL21 Star. Seule la construction avec GST s'exprime dans les deux
souches. Cette expression est exclusivement insoluble, sauf en BL21 Star
cultivée à 20°C où une bande de faible
intensité apparaît dans la fraction soluble.
En ce qui concerne la construction avec NusA, nous avons
rencontré des difficultés lors de l'étape de
transformation des BL21 Star par le plasmide d'expression. En effet,
malgré plusieurs essais, nous n'avons pas été en mesure de
transformer cette souche par notre vecteur d'intérêt. Ce
problème ne s'est pas posé avec la souche Rosetta, et
contrairement aux autres protéines de fusion, les taux d'expression
obtenus à 37°C et 20°C avec NusA sont significatifs dans les
fractions soluble et insoluble.
De manière intéressante, nous avons
constaté que la transformation des souches par les vecteurs d'expression
PROter induit une diminution importante de la croissance
bactérienne après induction par l'IPTG. Cette baisse de la
vitesse de croissance associée à une expression réduite
de la plupart des protéines de fusion est tout à fait
caractéristique de protéines hétérogènes
toxiques. Par conséquent, il nous est apparu nécessaire de tester
l'expression des protéines de fusion PROter en souche C41 qui
est adaptée à la production de ce type de
protéines.
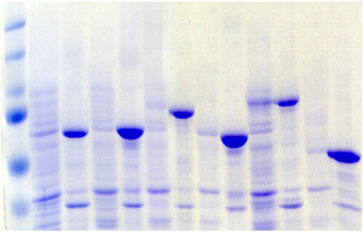
(kDa)
100
75
50
37
25
20
Gb1
S I
·
ZZ GST
S I S I
·
·
Trx
S I
·
MBP
S I
·
His
S I
(kDa)
100
75
50
37
25
20
Gb1
S I
·
ZZ GST
S I S I
·
·
Trx
S I
·
MBP
S I
·
His
S I
15
·
BL21 Star (37°C)
15
·
BL21 Star (20°C)
(kDa)
100
75
50
37
25
20
Gb1
S I
·
ZZ GST
S I S I
·
·
Trx
S I

·
MBP
S I
·
His
S I
NusA
S I
·
(kDa)
150
100
75
50
37
25
20
Gb1
S I
·
ZZ GST
S I S I
·
·
Trx
S I
·
MBP
S I
·
His
S I
NusA
S I
·
15
·
Rosetta (37°C)
15
·
Rosetta (20°C)
|
Gb1
|
ZZ
|
GST
|
Trx
|
MBP
|
His
|
NusA
|
|
S I
|
S I
|
S I
|
S I
|
S I
|
S I
|
S I
|
|
|
|
|
·
|
|
·
|
(kDa)
150
100
75
50
37
·
25
·
·
20
·
(kDa)
150
100
75
50
37
25
20
Gb1
S I
·
ZZ GST
S I S I
·
·
Trx
S I
·
MBP
S I
·
His
S I
NusA
S I
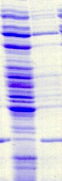
·
15 15
·
10 10

C41 (37°C)
·
C41 (20°C)
|
S
|
I
|
S
|
I
|
S
|
I
|
S
|
I
|
S
|
I
|
S
|
I
|
S
|
I
|
|
-
|
-
|
+/-
|
+
|
+/-
|
+++
|
-
|
-
|
-
|
-
|
-
|
-
|
ND
|
ND
|
|
-
|
-
|
+/-
|
+
|
+
|
++
|
-
|
-
|
-
|
-
|
-
|
-
|
ND
|
ND
|
|
-
|
-
|
-
|
-
|
+/-
|
++(+)
|
-
|
-
|
-
|
-
|
-
|
-
|
+
|
+(+)
|
|
-
|
-
|
-
|
-
|
-
|
+(+)
|
-
|
-
|
-
|
-
|
-
|
-
|
++
|
++
|
|
-
|
-
|
+/-
|
+
|
+
|
++(+)
|
+/-
|
++
|
+
|
+/-
|
-
|
-
|
+(+)
|
++(+)
|
|
-
|
-
|
-
|
-
|
+/-
|
++(+)
|
+
|
++(+)
|
+/-
|
-
|
-
|
-
|
+(+)
|
+(+)
|
PROter
Gb1 ZZ GST Trx MBP His NusA
BL21 Star
37°C
BL21 Star
20°C
Rosetta
37°C
Rosetta
20°C
C41
37°C
C41
20°C
Figure A4 : Analyse de
l'expression et de la solubilité des protéines de fusion
PROter dans 42 conditions expérimentales différentes.
Comme le met en évidence la Figure A4, on constate que la
souche C41 apporte une
amélioration significative des taux d'expression des
protéines hétérologues PROter. Ainsi, alors qu'en
souches BL21 Star et Rosetta l'expression des constructions Trx et MBP
n'était pas détectable, des bandes de surexpression
sont observables en C41 pour ces deux constructions, et notamment
pour le partenaire Trx où l'effet souche est assez spectaculaire.
En revanche, la production de PROter en fusion avec Gb1 et His
n'est toujours pas détectable
en souche C41. En ce qui concerne les constructions avec ZZ et
GST, les profils d'expression obtenus en souche C41 sont comparables à
ceux obtenus avec les deux autres souches.
Au vu de l'ensemble de ces résultats, il apparaît
que la nature du partenaire de fusion
et de la souche bactérienne est déterminante
pour l'expression et la solubilité des protéines
hétérologues PROter. En effet, seule la construction avec
NusA permet d'obtenir une expression suffisante de protéines
solubles en souches Rosetta ou C41. L'influence de la
température semble plus limitée, mais comme observé
précédemment, l'association souche Rosetta-température
de 20°C combinée à un partenaire de fusion efficace
conduit aux meilleurs taux d'expression de protéine soluble.
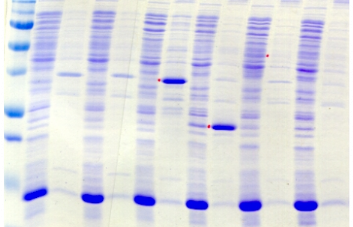
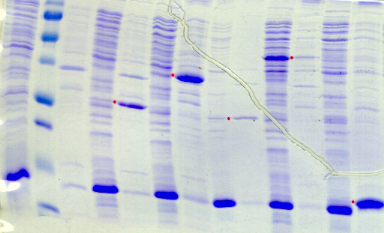
2.4) Le domaine PROinser
Les protéines hétérologues PROinser
présentent un profil d'expression qui ressemble davantage à
celui des protéines de fusion PROcatal et PROentier,
plutôt qu'à celui des protéines PROter. Ainsi, le
meilleur résultat est obtenu avec le partenaire MBP en souche
Rosetta cultivée à 20°C avec un taux d'expression de
protéines solubles tout à fait satisfaisant.
Comme le montre la Figure A5, les niveaux d'expression obtenus
sont très élevés pour l'ensemble des partenaires de
fusion, à l'exception de la construction avec Gb1, qui est peu ou non
exprimée. Cependant, cette production est majoritairement insoluble pour
la plupart des partenaires de fusion : c'est le cas notamment pour His, GST,
Trx, et ZZ. Aussi, dans le cas
de GST et Trx, une faible expression est presque
systématiquement observable dans la fraction soluble. A l'instar des
3 autres domaines PRODH, seules les constructions avec MBP
et NusA conduisent à une production significative dans la
fraction soluble.
(kDa)
100
75
50
37
Gb1
S I
ZZ GST
S I S I
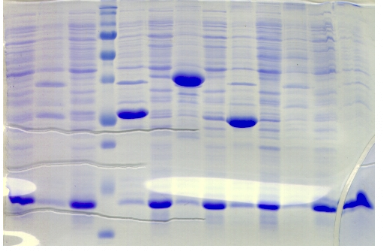

·
Trx
S I
MBP
S I
·
His
S I
NusA
S I
·
(kDa)
100
75
50
37
Gb1
S I
ZZ GST
S I S I

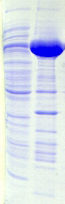
·
Trx
S I
MBP
S I
·
His
S I
NusA
Annexe : Criblage des conditions d'expression des
protéines PROentier, PROcatal, PROter, et PROinser
S I
·
25
·
·
·
20
25
·
·
·
20
15
·
10
BL21 Star (37°C)
15
·
10
BL21 Star (20°C)
(kDa)
100
75
50
37
Gb1
S I
ZZ GST
S I S I
·
Trx
S I
MBP
S I
·
His
S I
NusA
S I

·
(kDa)
100
75
50
37
Gb1 ZZ
S I S I
GST
S I
·
Trx
S I
MBP
S I
·
His
S I
NusA
S I

·
25
·
·
·
20
25
·
·
·
20
15
·
10
Rosetta (37°C)
15
10
·
Rosetta (20°C)
(kDa)
100
75
50
37
Gb1
S I
ZZ GST
S I S I
·
Trx
S I
MBP
S I
·
His
S I
NusA
S I
·
(kDa)

100
75
50
37
Gb1
S I
ZZ GST
S I S I
·
Trx
S I
MBP
S I
·
His
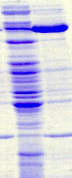
S I
NusA
S I
·
25
·
·
·
20
25
·
·
20
·
15
15
· 10
10
C41 (37°C)
·
C41 (20°C)
PROinser
BL21 Star
Gb1 ZZ GST Trx MBP His NusA
S I S I S I S I S I S I S I
37°C - +/- +/- +++(+) + ++++ + ++++
+ + +/- +++ +(+) +++(+)
BL21 Star
20°C - - - +++ + ++++ + +++ + + +/-
++(+) +(+) ++++
Rosetta
37°C - + - +++ + +++(+) + +++(+)
+(+) +(+) +/- ++(+) +(+) +++(+)
Rosetta
20°C - - +/- +++ + +++ + +++ ++(+) +
+/- +++ +(+) ++++
C41
37°C - - +/- +++ +/- +++ ND ND ++ +(+)
- +++ +/- +++
C41
20°C - - + ++(+) + +++ + + ++ + -
+++(+) +(+) +++(+)
Figure A5 : Analyse de
l'expression et de la solubilité des protéines de fusion
PROinser dans 42 conditions expérimentales différentes.
Annexe : Criblage des conditions d'expression des
protéines PROentier, PROcatal, PROter, et PROinser
L'analyse du criblage des conditions d'expression de PROinser met
donc une nouvelle
fois en évidence le rôle essentiel du
partenaire de fusion sur la solubilité des protéines
hétérologues. Aussi, contrairement aux résultats obtenus
avec les autres domaines PRODH, il
est plus difficile de mettre en évidence un effet
direct de la souche bactérienne ou de la température sur
l'expression et la solubilité des protéines de fusion PROinser.
Dans le cas de
la construction avec MBP, on constate toutefois que la
souche Rosetta donne de meilleurs résultats que les souches
BL21 Star et C41. Par ailleurs, même si un effet solubilisant
de l'association souche Rosetta-température de 20°C est
observé pour la construction avec MBP,
ce dernier ne se retrouve pas pour la construction avec NusA.
Références bibliographiques
| 


