|
BURKINA FASO
UNITE-PROGRES-JUSTICE
*************************************
MINISTERE DE L'ENSEIGNEMENT SUPERIEUR, DE LA RECHERCHE
SCIENTIFIQUE ET DE L'INNOVATION
***************************
UNIVERSITE JOSEPH KI-ZERBO

Unité de Formation et de Recherche en Sciences
de la Santé (UFR/SDS)
************************************
Année académique 2018 -
2019Mémoire n° 213
Comparaison de trois tests de détection rapide
des â-lactamases à Spectre Elargi dans les échantillons
d'hémocultures et d'urines
Présenté le 24 Juillet 2019 pour l'obtention du
Diplôme d'Etude Spécialisée deBiologie
Clinique
Par Hervé KAFANDO
Né le 17 Juin 1986 à
Guéswindé (BURKINA-FASO)
|
Directeur de mémoire : Pr Idrissa SANOU,
Professeur titulaire UFR-SDS/ U-JKZ
|
Jury
Présidente:Pr Rasmata
OUEDRAOGO, Professeur titulaire UFR-SDS/ U-JKZ
|
|
Co-directrice:Pr Béatrice BERÇOT,
PU-PH Université Paris VII, AP-HP
|
Membres :Pr Idrissa
SANOU
DrMahamoudou SANOU, maître de
conférences agrégé UFR-SDS/ U-JKZ
|
LISTE DES RESPONSABLES ADMINISTRATIFS ET DES
ENSEIGNANTS DE L'UFR/SDS ANNEE 2018-2019









DEDICACES
Je dédie ce travail à :
Ø A ma petite famille, mon fils Aaron et ma
femme Sandrine
Vous avez été un grand apport dans la
réalisation de ce travail, vous avez fait preuve de patience durant mon
absence pour le stage. Retrouvez ici, l'expression de mes sentiments les plus
sincères. Que Dieu nous garde toujours unis à jamais.
Ø A mes amis et camarades internes de
l'hôpital Saint Louis de Paris, en particulier Morgane PETIT,
Clément JANOT, Nicolas SALOUM.
Je ne peux trouver les mots justes et sincères pour
vous exprimer mon affection et mes pensées, vous êtes pour moi
tous des amis.
Ø A mes amis et camarades du DES
Ce travail est le couronnement du chemin que nous avons tous
emprunté en 2014, je vous remercie très sincèrement pour
votre parfaite collaboration. Vous êtes pour moi tous des amis.
REMERCIEMENTS
Ø Aux enseignants du D.E.S de biologie
clinique :
Merci pour vos enseignements, vos encouragements et votre
engagement pour la biologie clinique.
Ø A notre maître de stage et co-directrice du
mémoire, Pr Béatrice BERÇOT :
Chère maître, votre simplicité, votre
disponibilité, votre rigueur scientifique et vos soutiens multiples ont
permis la réalisation de ce travail. Merci pour tout et que Dieu vous
bénisse et vous donne une longue vie pleine de succès.
Ø A notre directeur de mémoire,Pr
Idrissa SANOU :
Votre simplicité, votre disponibilité et la
confiance dont vous avez placée en nous, ont facilité
l'élaboration de ce document. Je vous remercie très
sincèrement.
Ø A laprésidente du jury,Pr Rasmata
OUEDRAOGO/TRAORE :
Merci pour votre disponibilité à présider
ce jury.
Ø Aux membres du jury: Pr Idrissa SANOU et Dr
Mahamoudou SANOU:
Merci pour votre disponibilité à lire et
à critiquer ce document.
Ø A tout le personnel du laboratoire de
bactériologie de l'hôpital Saint Louis :
En particulier monsieur François
CAMELENA, merci pour les soutiens multiformes et pour la bonne
collaboration.
Table des matières
DEDICACES
xi
REMERCIEMENTS
xii
Liste des figures
xvi
Liste des tableaux
xvii
Liste des sigles et abréviations
xviii
INTRODUCTION/
ENONCE DU PROBLEME
1
PREMIERE
PARTIE: REVUE BIBLIOGRAPHIQUE
4
Chapitre 1 : Résistance bactérienne
aux antibiotiques
5
1. Mécanismes de résistance
bactérienne aux antibiotiques
5
1.1 Imperméabilité aux antibiotiques
5
1.2 Résistance par efflux
6
1.3 Modification de la cible
6
1.4 Modification des antibiotiques
7
2. â-lactamases des entérobactéries
7
2.1 â-lactamases à sérine
7
2.1.1 Pénicillinases
7
2.1.2 â-lactamases à spectre élargi
7
2.1.3 BLSE de types TEM et SHV
8
2.1.4 BLSE de type CTX-M
8
2.1.5 Carbapénèmases à
sérine
9
2.2 Métallo-â-lactamases
9
2.3 Oxacillinases
9
2.4 Céphalosporinases
10
3. Classification des entérobactéries en
fonction de leur phénotype de résistance
10
3.1 Groupe 0 : phénotype sensible
d'espèces dépourvues de gènes de â-lactamases
10
3.2 Groupe 1 : phénotype
céphalosporinase de bas niveau
11
3.3 Groupe 2 : phénotype
pénicillinase bas niveau
11
3.4 Groupe 3 : Phénotype
céphalosporinase de bas niveau
11
3.5 Groupe 4 : Yersinia enterocolitica et Serratia
fonticola
11
3.6 Groupe 5 : phénotype céfuroximase
12
3.7 Groupe 6 : Phénotype â-lactamase
à spectre étendu chromosomique
12
4. Impact économique et social de la
résistance bactérienne
12
Chapitre 2 : Outils de détection de la
résistance bactérienne
14
1.Antibiogramme
14
2. Outils de détection rapide
14
2.1 Tests immunochromatographiques
14
2.2 Tests chromogéniques
14
2.3 Tests de biologie moléculaire
15
DEUXIEME
PARTIE : NOTRE ETUDE
16
1.Objectif général
17
2.Objectifs spécifiques
17
3.Cadre d'étude
17
4.Type d'étude
17
5.Analyse des échantillons
17
5.1Matériel biologique
17
5.2Prise en charge des hémocultures positives
à Saint Louis (figure 2)
17
5.3Prise en charge des échantillons d'urines
à l'hôpital Saint Louis
18
5.4Test chromogénique de détection de la
résistance aux C3G
19
5.4.1Matériel nécessaire
19
5.4.2 Réalisation du test à partir des
flacons d'hémocultures
19
5.4.3Réalisation du test à partir des
échantillons d'urines
20
5.5Identification des espèces
bactériennes à partir des culots
20
5.5.1Matériel nécessaire
20
5.5.2Réalisation des identifications
21
5.6Test immunochromatographique de détection de
BLSE
21
5.6.1Matériel nécessaire
21
5.6.2Réalisation du test
22
5.7Analyse des échantillons sur l'automate
ePlex® de GenMark
22
5.7.1Matériel nécessaire
22
5.7.2Réalisation du test
24
6.Traitement des données
24
RESULTATS
25
1.Aspects épidémiologiques
26
1.1Total des échantillons analysés
26
1.2Espèces responsables d'ITU et de
bactériémie
26
1. 3 Résistance aux antibiotiques
28
1.3.1 Phénotypes de résistance des
entérobactéries isolées
28
1.3.2 Répartition des BLSE issues des
urocultures en fonction des services
30
2.Performances diagnostiques des tests
31
2.1Identification sur MALDI-TOF-MS à partir des
culots de produits
pathologiques.......................................................................................
31
2.2 Identification des espèces sur l'automate
ePlex® de GenMark
31
2.3 Détection des gènes de
résistance sur ePlex®
32
2.4 Détection des BLSE par les tests
immunochromatographiques
33
2.5 Mise en évidence de la résistance aux
C3G par le test â-LACTATM
33
2.6 Résultats comparés du diagnostic de
la résistance aux C3G par production de BLSE
35
DISCUSSION
38
1.Aspects épidémiologiques
39
1.1Répartition des échantillons
analysés
39
1.2Fréquence des germes responsables de
bactériémie
39
1.3Fréquence des germes responsables d'infection
du tractus urinaire
39
1.4Fréquence des BLSE parmi les
entérobactéries isolées dans les hémocultures
40
1.5Fréquence des entérobactéries
uropathogènes productrice de BLSE
40
2.Performances diagnostiques des tests
41
2.1Performances du MALDI-TOF-MS dans l'identification
d'espèces bactériennes à partir des culots de
centrifugation
41
2.2Performances de l'automate ePlex® pour
l'identification des espèces à partir des produits pathologiques
42
2.3Performances de l'automate ePlex® pour la
détection des gènes de résistance
42
2.4Performances du NG-Test-CTX-M MULTI dans la
détection des BLSE
42
2.5Performances du â-LACTATM test dans
la détection des BLSE
43
3.Avantages et limites des trois tests
étudiés
43
CONCLUSION
45
Recommandations
47
Perspectives
47
Références bibliographiques
48
Résumé
xix
LISTE DES FIGURES
Figure 1: Représentation schématique des
mécanismes de résistance aux antibiotiques..................
2
Figure 2: procédure de prise en charge des
hémocultures positives à Saint Louis
18
Figure 3: Processus d'identification sur MALDI-TOF SM .
19
Figure 4: Matériel nécessaire à la
réalisation du â-LACTATM Test
19
Figure 5: Matériel nécessaire à
l'identification des germes au spectromètre de masse
21
Figure 6: Matériel pour la réalisation du
test immunochromatographique NG-Test-CTX-M
22
Figure 7: Matériel nécessaire pour
l'analyse des échantillons sur ePlex®
23
Figure 8: Composition du panel BCID-GN de ePlex®
23
Figure 9: Répartition des phénotypes des
entérobactéries isolées dans les hémocultures
28
Figure 10: Répartition des phénotypes des
entérobactéries isolées dans les urines
29
Figure 11: Répartition des BLSE
uropathogènes selon les services
30
Figure 12: Comparaison des performances diagnostiques
des techniques de détection de la résistance aux C3G par
production de BLSE dans les urines.
36
Figure 13: Comparaison des performances diagnostiques
des techniques de détection de la résistance aux C3G par
production de BLSE dans les hémocultures
37
LISTE DES TABLEAUX
Tableau I: Récapitulatif des espèces
identifiées, ePlex® versus MALDI-TOF-MS à partir des urines
2
Tableau II: Récapitulatif des espèces
identifiées ePlex® versus MALDI-TOF-MS à partir des
hémocultures positives
27
Tableau III: Résultats de l'identification sur
MALDI-TOF-MS à partir des culots de centrifugation
31
Tableau IV: Performance diagnostiques de ePlex®
dans la détection des gènes de résistance
32
Tableau V: Résultats du NG Test CTX-M MULTI dans
la détection des BLSE
33
Tableau VI: Résultats du test â-LACTATM
pour la détection de la résistance aux C3G par production de BLSE
34
LISTE DES SIGLES ET
ABREVIATIONS
ATB : antibiotique
BGN : bacille à Gram
négatif
BLSE : â-lactamase à
spectre élargi
BLT : â-LACTATM
Test
C1G : céphalosporine de
première génération
G : céphalosporine de
deuxième génération
C3G : céphalosporine de
troisième génération
C4G : céphalosporine de
quatrième génération
CA-SFM : comité d'antibiogramme
de la société française de microbiologie
CTX-M : céphotaximase Munich
GES : guyana extended spectrum
HCASE : hyperproduction de
céphalosporinase
IMI : imipenem-hydrolysing enzyme
ITU : infection du tractus urinaire
KPC : Klebsiella
pneumoniaecarbapénémase
MALDI-TOF :Matrix Assisted Laser-
Desorption Ionisation-Time of Flight
MBLs : métallo
â-lactamases
PBN : pénicillinase de bas
niveau
PHN : pénicillinase de haut
niveau
PS : Phénotype sauvage
PTHN : pénicillinase de
très haut niveau
SARM : Staphylococcus aureus
résistant à la méticilline
SME : Serratia marcescens
enzyme
INTRODUCTION/ ENONCE DU
PROBLEME
Les bacilles à Gram négatif (BGN) en particulier
les entérobactéries sont de loin les germes responsables
d'infections du tractus urinaire (ITU) (Sarkiset al.,2017). Dans les
bactériémies à BGN les entérobactéries
occupent une place prépondérante(Melzeret
al.,2007 ; Peraltaet al 2012). Cependant, ces
bactéries deviennent de plus en plus une menace mondiale du fait de leur
multirésistance aux antibiotiques, essentiellement due à
l'acquisition de â-lactamases à spectre élargi (BLSE).
L'émergence et la dissémination des BLSE de type CTX-M à
l'échelle mondiale en sont la principale cause (Cantónet
al.,2016 ;Boutalet al.,2017). Ces enzymes confèrent
aux entérobactéries unerésistance à l'ensemble des
â-lactamines à l'exceptiondes céphamycines et des
carbapénèmes (Netgenet al.,2009).
Longtemps considérée comme un
phénomène hospitalier, la résistance par production de
BLSE est actuellement en plein essor en milieu communautaire. En effet, le
portage communautaire de BLSE est estimé à 70 %, 35 %
et 15 % respectivement en Asie, dans l'Est du Bassin
Méditerranéen et en Afrique (Woertheret al.,2013). Au
Burkina Faso, la prévalence du portage de BLSE était
estimée à 22% parmi des volontaires sains et 42% parmi les
hospitalisés (Ouedraogoet al., 2017)Les facteurs de risques
associés au portage de BLSE sont principalement les conditions
d'hygiène précaires et une consommation non négligeable
d'antibiotiques de qualité suboptimale (Woertheret al., 2013).
Une méta-analyse récente estimait que la prévalence
mondiale de BLSE se situait autour de 14 % et augmenterait en moyenne de
5,38 % par an (Karanikaet al.,2016)--.
Les bactériémiesà BLSE sont très
souvent associées à un mauvais pronostic comparativement aux
mêmes germes sensibles. Ce risque étant essentiellement dû
à l'instauration d'antibiothérapie probabiliste inadéquate
(Melzeret al.,2007 ; Schwaberet al., 2007).En pratique,
les méthodes traditionnelles de diagnostic bactériologique
mettent 48 à 72 heures pour l'obtention d'un antibiogramme et 20
à 30% des patients reçoivent une antibiothérapie initiale
inadéquate avec une augmentation du taux de mortalité
estimé à 7,6% pour chaque heure d'antibiothérapie efficace
retardée (Kumar A et al.,2006).
La résistance des entérobactéries aux
céphalosporines de troisièmegénération (C3G)qui
sont les molécules les plus couramment prescrites, pose
unproblème de choix des antibiotiques au coursdes traitements
probabilistesdes infections invasives. L'élaboration de techniques de
diagnostic rapide s'avère donc indispensable pour guider au mieux le
choix des antibiothérapies probabilistes.Plusieurs outils existent
actuellement surle marché mais très peu permettent à
partir des produits pathologiques d'identifier à la fois le
pathogène et les gènes de résistances. L'appareil
ePlex® de GenMark propose un panel d'identification de bacilles à
Gram négatif qui sont les plus souvent responsables d'ITU et de
bactériémies. Il permet en outre, l'identification des
gènes les plus couramment impliqués dans les
multirésistances (CTX-M, IMP, KPC, OXA, VIM, NDM).
L'objectif de ce travail était d'évaluer les
performances diagnostiques d'un test chromogénique, d'un test
immunochromatographique et d'un test de biologie moléculaire pour la
mise en évidence de la résistance bactérienne.
o PREMIERE PARTIE: REVUE
BIBLIOGRAPHIQUE
Chapitre 1 :
Résistance bactérienne aux antibiotiques
1. Mécanismes de
résistance bactérienne aux antibiotiques
Les principaux mécanismes de résistance
bactérienne aux antibiotiques sont :
l'imperméabilité, la résistance par efflux, la
modification de la cible et la modification des antibiotiques(figure 1).
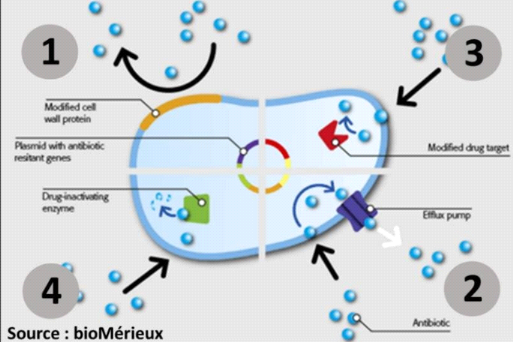
Figure 1: Représentation
schématique des mécanismes de résistance aux antibiotiques
(Boutaletal.,2017)
1.1 Imperméabilité
aux antibiotiques
Bon nombre d'antibiotiques ont des cibles intracellulaires, ce
qui implique nécessairement la pénétration à
travers la membrane externe et/ou la membrane cytoplasmique pour avoir un effet
antimicrobien. La capacité des bactéries à
développer des mécanismes pour bloquer la diffusion des
antibiotiques à travers leurs membranes est
l'imperméabilité. C'est un phénomène très
répandu chez les bactéries à Gram négatif pour
lesquelles cette diffusion est médiée par des porines notamment
pour des antibiotiques comme les tétracyclines, les fluoroquinolones ou
les â-lactamines (Pagèset al.,2008). La résistance
médiée par les porines peut résulter de plusieurs
phénomènes comme le changement de type, de
spécificité ou de niveau d'expression de la porine.
1.2 Résistance par
efflux
Les pompes à efflux ont pour rôle
principall'expulsion des déchets issus du métabolisme
bactérien. Elles peuvent également être impliquées
dans les phénomènes de résistance aux antibiotiques en
limitant l'accumulation de la molécule au contact de sa cible. Une
surexpression de ces pompes a un effet délétère sur
l'efficacité des antibiotiques (Yasufukuet al.,2011). Ces
pompes peuvent être spécifiques d'une ou plusieurs familles
d'antibiotiques (Poole K et al., 2005).
1.3 Modification de la cible
Plusieurs phénomènes peuvent être à
l'origine de la modification des cibles d'action des antibiotiques avec pour
conséquence une perte ou une baisse de l'affinité des
antibiotiques pour celles-ci.
o Une modification de la cible suite à une mutation
dans le gène correspondant : c'est le cas de la mutation pour les
gènes de la gyrase ou de la topoisomérase IV qui confère
la résistance aux fluoroquinolones(Hooperet al.,
2002) ;
o Une modification enzymatique de la cible: méthylation
du ribosome générant une résistance aux
macrolides(Leclercq et al., 2002) ;
o Une modification par le remplacement de la cible :
acquisition par S. aureus d'une PLP exogène (PLP2a)
portée par le gène mecA à l'origine de la
résistance à la méticilline. Cette PLP a une faible
affinité pour les â-lactamines rendant tous les membres de cette
famille inefficace contre Staphylococcus aureus résistant
à la méticilline (SARM) (Hiramatsuet al.,2013);
o Une protection de la cible : l'exemple type est la
résistance à la tétracyclineau cours de laquelle la
bactérie résistante produit des protéines (TetO et TetM)
homologues aux facteurs d'élongation impliqués dans la
synthèse protéique. Ces protéines se fixent au ribosome et
délogent la tétracycline tout en modifiant la conformation du
ribosome empêchant l'antibiotique de se refixer (Donhoferet
al.,2012). Une autre protéine qnrconfère
également la résistance aux quinolones en agissant comme un
homologue de l'ADN qui se fixe sur les sites de liaison à l'ADN de la
gyrase et de la topoisomérase IV. Le nombre de complexes ADN/gyrase est
ainsi limité ce qui diminue le nombre de complexes gyrase/ADN
clivé/quinolone délétère pour la bactérie
(Strahilevitzet al.,2009).
1.4 Modification des
antibiotiques
C'est un mécanisme enzymatique au cours duquel,
l'antibiotique est soit dégradé par hydrolyse (c'est le cas des
â-lactamases qui hydrolysent les â-lactamines), soit par
modification structurale des antibiotiques par méthylation,
phosphorylation ou adénylation.
2.â-lactamases des
entérobactéries
Le mécanisme de résistance le plus
partagé et le plus fréquent dans la famille des
entérobactéries est la production de â-lactamases
(Netgenet al., 2009). En effet, la production de â-lactamases
chez les entérobactéries a été décrite bien
longtemps. Toutefois la pression de sélection induite par l'utilisation
massive des antibiotiques a favorisé l'émergence de souches
multirésistantes (Drawzet al.,2010).
On distingue principalement deux types de classification des
â-lactamases, celle de Bush-Jacoby-Medeiros basée sur leurs
substrats et leurs inhibiteurs, et celle d'Ambler basée sur leur
homologie de séquence en acides aminés (Drawzet al.,
2010).La classification de Ambler sera utilisée dans la suite de ce
travail.
2.1 â-lactamases à
sérine
Ces enzymes appartiennent à la classe A de Ambler.
Toutes les â-lactamases à sérine ont un mode d'action
dépendant de l'acylation des â-lactamines, aboutissant à
l'hydrolyse du cycle â-lactame et àla perte d'activité de
l'antibiotique. L'enzyme est ensuite régénérée et
de nouveau active sur d'autres molécules d'antibiotiques (Drawzet
al., 2010).
2.1.1 Pénicillinases
Ce sont des enzymesde type plasmidique ou chromosomique,
généralement sensibles aux inhibiteurs (acide clavulanique,
tazobactam), leur spectre d'hydrolyse concerne les pénicillines et les
céphalosporines de première génération (C1G). Les
enzymes de type TEM et SHV sont courantes et souvent rencontrées en
clinique chez des souches de E. coli etdeK.
pneumoniaeresponsables d'ITU (Drawzet al., 2010).
2.1.2 â-lactamases
à spectre élargi
Les BLSE sont une grande famille
hétérogène d'enzymes bactériennes
découvertes dans les années 80 en France. Elles sont induites
soit par des plasmides, soit par des mutations du génome naturel. Les
deux mécanismes confèrent aux bactéries qui les
possèdent, la capacité d'hydrolyser une grande
variété de pénicillines et de céphalosporines.
Avant l'expansion des CTX-M, la majorité des BLSE était le
résultat de mutations génétiques de â-lactamases
naturelles, en particulier de TEM-1, TEM-2 et SHV-1(Netgenet al.,
2009).
Les â-lactamases naturelles sont très actives
contre les pénicillines et moyennement actives contre les C1G. Les
mutations génétiques à l'origine des BLSE
élargissent le spectre de ces enzymes et touchent également les
céphalosporines de troisième génération et les
monobactames. Les bactéries produisant une BLSE n'hydrolysent pas les
céphamycines ni les carbapénèmes et elles sont
inhibées par les inhibiteurs classiques de â-lactamases (acide
clavulanique, tazobactam et sulbactam)(Netgenetal.,2009). La
présence de BLSE est fréquemment associée à la
résistance aux fluoroquinolones.
2.1.3BLSE de types TEM et
SHV
La première â-lactamase plasmidique (SHV2)
hydrolysant les C3G fut isolée d'une souche de Klebsiella
ozaenae en Allemagne en 1983. Cette â-lactamase à spectre
élargi était dérivée de la
â-lactamaseà spectre étroit SHV1 par simple mutation qui
lui conférait un spectre d'hydrolyse assez large (Kliebeet
al.,1985). En 1984 en France, une autre â-lactamase plasmidique
dérivée de la â-lactamase à spectre étroit
TEM-1 hydrolysant le céfotaxime fut isolée de
Klebsiellapneumoniae(Sirotet al.,1987).Ces BLSE,
dérivées des â-lactamases à spectre étroit
conservaient la propriété d'être inhibées par les
inhibiteurs de â-lactamase comme l'acide clavulanique, le tazobactam et
le sulbactam.
2.1.4BLSE de type CTX-M
Les CTX-M dérivent des â-lactamases
chromosomiques d'espèces du genre Kluyvera
(entérobactéries non pathogènes). Ces â-lactamases
tiennent leur nom de par leur hydrolyse préférentielle du
céfotaxime par rapport à la ceftazidime « CTX » et
« M » pour leur lieu d'isolement (Munich). Elles ont
été isolées en Allemagne par Bauernfeind et al.
(1990) à partir d'une souche de E. coli résistante
au céfotaxime.
Les CTX-M constituent actuellement la majorité des BLSE
isolées dans le monde. En effet, elles ont été
retrouvées sur tous les continents à des fréquences
variables en fonction du temps du pays et du type de patients recrutés.
Elles sont également retrouvées en milieu communautaire
qu'à l'hôpital (Ruppéetal.,2010). Comme la
majorité des BLSE de classe A, les CTX-M confèrent une
résistance à toutes les â-lactamines hormis les
céphamycines et les carbapénèmes. La plupart des CTX-M
confère un haut niveau de résistance au céfotaxime et
à la ceftriaxone. Le niveau de résistance vis-à-vis des
céphalosporines de quatrième génération est
variable(Ruppéetal., 2010).
De nos jours, il existe plus de 90 CTX-M répartis dans
5 groupes(Ruppéetal., 2010) que sont :
-le groupe CTX-M-1 comprend les CTX-M-1, CTX-M-3, CTX-M-10 et
CTX-M-15 ;
-le groupe CTX-M-2 comprend les CTX-M-2, CTX-M-4, CTX-M-5,
CTX-M-6, CTX-M-7 et TOHO-1 ;
-le groupe CTX-M-8 comporte les CTX-M-8 et CTX-M-40 ;
-le groupe CTX-M-9 comporte les CTX-M-9, CTX-M-13, CTX-M-14,
CTX-M-17, CTX-M-19, CTX-M-21, CTX-M-27 et TOHO-2 ;
-le groupe CTX-M-25 comporte les CTX-M-25, CTX-M-26, CTX-M-41
et CTX-M-55.
2.1.5Carbapénèmases à sérine
Les carbapénèmases à sérines
regroupent des enzymes des familles NMC (not-metallo-enzyme carbapenemase), IMI
(imipenem-hydrolysing enzyme), SME (Serratia marcescens enzyme) dont
les gènes sont portés par le chromosome, et GES (Guyana
extendedspectrum) et KPC (Klebsiella pneumoniae carbapenemase) dont
les gènes sont portés par un plasmide. Ces enzymes ont un spectre
d'hydrolyse très large et touchent la quasi-totalité des
â-lactamines (pénicillines,céphalosporines, monobactames et
carbapénèmes)(Queenanetal., 2007). KPC est la
carbapénèmase la plus répandue de ce groupe.Elle hydrolyse
plus efficacement les C3G et est faiblement inhibée par les inhibiteurs
de â-lactamases (Papp-Wallaceetal.,2010).
2.2
Métallo-â-lactamases
Les Métallo-â-lactamases (MBLs) appartiennent
à la classe B de Ambler. Elles se caractérisent par un large
spectre d'hydrolyse vis-à-vis des â-lactamines et restent
insensibles aux inhibiteurs. A l'exception des monobactames, elles sont
capables d'hydrolyser toutes les classes de â-lactamines. Les
métallo-â-lactamases utilisentun ion (zinc
généralement) comme cofacteur lors de l'attaque
nucléophile du noyau â-lactame d'où leur nom
(Munitaetal.,2016). L'éthylène diamine tétra
acétique (EDTA), ayant un pouvoir chélateur inhibe l'action des
MBLs in vitro. IMP, VIM et NDM sont les MBLs les plus souvent
rencontrées chez les entérobactéries
(Munitaetal.,2016 ; Boutaletal., 2017).
2.3 Oxacillinases
Les oxacillinases appartiennent à la classe D de
Ambler. Elles confèrent plusieurs phénotypes de résistance
en fonction du type. Elles ont une activité pénicillinase mais
sont capables d'hydrolyser l'oxacilline, d'où leur nom. L'OXA 48 a une
activité carbapénèmase. Elle est uniquement
présente chez les entérobactéries. En France, 75 % des
déclarations d'entérobactéries productrices de
carbapénèmases (EPC) correspondent à des souches
productrices d'OXA-48 (Dortetet al., 2013).
2.4Céphalosporinases
Les â-lactamases de la classe C de Ambler sont des
céphalosporinases, ces enzymes sont insensibles aux inhibiteurs des
â-lactamases. Elles sont connues sous le nom AmpC, et leur site actif
contient une sérine (Harriset al.,2015). On distingue :
- les céphalosporinases chromosomiques non inductibles
chez les entérobactéries du groupe 1 (E. coli, Shigella
spp) : très peu ou pas exprimées, leur spectre d'hydrolyse
se limite aux pénicillines et aux C1G ;
- les céphalosporinases chromosomiques inductibles chez
les entérobactéries du groupe 3 (Enterobacter sp, Citobacter
freundii, Serratia marcescens, Morganella morganii, Hafnia alvei, Providencia
stuartii).Elles peuvent être hyper-produites suite à une
mutation du gène de régulation : on parle alors de
céphalosporinases déréprimées. Elles ont un spectre
d'hydrolyse très large, en plus des pénicillines et des C1G,
elles hydrolysent les G, C3G voire les C4G ;
- les céphalosporinases plasmidiques (AmpC) :
présentes sur des éléments mobiles
transférables,leur site actif contient une sérine. Ce sont des
céphalosporinases à large spectre. Elles sont également
actives sur l'aztréonam et insensibles aux inhibiteurs de
â-lactamases.
3. Classification des
entérobactéries en fonction de leur phénotype de
résistance
Les entérobactéries produisent naturellement
diverses â-lactamases ce qui permet de les classer en groupes
phénotypiques de résistance (Bonnet et al., 2012).
3.1 Groupe 0 :
phénotype sensible d'espèces dépourvues de gènes de
â-lactamases
Le groupe 0 est constitué de phénotype sensible
d'espèces dépourvues de gènes de â-lactamases.
Salmonella sp et Proteus mirabilis sont dépourvus de
â-lactamase à l'état sauvage et sont naturellement
sensibles aux aminopénicillines, carboxypénicillines,
uréïdopénicillines, à l'aztréonam, aux
céphalosporines et aux carbapénèmes. Les diamètres
d'inhibition de l'imipénème sont souvent réduits pour
P. mirabilis comme pour les autres Proteae(Bonnet et
al., 2012). La faible affinité de cette molécule
pour les PLP2 de ces espèces serait responsable de cette diminution.
Toutefois, elle est sans conséquence clinique contrairement à la
résistance acquise par mutation de PLP2.
La fréquence du phénotype sauvage chez
Proteus mirabilis est d'environ 50% en milieu hospitalier ; chez
Salmonella, elle est d'environ 90% avec une grande disparité
selon les sérogroupes (Chanalet al., 2000).
3.2 Groupe 1 :
phénotype céphalosporinase de bas niveau
Il est constitué de phénotype sensible
d'espèces produisant naturellement une céphalosporinase de classe
C. E. coli et Shigella spp sont naturellement sensibles aux
aminopénicillines, carboxypénicillines,
uréïdopénicillines, à l'aztréonam, aux
céphalosporines et aux carbapénèmes. Cependant, elles
produisent à très bas niveau une céphalosporinase
chromosomique non inductible de type AmpC qui peut entrainer chez certaines
souches une réduction de la sensibilité aux
aminopénicillines, à leurs associations au clavulanate et/ou aux
C1G. La fréquence du phénotype sauvage chez E. coli est
d'environ 50% en milieu hospitalier (Chanalet al., 2000).
3.3 Groupe 2 :
phénotype pénicillinase bas niveau
Klebsiella pneumoniae, Klebsiella oxytoca,
Citrobacterkoseri, Citrobacteramalonaticus et Escherichia hermanni
produisent naturellement et de façon constitutive des enzymes
chromosomiques de classe A sensibles aux inhibiteurs (SHV-1 ou LEN-1 pour
K. pneumoniae, OXY pour K. oxytoca, CKO pour C.
koseri, CdiA pour C. amalonaticuset HER-1 pour E.
hermanni). Elles confèrent une résistance patente aux
aminopénicillines et aux carboxypénicillines et souvent
inapparente aux uréïdopénicillines. Les associations
pénicillines-inhibiteurs sont actives (Bonnet et al., 2012).
Environ 70% des souches de K. pneumoniae, 75% des K. oxytoca
et C. koseri sont de phénotypes sauvages ou sensibles.
3.4 Groupe 3 :
Phénotype céphalosporinase de bas niveau
Les entérobactéries appartenant à ce
groupe réunissent des espèces productrices de
céphalosporinases de classe C (AmpC) chromosomiques et inductibles par
les â-lactamines. Ces céphalosporinases sont très
répandues chez Enterobacter cloacae, Enterobacter aerogenes,
Serratia marcescens, Citrobacter freundii, Morganella morganii, Hafnia alvei,
Providencia stuartii, Providencia rettgeri et Pantoeaagglomerans. Le
phénotype sauvage de ces espèces comprend une résistance
aux aminopénicillines, à leurs associations aux inhibiteurs des
â-lactamases et aux C1G (Bonnet et al., 2012).
La fréquence du phénotype sauvage est variable
selon l'espèce et la situation épidémiologique du lieu
considéré.
3.5 Groupe 4 : Yersinia
enterocolitica et Serratia fonticola
Yersinia enterocolitica et Serratia
fonticola produisent naturellement une céphalosporinase inductible
de classse C et une enzyme de classe A. Chez Yersinia enterocolitica,
cette dernière est une pénicillinase constitutive de classe
A tandis que chez Serratia fonticolal'enzyme de classe A est une
â-lactamase inductible (Bonnet et al., 2012).
Yersinia enterocolitica est résistante aux
aminopénicillines, à leurs associations avec l'acide
clavulanique, aux carboxypénicillines et aux C1G. La résistance
aux uréïdopénicillines n'apparait pas in vitro. Le
phénotype de résistance de Serratia fonticolaest
similaire. Cependant le céfuroxime n'est pas actif et la
résistance à l'association aminopénicillines-inhibiteurs
de â-lactamases normalement induite par l'AmpC n'est pas toujours
exprimée in vitro (Bonnet et al., 2012).
3.6 Groupe 5 : phénotype
céfuroximase
Proteus vulgaris et Proteus penneri
produisent naturellement une céphalosporinase de classe A inductible par
les â-lactamines appelée céfuroximase. Le phénotype
se caractérise par une résistance aux aminopénicillines,
aux C1G, aux G (céfuroxime, céfamadole) à l'exception des
céphamycines (céfoxitine) et une sensibilité aux
associations pénicillines-inhibiteurs de â-lactamases (Bonnet
etal., 2012).
3.7 Groupe 6 :
Phénotype â-lactamase à spectre étendu
chromosomique
Kluyvera ascorbata, Kluyvera cryocrescens, Kluyvera
georgiana, Rahnellaaquatilis, Citrobactersedlakii et Erwinia persicina
produisent naturellement des â-lactamases à spectre élargi
de classe A. Ces BLSE souvent exprimées à bas niveau
confèrent une résistance aux aminopénicillines, aux
carboxypénicillines, aux C1G et G. La résistance aux
uréïdopénicillines et aux C3G est souvent inapparente
(Bonnet etal., 2012).
4. Impact économique et
social de la résistance bactérienne
L'émergence et la dissémination des
bactéries multirésistantes (BMR) ont réduit
considérablement les possibilités thérapeutiques des
patients infectés par des BMR. Les antibiotiques de derniers recours,
notamment les carbapénèmes qui sont utilisés dans les
infections causées par des entérobactéries
multirésistantes connaissent aujourd'hui des limites du fait de
l'émergence et de la dissémination des
carbapénèmases à travers le monde. Cette impasse
thérapeutique à caractère mondial est à l'origine
de morbi-mortalité importante. En effet, Peraltaetal. (2012)
ont rapporté dans une étude multicentrique que les traitements
probabilistes étaient inadéquats dans près de 50% des cas
de bactériémie à BLSE avec un risque de mortalité
dans les 72 heures suivant le choc septique estimé à plus de 20%.
Dans les pays de l'Union Européenne, des Etats Unis et
du Thaïlande, le nombre de décès annuel lié au BMR
est estimé à plus de 23 000, 25000 et 38 000 avec un
coût global direct pouvant atteindre 20 milliards de dollars
(Kardaoe-Somaet al., 2017).
L'impact économique de la résistance
bactérienne n'est plus à démonter, il est tributaire des
coûts liés à la durée d'hospitalisation, des frais
inhérents aux médicaments ainsi que du coût de la
prévention. En effet, on estime à plus de 24 millions de
personnes dans le monde qui seraient réduites dans l'extrême
pauvreté d'ici 2030 à cause des BMR et le coût minimal
annuel pour freiner l'émergence et l'extension des BMR estimé
à 9 milliards de dollars (
https://genmarkdx.com/).
Selon les estimations de la banque mondiale, la
résistance bactérienne pourrait d'ici 2050 avoir des effets
dévastateurs sur l'économie mondiale d'une ampleur comparable
à ceux de la crise financière de 2008. Cette étude montre
également que la résistance microbienne pourrait accroître
la pauvreté en touchant surtout les pays démunis avec plus de 5%
du PIB et précipiter jusqu'à 28 millions de personnes dans la
pauvreté.
Chapitre 2 :Outils de
détection de la résistance bactérienne
1. Antibiogramme
C'est la méthode de référence pour la
détermination des phénotypes de résistance des
bactéries après isolement. Il peut se faire sur milieu
gélosé comme en milieu liquide (automatisé).
On distingue principalement 2 types sur milieu solide: la
diffusion en milieu gélosé et la méthode de gradient en
gélose (Etest).
La méthode en diffusion gélosée reste
largement utilisée et est basée sur la formation d'un gradient de
concentration en gélose de l'antibiotique (ATB) par diffusion radiale
à partir d'un disque imprégné de cet ATB et placé
sur gélose. Certains ATB diffusent mal en milieu gélosé et
le CA-SFMdéconseille la technique pour ces antibiotiques comme la
colistine, et la vancomycine (CA-SFM 2018).La méthode en diffusion
gélosée est applicable à la majorité des
bactéries. Il existe actuellement des systèmes de lecture
automatisée.
La méthode en gradient de concentration sur
gélose se fonde sur l'utilisation d'une bandelette au verso de laquelle
des spots en quantité croissante de l'ATB étudié sont
placés.Lorsque la bandelette est placée sur une gélose
préalablement ensemencée, un gradient continu d'ATB croissant du
bas vers le haut se crée. Après incubation, une zone d'inhibition
en forme d'ellipse est apparente permettant la lecture de la CMI à
l'intersection basse avec la bandelette. C'est une méthode
évaluée pour de nombreux ATB et largement utilisée au
laboratoire surtout quand la détermination de CMI est recommandée
(CA-SFM 2018).
2. Outils de détection
rapide
2.1 Tests
immunochromatographiques
Ce sont des tests qualitatifs rapides basés sur les
réactions antigènes-anticorps. On peut citer entre autres le test
Coris qui permet la détection des carbapénèmases (OXA
48/48 like, NDM, KPC et VIM) à partir des colonies, ainsi que le NG-Test
CTX-M MULTI pour la détection des BLSE type CTX-M qui sont les plus
répandues parmi les BLSE. Ce test permet la mise en évidence des
CTX-M des groupes 1, 2, 8, 9,25. La détection de la résistance
à la méticilline chez Staphylococcus aureus peut se
faire également par des tests immunochromatographiques qui mettent la
présence des PLP2a.
2.2 Tests
chromogéniques
Les tests chromogéniques les mieux connus sont des
tests complémentaires de l'antibiogramme pour rendre un résultat
fiable.Le plus utilisé est le test à la nitrocéfine, une
céphalosporine chromogène. La détection de production de
â-lactamase par un test chromogénique est recommandée par
le CA-SFM dès l'isolement de certaines bactéries comme
Haemophilus spp, Neisseria gonorrhoeae et Moraxella catarrhalis
(CA-SFM 2018).
Le â-LACTATM Test, basé sur
l'hydrolyse d'une céphalosporine chromogène (HMRZ-86) en
présence d'une â-lactamase. Ce testinitialement conçu pour
la détection de production de â-lactamasesur coloniea vu ses
applications dérivées à des produits pathologiques
notamment les hémoculturespositives et les culots urinaires.
La céphalosporine chromogène HMRZ-86 n'est pas
hydrolysée par lesâ-lactamases types SHV1 et TEM1, elle est
cependant sensible aux BLSE, carbapénèmases (KPC et MBLs) et aux
céphalosporinases acquises.
2.3 Tests de biologie
moléculaire
Les tests moléculaires de mise en évidence des
gènes de résistances sont très peu utilisés en
routine en raison de leur coût. Toutefois quelques-uns sont
utilisés notamment dans des situations d'urgences pouraider à la
décision d'antibiothérapie. Dans le cadre des PCR simplex, on
peut citer le CEPHEIDen technologie gèneExpert pour l'identification de
Staphylococcus aureus et son gène mecA dans les
bactériémies à staphylocoques. Des cassettes CARBA sont
également utilisées pour la mise en évidence des
gènes qui gouvernent l'expression des carbapénèmases.
Dans les PCR multiplex, outre le filmaray® qui a
déjà fait ses preuves et actuellement utilisé dans
certains laboratoires,l'appareil ePlex® de GenMark propose un panel
d'identification de BGN ainsi que les gènes de résistances
associés. En expérimentation sur les produits pathologiques, il
pourrait être l'un des outils de détection de la résistance
bactérienne dans un futur proche dans les laboratoires de routine.
DEUXIEME PARTIE : NOTRE
ETUDE
1.
Objectif général
Evaluer les performances
diagnostiques des 3 testspour la détection des BLSE à partir des
urines et des flacons d'hémocultures positives à BGN.
2.
Objectifs spécifiques
- Déterminer la sensibilité de l'automate
ePlex® pour l'identification des espèces et la détection des
gènes de résistance ;
- Evaluer les performances des cassettes
immunochromatographiques pour la détection des BLSE à partir des
échantillons d'urines et d'hémocultures ;
- Déterminer la sensibilité et la
spécificité du â-LACTATM test dans la mise en
évidence de la résistance aux C3G par production de BLSE.
3. Cadre d'étude
Le service de bactériologie de l'hôpital Saint
Louis a servi de cadre d'étude. L'hôpital Saint Louis est
situé au dixième arrondissement de la ville de Paris et constitue
avec l'hôpital Lariboisière et Fernand Vidal le Groupe Hospitalier
Universitaire Saint Louis-Lariboisière-Fernand Vidal, rattaché
à l'université Paris VII.
4. Type d'étude
Il s'agit d'une étude prospective qui s'est
déroulée du03 Mai au 28 Septembre 2018.
5. Analyse des échantillons
5.1 Matériel biologique
Les échantillons d'urines collectés dans des
tubes monovettesavec comme critères d'inclusion une leucocyturie =
100/mm3et la présence de BGN (>1 bacille par champs) ainsi
que les flacons d'hémocultures positives avec la présence de BGN
à l'examen microscopique ont servi à la réalisation de ce
travail.
5.2 Prise en charge
des hémocultures positives à Saint Louis (figure 2)
Les flacons d'hémocultures positives subissent les
étapes de l'examen cytobactériologique suivantes :
J0 : sont réalisés au premier jour de la
positivité d'un flacon d'hémoculture, l'examen microscopique
après coloration de Gram, l'ensemencement sur milieu
gélosé, l'identification au MALDI-TOF-MS et la réalisation
de l'antibiogramme à partir de la suspension d'hémoculture si
présence de BGN à l'examen microscopique.
J1 : rendu des antibiogrammes réalisés en
milieu solide et transmission de souches isolées (cocci à Gram
positif) pour l'antibiogramme en milieu liquide.
J2 : rendu des antibiogrammes des cocci à Gram
positif et conservation des souches.
30 min
24 h
24 h



10-15 min min
24 h
Equipe mobile
Figure 2: procédure de
prise en charge des hémocultures positives à Saint Louis
5.3 Prise en charge
des échantillons d'urines à l'hôpital Saint Louis
A l'hôpital Saint Louis, l'examen
cytobactériologique des urines est réalisé suivant les 3
étapes classiques que sont : l'examen direct, la culture et
l'antibiogramme.
Examen direct : la numération
des éléments cellulaires (hématies et leucocytes), ainsi
que la détection de bactéries dans l'urinesont
réalisées sur l'automate UF500i de bioMérieux.La
coloration de Gram n'est réalisée que sur les urines dans
lesquelles l'automate a détecté la présence de
bactéries.
Culture et identification : les
urines sont ensemencées sur milieu chromogène (gélose
URI4) et incubées à 37°C pendant 18 à 24h. Les
bactéries obtenues en culture sont identifiées par
spectrométrie de masse (VITEK2 SM de bioMérieux) comme suit:
Au moyen d'öses, les colonies bactériennes ont
été prélevées puis étalées sur les
cibles de la lame pour spectrométrie. La lame est ensuite mise à
sécher à l'étuve. Enfin, 1 ul de matrice a
été ajouté sur chaque dépôt et le tout a
été mis à sécher avant de passer la lame au
MALDI-TOF-MS.
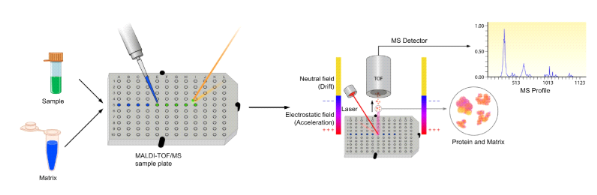
Figure 3: Processus
d'identification sur MALDI-TOF SM (Clark et
al.,2013).
Antibiogramme :l'antibiogramme en
milieu liquide sur VITEK2 a été le plus couramment
réalisé à l'hôpital Saint Louis. La méthode
par diffusion n'est effectuée que pour les cas nécessitant des
tests complémentaires afin d'étayer au mieux les
phénotypes de résistance.L'ADAGIO a été l'automate
de lecture utilisé pour l'antibiogramme en milieu solide.
5.4 Test
chromogénique de détection de la résistance aux C3G
5.4.1 Matériel nécessaire
La réalisation des tests chromogéniques a
nécessité l'utilisation d'une micro centrifugeuse pour
l'obtention des culots dans des tubes Eppendorf ainsi que des réactifs
R1 et R2 du â-LACTATM Test lot
numéro 64160447 pour la mise en évidence de la production de
â-lactamases. Le matériel utilisé pour la
réalisation du â-LACTATM Test est
représenté dans la figure 4.
A
B
C

Figure 4: Matériel
nécessaire à la réalisation du â-LACTATM
Test
A= réactifs R1 et R2, B= portoir
+ tubes Eppendorf, C= microcentrifugeuse
5.4.2 Réalisation du test
à partir des flacons d'hémocultures
Dans un tube Eppendorf, 1 ml de suspension de sang a
été transféré au moyen d'une pipette,50 ul de
triton 10% ont été ajouté puis laisser reposer pendant 10
minutes et passer au vortex pendant 5 secondes. Le mélange est ensuite
centrifugé à 13000 tours par minutes pendant 2 minutes.
Après élimination du surnageant, le culot est repris en
suspension dans 500 ul d'eau distillée par pipetage aller/retour.
Après une seconde centrifugation à 13000 tours par minutes
pendant 2 minutes, le surnageant est éliminé à
nouveau. Enfin, une goutte de réactifs R1 et une goutte de
réactifs R2 de â-LACTATMpréalablement sorti
à température du laboratoire sont ajoutées au culot et
remise en suspension. La lecture de la coloration du mélange à
T=15 minutes a été réalisée.
- coloration jaune : négatif
- coloration orangé : ininterprétable
-coloration rouge : Positif
5.4.3 Réalisation du test à partir des
échantillons d'urines
Les urines positives à BGN répondant aux
critères d'inclusions sus cités ont fait l'objet de test de mise
en évidence de bactéries résistantes aux
céphalosporines de 3ème génération par
production de béta-lactamases suivant le protocole ci-dessous.
Au total, 1,5 ml d'urine ont été
transférés à l'aide d'une pipette dans un tube Eppendorf,
puis centrifugés à 3000 tours par minute pendant 5 minutes.
Après élimination du surnageant, une goutte du réactif R1
et une goutte du réactif R2 du â-LACTATM
préalablement sortis à température du laboratoire
pendant 10 minutes ont été ajoutées au culot. Le
mélange est remis en suspension au moyen d'une pipette tout en
évitant la formation de mousse.
La lecture de la coloration du mélange a
été faite à T=15 minutes et T=30 minutes. Suivant les
recommandations du fabricant la lecture doit se faire à 15 minutes avec
l'interprétation sus citée.
5.5 Identification
des espèces bactériennesà partir des culots
5.5.1 Matériel
nécessaire
Le matériel ayant servi à l'identification des
espèces bactériennes à partir des culots d'urines et
d'hémocultures est représenté dans la figure 5.
A
B

Figure 5: Matériel
nécessaire à l'identification des germes au spectromètre
de masse
A= Pipette + matrice + lame de spectrométrie + E.
coli de contrôle, B = MALDI-TOF-MS,
5.5.2 Réalisation des
identifications
Le test au â-LACTATM était
systématiquement couplé à l'identification d'espèce
bactérienne à partir des culots en spectrométrie de masse
suivant la procédure ci-après.
Dans un tube Eppendorf, 1,5 ml d'urine ou de suspension
d'hémoculture ont été transférés à
l'aide d'une pipette,puis centrifugés à 3000 tours par minute
pendant 5 minutes. Après élimination du surnageant, le culot a
été repris dans 500 ul d'eau distillée puis remis en
suspension par pipetage aller/retour. Cette suspension est ensuite centrifuger
à 13000 tours par minute pendant 2 minutes, le surnageant est à
nouveau éliminé et le culot a été repris dans 50 ul
d'éthanol pure (éthanol 100%). Au moyen d'une pipette, 1 ul de la
suspensiona été déposé dans la cible puis
séché à l'étuve à 37°C. Enfin, 1 ul de
matrice a été ajouté sur chaque dépôt ; le
touta été mis à sécher avant de passer la lameau
MALDI-TOF-MS.
5.6 Test
immunochromatographique de détection de BLSE
5.6.1 Matériel
nécessaire
Les Cassettes CTX-M lot numéro 180604-01, une solution
d'extraction, des tubesEppendorf et des micropipettes de 100ul ont servi
à la réalisation de ce test (figure 6).
B
A

Figure 6: Matériel pour
la réalisation du test immunochromatographique NG-Test-CTX-M
A= cassette CTX-M MULTI ; B = réactif de lyse
5.6.2 Réalisation du test
La réalisation de test CTX-M a été faite
directement à partir des échantillons comme suit :
Dans un tube Eppendorf, 5 gouttes (environ 150ul) de
réactif et 5 gouttes d'échantillons (urines ou suspension
d'hémoculture) ont été mélangées au moyen
d'une pipette. Après homogénéisation du mélange au
vortex, 100ul ont été déposés au moyen d'une
pipette à usage unique destinée à cet effet sur la petite
rigole de la cassette immunochromatographique. Le mélange migre par
capillarité le long de la cassette, les résultats ont
été lus dans les 15 à 20 minutes suivant le
dépôt :
- une seule bande rouge apparait dans de la zone de
contrôle(C) : test négatif
- 2 bandes rouges apparaissent respectivement dans les zones
de C et dans la zone test (T) :test positif
- une seule bande rouge sur la zone T :test
ininterprétable
5.7 Analyse des
échantillons sur l'automate ePlex® de GenMark
5.7.1 Matériel
nécessaire
Les cartouches GenMark RUO lot numéro 53043525 et CE
lot 53111999, une pipette calibrée de 50ul, et l'automate ePlex® de
GenMark ont servi à l'identification d'espèces et de gènes
de résistance (figure 7).
B
A

Figure 7: Matériel
nécessaire pour l'analyse des échantillons sur
ePlex®
A= cartouche CE, B= automate ePlex®
La technologieePlex® de GenMark repose sur un principe de
PCR multiplex, elle est entièrement automatisée et permet
l'identification d'espèce et de gènes de résistance. Le
temps requis pour l'identification d'un germe est de 1h37 minutes. Cette
technologie propose actuellement trois panels : un panel Gram positif, un
panel de fungi et un panel Gram négatif, ce dernier est celui
utilisé dans notre étude, il permet la détection de 21
bactéries à Gram négatif et de 6 gènes de
résistance (figure 8).
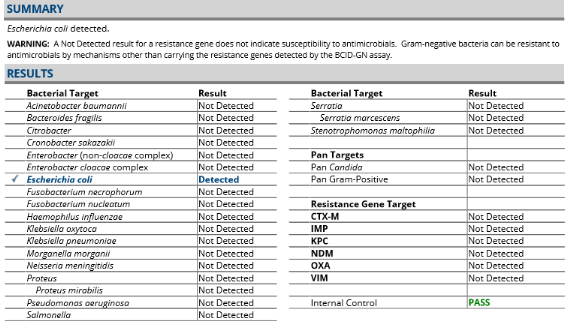
Figure 8: Composition du panel
BCID-GN de ePlex®
5.7.2 Réalisation du
test
La préparation et le chargement des cartouches sur
l'appareil ePlex® a suivi le protocole ci-contre.
Les cartouches ePlex® de GenMark ont été
ramenées à température ambiante du laboratoire avant leur
utilisation.Un code-barres d'identifiant échantillon correspondant au
code-barres de l'échantillon principal a été
appliqué sur la cartouche. Après homogénéisation de
l'échantillon par retournement du tube, 50 ul ont été
prélevés au moyen d'une pipette calibrée et
transférés dans l'orifice d'échantillonnage sur la
cartouche. Le bouchon a ensuite été tourné de 90°
dans le sens des aiguilles d'une montre et pressé pour fermer la
cartouche. Les codes-barres de la cartoucheont été balayés
au lecteur. Après une lecture réussie du code-barres de
numéro unique d'identification de la cartouche et du code-barres
d'identifiant échantillon, le lecteur émet un bip et la LED
clignote en vert pour indiquer la réussite de la lecture. La cartouche
est ensuite insérée dans l'une des baies prêtes,
indiquées par une LED blanche clignotante. Le système fait
correspondre cet identifiant échantillon avec la baie et lance
automatiquement le test.
6. Traitement des données
Les données issues de cette étude ont
été saisies sur Excel 2016. Les calculs statistiques pour
l'évaluation des performances des différentes techniques ont
été également réalisés sur Excel 2016. EPI
INFO version 7.2.2.6 a servi à la réalisation des comparaisons
entre les différentes valeurs avec P< 5% comme seuil de signification
statistique. Les références ont été
générées par Zotero-5.0.56.
RESULTATS
1. Aspects
épidémiologiques
1.1 Total des
échantillons analysés
Au total, 53 échantillons d'urines et 52
hémocultures non redondants ont été inclus dans cette
étude.
Parmi les 52 hémocultures positives, 47 (90,4 %)
flacons aérobies et 5 (9,6 %) flacons anaérobies ont
été enregistrés.Quant aux 53 échantillons d'urines,
ils sont issus de 29 femmes (55%) contre 24 hommes (45%), soit un sex-ratio
Homme/Femme de 0,83.
1.2 Espèces
responsables d'ITU et de bactériémie
Les échantillons positifs à BGN ont fait l'objet
d'identification directe sur ePlex®, et sur spectrométrie de masse
(MALDI-TOF-MS) à partir des colonies isolées en culture (tableau
1 et 2)
Tableau I: Récapitulatif des
espèces identifiées, ePlex® versus MALDI-TOF-MS à
partir des urines
|
Identification par automate ePlex® de GenMark
|
Identification par MALDI-TOF-MS après culture
|
Concordance d'identification
|
|
Espèces
|
Positif
|
Négatif
|
Echec
|
Positif
|
Négatif
|
ePlex® Vs Culture
|
|
Escherichia coli
|
33
|
2
|
2
|
37
|
0
|
33/35
|
|
Klebsiella pneumoniae
|
10
|
0
|
0
|
10
|
0
|
10/10
|
|
Klebsiella oxytoca
|
1
|
0
|
0
|
1
|
0
|
1/1
|
|
Enterobacter cloacae
|
2
|
0
|
1
|
3
|
0
|
2/2
|
|
Providencia rettgeri
|
Hors panel
|
|
1
|
0
|
-
|
|
Pseudomonas aeruginosa
|
0
|
0
|
1
|
1
|
0
|
0
|
|
Bacteroidesfragilis
|
1
|
0
|
0
|
-
|
1
|
-
|
|
Staphcoagnég
|
Pan Gram positive = 1
|
1
|
0
|
-
|
|
Strepto B
|
0
|
1
|
0
|
1
|
0
|
-
|
|
Total
|
47
|
4
|
4
|
55
|
1
|
46/48 (95,8%)
|
Au total, 56 souches bactériennes ont été
identifiées en cultures à partir des 53 urines. Les
entérobactéries représentaient 93 % des espèces
(52/56) dont le chef de fil (E. coli) représentait 66 % (37/56)
de l'ensemble suivi des espèces du genre Klebsiella qui
constituaient 19,6 % (11/56) des isolats.
TableauII: Récapitulatif des
espèces identifiées ePlex® versus MALDI-TOF-MS à
partir des hémocultures positives
|
Espèces
|
Identification par automate ePlex® de GenMark
|
Identification par MALDI-TOF-MS après culture
|
Concordance d'identification
|
|
Positif
|
Négatif
|
Echec
|
Positif
|
Négatif
|
ePlex® Vs Culture
|
|
Escherichia coli
|
20
|
1
|
0
|
21
|
0
|
20/21
|
|
Klebsiella pneumoniae
|
5
|
0
|
0
|
5
|
0
|
5/5
|
|
Klebsiella oxytoca
|
1
|
0
|
0
|
1
|
0
|
1/1
|
|
Enterobacter cloacae
|
3
|
0
|
0
|
3
|
0
|
3/3
|
|
Morganella morganii
|
1
|
0
|
0
|
1
|
0
|
1/1
|
|
Proteus mirabilis
|
1
|
0
|
0
|
1
|
0
|
1/1
|
|
Salmonella sp
|
1
|
0
|
0
|
1
|
0
|
1/1
|
|
Pseudomonas aeruginosa
|
6
|
0
|
1
|
7
|
0
|
6/6
|
|
Fusobacteriumnecrophorum
|
0
|
1
|
0
|
1
|
0
|
0/1
|
|
Stenotrophomonasmaltophilia
|
1
|
0
|
0
|
1
|
0
|
1/1
|
|
Acinetobacterbaumanii
|
2
|
0
|
0
|
2
|
0
|
2/2
|
|
Acinetobacterlwoffii,
Campylobacter coli,
Pseudomonas oryzyhabitans,
Pseudomonas stutzeri
|
Hors panel
|
1
1
1
1
|
0
|
-
-
-
-
|
|
Paenibacilusresidui
|
-
|
1
|
-
|
|
Bacillus subtilis
|
Pan Gram positive = 1
|
1
|
|
-
|
|
Bacillus licheniformis
|
Pan Gram positive = 1
|
1
|
0
|
-
|
|
Staphylococcus epidermidis
|
Pan Gram positive=2
Echec = 1
|
3
|
0
|
-
|
|
Staphylococcus hominis
|
Pan Gram positive= 1
|
1
|
0
|
-
|
|
Staphylococcus haemolyticus
|
Pan Gram positive= 1
|
1
|
0
|
-
|
|
Total
|
41
|
2
|
1
|
55
|
1
|
41/43 (95,3 %)
|
Au total, 56 souches bactériennes ont été
identifiées en culture à partir des 52 flacons
d'hémocultures dont 48 BGN, 3 BGP et 5 CGP. Les
bactériémies à BGN étaient essentiellement
dominées par les entérobactéries qui représentaient
environ 69 % (33/48) des cas, avec E. coli en tête43,7 %
(21/48).
1. 3 Résistance aux
antibiotiques
1.3.1 Phénotypes de
résistance des entérobactéries isolées
Les phénotypes de résistance des
entérobactéries isolés sont représentés dans
les figures 9 et 10. On enregistre 9 BLSE dans les bactériémies
contre 11 dans les ITU mais il n'existe aucune différence significative
dans la survenue d'une infection à BLSE en fonction du site d'infection
dans cette étude, P = 0,7.
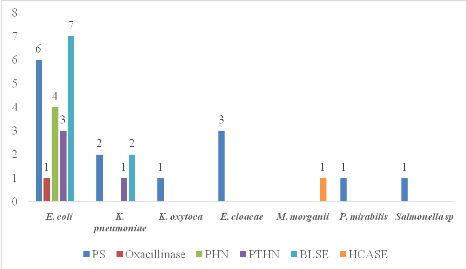
Figure 9: Répartition
des phénotypes des entérobactéries isolées dans les
hémocultures
Parmi les entérobactéries isolées dans
les hémocultures, 27,3% (9/33) était productrice de BLSE dont
7 E. coli et 2 Klebsiella pneumoniae.
.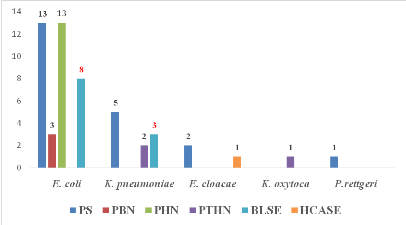
Figure 10: Répartition des
phénotypes des entérobactéries isolées dans les
urines
PS : phénotype sauvage
PBN : pénicillinase bas niveau
PHN : pénicillinase haut niveau
PTHN : pénicillinase de très haut niveau
BLSE : â-lactamase à spectre
élargi
HCASE : hyperproduction de céphalosporinase
Les entérobactéries uropathogènes
isolées, étaient productrices de BLSE dans 21,2 % (11/52). Au
sein des espèces majoritaires, 21,6 % (8/37) de E. coli contre
27,3 % (3/11) de Klebsiella spétaient productrices de
BLSE.Il y avait autant de souches sauvages que de souches productrices
de pénicillinases de haut niveau (35 %) au sein des E. coli
isolés.
Toutes les bactéries productrices de BLSEisolées
avaient une résistance associée aux quinolones (100% des BLSE
était résistant à l'ofloxacine). Cependant, elles ont
été toutes sensibles aux carbapénèmes (100% de
sensibilité vis-à-vis de l'ertapénème).
La prévalence d'entérobactéries
uropathogènes productrices de BLSE dans cette étude était
d'environ 21,2 %. Un patient sur cinq faisait donc une ITU à BLSE.
Plus de 27 % des klebsielles isolées étaient
productrices de BLSE contre 21,6 % des E. coli.
1.3.2 Répartition des
BLSE issues des uroculturesen fonction des services
La majorité des BLSE isolées était issue
du service de néphrologie. La répartition des BLSE

en fonction des services est présentée dans la
figure 11.
Figure 11: Répartition
des BLSE uropathogènes selon les services
Dans le service de néphrologie, 54,5% des ITU
étaient essentiellement dues à des bactéries productrices
de BLSE. Aucun patient des urgences médicales ne faisait une ITU
à BLSE.La forte prévalence des BLSE en néphrologie
pourrait s'expliquer par le long séjour des patients et le port des
sondes. Une intensification de la lutte contre la transmission des BLSE par des
activités de dépistage et d'isolement systématique des
patients dans ce service s'impose.
2. Performances diagnostiques des
tests
2.1 Identification
sur MALDI-TOF-MS à partir des culots de produits pathologiques
Les résultats de l'identification sur MALDI-TOF-MS
à partir des culots de centrifugation sont consignés dans le
tableau ci-dessous.
TableauIII: Résultats de
l'identification sur MALDI-TOF-MS à partir des culots de
centrifugation
|
Résultats identification
|
Échantillons d'urines
|
Hémocultures
|
P - value
|
|
Positif
|
9 (16,9 %)
|
32 (71 %)
|
< 0,001
|
|
Échec
|
44 (83,1 %)
|
13 (29 %)
|
|
Total
|
53 (100 %)
|
45 (100 %)
|
|
Tous les échantillons d'urines (100 %) ont
bénéficié d'un test d'identification sur MALDI-TOF-MS
à partir du culot. Des résultats mitigés ont
été enregistrés. En effet, 17% (9/53) d'identification
correcte ont été enregistré.
Quant aux hémocultures, l'identification à
partir des culots de suspensions a été réalisée sur
45 échantillons dont 71 % (32/45) d'identification correcte.
L'identification bactérienne par MALDI-TOF-MS à partir des
suspensions d'hémocultures a donné des meilleurs résultats
comparativement au culot urinaire P < 0,001.
2.2 Identification des
espèces sur l'automate ePlex® de GenMark
Parmi les bactéries isolées dans les
hémocultures, 44 BGN du panel, 4 BGN et 3 BGP hors panel GN de
ePlex® ainsi que 5cocci à Gram positif ont été
enregistrés. Dans les 44 BGN identifiés en culture faisant partir
du panel Gram négatif de ePlex®, 1 échec, 3 tests
négatifs ont été enregistrés et 41 bactéries
ont été détectées sur ePlex®, soit une
concordance de 95,3 % (41/43).
Dans les 53 échantillons d'urines, 56 souches
bactériennes ont été identifiées en culture dont
52BGN du panel GN de ePlex®, 1 BGN anaérobie, 1BGN hors panel et 2
cocci à Gram positifont été enregistrés.Parmi les
52 BGN identifiés en culture faisant parti du panel GN de ePlex®, 4
échecs ont été enregistrés, 2 tests négatifs
et 46 (95,8%)bactéries ont été identifiées sur
ePlex®.
Les concordances diagnostiques de l'automate ePlex® de
GenMark pour l'identification des espèces bactériennes
étaient de l'ordre de 95 % dans les deux produits pathologiques
étudiés (tableau 1 et 2).
2.3 Détection des
gènes de résistance sur ePlex®
Sur un total de 12 entérobactéries
uropathogènes résistantes aux C3G isolées dans les urines,
11 BLSEet 1 HCASE ont été enregistrées. 9/11 BLSE de type
CTX-Mont étédétectées sur ePlex®, 1
échec d'identification et 1 test négatif ont été
enregistrés.
Quant aux 9 entérobactéries productrices de BLSE
isolées dans les hémocultures, 8/9 BLSEde type CTX-M ont
été mise en évidence sur ePlex®. Un échec
d'identification d'un E. coli BLSE a également
été enregistré.
Les résultats des performances diagnostiques de
ePlex®, dans la détection des gènes de résistance
sont consignés dans le tableau 4.
TableauIV: Performance diagnostiques
de ePlex® dans la détection des gènes de
résistance
|
Echantillons d'urines
|
|
Résultats du test ePlex®
|
Présence de BLSE
|
Absence de BLSE
|
Total
|
|
Positif
|
9
|
1
|
10
|
|
Négatif
|
2
|
36
|
38
|
|
Total
|
11
|
37
|
48
|
|
Sensibilité= 81,8% VPP=90%
Spécificité=97,3% VPN=94,7%
|
|
Hémocultures
|
|
Résultats du test ePlex®
|
Présence de BLSE
|
Absence de BLSE
|
Total
|
|
Positif
|
8
|
0
|
8
|
|
Négatif
|
1
|
24
|
25
|
|
Total
|
9
|
24
|
33
|
|
Sensibilité= 88,9% VPP= 100%
Spécificité=100 % VPN= 96%
|
VPP= valeur prédictive positive ; VPN = valeur
prédictive négative
Les 2 bactéries productrices de BLSE résistantes
aux C3G dont aucungène de résistance n'a été mis en
évidence dans les urines sont des E. coli : l'une n'a pas
été détectée sur l'automate ePlex® et l'autre
a été détectée sans gène de
résistance associé.
2.4Détection des BLSE
par les tests immunochromatographiques
Tous les échantillons retenus ont fait l'objet de la
recherche de CTX-M par les cassettes immunochromatographiques. 10/11 desBLSE
issus des urines ont été positives au test CTX-M. La BLSE non
détectée avait un mécanisme de résistance autre que
la production de CTX-M.
Toutes les 9 BLSE issues des hémocultures ont
été détectées par les cassettes
immunochromatographiques. La sensibilité et la spécificité
diagnostiques des cassettes immunochromatographiquessont consignées dans
le tableau 5.
Tableau V: Résultats du NG Test
CTX-M MULTI dans la détection des BLSE
|
Echantillons d'urines
|
|
Résultats NG-Test-CTXM MULTI
|
Présence de BLSE
|
Absence de BLSE
|
Total
|
|
Positif
|
10
|
1
|
11
|
|
Négatif
|
1
|
40
|
41
|
|
Total
|
11
|
41
|
52
|
|
Sensibilité = 90,9 % VPP= 90,9
%
Spécificité= 97,6 %
VPN= 97,6 %
|
|
Hémocultures
|
|
Résultats NG-Test-CTXM MULTI
|
Présence de BLSE
|
Absence de BLSE
|
Total
|
|
Positif
|
9
|
0
|
9
|
|
Négatif
|
0
|
24
|
24
|
|
Total
|
9
|
24
|
33
|
|
Sensibilité = 100 % VPP= 100
%
Spécificité= 100 %
VPN= 100 %
|
Les entérobactériesrésistantes aux C3G
isolées avaient comme principal mécanisme de résistance la
production de BLSE de type CTX-M, 83,3 % (10/12) des cas dans les urines et
100% des cas dans les hémocultures.
2.5Mise en évidence de
la résistance aux C3G par le test â-LACTATM
Le test â-LACTATM a été
réalisé sur tous les échantillons d'urines et
d'hémocultures à partir du culot. Les résultats sont
consignés dans le tableau 6.
TableauVI: Résultats du test
â-LACTATM pour la détection de la résistance aux C3G par
production de BLSE
|
Echantillons d'urines
|
|
Résultats â-LACTAtest
|
Présence de BLSE
|
Absence de BLSE
|
Ininterprétable
|
Total
|
|
Positif
|
9
|
1
|
0
|
10
|
|
Négatif
|
2
|
40
|
0
|
42
|
|
Total
|
11
|
41
|
0
|
52
|
|
Sensibilité= 81,8 % VPP=90 %
Spécificité = 97,6 % VPN=
95,2 %
|
|
Hémocultures
|
|
Résultats â-LACTAtest
|
Présence de BLSE
|
Absence de BLSE
|
Ininterprétable
|
Total
|
|
Positif
|
9
|
0
|
0
|
9
|
|
Négatif
|
0
|
23
|
1
|
24
|
|
Total
|
9
|
23
|
1
|
33
|
|
Sensibilité = 100 % VPP = 100
%
Spécificité = 95,8% VPN=
100 %
|
Le test â-LACTATMa été positif
dans 10 cas sur 12 bactéries uropathogènes résistantes aux
C3G, avec 5 échantillons positifs en moins de 15 minutes et 5
échantillons positifs entre 15 et 30 minutes. Suivant les
critères établis par le fournisseur, la sensibilité du
test dans cette étude était de 45,5%. Selon les critères
de lectures utilisés dans cette étude, la sensibilité
diagnostic du test â-LACTATM était de 81,8 %.
Tous les échantillons d'hémocultures contenant
des BLSE ont été positifs au â-LACTATM test. Une
sensibilité de 100%, une spécificité de 95,8% et des
valeurs prédictives positives et négatives de 100% sont les
performances diagnostiques du â-LACTATM test pour la
détection des BLSE à partir des hémocultures ;
Le â-LACTATM test, couplé à
l'identification précoce par MALDI TOF MS permet une détection
précoce des BLSE dans les hémocultures.
2.6 Résultats
comparés du diagnostic de la résistance aux C3G par production de
BLSE
Les performances des trois tests pour la détection de
la résistance aux C3G par production de BLSE sont résumées
dans les figures 12 et 13 ;
Les performances diagnostiques de l'automate ePlex® sont
similaires à celles du â-LACTATM test. Le test
immunochromatographique a montré de meilleurs résultats.
Toutefois, les résultats de cette étude montrent que ces trois
tests sont suffisamment bons pour être recommandés dans la
détection rapide des BLSE dans les bactériémies et les
ITU.
BLT = â-LACTAtest
CTX-M = NG-test CTX-M MULTI
ePlex = automate ePlex®
C3GR : résistant aux céphalosporines de
3ème génération
C3GS : sensible aux céphalosporines de
3ème génération
HP = hors panel
Xr = gène de résistance
= négatif
+ = positif
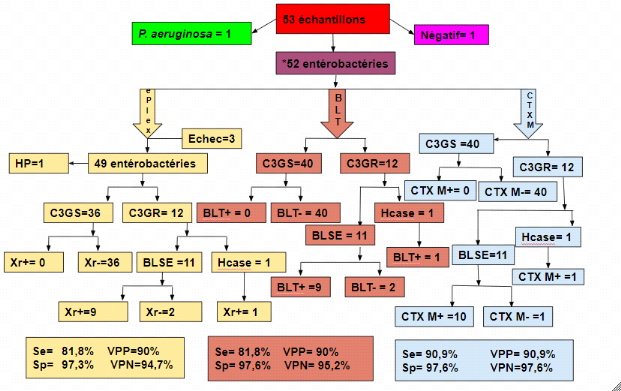
Figure 12: Comparaison des
performances diagnostiques des techniques de détection de la
résistance aux C3G par production de BLSE dans les urines.
* présence d'une (1) urine polymicrobienne (E.
cloacae et K. pneumoniae isolés dans un échantillon).
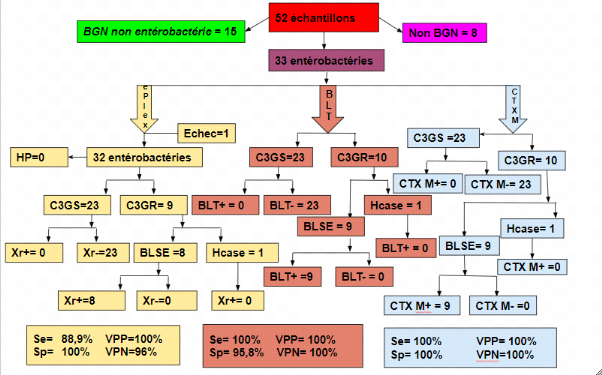
Figure 13: Comparaison des
performances diagnostiques des techniques de détection de la
résistance aux C3G par production de BLSE dans les
hémocultures
DISCUSSION
1. Aspects
épidémiologiques
1.1
Répartition des échantillons analysés
53 échantillons d'urines et 52 hémocultures non
redondants ont été analysés. Parmi les 52
hémocultures positives, 47 (90,4 %) flacons aérobies et 5 (9,6 %)
flacons anaérobies ont été enregistrés.La
fréquence élevée de la positivité des flacons
aérobies s'expliquerait par la prédominance des bactéries
aérobies ou anaérobies tolérantes en l'occurrence les
entérobactéries dans les bactériémies à
BGN.
Quant aux 53 échantillons d'urines, ils sont issus de
29 femmes (55%) contre 24 hommes (45%), soit un sex-ratio Homme/Femme de
0,83.Les ITU sont les infections bactériennes les plus communes chez la
femme, et un motif fréquent de consultation en médecine
générale. On estime que 75% des infections urinaires concernent
des femmes et plus de 50 % des femmes feront un jour un épisode d'ITU
(Foxmanet al., 2002 ; Merisier et al., 2014).
1.2
Fréquence des germes responsables de bactériémie
Au total, 56 souches bactériennes ont été
identifiées en culture à partir des 52 flacons
d'hémocultures dont 48 BGN, 3 BGP et 5 CGP. Les
bactériémies à BGN étaient essentiellement
dominées par les entérobactéries qui représentaient
environ 69 % (33/48) des cas, avec E. coli en tête 43,7 %
(21/48). Les entérobactéries constituent l'une des principales
étiologies des bactériémies potentiellement mortelle.
Mizrahiet al. (2018) ont rapporté que sur 335 épisodes
de bactériémies à BGN, 81,2% (272/335) était
causé par des entérobactéries avec E. coli en
tête 51% (171/335).
1.3
Fréquence des germes responsables d'infection du tractus urinaire
Au total, 56 souches bactériennes ont été
identifiées en cultures à partir des 53 urines. Les
entérobactéries représentaient 93 % des espèces
(52/56) dont le chef de fil (E. coli) représentait 66 % (37/56)
de l'ensemble suivi des espèces du genre Klebsiella qui
constituaient 19,6 % (11/56) des isolats. L'implication des
entérobactéries dans les ITU n'est plus à
démontrer. En milieu communautaire comme hospitalier, les
étiologies des ITU sont dominées par les
entérobactéries. La physiopathologie ascendante de l'ITU ainsi
que la forte colonisation du périnée par les
entérobactéries, associées aux facteurs spécifiques
d'uropathogénicité telles que les adhésines
bactériennes capables de se lier à l'épithélium
urinaire expliqueraient cette prédominance. Ces résultats sont
similaires à ceux décrits dans la littérature. En effet,
des auteurs avaient rapporté des prévalences de 80 à 92 %
d'entérobactéries uropathogènes avec 57 à 73 % de
E. coli (Sarkiset al., 2017 ; Hailajiet al.,
2016 ; Sbitiet al., 2017).
Dans cette étude, les espèces du genre
Klebsiella viennent en deuxième position après les
colibacilles dans les ITU. Ce même constat avait été fait
par Hailajietal. (2016)qui avaient rapporté que les klebsielles
représentaient 24 % des bactéries uropathogènes et
qu'elles venaient en seconde position après E. coli.
1.4
Fréquence des BLSE parmi les entérobactéries
isolées dans les hémocultures
Parmi les entérobactéries isolées dans
les hémocultures, 27,3% (9/33) était productrice de BLSE. Ces
résultats sont similaires à ceux rapportés par
Mainardiet al.ainsi que Walewski et al. (2015)qui ont
retrouvé qu'environ 30 % des entérobactéries
isolées dans les bactériémies était
résistantes aux C3G dont 17,8 à 24 % de BLSE.
1.5
Fréquence des entérobactéries uropathogènes
productrice de BLSE
Les entérobactéries uropathogènes
isolées, étaient productrices de BLSE dans 21,2 % (11/52). Au
sein des espèces majoritaires, 21,6 % (8/37) de E. coli contre
27,3 % (3/11) de Klebsiella sp étaient productrices de
BLSE. Il y avait autant de souches sauvages que de souches
productrices de pénicillinases de haut niveau (35 %) au sein des E.
coli isolés.
Toutes les bactéries productrices de BLSE
isolées avaient une résistance associée aux quinolones
(100% des BLSE était résistant à l'ofloxacine). Cependant,
elles ont été toutes sensibles aux carbapénèmes
(100% de sensibilité vis-à-vis de l'ertapénème).
La prévalence d'entérobactéries
uropathogènes productrices de BLSE dans cette étude était
d'environ 21,2 %. Un patient sur cinq faisait donc une ITU à BLSE. Un
constat similaire avait été rapporté par
Sarkisetal. (2017) au Liban qui avait montré que 23 % des
entérobactéries isolées étaient productrices de
BLSE. Toutefois la prévalence des BLSE dans les
entérobactéries uropathogènes est très
variée dans le temps et dans l'espace en fonction des études. En
effet, au cours des années 2004, les pays du sud de l'Europe
enregistraient des prévalences de BLSE pouvant aller jusqu'à 10 %
contre 5 % dans les pays du Nord (Nijssenetal., 2004). Des
études menées respectivement au Maroc et en Mauritanie entre 2013
et 2015 ont rapporté des prévalences de BLSE nettement
inférieures aux résultats de cette étude, allant de 10,25
à 13,77 % (Hailajietal., 2016 ; Sbitietal.,
2017). Ouedraogoet al. (2017) au Burkina Faso ont rapporté des
prévalences de 57,8% chez les entérobactéries
uropathogènes. La fréquence élevée des BLSE dans
cette étude pourrait s'expliquer par la spécificité de la
structure hospitalière. En effet, l'hôpital Saint Louis a une
cohorte importante de patients immunodéprimés dont la
majorité est sous antibioprophylaxie, ce qui serait sans doute à
l'origine de la sélection des bactéries productrices de BLSE.
Plus de 27 % des klebsielles isolées étaient
productrices de BLSE contre 21,6 % des E. coli, ces résultats
sont similaires à ceux rapportés par Hailajiet al.
(2016). Des prévalences plus élevées 62,7% de
Klebsiella BLSE contre 58,8% de E. coli BLSE ont
été retrouvées au Burkina Faso (Ouedraogoetal.,
2017).
2. Performances diagnostiques des
tests
2.1 Performances du
MALDI-TOF-MS dans l'identification d'espèces bactériennes
à partir des culots de centrifugation
Des résultats mitigés ont été
enregistrés lors de l'identification d'espèces
bactériennes à partir du culot urinaire. En effet, 17% (9/53)
d'identification correcte ont été enregistré. Toutefois,
des études de comparaison de la spectrométrie de masse aux
techniques classiques d'identification bactérienne ont conclu que la
technique MALDI-TOF-MS était une méthode prometteuse dans
l'identification des bactéries directement à partir du culot
urinaire obtenu après centrifugation à faible vitesse suivie
d'une centrifugation à grande vitesse. Cette technique serait plus
sensible si les urines sont mono microbiennes avec une bactériurie =
105(Ferreira etal.,2010). En outre, la technique pourrait
être optimisée par une précipitation à
l'éthanol des protéines du culot obtenu après la double
centrifugation, suivie d'une remise en suspension de celui-ci par de l'acide
formique et de l'acétonitrile(Ferreira et al., 2011).
L'établissement d'un protocole standard
d'identification des bactéries au MALDI-TOF à partir du culot
urinaire sur un nombre important d'échantillon s'avère donc
indispensable pour uniformiser les pratiques et optimiser cette technique.
Quant aux hémocultures, l'identification à
partir des culots de suspensions a été réalisée sur
45échantillons dont 71 % (32/45) d'identification correcte.
L'identification bactérienne par MALDI-TOF-MS à partir des
suspensions d'hémocultures a donné des meilleurs résultats
comparativement au culot urinaire, P < 0,001. Ces bonnes performances sur
les flacons d'hémocultures s'expliqueraient par la présence d'un
protocole standard validé ; en outre les hémocultures
étant des milieux d'enrichissement, la charge bactérienne serait
nettement plus élevée comparativement aux urines.Mizrahiet
al. (2018) ont rapporté que la technique MALDI-TOF-MS est
performante dans le diagnostic précoce des bactériémies
à BGN et contribue à une antibiothérapie précoce
adaptée avec une sensibilité diagnostique de 89,8 %.
2.2 Performances de
l'automate ePlex® pour l'identification des espèces à partir
des produits pathologiques
Les concordances diagnostiques de l'automate ePlex® de
GenMark pour l'identification des espèces bactériennes
étaient respectivement de 95,8 % (46/48) dans les urines contre 95,3%
(41/43) dans les hémocultures. Très peu de données sur cet
appareil dans l'analyse des urines existent dans la littérature.
Cependant, il pourrait être d'un grand intérêt dans la prise
en charge rapide des ITU. Casparet al. (2017) ont rapporté des
sensibilités de 90 % dans l'identification d'espèces
bactériennes à partir des flacons d'hémocultures positives
à BGN.
2.3 Performances de
l'automate ePlex® pour la détection des gènes de
résistance
Les Sensibilités et spécificités
diagnostiques de l'automate ePlex® pour la détection des BLSE
variaient respectivement entre 81,8 - 88,9 % et 97,3%- 100 %.
Aucune étude d'évaluation de cet appareil pour
la détection des gènes de résistance dans les urines n'a
été retrouvée dans la littérature. Cependant, une
sensibilité de 90 % avait été rapportée par
Casparetal.(2017) dans leur étude sur la détection de
BLSE à partir desflacons d'hémocultures positive à BGN.
Ces mêmes auteurs avaient montré que l'automate ePlex® offre
la possibilité d'une antibiothérapie efficace, précoce et
rationnelle tout en préservant les carbapénèmes qui sont
les antibiotiques de derniers recours.
Il ressort de cette étude que les performances
diagnostiques de l'automate ePlex® sont suffisamment bonnes pour son
utilisation dans la détection de la résistance bactérienne
directement à partir des produits pathologiques (urines et
hémocultures). Cependant le nombre de gènes inclus dans le panel
reste limité bien que ceux-ci soient les plus fréquemment
incriminés dans la multirésistance.
2.4 Performances du
NG-Test-CTX-M MULTI dans la détection des BLSE
Les paramètres sensibilité et
spécificité diagnostiques des cassettes NG-Test-CTX-M MULTI
étaient respectivement de 90,9 % et 97,6 % dans les ITU et 100% dans les
bactériémies. Le NG-Test CTX-M MULTI est d'un grand
intérêt dans la détection rapide des BLSE. C'est un test
simple à réaliser qui peut être pratiqué dans tous
les laboratoires et même dans le cabinet du médecin
généraliste. En outre, la majeure partie des BLSE isolées
actuellement en clinique sont de type CTX-M. Leur apparition dans ces
dernières décennies a inversé la tendance
épidémiologique faisant d'elles les plus répandues et les
plus fréquentes dans le monde entier, indépendamment du milieu
(hospitalier ou communautaire) (Ruppéet al., 2010).
2.5 Performances du
â-LACTATMtest dans la détection des BLSE
Suivant les critères établis par le fournisseur,
la sensibilité du test dans cette étude pour ce qui concerne les
échantillons d'urines était de 45,5 %. Selon les critères
de lectures utilisés dans cette étude, la sensibilité
diagnostic du test â-LACTATM était de 81,8 %. Jonas
et al. (2016) dans une étude d'optimisation de l'utilisation du
test â-LACTATM avaient trouvé une sensibilité
similaire de l'ordre de 81 %. Ces résultats confortent une fois de plus
que les conditions d'utilisation et d'interprétation du test
â-LACTATM dans la mise en évidence de la
résistance aux C3G dans les urines doivent être
réadaptées. Gallahetal. (2014) dans le même sens,
ont rapporté que tous les cas de tests positifs ou
ininterprétables étaient des BLSE.
Tous les échantillons d'hémocultures contenant
des BLSE ont été positifs au â-LACTATM test. Une
sensibilité de 100%, une spécificité de 95,8% et des
valeurs prédictives positives et négatives de 100 % sont les
performances diagnostiques du â-LACTATM test pour la
détection des BLSE à partir des hémocultures ; des
résultats similaires ont été rapportés par Walewski
et al. (2015) ainsi que Mizrahiet al. (2018) avec des
sensibilités et spécificités respectives de 89,3 - 95,7 %
et 100%. Le â-LACTATM test, couplé à
l'identification précoce par MALDI TOF MS permet une utilisation
efficace et rationnelle des antibiotiques dans les ITU et les
bactériémies à BGN.
3. Avantages et limites des trois tests
étudiés
Les tests étudiés présentent tous de
bonnes performances pour le diagnostic rapide des BLSE. Ils ont en commun, la
simplicité, la rapidité et la facilité d'exécution
mais aucun test n'est suffisamment parfait pour remplacer l'antibiogramme.
La technique de ePlex®, outre le diagnostic
d'espèces et des gènes de résistance présente
l'avantage d'être entièrement automatisée et s'affranchit
des erreurs dépendant des techniciens. Les résultats sont obtenus
en moins de 2 heures. Elle est facilement réalisable en période
de service à personnel réduit (garde, astreinte). Elle permet
également de prévoir le phénotype bactérien.
Cependant, cette technique a un coût très élevé
(environ 120 € par test) et implique la disposition de matériel
sophistiqué. C'est une technique réservée aux laboratoires
spécialisés. Le nombre de gène détecté est
assez limité et la présence d'un gène de
résistance n'est pas synonyme d'expression du gène.
La technique du â-LACTATM test est la moins
coûteuse comparée aux 2 autres. Elle est réalisable dans
tout laboratoire disposant d'une centrifugeuse. Les résultats sont
obtenus en moins de 30 minutes. Cependant, la lecture des résultats
à l'oeil nu est sujette à des erreurs. En plus une bonne
interprétation des résultats nécessite une identification
couplée du pathogène. Ce test présente une faible
sensibilité pour les entérobactéries du groupe 3. Un
résultat négatif de â-LACTATM test n'exclut pas
la présence de bactéries résistantes aux C3G.
Le NG-Test CTX-M MULTI présente l'avantage d'être
facilement praticable. Ce test ne nécessite pas l'utilisation de
matériel supplémentaire en dehors du kit fourni par le
fabriquant. Il est réalisable dans tous les laboratoires et peut
être pratiqué dans le cabinet du médecin
généraliste. Cependant, son coût est peu
élevé comparativement au â-LACTATMtest, il ne
permet de détecter que les BLSE de type CTX-M.
CONCLUSION
La résistance aux antibiotiques constitue un grand
enjeu de santé publique. La lutte contre la résistance
bactérienne implique nécessairement l'utilisation des outils de
diagnostic rapide.
Il ressort de cette étude que le panel Gram
négatif de ePlex® est bien adapté au diagnostic rapide des
entérobactéries et à l'identification des gènes de
résistance associés. Cette technique, entièrement
automatisée pourrait être d'un grand apport dans le diagnostic
rapide des ITU et des bactériémies surtout en période de
service à personnel réduit dans les hôpitaux. Toutefois, la
simplicité et le moindre coût des tests â-LACTATM
ainsi que des cassettes immunochromatographiques CTX-M MULTI font d'eux
des alternatives efficaces dans le diagnostic rapide des
entérobactéries productrices de BLSE. L'examen direct et la
culture demeurent indispensables pour l'amélioration de la
sensibilité et de la complémentarité de ces tests.
Recommandations
Ø Au chef de service de bactériologie de
l'hôpital Saint Louis de :
- Mettre à disposition des biologistes d'astreintes et
de gardes, l'automate ePlex® pour améliorer le diagnostic en
urgence des bactériémies et des ITU d'allures
sévères ;
- Améliorer le rendu des résultats des ECBU
à J0 en associant les cassettes CTX-M ou le â-LACTATM
test au service du jour.
Ø A tous les biologistes et techniciens de
laboratoire
Etendre les délais de lecture du
â-LACTATM test à 30 minutes pour le culot urinaire avec
considération de tout changement de coloration comme un résultat
positif.
Ø Aux autorités sanitaires du Burkina
Faso,
Intégrer les tests de diagnostic rapide de la
résistance bactérienne dans les laboratoires d'analyses
biomédicales.
Perspectives
Au vu des résultats de l'identification des
espèces à partir du culot urinaire, l'optimisation du
MALDI-TOF-MS pour l'identification des bactéries à partir des
urines sera envisagée à la suite de ce travail.
REFERENCES
BIBLIOGRAPHIQUES
1.Amzalag J, Mizrahi A, Naouri D, Nguyen JC, Ganansia
O, Le Monnier A. Optimization of the â LACTA
test for the detection of extended-spectrum- â
-lactamase-producing bacteria directly in urine samples. Infect
Dis.2016 ; 48 (9) : 695?8.
2. Bauernfeind A, Schweighart S, Grimm H. A
new plasmidiccefotaximase in a clinical isolate of Escherichia coli.
Infection. 1990 ; 18 (5) : 294?8.
3. Bonnet R. â-lactamines et
entérobactéries. In Antibiogramme, 3ème ed.,
ESKA. 2012: p 165-188.
4. Boutal H. Développement et
validation de tests de détection rapide de la résistance aux
antibiotiques.Thèse. Université Paris-Saclay, 2017 :245p.
5. Cantón R, Coque TM. The CTX-M
â-lactamase pandemic. Curr Opin Microbiol. 2006 ; 9 (5) :
466?75.
6. Caspar DY. Apports des panels BCID
à la prise en charge du sepsis. : RICAI 2017.
7. Chanal C, Bonnet R, De Champs C, Sirot D, Labia R,
Sirot J. Prevalence of â-lactamases among 1072 clinical strains
of Proteus mirabilis: 2 years survery in French hospital.
Antimicrob. Agents Chemother.2000. 44: 1930-1935.
8. Clark AE, Kaleta EJ, Arora A, Wolk DM.
Matrix-Assisted Laser Desorption Ionization-Time of Flight Mass Spectrometry: a
Fundamental Shift in the Routine Practice of Clinical Microbiology.
ClinicalMicrobiologyReviews. 2013;26(3):547-603.
9. Comité de l'Antibiogramme de la
Société Française de Microbiologie: Recommandations de
fevrier 2018.
10. Costelloe C, Metcalfe C, Lovering A, Mant D, Hay
AD. Effect of antibiotic prescribing in primary care on antimicrobial
resistance in individual patients : systematic review and meta-analysis.
BMJ. 2010 ; 340 : c2096?c2096.
11. Donhofer A, Franckenberg S, Wickles S,
Berninghausen O, Beckmann R, Wilson DN. Structural basis for
TetM-mediated tetracycline resistance. ProcNatlAcad Sci. 2012 ; 109
(42) :16900?5.
12. Dortet L, Poirel L, Nordmann P.
Épidémiologie, détection et identification des
entérobactéries productrices de
carbapénèmases Feuill De Biol. 2013.
13. Drawz SM, Bonomo RA. Three Decades of
-Lactamase Inhibitors. ClinMicrobiol Rev. 2010 ; 23 (1) : 160?201.
14. Ferreira L, Sanchez-Juanes F, Gonzalez-Avila M,
Cembrero-Fucinos D, Herrero-Hernandez A, Gonzalez-Buitrago JM, et
al., Direct Identification of Urinary Tract Pathogens from Urine
Samples by Matrix-Assisted Laser Desorption Ionization-Time of Flight Mass
Spectrometry. J ClinMicrobiol.2010 ; 48 (6) : 2110?5.
15. Ferreira L, Sánchez-Juanes F,
Muñoz-Bellido JL, González-Buitrago JM. Rapid method for
direct identification of bacteria in urine and blood culture samples by
matrix-assisted laser desorption ionization time-of-flight mass spectrometry:
intact cell vs extraction method.ClinMicrobiol Infect.2011 ; 17 (7) :
1007?12.
16. Foxman B. Epidemiology of urinary tract
infections : incidence, morbidity, and economic costs. Am J Med. 2002
; 113 (1) : 5?13.
17. Gallah S, Decre D, Genel N, Arlet G. The
-Lacta Test for Direct Detection of Extended-Spectrum- -Lactamase-Producing
Enterobacteriaceae in Urine. J ClinMicrobiol. 2014 ; 52 (10)
: 3792?4.
18. Hailaji NSM, Ould Salem ML, Ghaber SM.La
sensibilité aux antibiotiques des bactéries uropathogènes
dans la ville de Nouakchott - Mauritanie. Prog En Urol.2016 ; 26 (6) :
346?52.
19. Harris P. Clinical Management of
Infections Caused by Enterobacteriaceae that Express Extended-Spectrum
â-Lactamase and AmpC Enzymes. SeminRespirCrit Care Med. 2015 ;
36 (01) : 056?73.
20. Hiramatsu K, Ito T, Tsubakishita S, Sasaki T,
Takeuchi F, Morimoto Y, et al., Genomic Basis for
Methicillin Resistance in Staphylococcus aureus. Infect
Chemother. 2013 ; 45 (2) :117.
21. Hooper DC. Fluoroquinolone resistance among Gram-positive
cocci. Lancet Infect Dis. 2002 ; 2 (9) : 530?8.
22. Karanika S, Karantanos T, Arvanitis M, Grigoras C,
Mylonakis E. Fecal Colonization With Extended-spectrum
Beta-lactamase-Producing Enterobacteriaceae and Risk Factors Among
Healthy Individuals: A Systematic Review and Metaanalysis. Clin Infect
Dis. 2016 ; 63 (3) : 310?8.
23. Kardaoe-Soma L. Coût de la
résistance bactérienne. la santé
publique.Séminaire DESC d'Infectiologie. 2017 :36.
24. Kliebe C, Nies BA, Meyer JF, Tolxdorff-Neutzling
RM, Wiedemann B. Evolution of plasmid-coded resistance to
broad-spectrum cephalosporins. Antimicrob Agents Chemother. 1985 ; 28
(2) : 302-307.
25. Kumar A, Roberts D, Wood KE, Light B, Parrillo JE,
Sharma S, et al., Duration of hypotension before initiation
of effective antimicrobial therapy is the critical determinant of survival in
human septic shock : Crit Care Med. 2006 ; 34 (6) : 1589?96.
26. Leclercq R. Mechanisms of resistance to
macrolides and lincosamides: nature of the resistance elements and their
clinical implications. Clin Infect Dis. 2002 ; 34 (4) : 482-492.
27. Mainardi J-L, Compain F, Bensekhri H, Lavollay M,
Rostane H. â LACTA test for rapid detection of
Enterobacteriaceae resistant to third-generation cephalosporins from
positive blood cultures using briefly incubated solid medium cultures.
Journal of Medical Microbiology. 2015;64(10):1256-9.
28. Melzer M, Petersen I. Mortality following
bacteraemic infection caused by extended spectrum beta-lactamase (ESBL)
producing E. coli compared to non-ESBL producing E. coli.
J Infect. 2007 ; 55 (3) : 254?9.
29. Merisier S. Le médecin
généraliste et les infections urinaires basses communautaires
(cystites simples), chez la femme de 15 à 75 ans : évaluation des
pratiques professionnelles en fonction des nouvelles recommandations du SPILF
de juin 2014. 2017 ; 85.
30. Mizrahi A, Amzalag J, Couzigou C, Péan De
Ponfilly G, Pilmis B, Le Monnier A. Clinical impact of rapid bacterial
identification by MALDI-TOF MS combined with the bêta-LACTATM
test on early antibiotic adaptation by an antimicrobial stewardship team in
bloodstream infections. Infectious Diseases. 2018;50(9):668-77.
31. Munita JM, Arias CA. Mechanisms of
Antibiotic Resistance. In : Kudva IT, Cornick NA, Plummer PJ, Zhang Q,
Nicholson TL, Bannantine JP, et al., Editeurs. Virulence Mechanisms
of Bacterial Pathogens, Fifth Edition. Am Society of Microbiol; 2016 ;
481?511
32. Naber KG, Schito G, Botto H, Palou J, Mazzei
T. Surveillance Study in Europe and Brazil on Clinical Aspects and
Antimicrobial Resistance Epidemiology in Females with Cystitis (ARESC):
Implications for Empiric Therapy. EurUrol. 2008;54 (5):1164?78.
33. Netgen. Que signifie
«?bêtalactamases à spectre élargi?» en pratique ?
Rev Med Suisse. - www.revmed.ch - 2009.
34. Nijssen S, Florijn A, Bonten MJM, Schmitz FJ,
Verhoef J, Fluit AC. Beta-lactam susceptibilities and prevalence of
ESBL-producing isolates among more than 5000 European Enterobacteriaceae
isolates. Int J Antimicrob Agents. 2004 ; 24 (6) : 585?91.
35. Ouedraogo A-S. Prévalence,
circulation et caractérisation des bactéries
multirésistantes au Burkina Faso. Thèse, Université de
MONTPELLIER 2017 :191p.
36. Pagès J-M, James CE, Winterhalter
M. The porin and the permeating antibiotic : a selective diffusion
barrier in Gram-negative bacteria. Nat Rev Microbiol. 2008 ; 6 (12) :
893?903.
37. Papp-Wallace KM, Bethel CR, Distler AM, Kasuboski
C, Taracila M, Bonomo RA. Inhibitor Resistance in the
KPC-â-Lactamase, a Preeminent Property of This Class A â-Lactamase.
Antimicrob Agents Chemother. 2010 ; 54 (2) : 890?7.
38.Paterson DL, Bonomo RA. Extended-Spectrum
â-Lactamases : a Clinical Update. ClinMicrobiol Rev. 2005 ; 18
(4) : 657?86.
39. Peralta G, Lamelo M, Álvarez-García
P, Velasco M, Delgado A, Horcajada JP, et al., Impact of
empirical treatment in extended-spectrum beta-lactamase-producing
Escherichia coli and Klebsiella spp. bacteremia. A
multicentric cohort study. BMC Infect Dis. 2012; 12 (1).
40. Poole K. Efflux-mediated antimicrobial
resistance. J AntimicrobChemother. 2005 ; 56 (1) : 20?51.
41. Queenan AM, Bush K. Carbapenemases: the
Versatile -Lactamases. Clin MicrobiolRev. 2007 ; 20 (3) : 440?58.
42. Ruppé E.
Épidémiologie des bêta-lactamases à spectre
élargi?: l'avènement des CTX-M. Antibiotiques. 2010 ; 12
(1) : 3?16.
43. Sarkis P, Assaf J, Sarkis J, Zanaty M, Rehban
R. Profil de résistance aux antibiotiques dans les infections
urinaires communautaires au Liban. Prog En Urol. 2017 ; 27 (13) : 727.
44. Sbiti M, Lahmadi khalid, louzi L. Profil
épidémiologique des entérobactéries
uropathogènes productrices de bêta-lactamases à
spectre élargi. Pan Afr Med J. 2017 ; 28.
45. Schwaber MJ, Carmeli Y. Mortality and
delay in effective therapy associated with extended-spectrum -lactamase
production in Enterobacteriaceae bacteraemia: a systematic review and
meta-analysis. J AntimicrobChemother. 2007 ; 60 (5) : 913?20.
46. Sirot D, Sirot J, Labia R, Morand A, Courvalin P,
Darfeuille-Michaud A, et al., Transferable resistance to
third-generation cephalosporins in clinical isolates of Klebsiella
pneumoniae?: identification of CTX-1, a novel â-lactamase. J
AntimicrobChemother. 1987 ; 20 (3) : 323?34.
47. Strahilevitz J, Jacoby GA, Hooper DC, Robicsek
A. Plasmid-Mediated Quinolone Resistance : A multifaceted Threat.
ClinMicrobiol Rev. 2009 ; 22 (4) : 664?89.
48. The-War-on-Drug-Resistance_PPC-Fall-2017. Disponible sur :
https://genmarkdx.com/wp-content/uploads/2018/06/.
49. Walewski V, Podglajen I, Lefeuvre P, Dutasta F,
Neuschwander A, Tilouche L, et al. Early detection with the
â-LACTATM test of extended-spectrum
â-lactamase-producing Enterobacteriaceae in blood cultures.
Diagnostic Microbiology and Infectious Disease. 2015;83(3):216-8.
50. Woerther P-L, Burdet C, Chachaty E, Andremont
A. Trends in Human Fecal Carriage of Extended-Spectrum
â-Lactamases in the Community : Toward the Globalization of CTX-M.
ClinMicrobiol Rev. 2013 ; 26 (4) : 744?58.
51. Yasufuku T, Shigemura K, Shirakawa T, Matsumoto M,
Nakano Y, Tanaka K, et al., Correlation of
Overexpression of Efflux Pump Genes with Antibiotic Resistance in
Escherichia coli Strains Clinically Isolated from Urinary Tract
Infection Patients. J Clin Microbiol. 2011 ; 49 (1) :189?94
RESUME
Les entérobactéries sont les principaux germes
responsables de bactériémies et d'infections du tractus urinaire.
Ces bactéries sont devenues une menace mondiale du fait de leurs
multirésistances aux antibiotiques, essentiellement due à
l'acquisition de â-lactamases à spectre élargi. Le but de
ce travail est d'évaluer d'une part, les performances diagnostiques de
l'automate ePlex® dans l'identification des bacilles à Gram
négatif et les gènes de résistances associés et
d'autres part évaluer les performances du NG-Test-CTXM MULTI et
â-LACTATMtest dans la détection des BLSE à
partir des échantillons d'urines et d'hémocultures.
Sur une période de cinq mois, les automates ePlex®
et MALDI-TOF-MS ont été utilisés pour l'identification des
bactéries à partir des échantillons d'hémocultures
et d'urines positives à bacilles à Gram négatif à
l'examen microscopique. La détection des BLSE a été
réalisée par l'automate ePlex® et par les NG-Test-CTXM MULTI
et â-LACTATM Test.
Au total, 53 échantillons d'urines et
52 hémocultures ont été analysés, les
entérobactéries représentaient respectivement 93 % (52/56)
et 59 % (33/56) des identifications avec Escherichia coli en
tête, 66 % (37/56) et 37,5 % (21/56) dans les ITU et les
bactériémies. Les entérobactéries productrices de
BLSE étaient de 21,2 % (11/52) dans les ITU et 27,3% (9/33) dans les
bactériémies.Les concordances d'identification entre ePlex®
et MALDI-TOF-MS après isolement étaient d'environ 95 % dans les
deux types d'échantillons. Les performances diagnostiques de l'automate
ePlex® pour la détection des BLSE sont similaires à celles
du â-LACTATM test avec des sensibilités de 81,8
à 100 % et des spécificités de 95 à 100%. Le
NG-Test-CTX-M MULTI a montré de meilleurs résultats avec des
sensibilités allant de 90,9 à 100 % et des
spécificités de 97,6 à 100 %. Le panel Gram négatif
de ePlex® est bien adapté au diagnostic rapide des
entérobactéries et la mise en évidence des gènes de
résistance associés. Toutefois, la simplicité et le
moindre coût des tests â-LACTATMainsi que des
NG-Test-CTX-M MULTI font d'eux des alternatives efficaces dans le diagnostic
rapide des entérobactéries productrices de BLSE.
Mots clés :BLSE, CTXM,
â-LACTAtest, ePlex®.
ABSTRACT
Enterobacteria are the main germs responsible for
bloodstreams and urinary tract infections. These bacteria became a global
threat because of their multidrug resistance, essentially due to the Extended
Spectrum Betalactamase (ESBL) acquisition. The purpose of this study is to
evaluate on the one hand, the diagnostic performances of the ePlex® PLC
from urines and blood cultures samples for Gram negative bacilli (BGN) and
associated resistances genes identification and, on the other hand, to evaluate
the performance of NG-Test-CTXM MULTI and â-LACTATM test in
the detection of ESBL from urine and blood cultures samples.
During five months, an identification has been achieved by
ePlex® and by MALDI-TOF-MS, from the positive urines and blood cultures
with BGN to the microscopic exam. The detection of the ESBL has been achieved
by ePlex®, NG-Test-CTXM MULTI and â-LACTATM Test.
On 53 samples of urines and 52 blood cultures analyzed, the
enterobacteria represented 93 % (52/56) and 59 % (33/56) respectively, with 66
% (37/56) and 37,5 % (21/56) of E. coli in UTI and bacteremia.
ESBL-producing enterobacteria were 21.2% (11/52) in the UTI and 27.3%
(9/33) in the bacteremia. The corresponding identification between ePlex®
and MALDI-TOF-MS were approximately 95 % in both types of samples.
The diagnostics performances of the ePlex® PLC for ESBL
production are similar to that of the â-LACTATM test with
sensitivities of 81.8 to 100% and specificities of 95 to 100%. The
NG-Test-CTX-M MULTI showed better results with sensitivities ranging from 90.9
to 100% and specificities from 97.6 to 100%. The Gram-negative panel of
ePlex® is adapted to the rapid diagnosis of the enterobacteria and their
genes of resistance. However, the simplicity and the lower cost of
â-LACTATM test as well as NG-Test-CTX-M MULTI are both
coalesces for efficient alternatives in the rapid detection of ESBL.
Key words :ESBL, CTXM, â-LACTAtest,
ePlex®

| 


