2.2.5 Conclusion
Nous avons introduit le concept, les stratégies de
criblage virtuel. Ce dernier est une approche informatique visant à
prédire des propriétés de librairies de molécules.
Avec l'essor considérable de données expérimentales
publiquement disponibles, cette discipline a enregistrédes
progrès considérables quant au débit, la qualitéet
la diversitédes prédictions possibles. Un inventaire des
applications du criblage »in-silico» est donné, tout
en gardant
8
une attention particulière à des cas concrets
d'utilisation ainsi qu'au développements futur. Le criblage virtuel
fournit une solution complémentaire pour le criblage virtuel à
haut débit »HTS», oùil comprend des techniques
de calcul novatrices. L'avantage du criblage virtuel »in-silico»
est donc de fournir une petite liste de molécules à tester
expérimentalement et ainsi réduire les coûts et gagner du
temps. On peut aussi explorer rapidement de nombreuses molécules pour se
focaliser ensuite, au niveau expérimental, sur les molécules les
plus intéressantes. Les difficultés intrinsèques aux
techniques à haut débit ainsi que celles rencontrées lors
des étapes d'optimisation des molécules chimiques, ont
encouragéle développement de nouvelles approches, telles que les
techniques de criblage virtuel par docking moléculaire.
9
2.3 Docking
2.3.1 Introduction
La modélisation de la structure d'un complexe
protéine-ligand est très importante pour la compréhension
des interactions de liaison entre un composépotentiel
»ligand» et sa cible thérapeutique
»protéine», et pour la conception de
médicaments à base de structure moderne.
Le docking ou »amarrage, arrimage» est une
procédétrès utile qui vise à prédire
l'inter-action potentiel de la structure d'un complexe moléculaire
à partir des petites molécules dans les sites de liaison de
protéine afin d'accélérer la recherche et la
découverte de nouveaux médicaments in-silico
(c-à-d à l'aide d'ordinateur), »Le docking
in-silico est la détermination de la structure 3D des complexes
protéiques à l'échelle atomique, qui permet de mieux
comprendre la fonction biologique de ces complexe [1]». Plus
précisement, le docking consiste à trouver la meilleure position
d'un ligand (petite molécule) dans le site de liaison d'un
récepteur (protéine) de façon à optimiser les
interactions avec un récepteur, évaluer les interactions
ligand-protéine de façon à pouvoir discriminer entre les
positionnement observées expérimentalement et les autres. De
façon générale, le docking a pour but de simuler
l'interaction entre les molécules in-silico, et les
résultats obtenus servent à prédire la structure et les
propriétés de nouveaux complexes [3].
Historiquement, les premiers outils de docking
obéissaient au principe dit: »lock-and-key» (principe
clef-serrure), selon lequel le ligand qui représente la clef, est
complémentaire au niveau géométrique du site actif du
récepteur, qui représente la serrure [Yuriev et al., 2011]. Les
ligands sont des petites molécules destinées à inhiber
l'activitéd'une protéine, qui constitue le récepteur. Il
permet aussi, de prédire la structure intermoléculaire entre deux
molécules en une structure tridimensionnelle 3D, les modes de liaison ou
les conformations possibles d'un ligand à un récepteur, et de
calculer l'énergie de liaison. La technique de docking prévoit
également la résistance de la liaison, l'énergie du
complexe, les types de signaux produits et estime l'affinitéde liaison
entre deux molécules. Elle joue un rôle très important dans
l'aide à la décision, afin de déterminer quel ligand
candidat interagira le mieux avec un récepteur protéine cible
[15].
Le docking protéine-ligand est utilisépour
vérifier la structure, la position et l'orientation d'une
protéine quand elle interagit avec les petites molécules comme
les ligands. Son but
est de prédire et de classer les structures
résultant de l'association entre un ligand donnéet une
protéine cible d'une structure 3D connue.
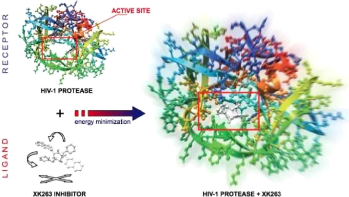
10
FIGURE 3 - Docking protéine-ligand
Le docking ligand-protéine reste donc la méthode
la plus souvent employée, car elle permet une évaluation rapide
de bases de milliers, voire de millions de molécules.
En principe, un programme de docking doit être capable
de générer les modes de liaison attendus pour des ligands dont la
position adoptée au sein du site actif est connue dans un temps
raisonnable. Pour cela, il est nécessaire que l'algorithme de recherche
confor-mationnelle puisse explorer l'espace conformationnel le plus
exhaustivement possible et de façon efficace. Classiquement, on juge la
qualitédu docking en mesurant le RMSD (Root Mean Square Deviation)
sur les atomes entre la pose obtenue en docking, et la pose
observée expérimentalement si elle existe.
| 


