2.3.4 Outils de Docking
A l'heure actuelle, plus de 30 programmes de docking
moléculaires (commerciaux ou non) sont disponibles [6]. Les plus
fréquemment cités sont respectivement : AutoDock [9], GOLD,
FlexX, DOCK et ICM. Ils permettent notamment un criblage rapide de vastes
librairies de composés. Ces programmes reposent le plus souvent sur des
algo-
rithmes spécifiques (Algorithme
génétique, Recuit Simulé...), leur protocole est
composéde 2 étapes essentielles Docking/Scoring.
Pour accomplir la tâche de docking, les outils
d'amarrage moléculaire vont générer une série de
poses différentes de liaison au ligand et en utilisant une fonction de
notation »scoring» pour évaluer les affinités
de liaison de ligand pour les poses générées afin de
déterminer le meilleur mode de liaison.
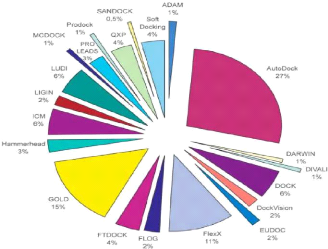
13
FIGURE 6 - Comparaison des programmes de
docking [16]
Comme la figure ci-dessus montre, le programme AutoDock est le
plus citéet le plus utiliséparmi les autres programmes de
docking.
2.3.5 Conclusion
Le processus de docking est l'un des premières
étapes dans la conception de médicaments, il consiste à
faire interagir une petite molécule organique avec un récepteur,
généralement de nature protéique. En conséquent, le
plus grand avantage des méthodes de docking protéine-ligand est
qu'ils peuvent proposer des hypothèses structurelles sur la façon
dont une petite molécule peut interagir avec sa cible
macromolécule. Des études ont montréque certains
algorithmes de docking sont plus fiables que d'autres pour reproduire le mode
de fixation expérimentale de ligand. La contrepartie de ces techniques
est généralement une hausse des temps de calcul et des
ressources. A l'inverse, un projet impliquant le criblage virtuel de millions
de produits ne pourra pas être accompli avec ce type d'algorithme mais
plutôt des codes plus simples, dans lesquels les approximations
engendrent un gain de temps de calcul et d'argent. Le nombre de programme de
docking actuellement disponibles est élevéet n'a
cesséd'augmenter au cours des dernières décennies. Les
exemples suivants présentent un aperçu des programmes les plus
communs de docking protéine-ligand (LigandFit, FlexX, AutoDock). Dans ce
travail nous avons utiliséle programme AutoDock.
Le docking est un type d'application facilement distribuable
sur une grille. De sorte que, de nombreuses ressources de calcul et de stockage
ont étémises à disposition par le projet EGEE
(Enabling Grids for E-sciencE), qui est financépar la
commission européenne et qui a pour but de construire sur les plus
récentes avancées des technologies de grille et de
développer un service d'infrastructure de grille disponible 24h/24h.
14
| 


