2.2 Direct Viable Count (DVC)
La méthode Direct Viable Count (DVC) a
été décrite par l'équipe de Kogure K. et al. en
1979 (Kogure K. et al, 1979 [19]). Cette méthode consiste à
incuber les cellules d'un échantillon à étudier,
éventuellement après purification ou extraction de la matrice
d'origine, dans un milieu contenant de l'extrait de levures et un inhibiteur de
synthèse d'ADN. L'antibiotique utilisé est l'acide nalidixique,
il s'agit d'un bactériostatique de la famille des quinolones, il inhibe
l'action de la girase empêchant par ce biais la synthèse d'ADN.
Les bactéries viables et sensibles à l'acide nalidixique, ne
pouvant plus se diviser, vont utiliser l'extrait de levures présent dans
le milieu comme substrat et avoir une forme allongée. Ce
caractère morphologique permet la différenciation et la
numération des germes viables.
Le problème est la sensibilité de la
méthode, faussée par les souches naturellement résistantes
à cet antibiotique. C'est pourquoi de nombreuses équipes ont
travaillées sur la méthode DVC en remplaçant l'acide
nalidixique par des cocktails d'antibiotiques. Dans leur article, «
Ecological Implications of an Improved Direct Viable Count Method for Aquatic
Bacteria » paru en 1997, Fabien Joux et Philippe LeBaron de l'observatoire
océanologique de Banyuls sur mer, montrent l'intérêt de
l'utilisation d'un cocktail d'antibiotiques dans l'application de la
méthode Direct Viable Count sur des échantillons marins (Joux F.,
LeBaron P., 1997, [15]). Dans un premier temps ils comparent la
sensibilité de 100 souches bactériennes isolées de
différents environnements marins côtiers à quatre
inhibiteurs de synthèse d'ADN (ciprofloxacine, acide nalidixique, acide
piromidique, acide pipemidique), et d'un inhibiteur de division cellulaire
(cephalexine).
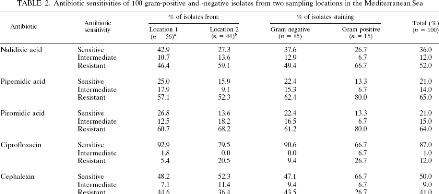
Le tableau 2 nous indique que seulement 36% des souches
testées sont sensibles à l'acide
nalidixique. Ces
résultats montrent donc que l'action de l'acide nalidixique seul est
insuffisante

pour bloquer la croissance de toutes les bactéries d'un
échantillon marin. Ceci implique un biais important dans les
résultats d'une analyse par la méthode DVC.
Tableau 2 : Sensibilité de 100 souches isolées
d'échantillons d'eau de mer (sur deux sites
Méditerranéens) à différents antibiotiques. (Joux
F., LeBaron P., 1997, [15])

Les auteurs ont alors testé le cocktail d'antibiotiques
suivant : acide nalidixique (20ug/mL), acide piromidique (20ug/mL), acide
pipemidique (10ug/mL), ciprofloxacine (0,5ug/mL) et cephalexine (10ug/mL).
La figure 6 présente l'évolution de la
population bactérienne dans un échantillon d'eau de mer
supplémentée avec 50mg/L d'extrait de levures, couplé dans
un cas avec l'acide nalidixique et dans l'autre avec le cocktails
d'antibiotiques. Les résultats montrent l'efficacité du cocktail
d'antibiotiques à inhiber la flore des échantillons.


Figure 6 : Réponse d'un échantillon naturel d'eau
de mer à l'ajout d'extrait de levures (50mg/L) et
d'antibiotiques.
Barre noire : nombre de bactéries totales sans
antibiotique ; barre blanche : nombre de bactéries totales avec
acide
nalidixique, barre grise : nombre bactéries totales avec le
cocktail antibiotique. Les échantillons présentant
des
croissance sont annotés par des astérisques. (Joux F.,
LeBaron P., 1997 [15])
La combinaison de plusieurs antibiotiques permet d'inhiber la
division et donc la prolifération des bactéries pendant 18h
d'incubation (statique et à 20°C dans l'obscurité). Ceci
permet de prolonger le temps d'incubation et donc le temps d'élongation
des bactéries lors du test DVC. La figure 7 illustre ces
résultats.

Figure 7 : Observation en microscopie à fluorescence,
coloration au DAPI. (A) aspect des bactéries avant incubation,
(B)
aspect après 6h d'incubation avec acide nalidixique, (C) aspect
après 18h d'incubation avec cocktail
d'antibiotiques. Les
flèches indiquent les cellules viables ayant métabolisées
l'extrait de levure (Joux F., LeBaron P.,
1997 [15]).
La méthode DVC permet de dénombrer les cellules
viables par microscopie. Cependant elle est
limitée par la
sensibilité des bactéries aux antibiotiques, certaines
résistantes, d'autres trop
sensibles. De plus, certaines
espèces bactériennes peuvent présenter naturellement un

polymorphisme visible au microscope, par exemple un bacille
à GRAM négatif peut se trouver dans la même culture en
forme de coccobacille ou en forme de très long bacille.
La méthode DVC, optimisée par l'utilisation de
cocktails d'antibiotiques, permet d'étudier la viabilité des
cellules de certains échantillons. La limite de cette méthode est
la différence de sensibilité des bactéries aux
antibiotiques, son utilisation doit être précédée
d'études préliminaires sur la sensibilité aux
antibiotiques des souches étudiées.
| 


