3.4 Les résultats finaux
La tâche principale de ce travail est de proposer au
final la meilleure combinaison de paramètres permettant d'avoir le
meilleur ajustement parallèle par génotype. Pour trouver la
meilleure combinaison paramétrique par génotype, il faut donner
des tailles de

49
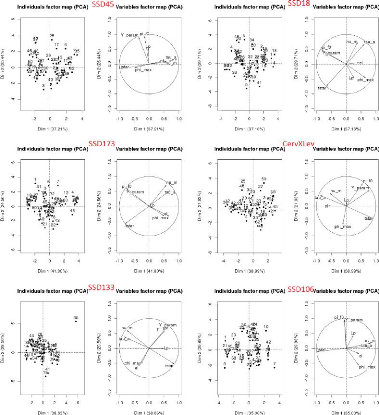
FIGURE 3.14 Les graphes des corrélations entre les huit
paramètres par génotype.

FIGURE 3.15 Les graphes des corrélations entre les
huit paramètres par génotype et sur l'ensemble des
génotypes.
50
Varlablea factor map (PCA)
|
Y -
|
individuels
factor map (PCA)
|
|
|
|
39
0 42
· GB -1
·I- ~8-_
. 23
·
P
|
1.0 -0.5 0.0 0.5
|
|
|
-4 -2 0 2 4
Dim 1 (32.37%)
Dim1 (3022%)
CeNI I Variables factor map (PCA)
-1.0 -0.5 0.0 0.5 1.0
Dim 1 (30.22%)
Celle-ci est une sous figure
Individuals factor map (PCA) Levovil

Dim 1 (32.37%)
-2 0 2 4
1,0 -0.5 0.0 0.5 1.0
-1,0 -0.5 0.0 0.5 1.0 4 -2 0 2 4
Dim 1 (37.21%)
Dim 1 (37.21%)
Dim, (37.18%)
Dim 1 (37.18%)
48
1 8 7
31 3:
a2 12 4
· as'2 ).6° âti
5073
.2 0 2 4
Dim, (41.80%)
1.0 -2 5 0.0 0.5
Dim 1 (38.99%)
1.0
1.0 -0.5 OD 0.s 1.0 4 -2 0 2
Dim, (41.80%) Dim1 (38.99%)
Individuals factor map (PCA)
SSD45 Variables factor map (PCA)
Individual, factor map (PCA)
SSD18 Variables factor map (PCA)
SSD173
Individual, factor map (PCA) Variables factor map
(PCA)
CervXLev
Individual, factor map (PCA) Variables factor map
(PCA)
Individuals factor map (PCA)SSD133 Variables
factor map (PCA)
Individuels factor map (PCA)
SSD1Oâyariablea factor map (PCA)
Dim 1 (38.83%)
21994 10 23 4.0.t
9~~ 42
2145 3
· 33 u.
I
2 0 2 4
Dim, (35 00%)
1.0 -05 0.0 0.5 1'0
Dim 1 (35.00%)
24
É
E
~0 iT843
21 22
·
·
25 50
45 27
· ~°
· 33
qqaa
1 zz y
3fi2 2
·reeïT
·9 14b2i~ 7s ~
·
· 4 353
·
·
·
·
· 34 2, 2
Al2 6 32 a§
,,p4,~~ L+tA9
5
· 41
L'ensemble des génotypes
Individual, factor map (PCA) Variables factor map
(PCA)
p\(2
tau_ Y.peram
51
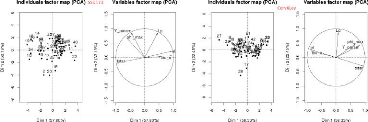
FIGURE 3.16 - Les graphes d'analyse en composantes principales
entre les huit paramètres par génotype et sur l'ensemble des
génotypes.
I l I l
4 -2 0 2 4
Dire 1 (27.87%)
1.0 -0.5 0.0 0.5 1.0
Dim 1 (27.87%)
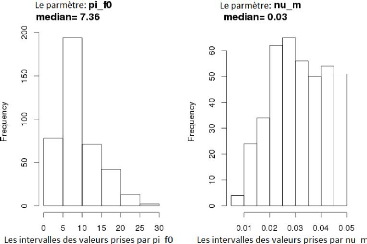
52
FIGURE 3.17 Les histogrammes obtenus pour num et pi_f0.
génération/population permettant à
l'algorithme NSGA-II de bien explorer l'espace de recherche vers le meilleur
front de Pareto possible. Après plusieurs tests, qui ont
été faits durant ce travail sur l'ensemble des génotypes,
le choix le plus judicieux qui a été défini est d'appeler
NSGA-II sur une génération de taille 250 et une population de
taille 400 par génotype. Avec ces tailles (voir la Table3.5),
on a fait 20 estimations par génotype (la Figure3.22
pour le génotype SSD45).
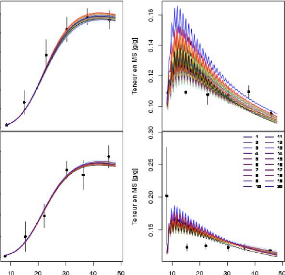
Pour faire le graphique associé à ces 20
meilleurs compromis, on a appliqué le principe de cluster (vu en
sectin2.1.1) sur les nuages de points de données
observées (MFobs, MSobs) en remplaçant chaque cluster par sa
moyenne et son écart-type (le point segment) puis on trace les 20
courbes associées aux MFpred et MSpred. Même chose pour les sous
graphiques associés aux teneurs en matière sèche (MS/MF)
en condition témoin et stress.
Sur le graphique associé à ces 20 meilleurs
compromis, on trouve que les courbes simulent presque les points-segment des
données observées de la même manière, ce qui prouve
la convergence de ces compromis vers la même zone du meilleur compromis
possible.
Au niveau des courbes des teneurs, on voit qu'elles n'ajustent
pas bien les données dans un premier temps et cela vient du fait que les
poids du fruit (MF, MS) sont très petits au début (assez
proches). Mais dès que ces poids commencent à croître, les
teneurs prédites simulent relativement bien les teneurs
observées.
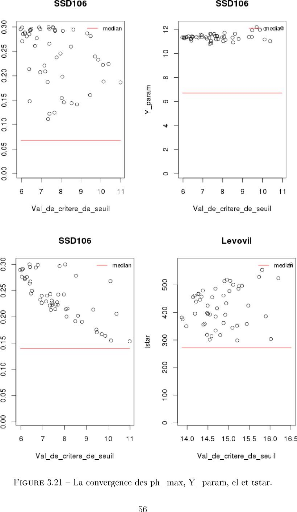
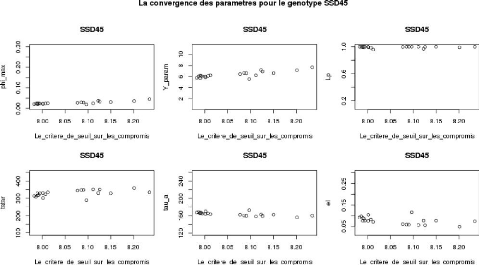
Sur chacun des sous graphiques associés à la
convergence des paramètres (toujours la Figure3.22), on
voit bien que chaque paramètre converge presque vers la même
valeur (ou disons reste restreint dans le même petit intervalle), ce qui
nous permet de
|
ol
|
Levovll
|
|
Levovll
|
|
Coral!
|
ô
|
Cervll
|
o -- median
so
o o °o 0
° ô 0
0
tô
|
° --maan
0 0
0 00
°°° ° 0
0 0 °
°o a o0
|
-- median
ô
0 0 0
0 0 0
° 0 0 ° o
0 0
0
|
oe -- median
oao 0
0 0 °
o 0
o$
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
o
|
|
8 10 12 14 18
|
8 10 12 14 18
|
11 12 13 14
|
11 12 13 14
|
|
Val de critere de seuil Val de critere de seuil
|
Val_de_critere_de_seu I I
|
Vol_de_critere_de_seu I I
|
SSD173 SSD173 CervXLev CervXLev

median
median
0 0° o 0
o° 0
8
°°
p0 0 0
00 0
° o
0 0
08 ° ZP°0 o g2/0 0
00
0 0
median
o
00 8 0
o o A
°° O
0
0
0 00ô0ô
~o
° 0
.0.4%
GO ° ° o0 0
° ° o
13 14 15 16 17 18 19
Vol_de_critare_de_seu i l
13 14 15 18 17 18 19
V al_de_critere_de_seuil
9 8 10 12 14 8 8 10 12 14
Val_de_critere_de_seu i l Vol_de_critere_de_seu i l
E o
SSD133
-- madian
oYd g~o
moo®
° 0 0
9 10 11 12 13
Val_de_critere_de_seu I I
SSD133

00 0
ô °
0 0
o °9 0 0o 0ô 0 0
o 0
0 o
o
median
9 10 11 12 13
V al_de_critere_de_sauil
SSD106
median
04 o° °
° ° o
0
10 12
Val_de_critere_de_seu I I
SSD106

median
0 °
8 10 12
Val_de_critere_de_seu il
53
FIGURE 3.18 La convergence des num et pi f0.





Levavll
Cervll
50 150 250
0 05 0 15
0 05 0 20
0.83
phi_max
phi_max
0.62
0.34
:fl
Y_param
0.70
Y_param
0.51
KIM
Lp
Lp
0.99
0.93
0.87
0.86
tatar
tatar
0.81
tau_a
tau_a
0.62
el
el
0 05 0 15
SSD45
SSD18
0.87
phi_max
0.81
phi_max
0.48
0.50
com
0.40
0 35
Y_param
Y_param
0.61
0.50
0.52
0.54
044
0.44
0.49
8 ° m8
;bEAe a Wi-rr~-rr
Lp
Lp
0.98
0.91
0.98
0.77
tatar
tatar
0.83
tau_a
tau_a
0.63
el
el
SSD173
CervXLev
150 250 950
005 0 15 025
0.81
phi_max
phi_max
0.71
054
0.43
0.40
0.43
0.40
Y_param
Y_param
0.49
0.59
0.55
0.57
v ldI
°ôd
oo 8
o. o. o oo t,o o
Lp
Lp
054
0.46
ria
0.99
0.98
0.88
0.83
tatar
tatar
0.81
tau_a
tau_a
0.72
el
el
SSD133
SSD106
100 200

0 15 0 25
0.88
phi_max
phi_max
0.44
0,42
0.49
0.40
Y_param
0.71
0.70
Y_param
0.61
0.64
052
0.47
044
Lp
Lp
0.52
0.46
â
· e
002 001 014 04 Od 01 10
0.93
0.84
0.83
0.80
tatar
tatar
tau_a
tau_a
r
el
el
200 240
120 150 200
L'ensemble des genotypes
phi_max
0.64
Y_param
0.58
0 07
!>
i
Lp
0.80
tatar
rr 711
tau_a
el
·
00 04 08
54
FIGURE 3.19 Les graphes des corrélations entre les six
paramètres par génotype et sur l'ensemble des
génotypes.
Individuals factor map (PCA) CONS Variables factor map
(PCA)
Individual. factor map (PCA) sols Variable. factor map
(PCA)
|
|
|
Dim 1 (80.78%)
|
|
Dim 1 (83.]5%)
|
|

Individuals factor map (PCA) Levovil
Variables factor map (PCA)
38 31
.43
·
24 10
·
· 512 54),
4,21
331.- 1,! -2 - 3.
·zé
·
1 ..138
48
· 4 08 .0
· ,Ar5
·
r
.25
-4 -2 0 2
Dlm 1 (48.94%)
1.0 -05 00 0.5 1.0
Dim 1 (48. 94%)
Indlvlduala factor map (PCA) 555 ° Variables factor
map (PCA)
1.0 -0.5 00 0.5 1.0
Dim 1 (5792%) Dim 1 (5792%)
1.0 -0.9 00 0.5 110
Dim 1 (58.33%)
8 -0 -2 0 2 4 Dim 1 (58.33%)
Indlvlduala factor map (PCA) 55 c^ Variables factor map
(PCA)
Individual. factor map (PCA) Variable. factor map
(PCA)
EervxLev
Dim 1 (57.80%)
Dim 1 (5].80%)
10 -5 5 00 0.5 1.8
Indlvlduala factor map (PCA) 554133
Variables factor map (PCA)
Individuals factor map (PCA) OSE100
Variable. factor map (PCA)
E ô 9
-8 -4 -2 0 2 4 Dim 1 (59.29%)
1.0 -0.5 0.0 0.s 10
Dim 1 (59.29%)

-4 -2 0 2 4
Dim 1 (52.83%)
Yyera
E
1.0 -0.s 00 5.s 10
Dim 1 (52 83%)
N
5-
L'ensemble des génotypes
Indlvlduala factor map (PCA) Variables factor map
(PCA)
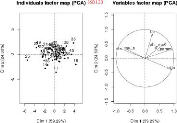
FIGURE 3.20 - Les graphes d'analyse en composantes principales
entre les six paramètres par génotype et sur l'ensemble des
génotypes.
-4 -2 0 2 4
|
Dim 1 (3829%)
|
55
|
1.0 -0.5 0.0 0.5 1.0
Dlm l (38.29%)
|
SSD106
I I I I
6 7 8 9 10 11
Val de critere de seuil Val de critere de seuil
SSD106 Levovil
a
0 D -- median
o
C}
in
C1--
6 e 00D o0
Q 00 O
N - 0 0
d 0 O
o o
0
a -
d Î I I I I I
6 7 8 9 10 11
14.0 14.5 15.0 15.5 16.0 16.5
Val de critere de seuil Val de critere de seuil
FIGURE 3.21 -- La convergence des ph max, Yparam, el et tstar.
56
1
9
6 7 8
SSD106
Q
median
·
0o
o
CV
Q
a
{V --
m 0
D 00
0 p0
Q
0 0 00
o
00
a
d
o
a
d
114-7
d
LU
a
0
d
edia0
Vie: Opt
0 O OD
ç
·
2 POP
a,
cv
0
CO -
a --
06, 0
o
c
o
o
o
0 a -
mediar
o
0
m 0 0 0
· 0 ki0
o 0 co
o
CV
a
a -
0 a -
57
|
Les paramètres de NSGA-II
|
Les valeurs des paramètres de NSGA-II
|
|
idim
|
6
|
|
odim
|
2
|
|
generations
|
250
|
|
popsize
|
400
|
|
cprob
|
0.9
|
|
cdist
|
20
|
|
mprob
|
0.1
|
|
mdist
|
20
|
TABLE 3.5 Tableau des valeurs de paramètres de NSGA-II
pour les résultats finaux.
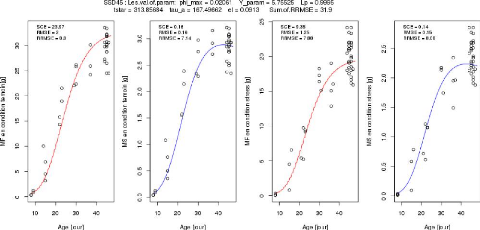
sélectionner le meilleur de ces 20 compromis ayant la
meilleure combinaison possible de paramètres, c'est-à-dire qui
permet d'avoir le meilleur ajustement parallèle
(Figure3.23).
Pour résumer, on appliquera le même traitement
(20 estimations par génotype, puis la sélection du meilleur des
20 meilleurs) sur l'ensemble des génotypes, qui seront traités
dans le grand document.
Remarque 3.3. Sur les résultats
finaux de 37 génotypes qui ont été déjà
traités, on a refait encore l'analyse de la variabilité des
paramètres et la Figure3.25 confirme
l'invalidité des corrélations trouvées dans la
Figure3.24. Ce qui nous confirme encore ce qui a
été trouvé en sectin3.3 pour ces
corrélations.
Le graphique associe aux 20 estimations pour le genotype
SSD45
10 20 30 40 50
10 20 30 40
50
O
o
N
O
Teneur en MS [gig]
O
o
o O
If1
0 N
o
Teneur en MS [gr'g]
o
|
|
|
|
|
|
|
|
|
|
|
|
|
o
N
LL
· -
0
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
u-
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
O
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
I I I I
|
|
|
|
10 20 30 40 50
|
o ri
N
o
_ N co 0
T2 r
o
o
O
o
N
o
N
~
· r
o
o
o
o
aari D 6 O O 4
La convergence des pa ra metres pour le genotype
SSD45
o
o
O
o
E m
N -
0% ô°%
o
0 N
o
o
I
E
E
SSD45
SSD45
6.00 8.05 8.10 8.15 8.20
Le_crite re_de_se u il_su r_les_co m pro m s
SSD45
D
o
o -
6.00 8.05 2.10 8.15 8.20
Le_crite re_de_se u il_su r_les_co m pro m s
O
N -
8.00 6.05 6.10 6.15 8.20
Le_crite re_de_se u il_su r_les_ca m prom s
8.00 6.05 6.10 6.15 8.20
Le_crite re_de_se u il_su r_les_ca m prom s
SSD45
N
O
o
40 0on 0
pdD 0
o
ow O 4 O 0 o
cktEtop
SSD45
8.00 2.05 8.10 8.15 8.20
Le_crite re_de_se u il_su r_les_ca m prom s
SSD45
8.00 8.05 8.10 8.15 8.20
.131
Le_crite re_de_se u il_su r_les_ca m prom s
O
6
N -
0
0
0 -
o
m
0 4 O a
m
0
N _ ô
58
FIGURE 3.22 Les 20 meilleurs compromis pour SSD45.
00
4 d
SCE-L35RMSE-125 RRMSE-71!
55045 : Les.val.of.param: phi_rrax = 0.02061 Yjram =
5.75525 Lp = 0.9995
tstar = 313.85684 tau a = 16749662 el = 0.0913 Sum.of.RRMSE =
31.9
10 20 30 40
Age [our]
10 20 30 40 10 20 30 40
Auge []our] Age Dur]
MF en condition temoin[g]
MS en condition temoin [g]
MF en condition stress [g]
Age [pur]
MS en condition stress [g]
10 20 30 40
D
u_
u_ en-
teneur simule
Le genotype 991345 en condition temoin
Le genotype SSD45 en condition stress
teneur simule
tn
o _
a I I I I
59
10 20 30 40 50 60 10 20 30 40 50 60
Temps [jour] Temps [jour]
FIGURE 3.23 Le meilleur des 20 meilleurs compromis pour SSD45.
Sur 37 genotypes

phi_max
0 4 e
I I L
0.62
Y_param
0.00 0.15 0.30
0.89
_o
0.0 0.4 0.8
150 250 350
tstar
0.27
el
0.00 0.15 0.30
I
0.46
0.49
O
O
0 200 400
11 I I I 1
0.37
0.47
Lp
5 15
aiG
Individuals factor map (PCA) Variables factor map
(PCA)
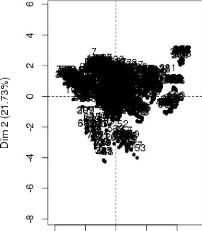
I I I I
Dim 2 (21.73%)
0
cv
00 --
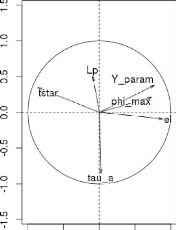
L(
rn
L(.
Lp
Y_param
tstar
ph.iimax
ta __a
I I
-4 -2 Cl 2 4 -1.0 -0.5 0.0 0.5 1.0
OH 1 (44,47%) Dim 1 (44.47%)
60
FIGURE 3.24 -- Les graphes des corrélations et
d'analyse en composantes principales entre les six paramètres sur 37
génotypes.
61
SSD1O6
|
|
SSD1O6
|
|
|
|
|
-
ô
u,
N --
O O
N --
O
ce
E _
ç O
Q
O O
u,
4 --O
O
O 0
|
medi
|
E
a
Q
}
|
0
03
m
N --
O -
|
(=MO ® n media
|
|
|
|
I I I 1 I I
|
|
5.94 5.98 6.02
|
5.94 5.98 6.02
|
|
Val de critere de seuil
|
Val de critere de seuil
|
|
SSD1O6
|
Levovil
|
|
0 en --ô
u,
|
|
|
0
O --Ln
|
|
q 0 median
q 0 c7
|
median
|
|
N --
|
|
|
|
|
|
O
|
|
|
|
|
|
|
|
O -
|
D
|
|
0
|
|
|
|
0
|
|
--
O
|
|
|
|
|
|
|
|
O
|
|
|
|
|
O -
|
|
|
Ln
|
|
ce
|
|
|
|
O
|
|
.N.
|
|
|
|
|
|
O
|
|
|
O
|
|
|
O --
N
|
|
|
4
u,
O --
|
|
|
O
O -
|
|
|
O
|
|
|
|
|
|
4
|
|
|
|
|
|
O -
|
|
|
O -
|
|
|
O
|
|
|
|
I I I I I
|
5.94 5.98 6.02 13.80 13.90 14.00
Val de critere de seuil Val de critere de seuil
FIGURE 3.25 -- La convergence des ph max, Yparam, el et tstar
dans les résultats finaux.
62
| 


