II.2) Transformation par éléctroporation
de la souche KM71H de Pichia pastoris :
Les levures KM71H ont été
sensibilisées par le sorbitol pour subir une
éléctroporation en présence du plasmide recombinant
linéarisé.
L'insertion du gène d'intérêt dans le
génome de Pichia
pastoris se fait par recombinaison homologue.
Après étalement sur le milieu de culture
YPDS+Zéocine les boites sont incubées dans l'étuve
à 30°C pendant trois à quatre jours.
Les clones qui poussent sur zéocine sont
utilisés pour la production de la protéine recombinante.
II.3) Production de la protéine recombinante
CFP10 :
Le gène Rv3874 est cloné en aval d'une
séquence dite á factor qui code pour un peptide signal servant
à la sécrétion de la protéine recombinante dans le
milieu extracellulaire.
Les surnageants de culture de la production, après une
induction avec le méthanol 1% pendant 24, 48 et 72 heures, sont
analysés sur gel de polyacrylamide SDS-PAGE 15% dans des conditions
dénaturantes en présence de â2
mercaptoéthanol. Ce mélange subit également une
dénaturation à la chaleur, 100°C pendant 5 minutes.
L'analyse des deux premières inductions ne montre rien
par contre on observe une bande concernant l'analyse de la troisième
induction (72 heures).
1
15kDa
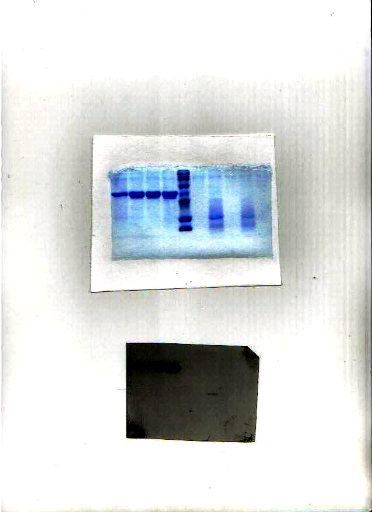
Figure9 : Analyse sur SDS-PAGE 15% du surnageant
de culture après 72 heurs de la
production de CFP10.
1 : Marqueur de poids moléculaire RPN 800.
2 : Protéine recombinante CFP10.
Le poids moléculaire théorique de la
protéine CFP10 est de 10 KDa. En ajoutant le poids moléculaire de
myc épitope et de la queue histidine plus les modifications post
traductionnelles réalisées par Pichia pastoris on
s'attend à une taille plus grande ce qui explique que la bande au niveau
de la figure 9 est à peu prés de 15 KDa.
II.4) Identification de la protéine recombinante
par immunoblot blot :
A fin de confirmer l'identité de la
protéine révélée au niveau du gel (fig 9) nous
avons réalisé la méthode de western blot en utilisant
l'anti myc HRP, un anticorps couplé à la peroxydase qui
reconnaît spécifiquement l'épitope myc
CFP 10
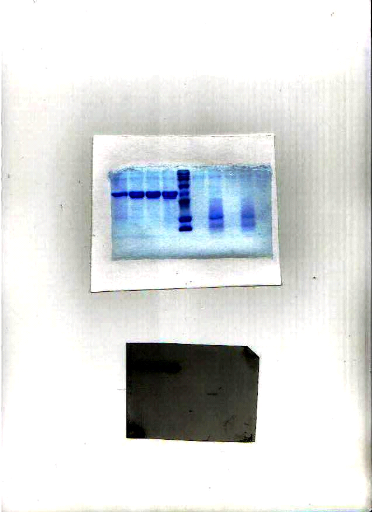
Figure 10 : Révélation par ECL du
wester blot réalisé sur la protéine CFP10 en utilisant
l'anti myc HRP.
La durée d'exposition de film en autoradiographie = 5
minutes.
L'anticorps Anti-myc reconnaît de façon
spécifique l'épitope myc. Cette reconnaissance montre que la
protéine produite contient bien l'epitope myc.
La tuberculose constitue un réel problème
de santé publique à l'échelle mondiale.
Les recherches ne cessent de progresser par l'approche
moléculaire de la tuberculose ce qui peut aider à
l'amélioration du traitement et de la prévention de cette
maladie.
Le séquençage total, du génome de M.
tuberculosis H37Rv en 1998 a constitué un tournant qui ouvre
plusieurs voies de recherche et permis d'identifier de nombreux gènes
dont les fonctions reste mal connues.
Pour découvrir le rôle de ces gènes les
chercheurs doivent passer par les étapes de clonage et d'expression
afin de convertir l'information génétique en une protéine
biologiquement active
à partir de laquelle on peut appliquer plusieurs tests
afin de dégager les caractéristiques de la protéine et
donc savoir l'intérêt du gène en question.
Au cours de ce travail nous avons réussi à
cloner le gène Rv3874, qui code pour la protéine CFP10, dans le
vecteur pPICZáA destiné à la transformation de la levure
Pichia pastoris qui est un excellent système de production des
protéines hétérologues vu la croissance rapide de la
levure qui se maintient en fermenteur et qui pousse sur des milieux simples.
Par ailleurs, il s'agit d'un système eucaryote qui réalise,
normalement, les modifications post- traductionnelles. Pour ce qui est du
clonage de gène Rv3874 les résultats on étés
confirmés par colonie PCR, par carte de restriction et par
séquençage automatique.
Nous avons choisi de produire la protéine recombinante
sous forme sécrétée soluble dans le surnageant de culture
ce qui facilite sa purification puisque, la levure Pichia pastoris
sécrète de très faibles quantités de
protéines autres que la protéine d'intérêt.
La protéine CFP 10 a été produite dans un
premier temps dans E-Coli. Ce systéme de production
présente des limitations puisque la bactérie est incapable de
réaliser les modifications post traductionnelles qui peuvent être
importantes pour l'activité biologique de la protéine.
De plus E-Coli, sécrète très mal
les protéines et si la protéine recombinante est exprimée
dans le cytoplasme il faut casser la membrane plasmique pour y accéder,
ce qui nécessite une purification très poussée.
On peut signaler aussi q'une autre protéine appartenant
au complexe de M. tuberculosis qui est la CFP32 et qui était le
sujet d'un DEA d'un étudiant au laboratoire a été produite
avec succès dans la levure Pichia pastoris avec un rendement
appréciable, de l'ordre de 0,5g/l. (5).
Donc, au cours de ce travail nous avons montré que
Pichia pastoris peut être utilisée comme système de
production des protéines de Mycobactérium
tuberculosis.
Nous avons réussi à produire sous forme
recombinante la protéine CFP10-myc-6his et nous avons identifié
la protéine recombinante par western-blot en utilisant l'anticorps
marqué anti-myc HRP.
Finalement on peut enrichir ce travail en réalisant la
purification de cette protéine et également développer un
test ELISA qui sera utilisé pour la recherche d'anticorps anti-CFP10
dans le sérum des patients tuberculeux.
Références bibliographiques
B
1. Behr, M. A., and Small, P.M. (1999). A historical and
molecular phylogeny of BCG strains. Vaccine, 17, pp: 915-922.
2. Betton J. M . (2005). Repliement et production de
protéines recombinantes. Médecine sciences Vol 21 : 6-7.
3. Brosch, R ; Durieux, P ; Gauselus, J ;
Guérin, N ; Infuso, A ; Jarlier,V ; Marchal, G ;
Zylberman, P. (2004). La place de la vaccination dans le contrôle de la
tuberculose en France ; expertise collective. Vol 2 : 25-42.
4. Benabdesselem, C. (2003) Clonage et expression dans
Pichia pastoris, et caractérisation d'une forme recombinante de
la protéine CFP32 spécifique du complexe mycobactérium
mtuberculosis ; Mastère de biotechnologie et immunologie
appliquées aux maladies trasmissibles : Faculté de
pharmacie-Monastir ; Tunisie.
C
5. CereghinoJ.L., Cregg J.M. (2000) Heterologous protein
expression in the methylotrophic yeast Pichia pastoris. FEMS MICROBIOLOGY
REVIEWS 24: 45-66.
6. Cole, S. T., R. Brosch, J. Parkhill, T. Garnier, C.
Churcher, D. Harris and B. G. Barrel. (1998). Deciphering the biology of
mycobactérium mtuberculosis from the complete genome sequence.
Nature, 393, pp: 537-544.
7. Cregg, J, M., Barringer, K. J., Hessler, A. Y. and Madden,
K, R (1985). Pichia pastoris as a hot system for transformation. Mol.
Cell. Biol; 5:3376-3385.
8. Cregg, J.M., Vedvick, T.S., and Raschke, W.C. (1993) Recent
advances in the expression of foreing genes in Pichia pastoris. Biotechnology
11: 905-910.
D
9. Daly, R. and Hearn, T.W.M (2005) Expression of heterologous
proteins in Pichia pastoris: a useful experimental tool in protein
engineering and production. J. Mol. Recognit. 18: 119-138.
G
10. Grosset J. place des examens microbiologiques et
anatomopathologiques dans la décision diagnostique et
thérapeutique. Med Mal Infect 1995 ; 25 : 327-333.
H
11. Hu, Y., Meng, X.L., Xu, J.P., Lu, W., Wang,J. (2005)
Cloning and expression of earthworm fibrinolytic enzyme PM246 in
Pichia pastoris. Prot. Exp. Purif. 43 pp 18-25.
I
12. Issa, I. (2004). Production par un nouveaux vecteur
navette de protéine de fusion avec la GST dans la levure Pichia
pastoris: cas de l'interféron á. -Tunis :
Faculté des sciences, -DEA en microbiologie.
13. Issar, S. (2003). Mycobactrium tuberculosis pathogenesis
and molecular determinants of virulence. Clinical Microbiology Reviews vol.16,
No3, pp: 464.
L
14. Lipsitch, M. (2001). The rise and fall of antimicrobial
resistance. Microbiology, 148, pp:2967-2973.
M
15. Montgomery, S. A. (2004). Transgenic Animals: Walking
Bioreactors. BioProcess INTERNATIONAL Chap 4: 40-50.
R
16. Rosin, L. J. (2004). Transgenic plants: biofarming of the
future. BioProcess INTERNATIONAL Chap5: 52-61.
S
17. Scott, C. (2004)a. Microbial fermentation: the oldest form
of Biotechnology. BioProcess INTERNATIONAL Chap1: 8-20.
18. Scott, C. (2004)b. Animal cell culture: High maintenance
but Worth the trouble. BioProcess INTERNATIONAL Chap2: 22-32.
19. Sue Marcauley-Patrick., Mariana L. Fazenda., Brian McNeil
& Linda M. Harvey. (2005). Heterologous protein production using the Pichia
pastoris expression system.Wiley Inter Science Review. Yeast. 22: 249-270.
T
20. Tschopp, J. F., Sver low, G., Kosson, R., Croig, W and
Grinna, L. (1997). High level secretion of glycosylated invertase in the
methylotrophic yeast, Pichia pastoris Bio/Technology; 5: 1305-1308.
V
21. Van Crevel., R., Ottenhoff, T.M and Van Der Meer, J.M.
(2002). Innate Immunity to Mycobacterium tuberculosis. Clinical Microbiology
Reviews, 15 N° 2, pp: 294-309.
W
22. Wegner, G.H; Harder, W. 1986. Methylotrophic
yeast-microbial growth on C1 compounds. In: Van Versveld, H.W.and
Duine, J.A.,(Eds), Proceedings of the 5th International symposium.
Martinus Nijhoff, Derdecht, pp: 139-149.
| 


