CHAPITRE 4. MÉTHODOLOGIE ET
IMPLÉMENTATION
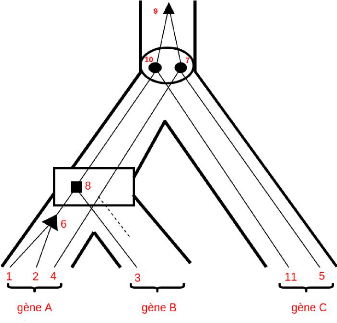
FIGURE 4.6 - Extrait d'arbre de protéines
qui respectent nos contraintes.
Les principales méthodes de regroupement de
protéines présentes dans la littérature sont
décrites à la figure 4.7. Elles sont basées sur les
méthodes par partition-nement [23], les méthodes
hiérarchiques [7] et les méthodes utilisant des graphes [30].
Toutes ces méthodes approchent le problème posé en
4.2.4.2, mais présentent des limites majeures qui nous ont conduits
à développer notre propre méthode de regroupement.
D'abord, les méthodes de regroupement
hiérarchique, bien qu'étant les plus utilisées en
bio-informatique du fait de leur complexité quadratique, conduisent
automatiquement à des regroupements sans chevauchement. Ensuite, les
approches par partitionnement quant à elles sont très efficaces,
car chaque élément à classer est mis dans la meilleure
classe préexistante. Mais leur principale limite est qu'elles exigent
élément puisse appartenir à plus d'un
groupe, par opposition au regroupement sans chevauchement qui ne le permet
pas
35
CHAPITRE 4. MÉTHODOLOGIE ET
IMPLÉMENTATION
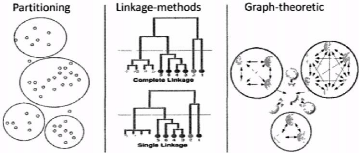
FIGURE 4.7 - Les trois principales catégories
d'algorithmes utilisées pour le regroupement des séquences de
protéines
de connaître au préalable le nombre de groupes,
alors que dans notre cas ce nombre doit être découvert. Enfin, les
approches basées sur la théorie de graphe conduisent
également à des regroupements sans chevauchement. Bien qu'il
existe des cas rares où l'on peut obtenir des regroupements avec
chevauchement.
4.2.4.3 Regroupement avec chevauchement
hiérarchique
Idée de base. L'idée de base de
notre méthode de regroupement avec chevauchement est de proposer une
version modifiée du regroupement hiérarchique (qui est à
la base du regroupement sans chevauchement) pour créer des regroupements
avec chevauchement. Tel que chaque groupe contient au plus une protéine
par gène tout en permettant qu'une protéine puisse appartenir
à plus d'un groupe. La figure 4.8 illustre graphiquement la
différence majeure entre le regroupement sans chevauchement et le
regroupement avec chevauchement que nous choisissons comme approche.
Présentation globale du modèle
Soit n le nombre de protéines et á un
seuil de similarité. Initialement, chaque protéine forme un
groupe. La méthode va, de manière récursive, choisir
à chaque étape les deux groupes les plus proches, les fusionner
afin d'en former un nouveau. On choisit deux groupes à fusionner si et
seulement si le score de similarité entre eux est inférieur au
seuil á. Le score de similarité pour deux groupes A
et B est calculé ainsi comme suit (D étant
la matrice de similarité calculé à l'étape 3) :
A' ? A - B
36
| 


