2) Prédiction du peptide signal de la
séquence PRODH humaine
Nous avons montré que la production de la
protéine PRODH humaine chez E. coli
conduit à l'unique formation de corps d'inclusion. La
stratégie d'étude structurale de PRODH
qui a été initialement définie repose sur
une recherche de domaines structurés à partir de la
protéine entière repliée. Cette protéine de 70
kDa n'étant pas exprimée sous forme soluble chez E.
coli, nous avons été contraint de modifier notre
stratégie de départ. Nous avons ainsi entrepris d'identifier et
de produire la forme mature de cette protéine humaine mitochondriale.
2.1) Les messages d'adressage mitochondrial
La proline déshydrogénase humaine est une
protéine mitochondriale encodée par un gène porté
par de l'ADN nucléaire. Comme toutes les protéines
mitochondriales synthétisées
au niveau du ribosome du cytoplasme, elle possède dans sa
séquence primaire l'information
qui lui permet de rejoindre et d'intégrer la
mitochondrie (pour revue : Pfanner & Neupert,
1990). Cette information, appelée message
d'adressage ou peptide signal, est toujours localisée dans
l'extrémité N-terminale de la séquence. Ces motifs sont au
nombre de 1 ou 2 dans la séquence primaire en fonction de la
localisation précise de la protéine dans la mitochondrie.
Ils n'intègrent pas la forme mature de la protéine et sont
clivés après leur entrée dans la mitochondrie. D'un
point de vue structural, les peptides signaux sont des hélices
amphipatiques de longueur variable qui comportent, une
région N-terminale chargée positivement, une région
centrale très hydrophobe, et une région C-terminale
plutôt neutre
(Nielsen et al., 1997). Ils se caractérisent
également par la présence dans leur extrémité C-
terminale d'un résidu neutre de type alanine en
position -3 et -1 du site de clivage. La présence de tels
motifs à caractère hydrophobe dans une région qui
n'appartient pas à la protéine mature peut favoriser
la formation de corps d'inclusion dans le cas d'une
surexpression chez E. coli. L'identification de ces peptides signaux
s'avère donc utile dans l'optique d'améliorer les profils
d'expression de PRODH recombinante.
2.2) Résultats de la prédiction
Plusieurs programmes bio-informatiques disponibles sur le
web ont été récemment développés avec
pour objectif la prédiction de peptides signaux à partir
d'une séquence primaire. A ce jour, il n'existe pas de
séquence consensus de peptide signal d'adressage mitochondrial.
Aussi, ces programmes utilisent les caractéristiques topologiques de ces
motifs (citées ci-dessus) pour prédire leur présence
dans une séquence. Nous avons soumis la séquence de PRODH
humaine à plusieurs de ces programmes. Les résultats
obtenus sont reportés dans le tableau suivant :
|
Programme
|
Longueur du peptide signal N-terminal
|
|
Predotar (Small et al., 2004)
|
non détecté
|
|
iPSORT (Bannai et al., 2002)
|
30
|
|
MitoProt II 1.0 (Claros & Vincens, 1996)
|
49
|
|
PSORT II (Nakai & Horton, 1999)
|
57
|
|
TargetP V1.0 (Emanuelsson et al., 2000)
|
114
|
|
SignalP V1.0 (Nielsen et al., 1997)
|
35 et 24
|
Tableau 2.1 : Bilan de l'analyse de
la séquence primaire de PRODH humaine par 6 logiciels
de prédiction de peptide signal d'adressage
mitochondrial.
De manière inattendue, les prédictions obtenues
sont très différentes d'un programme
à l'autre. Ainsi, la présence d'un message
d'adressage mitochondrial est bien prédite chez PRODH par la
majorité des logiciels, mais la longueur du peptide signal
varie de manière significative selon le programme de 30 à 114
résidus. La figure 2.2 présente le diagramme de prédiction
obtenu avec le logiciel SignalP qui est présenté comme
l'algorithme le plus fiable d'après une récente étude
comparative (Emanuelsson et al., 2001). Ce diagramme suggère la
présence de 2 peptides signaux dans la région N-terminale de
PRODH, qui pourraient peut- être expliquer la divergence des
résultats obtenus avec les autres programmes. Le premier est
prédit au niveau des 35 premiers résidus et le
second serait situé entre les résidus 65 et 88.
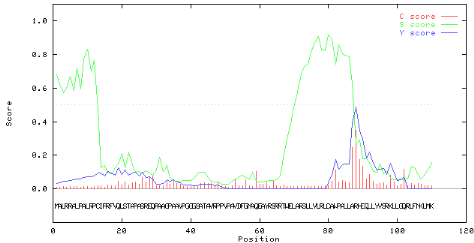
Figure 2.2 : Diagramme de
prédiction de peptide signal de PRODH par le logiciel SignalP.
La prédiction de l'existence de 2 peptides
signaux dans l'extrémité N-terminale de PRODH humaine est
surprenante dans la mesure où les protéines mitochondriales
destinées à intégrer la matrice ne contiennent qu'un seul
de ces motifs (Pfanner & Neupert, 1990). Nous avons soumis la
séquence primaire de PRODH humaine au logiciel HMMTOP (Tusnady &
Simon, 1998) qui permet de prédire les hélices transmembranaires.
De manière intéressante, l'algorithme HMMTOP suggère la
présence d'une hélice transmembranaire unique entre les
résidus 70 et 87, c'est-à-dire au niveau du second
peptide signal potentiel. Dans leur étude comparative, Emanuelsson
et al., ont mis en évidence la grande difficulté des
programmes de prédiction de peptide signal à distinguer les
motifs transmembranaires des hélices d'adressage mitochondrial
(Emanuelsson et al., 2001). La prédiction d'un second peptide signal
potentiel n'est donc pas surprenante et correspond plus vraisemblablement
à un segment hydrophobe.
Au final, nous avons utilisé une partie des
résultats de SignalP pour estimer la longueur du peptide signal
N-terminal de PRODH à 35 résidus. La longueur moyenne
d'un signal d'adressage mitochondrial étant de 23 résidus, cette
prédiction semble tout à fait réaliste.
L'analyse de la séquence de PRODH humaine par des outils
bio-informatiques a donc permis de prédire la présence d'un
peptide d'adressage mitochondrial dans l'extrémité
N-terminale 1-35 de la protéine. Ce motif
à caractère hydrophobe n'incluant pas la forme mature de
la protéine, nous avons par conséquent entrepris
de réaliser une nouvelle construction PRODH délestée
de ce peptide signal. Cette forme mature est nommée PRO564
et correspond à la région 37-600 de PRODH humaine
(66 kDa).
| 


