2) La protéine KIN17
Le Laboratoire de Génétique de la
Radiosensibilité (LGR) du CEA de Fontenay-aux- Roses s'intéresse
aux effets biologiques produits par les stress génotoxiques sur les
cellules, et notamment à la réponse des cellules de
mammifères aux radiations ionisantes (RI). En 1991,
les chercheurs du LGR ont découvert une nouvelle
protéine nucléaire de 45 kDa appelée KIN17 (Angulo
et al., 1991). Les fonctions précises et les partenaires
protéiques de cette protéine, uniquement présente
chez les organismes eucaryotes et exprimée de manière
ubiquitaire, sont à ce jour inconnues. Cependant, un certain
nombre d'études génétiques in vitro et
in vivo ont permis de caractériser l'implication de
cette protéine dans différents mécanismes
nucléaires majeurs, et notamment la réplication et la
réponse cellulaire aux dommages de l'ADN.
2.1) Les propriétés de la protéine
KIN17
La protéine KIN17 a été initialement
identifiée sur la base de sa capacité à réagir
avec
les mêmes anticorps que ceux dirigés contre RecA,
une protéine bactérienne impliquée à la fois dans
la réparation et la réplication de l'ADN (Angulo et al.,
1991). KIN17 possède effectivement dans sa séquence une
région d'environ 40 résidus qui présente 47 %
d'identité avec un segment situé en C-terminal de RecA.
De manière intéressante, dans RecA, cette région est
impliquée dans la régulation de la liaison de la
protéine à l'ADN et dans le mécanisme SOS, une des
réponses aux lésions de l'ADN chez les organismes
procaryotes (Kurumizaka et al., 1996).
2.1.1) Implication de KIN17 dans la
réplication et la réponse aux dommages
de l'ADN
Le gène humain encodant KIN17, situé sur
le chromosome 10, fait partie de l'ensemble des gènes de
l'organisme très faiblement exprimés. Cependant, les
cellules en division rapide contiennent 3 fois plus d'ARNm KIN17 que les
cellules au repos (Kannouche
et al., 1998). De plus, la protéine KIN17 est
distribuée dans les noyaux des cellules de mammifères
sous forme de structures discrètes appelées «
foyers intra-nucléaires » (Kannouche et al., 1997). Ce type de
foyers reflète la compartimentation de l'ADN lors des processus
complexes comme la réplication, la réparation, la
transcription, ou l'épissage. Toutes ces observations suggèrent
fortement que KIN17 appartient à un réseau de protéines
intranucléaires requises lors de la prolifération cellulaire.
L'irradiation par des rayons UV ou ã et des
radiations ionisantes provoquent une augmentation significative de la
concentration d'ARNm KIN17 dans les 13 heures qui suivent l'irradiation de
cellules de souris en culture (Kannouche et al., 2000 ; Biard et
al., 2002). Cette augmentation s'accompagne d'une accumulation de la
protéine dans le noyau des cellules. KIN17 est donc
régulée de manière positive suite à une
exposition à des sources rayonnantes qui induisent des dommages de
l'ADN. De manière intéressante, cette régulation positive
après irradiation par des UV-C dépend de la
présence des protéines XPA et XPC (Masson et al., 2003). Ces
deux protéines sont impliquées dans les mécanismes de
réparation
de l'ADN par excision de nucléotide (NER) (Wakasugi
& Sancar, 1999). Sur la base de ces observations, il apparaît donc
que la protéine KIN17 est impliquée dans la
réponse aux dommages cellulaires de l'ADN.
Par ailleurs, des études de gel filtration,
réalisées sur un extrait cellulaire humain de protéine
totale, ont révélé la présence de KIN17 dans 3
complexes multi-protéiques de très haute masse
moléculaire (respectivement : 400 kDa, 600 kDa, et 1800 kDa)
contenant la protéine de réplication RPA (Miccoli et al.,
2005). De plus, une interaction physique entre KIN17 et l'antigène
T du virus SV40 a été démontrée (Miccoli et al.,
2002). Ces observations confortent l'hypothèse de l'implication de KIN17
dans la réplication de l'ADN.
2.1.2) Liaison de KIN17 aux acides
nucléiques
En 1994, Mazin et al., ont mis en
évidence la capacité de KIN17 à lier l'ADN et
l'ADN courbe in vivo et in vitro chez l'homme et la souris
(Mazin et al., 1994). Ainsi, une fraction de KIN17 est fortement et directement
associée avec l'ADN chromosomique dans les cellules humaines (Biard et
al., 2002). L'ADN courbe est une forme de l'ADN, riche en bases adénine,
retrouvée dans les sites de recombinaison illégitime des cellules
de mammifère. Chez
les organismes procaryotes, l'ADN courbe occupe
une fonction importante dans la transcription des gènes
(Nishikawa et al., 2003). L'affinité de KIN17 pour cette forme
de l'ADN a été confirmée in vivo en surexprimant
la protéine KIN17 de souris chez la bactérie
E. coli (Timchenko et al., 1996). Dans cette
étude, il est montré que KIN17 est capable de complémenter
les fonctions du facteur de transcription H-NS, qui lie l'ADN courbe
et contrôle l'expression d'au moins 36 gènes.
Par ailleurs, une étude protéomique à grande
échelle a mis en évidence la présence de
KIN17 dans le spliceosome humain, un large complexe
protéine-ARN (Rappsilber et al.,
2002). L'interaction de KIN17 avec l'ARN a été
récemment caractérisée par Pinon-Lataillade
et al., qui ont montré d'une part, que la
protéine KIN17 de souris était capable de fixer l'ARN
in vivo, et d'autre part, que les
protéines humaines et de souris reconnaissent de manière
directe différents types d'homopolymères d'ARN in vitro
(Pinon-Lataillade et al., 2004).
2.2) Organisation des domaines structuraux de KIN17
Depuis sa découverte en 1991, parallèlement aux
études génétiques, la protéine KIN17
a fait l'objet d'analyses bio-informatiques qui ont
permis de caractériser une organisation segmentée en
domaines structuraux (Tissier et al., 1995 ; Pinon-Lataillade et al.,
2004 ; Ponting, 2002). Sur la base de ces analyses, les chercheurs du LGR ont
caractérisé quelques propriétés de ces domaines qui
sont présentées dans ce paragraphe.
La protéine KIN17 humaine est un polypeptide de
393 acides aminés. Comme le montre l'alignement de la Figure
1.2, KIN17 est remarquablement conservée de la levure
jusqu'à l'homme dans la moitié N-terminale correspondant aux
résidus 1-165 de la séquence humaine. Dans cette
région, les pourcentages d'identité et de
similarité atteignent respectivement 14 % et 22 %. Cependant, les
séquences de KIN17 ne s'alignent pas dans la
moitié C-terminale. En effet, la séquence de KIN17
ne compte qu'entre 250 et 300 résidus
chez les eucaryotes inférieurs telle que la levure,
alors qu'elle est proche de 400 résidus chez tous les eucaryotes
supérieurs. Ceci laisse supposer l'existence d'un domaine
structural supplémentaire chez les eucaryotes supérieurs.
Human
1-MGK-SDFLTPKAIANRIKSKGLQKLRWYCQMCQKQCRDENGFKCHCMSESHQRQLLLASENPQQFMDYFSEEFRNDFLEL
Mouse
MGK-SDFLSPKAIANRIKSKGLQKLRWYCQMCQKQCRDENGFKCHCMSESHQRQLLLASENPQQFMDYFSEEFRNDFLEL
CAEEL
MGK-HEKGSSKDLANRTKSKGLQKLKFFCQMCQKQCRDANGFKCHLTSEAHQRQLLLFAENSNSYLRQFSNDFEKNFMQL
Drosophila
MGR-AEVGTPKYLANKMKSKGLQKLRWYCQMCEKQCRDENGFKCHTMSESHQRQLLLFADNPGKFLHSFSKEFSDGYMEL
Arabidopsis
MGK-NDFLTPKAIANRIKAKGLQKLRWYCQMCQKQCRDENGFKCHCMSESHQRQMQVFGQNPTRVVDGYSEEFEQTFLDL
POMBE
MGR-AEAGTPKAISNALKSKGLQRLRWYCSACQKQMRDENGFKCHTQSEGHIRQMNVIAMNPGKRIQDFSNQFLRDFISL
Cerevisiae
---MADYDSAKYWSKQGARRGLQKTRYYCQICQRQCKDANGFQSHNKSPSHLRKISQVTAEDAR---RYNIQFEKGFLQL
: : :** : :::*. *::. :* **:: * : .* :. * ::
Human
80-LRRRFGTKRVHNNIVYNEYISHREHIHMNATQWETLTDFTKWLGREGLCKVDETP-KGWYIQYIDRDPETIRRQLELEKK
Mouse
LRRRFGTKRVHNNIVYNEYISHREHIHMNATQWETLTDFTKWLGREGLCKVDETP-KGWYIQYIDRDPETIRRQLELEKK
CAEEL
LRTSYGTKRVRANEVYNAFIKDKGHVHMNSTVWHSLTGFVQYLGSSGKCKIDEGD-KGWYIAYIDQ--EALIRKEEDQRK
Drosophila
LRRRFGTKRTSANKIYQEYIAHKEHIHMNATRWLTLSDYVKWLGRTGQVIADETE-KGWFVTYIDRSPEAMERQAKADRK
Arabidopsis
MRRSHRFSRIAATVVYNEYINDRHHVHMNSTEWATLTEFIKHLGKTGKCKVEETP-KGWFITYIDRDSETLFKERLKNKR
POMBE
LRTAHGEKKIHFNQFYQEYIRDKNHVHMNATRWHTLSEFCKFLGRQGMCRVEENE-KGFFISYIDKNPANILRNEANKKR
Cerevisiae
LKQRHGEKWIDANKVYNEYVQDRDHVHMNATMHRSLTQFVRYLGRAGKVDVDMDI---------DDTSENVEGPLLIRIH
: . . .* : : * *:*:* :*: : * :
Human
159-KKQDLDDEEKTAKFIEEQVRRGLEG-KE------------------QEVPTFTELSREN------------DEEKVTFNL
Mouse
KKQDLDDEEKTAKFIEEQVRRGLEG-KE------------------QETPVFTELSREN------------EEEKVTFNL
CAEEL
QQQEKDDEERHMQIMDGMVQRGKEL-AGD----------------DEHEYEATELIRDT------------PDQKIQLDL
Drosophila
EKMEKDDEERMADFIEQQIKNAKAKDGEE----------------DEGQEKFTELKRE-------------ENEPLKLDI
Arabidopsis
VKSDLAEEEKQEREIQRQIERAAEKLNGG-----------GGEGETSGNDEVVDDGDDERKKDEDLR--LKSGVKVGFAL
POMBE
ERQEKSDEEQRLRLLDEQIKRA----------------------------------------------------------
Cerevisiae
PSSLSSPSE------DGMLRSQ----------------------------------------------------------
: :
Human
208-SKGACSSSGA----TSSKSSTLGPSALKTIGSS--------------------------ASVKRKESSQSSTQSKEKKKK
Mouse
NKGAGGSAGA----TTSKSSSLGPSALKLLGSA--------------------------ASGKRKESSQSSAQP--AKKK
CAEEL
NLGILDRKLD----VKSGVASAKISIFDMPKVKKEDPDEPG------------PSQPSRKSGKKRSRSRSPAAK--KFSK
Drosophila
RLEKKFQPDT----VLGKSALAKRPAPEAEE---------------------------KVFKKPKSVAGDSQTR------
Arabidopsis
GGGVKQVATGK---ERGESSKLLFGDEENDKVERGEK-------------------------RKRSGDSGRSEK----ER
POMBE
---------------YESAQNNEDNKDGSSREQ--------------------------P--VLHEIDLSKKGN------
Cerevisiae
-------------------QEEQE--------------------------------------------------------
Human
258-KSALDEIMEIE-EEKKRTA------------------------RTDYWLQPEIIVKIITKKLGEK-YHKKKAIVKEVIDK
Mouse
KSALDEIMELE-EEKKRTA------------------------RTDAWLQPGIVVKIITKKLGEK-YHKKKGVVKEVIDR
CAEEL
KSALDEIKEMEERKKERKN------------------------RKDYWMREGIVVKVITKSLGSE-YYKAKGVVRKVVDD
Drosophila
-SVLDEIIKQEESKKERAN------------------------RKDYWLHKGIVVKFISKSMGEK-FFKQKAVVLDVIDR
Arabidopsis
RSALDELMKEEEKKKERMN------------------------RKDYWLFEGIIVKVMSKALAEKGYYKQKGVVKKVIDN
POMBE
PIQLNLSSSSDSHSAQNEFF----------------------------QTRNTPTFSFSSSSSQTSLKHKPKNVFAELNK
Cerevisiae
-VIAAELLKRQLNRAKRQT--------------------------------------------EKVYQPEMKSEISGD--
.
Human
312--YTAVVKMIDSG----DKLKLDQTHLETVIPAPGKRILVLNGGYRGNEGTLESINEKTFSATIVIETGPLKGRRVEGIQY
Mouse
-YTAVVKMTDSG----DRLKLDQTHLETVIPAPGKRVLVLNGGYRGNEGTLESINEKAFSATIVIETGPLKGRRVEGIQY
CAEEL
-YTAQVKLDD-G----TVVKLDQEHVETVIPSLGRQMMIVNGAYRGQEATLESIDEKRFSLRLKIASGPTRGRQID-VPY
Drosophila
-YQGKIKFLETG----EKLKVDQAHLETVIPALDKPVMVVNGAYRGSEALLRKLDERRYSVSVEILHGPLKGRIVDNVQY
Arabidopsis
-YVGEIKMLDSK----HVLRVDQKELETVLPQIGGMVKIVNGAYRGSNARLLGVDTEKFCAKVQIEKGVYDGRVIKSIEY
POMBE
---------------SRKKNNKDSLDQGQNVKRPRSAVEDIIAQETMRE-------KRRNIKL-----------------
Cerevisiae
----------------STLKRVQVTFHG-NGRVNKKKKKVPPRKDGIKFR------------------------------
:
Human
387-EDISKLA-----------------------------
Mouse EDISKLA----------------------------- CAEEL
EDASKLA----------------------------- Drosophila
EDISKLHGA--------------------------- Arabidopsis
EDICKLA----------------------------- POMBE ------------------------------------
Cerevisiae ------------------------------------
Figure 1.2 : Alignement de 7
séquences de KIN17 réalisé avec l'aide du logiciel
clustalw. Les séquences alignées sont relatives aux
espèces eucaryotes humaine (Human, 393 résidus), Mus
Musculus (Mouse, 391 réidus), Caenorhabditis elegans (CAEEL,
404 résidus), Drosophila Melanogaster (Drosophila, 390
résidus), Arabidopsis thaliana (Arabidopsis, 411 résidus),
Schizosaccharomyces pombe (POMBE, 304 résidus), et Saccharomyces
cerevisiae (Cerevisiae, 232 résidus). Les résidus sont
colorés en rouge lorsqu'ils sont conservés (sigle *), en
vert lorsqu'il sont fortement similaires (sigle :), et en bleu
lorsqu'ils sont faiblement similaires (sigle .). La numérotation est
relative à la séquence humaine.
L'utilisation des programmes SMART (Schultz et al.,
1998) et Pfam (Finn et al., 2006)
de reconnaissances de domaines suggère l'existence de 4
motifs dans la séquence primaire de
KIN17 humaine :
- Un motif « doigt de zinc » également
appelé C2H2 (résidus 28-50)
- Un domaine de repliement FF (résidus 50-150)
- Une séquence signale de localisation nucléaire
(région 239-256)
- Un motif KOW (335-373)
? Les motifs « doigts de zinc » sont des domaines de
liaison aux acides nucléiques (ADN et ARN). Ils se caractérisent
par la présence d'un ion zinc complexé entre 2 résidus
cystéine et 2 résidus histidine (Figure 1.3). Lorsqu'ils ne sont
pas retrouvés en plusieurs exemplaires dans une séquence, la
liaison à l'ADN n'est en général pas
spécifique d'une séquence nucléotidique donnée
(Böhm et al., 1997). La présence des 4 résidus
ultra conservés C28, C31, H44, et H50 (relatifs à la
séquence humaine) suggère fortement que la région
N-terminale de KIN17 comporte un motif C2H2 de la
levure jusqu'à l'homme. Cette hypothèse a été
confirmée par Mazin et al., qui ont montré que le «
doigt de zinc » potentiel
de la protéine KIN17 de souris était capable de
lier l'ADN de manière dépendante aux ions
Zn2+ (Mazin et al., 1994).






























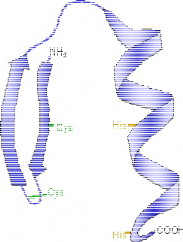
Figure 1.3 : Représentation
schématique d'un motif « doigt de zinc »
? Entre les résidus 50 et 150, toutes les
protéines KIN17 possèdent une région, qui, chez la levure
Schizosaccharomyces pombe et le ver Caenorhabditis
elegans, est prédite comme adoptant un repliement de type FF par
le programme SMART. Les domaines de repliement FF sont des modules
de liaison à des peptides phosphorylés trouvés dans
de nombreuses protéines eucaryotes comme le facteur de
transcription CA150 (Allen et al., 2002). Ceci suggère que les
protéines KIN17 pourraient posséder un domaine de liaison
à un motif phosphorylé. Cependant, ce type de repliement n'est
pas prédit chez toutes les espèces qui ne contiennent pas tous
les résidus décrits comme importants pour la structure en 3
hélices des domaines FF. D'autre part, la surexpression de
plusieurs formes tronquées de KIN17 de souris chez E. coli
a montré que la région 71-281 de KIN17 (relative à la
séquence humaine) comportait un second domaine de liaison à l'ADN
(Mazin et al., 1994). Or, les modules FF ne sont pas des domaines de
liaison à l'ADN. Sur la base de ces résultats,
l'existence d'un domaine FF chez KIN17 apparaît donc
hypothétique.
? Enfin, les logiciels de détections de domaines ont mis
en évidence l'existence d'un domaine additionnel d'environ 100 acides
aminés contenant un motif appelé KOW (Kyrpides et
al.,
1996) dans la région C-terminale de KIN17 humaine absente
chez les eucaryotes inférieurs.
Ce module, également prédit dans les
séquences KIN17 de ver, de plante, et de mammifère,
est retrouvé dans une sous-famille des domaines
TUDOR (Selenko et al., 2001). Les domaines TUDOR sont présents dans
des protéines s'associant à l'ARN. Cependant, le rôle
biologique des domaines TUDOR à motif KOW demeure obscur. De
manière intéressante, les
chercheurs du LGR ont montré que la région
C-terminale de KIN17 contenant le module
KOW est impliquée dans la liaison à l'ARN
(Pinon-Lataillade et al., 2004). Ceci conforte la prédiction des
logiciels SMART et Pfam. Il est également à
noter que la région C-terminale de KIN17 n'apparaît pas
impliquée dans la liaison à l'ADN (Mazin et al., 1994).
| 


