2.2) Détermination de la structure secondaire et de
la topologie
L'extraction des paramètres structuraux RMN est
la méthode classique qui permet d'identifier rapidement la
structure secondaire d'une protéine. L'analyse consensus de ces
paramètres conduit à l'obtention des premiers
éléments de structure tertiaire, utilisés par le
programme d'attribution automatique en amont du calcul de la structure.
2.2.1) Les déplacements chimiques
secondaires
Le déplacement chimique des noyaux de la chaîne
principale d'un résidu est influencé
par plusieurs facteurs dont la structure secondaire dans
laquelle celui-ci est engagé. Wishart et Sykes ont mis au point
une méthode qui permet d'apprécier les types, et les positions
dans la séquence, des éléments de structure secondaire
à partir du déplacement chimique des noyaux
13CO, 13Cá, 13Câ, et
1Há (Wishart et al., 1992 ; Wishart & Sykes, 1994). La
procédure consiste
à calculer la différence entre le
déplacement chimique expérimental (äobs) et une valeur
de référence (äref) correspondant au même
noyau pour le même acide aminé dans une conformation
aléatoire. La valeur obtenue est appelée déplacement
chimique secondaire (CSD
ou Chemical Shift Deviation). Les indices CSD n'ont
pas la même signification selon la nature du noyau. Ainsi, pour le
proton 1H, une succession d'indices supérieurs à 0.1
ppm en valeur absolue reflète une structure secondaire stable et
le signe en précise le type : positif pour un feuillet â, et
négatif pour une hélice á ou 310. Dans le cas
des 13Cá, il s'agit
exactement du contraire, et la valeur significative de CSD est de
1 ppm.
2.2.2) La constante de couplage
3JHN-Há
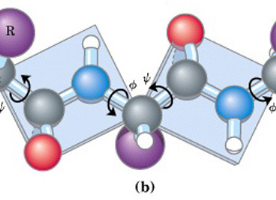
Les angles et sont deux des trois angles de
torsion qui décrivent le squelette peptidique d'une protéine.
La combinaison d'effets stériques, au sein d'un même
résidu, ou entre les chaînes latérales, ne permet
à ces angles de prendre que certaines valeurs qui
définissent les différents types de structure secondaire. La
constante de couplage 3JHN-Há est reliée
indirectement à l'angle dièdre via l'équation de
Karplus (2.3) avec les coefficients semi-empiriques A, B, et C (Karplus,
1959). Plusieurs jeux de coefficients ont été proposés, et
ceux utilisés par Pardi et al., sont les plus
communément admis (2.4) (Pardi et al., 1984). La résolution de
l'équation fait apparaître que les valeurs idéales de
constante 3JHN-Há sont de 4Hz pour l'hélice
á (-57°), et de 9 Hz pour le feuillet â
antiparallèle (-139°). Plusieurs études statistiques,
comme celle menée par Smith et al., sur 85
structures résolues par radiocristallographie, ont cependant
conduit à considérer des valeurs corrigées de
3JHN-Há (Smith et al., 1996). Sur la base de ces
études, il est généralement admis que plusieurs valeurs
de 3JHN-Há consécutives
inférieures à 5.5 Hz indiquent une conformation
hélicoïdale, alors que
des valeurs supérieures à 8 Hz reflètent une
structure en feuillet ou étendue.
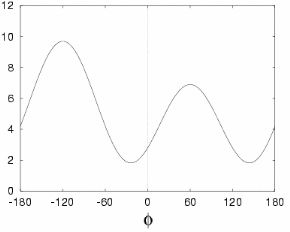
O C) 3JHN-Há = 6,4.cos2
è - 1,4.cos è + 1,9
A) N
C'
C'
Cá N
10
8
J
3
HN-Há 6
avec è = |-60°| (2.4)
O [Hz]
4
3JHN-Há = A.cos2
è - B.cos è + C
B) avec è = | - 60°|
(2.3)
2
-180 -120 -60
0 60
[°]
120
180
Figure 3.9 : Estimation de
l'angle dièdre à partir de la constante de
couplage 3JHN-Há.
A) Définition des angles de torsion et
du squelette peptidique. B) Relation de Karplus
liant la constante 3JHN-Há aux angles
è et . C) Résolution de l'équation de Karplus
selon les coefficients de Pardi.
Dans le cas des protéines, les constantes de
couplage 3JHN-Há sont généralement
estimées à partir de l'expérience 3D HNHA, qui met
en évidence la corrélation intra- résiduelle entre un
groupement amide (1HN, 15NH) et son proton alpha
(1Há). Sur les spectres correspondants, le rapport
d'intensité entre le pic croisé négatif (Ic) et
le pic diagonal positif
(Id) est relié à la constante
3JHN-Há via une constante å (Vuister
& Bax, 1993) :
Ic / Id = -tan2 (2ðå .
3JHN-Há )
(2.5)
2.2.3) Recherche des nOe
caractéristiques
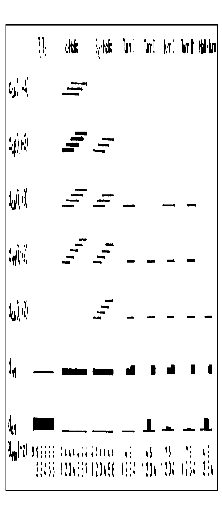
Comme suggéré dans le paragraphe 2.1.2, chaque type
de structure secondaire donne naissance à des pics nOe
spécifiques en nature et en intensité sur les spectres NOESY.
Aussi,
la méthode classique proposée par
Wüthrich pour identifier les éléments de structure
secondaire consiste à reporter sur un diagramme les nOe
caractéristiques entre protons appartenant à des résidus
proches dans la séquence (Wüthrich, 1986) (Figure 3.10A). Ces nOe
sont facilement identifiables sur les spectres NOESY-HSQC éditée
15N, et un simple examen
de ce diagramme suffit en théorie à
discerner de manière univoque les différents types de
conformation au sein de la protéine.
A) B)
H
H
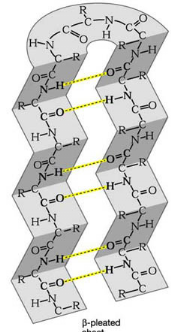
Figure 3.10 : Identification des
éléments de structure secondaire et de la topologie
d'une
protéine à partir de l'attribution de
pics nOe spécifiques. A) Diagramme des nOe
caractéristiques des différents types de structure
secondaire selon Wüthrich (Wüthrich,
1986). B) Description schématique d'un feuillet
â antiparallèle. Les liaisons hydrogène sont
représentées par des pointillés jaunes.
Les traits rouges représentent le type de connectivité longue
distance recherchée.
Une fois les éléments de structure secondaire
identifiés à partir du résultat consensus
de l'analyse des paramètres structuraux, la
topologie de la protéine est déterminée en
recherchant des effets spécifiques à longue distance de
type HN-HN, HN-Há, et Há-Há au niveau des
résidus n'adoptant pas de structure hélicoïdale. La
caractérisation de ces effets permet d'une part, de distinguer les
régions étendues de celles impliquées dans un feuillet
â et d'autre part, de caractériser l'organisation des brins
au sein des feuillets (parallèle ou antiparallèle) (Figure
3.10B). Ces éléments de structure tertiaire facilement
identifiables sont extrêmement précieux car ils fournissent
les premières informations du repliement de la protéine, et
vont guider le programme d'attribution automatique des nOe.
2.2.4) Le nOe hétéronucléaire
1H-15N
Comme nous l'avons évoqué
précédemment, le phénomène de relaxation
croisée entre deux spins dépend essentiellement de la
distance r entre les deux noyaux, et du temps de
corrélation effectif ôc entre les deux spins,
c'est-à-dire de l'amplitude de leur mouvement. Dans
l'approximation d'une distance fixe entre les atomes 15NH et
1HN (rHN ~ 1.01 Å), le paramètre nOe
hétéronucléaire 1H-15N ne
dépend plus que des mouvements rapides de la liaison NH-HN (de
la picoseconde à la nanoseconde). D'autre part, ce type
d'effet est indépendant des protons environnants (Kay et al., 1989).
Par conséquent, la mesure du nOe
1H-15N peut se révéler
très utile pour distinguer les parties flexibles de la protéine,
telles que
les boucles exposées ou les parties
déstructurées (mouvements rapides), de la partie
repliée
(mouvements plus lents).
En pratique, ce paramètre dynamique est
quantifié à partir de deux expériences 2D similaires
qui permettent d'observer la corrélation scalaire
1H-15N de chaque résidu. Le premier spectre
est enregistré avec une saturation préliminaire des
1H qui engendre un transfert d'aimantation vers le 15N
via le couplage dipolaire. Le second spectre est obtenu par
la même séquence d'impulsion, mais en absence de
saturation des 1H (Farrow et al., 1994).
Pour chaque pic de corrélation, le rapport entre
l'intensité du pic saturé (Isat) et l'intensité
du
pic non saturé (Iref) détermine
alors le nOe hétéronucléaire (2.6). L'incertitude
sur chaque valeur est calculée à partir des erreurs de mesure
des intensités qui sont estimées par le bruit spectral sur chacun
des deux spectres.
nOe (1H-15N) =
Isat
Iref
(2.6)
Les régions dans lesquelles les résidus ont une
valeur de nOe 1H-15N supérieure à 0.5
sont
considérées comme rigide, entre 0.2 et 0.5
comme relativement flexibles (boucles), et les acides aminés pour
lesquels ce nOe est inférieur à 0.2 ou négatif
appartiennent à des régions déstructurées de la
protéine. Dans l'optique du calcul de la structure, la mesure
du nOe hétéronucléaire fournit une information
supplémentaire car elle permet d'identifier avec précision
les résidus non structurés des extrémités N- et
C-terminales. Aussi, sur les spectres
de type NOESY, les nOe homonucléaires 1H
observables correspondent à une moyenne
pondérée des nOe correspondants à chaque
population de l'espace conformationnel. Dans le
cas de résidus en échange conformationnel
très rapide, le poids de chaque population est
faible et les contacts de type longue distance sont incompatibles
avec une telle flexibilité. Par
conséquent, la connaissance de ces résidus peut
être utilisée par le programme d'attribution automatique des pics
nOe afin de restreindre le nombre de possibilités d'attribution et donc
de faciliter celle-ci.
| 


