2.3) Recueil des contraintes structurales
Les contraintes expérimentales de distance et d'angle
dièdre utilisées pour le calcul de
la structure de K2 sont issues de quatre observables RMN :
l'effet Overhauser nucléaire 1H, le couplage scalaire, le
déplacement chimique, et les vitesses d'échange des protons
amides.
2.3.1) Collecte des pics nOe
La grande majorité des contraintes de distance est obtenue
à partir des expériences 3D NOESY-HSQC éditée
15N, et 13C. Les pics de corrélation dipolaire
1H-1H sont sélectionnés
sur les spectres, puis leur volume est mesuré
à l'aide du logiciel Felix (Accelrys). La démarche
consiste dans un premier temps à définir des «
boites » d'intégration autour de chaque pic nOe. Avant de
mesurer le volume de ces boites, leur taille doit être
optimisée, l'une après l'autre, afin d'éviter
d'intégrer du bruit spectral ou un pic voisin, qui conduirait à
des distances inter-atomiques erronées (souvent sous-estimées).
Cette étape est donc cruciale dans l'optique de préparer un jeu
de contraintes expérimentales de qualité. Selon la
stratégie définie, l'attribution des pics nOe n'est pas
nécessaire à ce stade de l'étude. Seuls les nOe
inter-résidus qui traduisent la topologie des feuillets
â sont attribués et convertis en
contraintes de distance.
2.3.2) Les contraintes d'angle dièdre
Le déplacement chimique des noyaux
13Cá, 13Câ, 13CO,
1Há, et 15N peut être utilisé
pour déterminer les angles et sur la base d'une
prédiction empirique réalisée par le logiciel TALOS
(Cornilescu et al., 1999). Ce programme recherche dans une base de
données, actuellement constituée de 78 protéines, des
triplets de résidus consécutifs ayant une homologie de
déplacements chimiques et de type de résidu avec les
triplets de la protéine étudiée. Pour chaque triplet de
la protéine, les 10 tripeptides les plus proches en séquence
et
en déplacements chimiques sont sélectionnés.
Aussi, si les angles et des résidus centraux
de 9 de ces 10 triplets sont similaires, la prédiction est
considérée comme fiable. Leur valeur
moyenne et l'écart type correspondant sont alors
utilisés pour constituer les contraintes
d'angle dièdre sous forme d'un intervalle de valeurs
permises.
Pour les résidus dont la prédiction TALOS
n'est pas jugée satisfaisante, le jeu de données angulaires
peut être complété à partir de la constante de
couplage 3JHN-Há pour des valeurs inférieures
à 5.5 Hz ou supérieures à 8 Hz (cf. §
2.2.2). L'intervalle des valeurs permises d'angle est alors déduit
de la courbe de Karplus intégrée par les coefficients de
Pardi (Tableau 3.1) :
|
3JHN-Há
(Hz)
|
contrainte sur l'angle
(°)
|
|
3J > 9.0
8.0 < 3J < 9.0
5.5 < 3J < 8.0
5.0 < 3J < 5.5
3J < 5.0
|
-120 #177; 30
-120 #177; 40
Ø
-70 #177; 30
-60 #177; 30
|
Tableau 3.1 : Intervalles de
contraintes sur l'angle en fonction de la constante
3JHN-Há.
2.3.3) Les contraintes de distance déduites des
liaisons hydrogène
Dans toute solution aqueuse, l'autodissociation de l'eau en
ions H3O+ et OH- catalyse l'échange des
protons amides avec les protons de l'eau (Englander et al., 1972).
Selon ce principe, l'incubation d'une protéine dans une solution
à 99% d'eau lourde va mener à la substitution progressive
de la majorité des protons 1HN par le deutérium de
l'eau lourde. La cinétique de cet échange dépend d'une
part, de l'accessibilité au solvant et d'autre part, de la liaison
hydrogène dans laquelle un proton amide peut être impliqué
en se liant à un oxygène
de carbonyle. Un proton 1HN exposé au
solvant et non engagé dans une liaison hydrogène
s'échangera très rapidement, alors que dans
le cas d'un proton amide impliqué dans une liaison
hydrogène, la vitesse d'échange sera d'autant plus lente que
cette liaison est enfouie dans le coeur hydrophobe de la protéine.
Par conséquent, une expérience de spectroscopie
d'échange 1H-2D suivie sur spectres HSQC
1H-15N peut permettre de localiser une partie des protons
amides impliqués dans une liaison hydrogène. Ces liaisons sont
spécifiques de chaque structure secondaire. Aussi, la connaissance
de la topologie de la protéine est utilisée
conjointement pour identifier les deux atomes
impliqués dans chaque liaison. Cette donnée
structurale est finalement convertie en contraintes de distance
de la manière suivante :
|
2.1 Å < dHN-O (i, i-4) < 2.5 Å
|
et
|
3.1 Å < dN-O (i, i-4) < 3.5 Å
|
dans une hélice á
|
|
2.1 Å < dHN-O (i, i-3) < 2.5 Å
|
et
|
3.1 Å < dN-O (i, i-3) < 3.5 Å
|
dans une hélice 310
|
2.1 Å < dHN-O (i, j) < 2.5 Å et 3.1 Å
< dN-O (i, j) < 3.5 Å dans un feuillet â
3) Modélisation Moléculaire sous
contraintes RMN
Les contraintes structurales issues de
l'interprétation des paramètres RMN ne permettent pas
d'accéder directement à la structure tridimensionnelle de
la protéine. Ces données expérimentales sont introduites
dans des procédures de modélisation moléculaire afin
de construire des modèles à l'échelle
atomique. Il s'agit de la modélisation moléculaire sous
contraintes RMN, étape indispensable pour obtenir une
représentation graphique de la structure 3D d'une protéine.
La méthode s'appuie sur des calculs
théoriques de mécanique moléculaire et de dynamique
moléculaire pour déterminer un ensemble de conformations, qui
correspondent à des minima énergétiques, et qui sont en
accord avec les données expérimentales RMN. Dans
le cadre de l'étude de la protéine K2,
nous avons utilisé le logiciel de modélisation CNS
adapté au traitement de données expérimentales
obtenues par radiocristallographie ou par RMN. Les principes de
mécanique et dynamique moléculaire sur lesquels repose ce
logiciel seront évoqués en première partie de ce
paragraphe. La seconde partie sera consacrée à la
présentation du programme d'attribution automatique des pics nOe,
qui fonctionne en interface avec le logiciel CNS.
3.1) Principe de la mécanique moléculaire
adaptée aux systèmes biologiques
3.1.1) Notion de champ de force
Une grande part des systèmes auxquels la
modélisation moléculaire s'intéresse ont une taille bien
trop importante pour pouvoir être étudiés par des
méthodes classiques de mécanique quantique de type ab
initio ou semi-empiriques. En effet, malgré le
perpétuel développement du domaine de l'informatique et
notamment de la capacité de calcul, les
équations de mécanique quantique, utilisées
pour représenter la fonction d'énergie de petites
molécules, ne peuvent être résolues
dans le cas de macromolécules. C'est pourquoi, les simulations
de mécanique et dynamique moléculaire s'appuient sur une
représentation simplifiée de la fonction d'énergie
appelée « champ de force ». Selon ce principe, à chaque
état conformationnel correspond une énergie potentielle
empirique définie par le champ de force, et qui est fonction
des interactions attractives et répulsives entre les atomes. La
conformation la plus stable est alors associée à l'état
pour lequel la fonction d'énergie est la plus basse. Cependant, et avec
ce seul terme d'énergie potentielle, une protéine qui fait
l'objet d'une simulation de mécanique moléculaire peut adopter
une multitude de conformations qui correspondent à des minima
énergétiques locaux. L'intégration de contraintes
expérimentales dans les protocoles de modélisation
moléculaire permet de restreindre l'espace conformationnel à
explorer, et de favoriser des configurations qui sont en accord avec
les données expérimentales. Pour prendre en compte ces
données, le champ de force est alors constitué, d'un
terme d'énergie potentielle empirique Epot
représentant les contraintes physiques internes à la
molécule entre atomes liés et non liés, et d'un
terme énergétique supplémentaire Econt qui
tient compte des contraintes expérimentales (2.7). C'est le cas
du champ de force du programme CNS qui sera présenté
succinctement dans le paragraphe 3.2.
E = Epot + Econt
3.1.2) Les algorithmes de minimisation
(2.7)
Une fois le champ de force choisi, l'objectif de la
modélisation moléculaire sous
contraintes est de déterminer les conformations
associées aux surfaces d'énergie les plus basses, et
satisfaisant un maximum de contraintes RMN. Pour cela, il existe de
nombreux algorithmes qui se différencient par leur efficacité et
leur rapidité d'exécution.
Une minimisation d'énergie, par des algorithmes dits
de mécanique moléculaire, est
un processus itératif qui a pour but d'optimiser la
géométrie d'une molécule à partir d'une
conformation initiale. Tous les paramètres définissant la
géométrie du système (degrés de
libertés) sont systématiquement modifiés par petits
incréments jusqu'à ce que l'énergie atteigne un
minimum, ou pendant un nombre de pas fixé préalablement. Les
algorithmes de minimisation les plus couramment utilisés recherchent un
minimum énergétique en se basant
sur le gradient de la surface d'énergie
potentielle, c'est à dire sur sa dérivée
première ou
seconde par rapport aux coordonnées atomiques.
Ce gradient indique la direction du
minimum, tandis que sa valeur renseigne sur la « pente
» locale de la fonction d'énergie. C'est
Energie potentielle
le cas de la méthode de la plus grande pente
(steepest descent ; Arkfen, 1985), efficace lorsque la
conformation est proche de la structure initiale, et de la
méthode de gradient conjugué Powell (Powell, 1977),
utilisée dans le protocole CNS, et plus adaptée aux
systèmes dont le minimum énergétique est proche.
Figure 3.11 : Illustration
de l'inconvénient des algorithmes de minimisation par la
représentation schématique d'une surface d'énergie
potentielle. La boule grise représente l'énergie potentielle
d'une conformation initiale. La minimisation de l'énergie en se
basant
sur des gradients de surface ne permet de
déterminer qu'un minimum local proche de la structure de
départ et souvent différent du minimum global.
Même si les algorithmes de minimisation basés sur
le gradient de l'énergie potentielle permettent d'optimiser rapidement
la géométrie d'un édifice moléculaire, ils
conduisent le plus souvent au minimum énergétique local, le plus
proche de la structure de départ, et très souvent
éloigné du minimum global (Figure 3.11). Par conséquent,
ces méthodes de calcul ne constituent certainement pas l'outil principal
pour déterminer la structure d'une protéine par
modélisation sous contraintes. La solution idéale serait
une méthode capable d'explorer efficacement l'espace conformationnel
d'un composé, c'est-à-dire l'ensemble des conformations qui lui
sont accessibles. La manière la plus simple et la plus
sûre serait de générer et d'optimiser des
modèles en combinant toutes les possibilités
conformationnelles imaginables. Cette approche, appelée recherche
systématique, est cependant difficilement envisageable dans le cas
de l'étude de macromolécules, où les temps de
calcul exigés constitueraient alors un facteur limitant.
3.1.3) La dynamique moléculaire
La dynamique moléculaire est une des méthodes
qui permet d'explorer l'espace conformationnel d'une structure complexe.
Elle a pour objectif de simuler les mouvements
internes d'une molécule en fonction du temps par
le calcul du déplacement de chacun des
atomes. Elle repose sur les principes de la mécanique
classique Newtonienne et la trajectoire
de chaque atome est définie par la relation
fondamentale de la dynamique (2.8), qui relie la force Fi
s'exerçant sur chaque atome à sa masse mi, son
accélération ai, et sa position ri en fonction
du temps t.
? ?
Fi = miai =
mi
i
d2r?
dt2
(2.8)
?
Fi = -
?
dEpot
i
dr?
(2.9)
?r(t+ät) = 2?r(t)
-
?r(t - ät) + ät2
a?(t) (2.10)
?v(t+ät) = ?v(t) +
0.5ät[a?(t) +
a?(t+ät)]
(2.11)
La force Fi étant
reliée à la fonction d'énergie potentielle (2.9), il est
donc possible de
calculer à tout instant t
l'accélération ai s'exerçant sur chaque
atome. Plusieurs algorithmes mathématiques sont proposés pour
résoudre ces équations. Parmi ceux-ci, figure celui de
Verlet (Verlet, 1967) dans lequel est utilisé un développement en
série de Taylor du second ordre du vecteur position. Ainsi,
connaissant la position à l'instant t, il est alors
possible d'obtenir les positions r(t+ät) (2.10) et les vitesses
v(t+ät) (2.11) des différents atomes. Cet algorithme
implique un pas d'intégration ät très court de
l'ordre de la femtoseconde (10-15 s) afin de maintenir une
trajectoire stable.
Dans une simulation de dynamique moléculaire,
l'énergie cinétique totale d'un système de N
atomes peut être reliée à la température T
via la constante de Boltzman kB :
Ecinétique
= 1
Ó
i 2
2
mivi =
3
2 NkBT
(2.12)
Cette équation introduit la notion de
température au sein des méthodes de dynamique
moléculaire. Plus cette température est
élevée, plus la vitesse communiquée aux atomes sera
importante. Par l'intermédiaire d'une hausse « fictive » de
température, il est alors possible de fournir au système une
certaine quantité d'énergie cinétique qui lui permet
de franchir les barrières d'énergie potentielle, et donc
d'explorer un espace conformationnel beaucoup plus large. Si la
température est suffisamment importante, une molécule est alors
potentiellement capable d'adopter toutes les conformations possibles.
Une simulation de dynamique
moléculaire se réalise généralement
en trois temps : après avoir attribué une vitesse initiale
aléatoire aux atomes par une distribution de type
Maxwell-Bolzman, une augmentation de
température est simulée afin d'atteindre rapidement
une valeur finale choisie (thermalisation).
La température est alors maintenue à cette valeur
constante afin de répartir l'énergie cinétique
sur toute la molécule (équilibration). Finalement,
la configuration du système est enregistrée à intervalles
de temps réguliers durant une phase de recueil de données
(collecte).
3.1.4) Le recuit simulé
Le recuit simulé est une méthode de dynamique
moléculaire couramment utilisée pour déterminer la
structure d'une protéine par modélisation sous contraintes
(Figure 3.12). Le nom de ce protocole s'inspire de la technique industrielle
de « recuit » qui consiste à refroidir très lentement
un matériau fondu, afin de lui permettre d'organiser au mieux
sa structure moléculaire.
1) thermalisation et équilibration
Energie potentielle
2) refroidissement
3) minimisation
1000 K
100 K
Figure 3.12 : Illustration d'une
procédure de recuit simulé par la représentation
schématique
d'une surface d'énergie potentielle. La
dynamique à haute température permet d'explorer l'espace
conformationnel et de s'éloigner de la structure initiale (non
représentée). Les structures générées
sont lentement refroidies de manière à occuper les zones
stables de la surface d'énergie. Une dernière étape de
minimisation permet d'optimiser les conformations
et d'obtenir finalement des minima énergétiques
plus profonds.
La méthode du recuit simulé consiste dans un
premier temps à mener une dynamique à haute température
(de 100 à 1000 K) à partir d'une structure initiale
aléatoire étendue préalablement minimisée. Cet
apport d'énergie cinétique permet d'explorer l'espace
conformationnel et de se dégager des minima locaux proches des
structures initiales. Après une période d'équilibration,
la température est lentement diminuée jusqu'à 100
K afin de réduire l'énergie cinétique et de
contraindre le système à occuper les états de plus
basse
énergie. On peut comparer schématiquement cette
étape à la phase de refroidissement de la
technique industrielle de « recuit » où une
diminution très lente de la température conduit à
l'obtention de l'espèce moléculaire la plus stable et qui
possède une énergie libre minimale. Finalement, les
dernières étapes du recuit simulé consistent à
minimiser puis à collecter les structures ainsi calculées. Il est
cependant important de noter que cette méthode ne garantit pas
l'identification du minimum énergétique global, mais s'en
approchera probablement dès lors que le jeu de contraintes
expérimentales est optimisé (diminution cohérente des
degrés de
liberté), et qu'un ensemble suffisant de configurations
est collecté.
3.2) Le logiciel CNS
Le logiciel CNS (Cristallography and NMR system ; Brünger et
al., 1998) est dédié à
la détermination et au raffinement de structures
tridimensionnelles à partir de données expérimentales
obtenues par RMN ou radiocristallographie. Il repose sur les principes
de mécanique et dynamique moléculaire et notamment celui du
recuit simulé.
3.2.1) Description du champ de force
Comme nous l'avons évoqué
précédemment, pour tenir compte des contraintes
expérimentales, les champs de force utilisés par le
programme CNS sont composés d'un terme d'énergie potentielle
empirique Epot, et d'un terme supplémentaire Econt
qui permet de contrôler le respect de ces contraintes :
E = Epot + Econt
a) La fonction d'énergie potentielle
Epot
(2.13)
L'énergie potentielle empirique d'une
molécule est définie par l'ensemble des
composantes qui décrivent sa géométrie. Elle
est la somme de deux termes, l'un représentant
les interactions entre atomes liés Eliés
et l'autre, les interactions entre atomes non liés
Enon_liés :
Epot = Eliés +
Enon_liés
(2.14)
La description succincte des différents termes de la
fonction Epot est réalisée à
partir
des composantes du champ de force CHARMM (Brooks, 1983) dont
dérive le champ de force
CHARMM22 utilisé pour le raffinement des structures
finales sélectionnées.
Terme des atomes liés :
Eliés =
Eliaisons +
Eangles
+ Eimpropres
+ Edièdres
(2.15)
2 Ó 2 Ó 2 Ó
Eliés =
Ó kr (r - r0) +
kè (è - è0) +
kù (ù - ù0) +
k (1 +
cosn)













r è ù
Les termes énergétiques associés aux
interactions entre atomes liés permettent en
premier lieu de maintenir la géométrie covalente
de la molécule. Ils rendent compte du coût
énergétique de l'élongation des liaisons covalentes
(r), des fluctuations, de l'angle è formé par
deux liaisons covalentes, de l'angle dièdre impropre ù
pour maintenir la chiralité d'un groupe d'atomes, ou bien encore
de l'angle de torsion formé par un groupe de quatre atomes (n
correspondant à la période de la fonction cosinus). Les
trois premiers termes de la fonction Eliés sont des potentiels
harmoniques centrés sur une position d'équilibre de
géométrie idéale
(r0,
è0, et
ù0). Ces valeurs de
référence dérivent de modèles
moléculaires obtenus, soit
expérimentalement par diffraction des rayons X ou
de neutrons, soit par des calculs théoriques de type ab
initio. Les différentes valeurs des constantes de force
kr, kè, et kù sont quant à
elles obtenues à partir d'analyses vibrationnelles de
molécules en phase gazeuse. Ainsi, par cette représentation de
la fonction d'énergie Eliés, la géométrie
moléculaire peut être considérée comme une
série d'oscillateurs composés de billes et de ressorts. La somme
des contributions de ce terme traduit alors les écarts des
structures calculées par rapport à des
géométries de référence.
Terme des atomes non liés :
Enon_liés =
Evan_der_Waals
+ Eélectrostatique
(2.16)
r
Enon_liés =
Ó
i,j
Aij
-
12
ij
Bij
6
r
ij
+ Ó
i,j
1
4ð år
. qiqj
å0 rij




qi




rij qj
Le terme d'énergie des interactions entre
atomes non liés Enon_liés est
la somme des
contributions correspondant aux interactions de type
électrostatiques et de van der Waals entre couples d'atomes. En
mécanique moléculaire, chaque atome est considéré
comme une masse possédant une charge ponctuelle. Selon ce principe,
l'interaction électrostatique entre deux charges partielles qi
et qj distantes de rij est définie par un
potentiel de type coulombien. L'intensité de l'interaction dépend
notamment de la distance rij, et de la constante
diélectrique
du milieu år. Le
paramètre år sert à
reproduire les effets d'écran du solvant entre les charges.
Aussi, un choix judicieux de la valeur år
permet de mimer de manière implicite l'effet du solvant et
de prendre en compte sa présence dans le calcul des interactions
électrostatiques.
Les interactions de van der Waals traduisent l'attraction
et la répulsion entre deux atomes séparés par une
distance rij et constituant un dipôle. Le terme
d'énergie correspondant
6
est un potentiel de Lennard-Jones qui contient une
composante attractive en 1/rij
12
due aux
forces de dispersion de London, et une composante
répulsive en 1/rij
due aux couches
électroniques des atomes les plus proches. Les
coefficients Aij et Bij dépendent de la nature des
atomes. Lorsque la distance rij est inférieure à la
somme des rayons de van der Waals, c'est le terme répulsif qui
prédomine et inversement pour une distance supérieure,
c'est le terme attractif qui agit principalement.
Selon la description des termes de la fonction
Enon_liée, des interactions de type électrostatiques
et de van der Waals peuvent en théorie être observées entre
deux atomes très éloignés en distance. La prise en
compte de l'ensemble des combinaisons possibles entre paires d'atomes
d'une macromolécule impliquerait un temps de calcul
considérable. Pour réduire la durée de ces calculs,
il est possible de limiter la portée des interactions en
définissant un « seuil de coupure ». Selon ce principe,
seules les interactions entre atomes séparés par une
distance inférieure à ce seuil seront prises en compte par le
champ de force. Aussi, pour éviter des discontinuités
d'énergie induites par l'introduction de ces seuils, la fonction
d'énergie Enon_liée est multipliée par une
fonction d'amortissement ou de « switch » (Brünger, 1992). Ce
type de fonction permet de faire tendre progressivement les
énergies électrostatiques et de van der Waals vers zéro,
et donc d'éviter des coupures brusques pouvant
générer des erreurs de calcul.
b) La prise en compte des contraintes RMN
Les contraintes expérimentales, introduites dans les
protocoles de recuit simulé sous forme d'intervalles de valeurs
permises, sont associées à des termes
énergétiques supplémentaires dans le champ de force de
CNS. Ces termes traduisent le degré de prise en compte de ces
données. Le non respect d'une contrainte conduit à une
pénalité énergétique, appelée violation, qui
informe de l'écart entre une valeur de distance ou d'angle dièdre
dans une structure calculée, et l'intervalle de valeurs
autorisées correspondant.
Terme des contraintes de distance :
Le logiciel CNS propose plusieurs types de fonctions
potentielles pour exprimer le
terme énergétique des contraintes de distance
EnOe. Dans le cadre du calcul de la structure du domaine K2, nous
avons utilisé les deux fonctions suivantes :
· Le potentiel carré (square) :
EnOe = KnOe . ä
n
avec :
ä = rmin - d pour d <
rmin
ä = d - rmax pour d >
rmax
(2.17)
où rmin et
rmax représentent les limites de
l'intervalle de distances permises pour une contrainte
donnée, d est la distance correspondante dans le
modèle, KnOe est la constante de force, et n
est généralement égal à 2.
Notons par ailleurs que le terme EnOe est
systématiquement nul lorsque la valeur de distance d est
encadrée par les valeurs rmin et rmax.
· Le potentiel biharmonique adoucie (softsquare)
Lorsque la distance d mesurée dans un
modèle se situe nettement en dehors de l'intervalle de valeurs
permises, le potentiel carré conduit à une
pénalité énergétique importante qui
pénalise le système et ralentit la convergence des structures.
Pour éviter que
les énergies de contraintes ne soient trop
élevées, le potentiel biharmonique adoucie est utilisé
lorsque l'écart entre la distance d et
l'intervalle de valeurs atteint un certain seuil. L'introduction
d'une asymptote à la courbe (kasym), et des constantes
a et b qui assurent la continuité de potentiel,
permet ainsi d'adoucir la fonction EnOe dans les cas extrêmes
(2.18).
EnOe = a + b/ä +
kasym.ä (2.18)
Terme des contraintes angulaires :
Le terme énergétique Edih
lié aux contraintes angulaires (ou ) est
représenté par une
fonction potentielle carrée comparable à celle
définie ci-dessus et de la forme suivante :
Edih = Kdih
( - x )2
(2.19)
avec x la limite minimum ou
maximum de l'intervalle de valeurs autorisées pour une
contrainte donnée, la valeur d'angle dans le
modèle, et Kdih la constante de force associée.
3.2.2) Description du protocole de recuit
simulé
Le paragraphe suivant décrit les différentes
étapes de la simulation de dynamique moléculaire
utilisée pour calculer la structure du domaine K2 de la protéine
humaine KIN17.
a) Génération des structures initiales
aléatoires
L'étape préalable aux calculs de dynamique
moléculaire consiste à générer des structures
initiales par tirage aléatoire des angles et . La topologie
de la protéine est obtenue à partir d'un champ de force
simplifié défini par les paramètres standard des fichiers
parallhdg et topallhdg. Le fichier topallhdg
décrit tous les acides aminés avec le type d'atome,
de liaison, et d'angle qui les constituent, ainsi que la charge partielle et la
masse des atomes. Ce fichier est utilisé par CNS pour créer le
fichier de topologie PSF spécifique à la séquence
du domaine K2. Les paramètres du champ de force sont
définis dans le fichier parallhdg qui contient toutes
les valeurs d'équilibre des liaisons et des angles de valence
dièdres et impropres relatives aux différents termes
énergétiques. La fonction d'énergie
potentielle Epot de ce champ de force simplifié
est de la forme suivante :
Epot = Eliaisons +
Eangles + Eimpropres + Erepel
(2.20)
Cette fonction exclue donc les termes
Edièdres et
Eélectrostatique uniquement pris en compte
dans
le champ de force CHARMM22 utilisé pour le
raffinement des structures. Par ailleurs, le terme des interactions de
van der Waals est remplacé par un potentiel continu purement
répulsif (Erepel) similaire à la composante
répulsive du potentiel de Lennard-Jones. Ce terme
est associé à une constante de force
kvd_Waals, et à un facteur multiplicateur
du rayon minimum
de van der Waals krepel.
b) Recuit simulé
Lors du recuit simulé, la fonction d'énergie du
système est recalculée après chaque pas
de simulation à partir de la position des atomes via les
équations de Newton. Les positions des protons géminés
sont régulièrement échangées pendant la
procédure (coordonnées et vitesses)
de manière à choisir la configuration dans laquelle
l'énergie est minimum. Il en est de même pour les
méthyles chiraux. Les valeurs des principaux
paramètres utilisés pendant la
simulation de dynamique moléculaire sont reportées
dans le Tableau 3.2.
|
Recuit simulé
|
T° [K]
|
Nombre
de pas
|
kvd_Walls
|
krepel
|
knOe
|
kdih
|
kr
|
kè
kù
|
|
i) Minimisation
ii) Chauffage
iii) Echantillonnage
iv) Refroidissement
v) Minimisation
|
-
2000
entre 1000
et 2000
1950?100
-
|
50
1300
2500
1000
2500
|
0.003
0.003?4
4
|
0.9
0.9?0.75
0.75
|
10?50
50
50
|
5
5
200
|
1000
1000
1000
|
500
500
500
|
Tableau 3.2 : Evolution des
principaux paramètres au cours du recuit simulé. Les
constantes
de force sont exprimées en kcal.mol-1.rad-2
à l'exception de kr et knOe
exprimé en kcal.mol-1.Å-2.
Le protocole de recuit simulé peut être
décomposé en 5 étapes distinctes :
i) La première étape consiste en une
minimisation de la structure initiale étendue par
un algorithme de type Powell.
ii) Le recuit simulé débute par une dynamique de
type Verlet où le système est porté à une
température de 2000 K. Les constantes de force relatives aux termes
d'énergie des élongations de distance, des angles de
valence, et des angles impropres sont maintenues à une forte
valeur pendant toute la durée de la simulation afin de
conserver la géométrie covalente de la protéine. A
contrario, la valeur de constante de force kvd_Waals est dans un
premier temps choisie faible dans le but d'autoriser les atomes
à s'interpénétrer, ce qui permet d'explorer l'espace
conformationnel. Aussi, le facteur krepel est fixé
à 0.9 afin d'éviter le collapse de la protéine.
Lors de cette première phase de chauffage, les
contraintes expérimentales sont prises en compte par introduction
des termes EnOe et Edih. Les contraintes de distance sont
par ailleurs
privilégiées via une valeur de
knOe progressivement augmentée de 10
à 50.
iii) La troisième étape du protocole est une phase
d'échantillonnage où la température
oscille entre 1000 et 2000 K. La constante kvd_Waals est
progressivement augmentée de
0.003 à 4 pour restreindre la
pénétration des atomes alors que le facteur
krepel est
diminué de 0.9 à 0.75.
iv) La dernière étape de dynamique consiste en un
refroidissement du système de 1950
à 100 K où les contraintes d'angles dièdres
sont pleinement prises en compte via une valeur de constante de force
fixée à 200.
v) La simulation s'achève par une
minimisation des structures calculées par un
algorithme de type Powell.
3.3) Le programme d'attribution automatique des pics nOe
du LSP
Développé récemment au Laboratoire de
Structure des Protéines du CEA de Saclay (LSP), le programme
d'attribution automatique des nOe est un module de gestion de scripts et
logiciels dédiés à la résolution de
structures tridimensionnelles de biomolécules par RMN (Savarin et
al., 2001). Outre l'attribution des pics nOe, les quatre autres principales
fonctions
du programme sont : la conversion des données
expérimentales en contraintes, l'utilisation et
le contrôle des calculs de CNS, l'exploitation des fichiers
de sortie de CNS, et l'analyse des structures. La succession de ces
différentes actions constitue un cycle de calcul.
L'attribution automatique des nOe est un processus qui n'est
efficace que s'il est mené
de manière itérative. De ce fait, le
programme procède au début de chaque cycle à une
nouvelle attribution qui dépend des modèles de plus basse
énergie de l'itération précédente.
La répétition des cycles de calcul permet
d'optimiser l'attribution des pics au fil des itérations, et
conduit à une convergence des structures vers un repliement
unique. Les paragraphes suivants sont consacrés à la
description des méthodologies utilisées par le programme
pour attribuer et convertir les volumes des pics nOe en contraintes de
distance.
3.3.1) Gestion des contraintes de distance
a) Notion de contrainte de distance
ambiguë
Comme nous l'avons évoqué dans le chapitre 2.1.1,
le volume d'un pic de corrélation dipolaire Vij entre deux
protons i et j peut être relié à la
distance rij séparant les deux noyaux
via un facteur de calibration (ou de
référence) Rcal :
rij = Rcal
1
Vij
1/6
(2.21)
La distance rij constitue
alors une contrainte de distance non ambiguë. En revanche,
lorsque
plusieurs attributions sont possibles pour un
même pic, le programme d'attribution automatique considère
le volume de ce pic Vx comme la somme des contributions dipolaires
entre plusieurs paires de protons :
Vx =
n
6
1
r
Ó
x=1 x
6
Rcal
(2.22)
avec rx l'ensemble des distances entre paires d'atomes
i et j relatif au n nombre d'attributions possibles.
Selon ce principe, la contrainte de distance ambiguë r introduite
par Nilges en 1995
(Nilges, 1995) peut s'écrire de la manière suivante
:
n 1 -1/6
6
r
r = Ó
x=1 x
Rcal
(2.23)
Cette notion de contrainte ambiguë est fondamentale car elle
va permettre d'intégrer au calcul
de la structure des informations nOe dont l'attribution
est incertaine. La gestion des attributions ambiguës et non
ambiguës est un des principes sur lequel repose la stratégie du
programme d'attribution automatique.
b) Calibration des distances inter-atomiques
La conversion des volumes nOe en distance nécessite de
déterminer préalablement la valeur du facteur de calibration
Rcal relative à chaque expérience NOESY-HSQC. La
méthode classique consiste à relever le volume de plusieurs pics
référents Vref qui correspondent à la
corrélation de 2 protons géminés aliphatiques, ou
de 2 protons aromatiques appartenant au même cycle, et dont la
distance rref les séparant est connue et fixe. La valeur de
Rcal peut alors être obtenue par la relation suivante :
Rcal =
rref
1/6
Vmoy
(2.24)
avec Vmoy la moyenne des
volumes Vref. Cette méthode manuelle
et simple permet une
première estimation approximative de la valeur de
facteur de calibration utilisée lors des premières
itérations. Le programme d'attribution automatique offre la
possibilité d'obtenir une calibration plus exacte à partir
des premières structures repliées de la protéine.
La procédure consiste à comparer les distances calculées
à partir du volume nOe (rnOe) avec ces mêmes
distances mesurées dans les modèles
(robs) (2.25). Selon ce principe, une valeur de
rapport C proche de 1 indique que la valeur de facteur
de calibration Rcal utilisée est adaptée à
la quantification des volumes nOe. Dans le cas contraire, le
paramètre Rcal doit être
réajusté
selon que la valeur de C est inférieure ou
supérieure à 1. Cette méthode est avantageuse car elle
donne la possibilité d'affiner la calibration au fil des
itérations.
Óij
C =
Óij
nOe
rij
obs
rij
(2.25)
c) Les intervalles de distances permises
Comme nous l'avons évoqué dans le paragraphe
2.1.1, le rapport de proportionnalité entre le volume nOe et la
distance inter-atomique 1H repose sur l'approximation d'un
mouvement isotrope de la molécule étudiée (ôc
moyenné). Aussi, la différence de ôc
résultant
de mouvements internes de la protéine, et le
phénomène de diffusion de spin, rendent la quantification
du nOe approximative. Pour tenir compte de cette imprécision,
les distances inter-protons sont classiquement introduites dans les
protocoles de calcul CNS sous forme
d'un intervalle de distances permises :
rij - ? < rij < rij + ?
(2.26)
avec ? représentant l'erreur sur la contrainte de
distance rij. Le programme d'attribution automatique propose
à l'utilisateur 3 expressions distinctes pour calculer le
paramètre ? :
mode 1 : ? = 0.25.rij
mode 2 : ? = 0.125.(rij)2
mode 3 : ? = 0.15.rij
(2.27)
Ces expressions peuvent être utilisées pour
définir l'erreur sur les contraintes de distance entre protons de
la chaîne principale (variable ER_MODE_SQE), et sur les
contraintes d'autre nature (variable ER_MODE). Dans le cas de
l'étude du domaine protéique K2, l'incertitude ?
sur la distance rij séparant deux
protons de la chaîne principale a été estimée
à 25% de rij (ER_MODE_SQE = 1). L'amplitude
des mouvements de chaîne latérale pouvant influer de
manière significative sur l'intensité du nOe, l'erreur sur
la distance séparant un proton de chaîne latérale
avec un autre noyau est généralement plus importante et a
été estimée à 12.5%
du carré de la distance rij (ER_MODE =
2).
Par ailleurs, les intervalles de valeurs permises doivent
tenir compte de la réalité physique du nOe. Ainsi, le
programme tolère une limite maximale définie par la
variable
DRMNMAX et contrôlée par le mode
DRMNMAX_MODE. Le mode suppression conduit au
rejet d'une contrainte dont la limite maximale est
supérieure à la valeur de DRMNMAX. Pour notre part, la
valeur de DRMNMAX a été fixée à 5.3
Å, et nous avons préféré utiliser le mode
truncature qui réduit systématiquement la limite
maximale d'une contrainte à la valeur de DRMNMAX lorsque
celle-ci est supérieure à DRMNMAX.
3.3.2) Préparation des données
initiales
L'utilisation du programme d'attribution automatique du LSP
nécessite dans un premier temps de préparer un certain nombre
de données initiales contenant :
- Une table des déplacements chimiques des noyaux
1H, 15N, et 13C de la protéine pour
chaque expérience NOESY-HSQC éditée 15N, et
13C.
- Une liste des pics nOe de chaque expérience où
sont reportés le volume de chaque pic
et ses coordonnées en déplacement chimique.
- Un fichier de contraintes de distances non ambiguës
attribué manuellement et qui renseigne sur la topologie de la
protéine (fichier ss.cons).
- Un fichier de contraintes dihédrales sur les angles et
Le fichier de topologie, appelé ss.cons,
contient des informations de distance qui renseignent sur la structure
secondaire et tertiaire de la protéine. Ces éléments
obtenus après analyse des paramètres structuraux RMN indiquent
principalement, la topologie des feuillets
â déduite des nOe caractéristiques
inter-brins, et la formation de liaisons hydrogène déduites des
expériences de spectroscopie d'échange. Il est à
noter que les fichiers de contraintes dièdres et de topologie ne
sont pas utilisés de manière directe pour attribuer les pics nOe
dans
le programme d'attribution automatique. Ces
données sont converties au format CNS, et systématiquement
introduites dans les protocoles de calcul CNS à chaque itération,
et quelles que soient les attributions choisies. Par conséquent,
un certain nombre d'informations de structure tertiaire sont
dupliquées dans les fichiers de contraintes de CNS.
3.3.3) Stratégie d'attribution des
pics
a) Collecte des possibilités
d'attribution
La première phase du processus d'attribution
automatique consiste à comparer les valeurs des tables de
déplacements chimiques de la protéine aux déplacements
chimiques des
pics nOe afin d'établir la liste de toutes les
attributions possibles de chaque pic. Pour cela, un
intervalle de tolérance est défini par
l'utilisateur pour chacune des trois coordonnées des nOe.
Les gammes de tolérance classiquement testées
sont : #177; 0.02 ppm dans la dimension 1H
d'acquisition, #177; 0.25 ppm sur la fréquence de
résonance de l'hétéroatome 15N ou
13C, et
#177; 0.04 ppm dans la dimension 1H
indirecte. Les nOe pour lesquels aucune possibilité d'attribution
n'est déterminée constituent la liste des non
attribués, et sont soumis à un nouveau processus
d'attribution lors de l'itération suivante.
Par ailleurs, il est parfois constaté pour certains
protons une légère différence entre la valeur de
déplacement chimique définie dans les tables et la valeur
réelle sur les spectres NOESY-HSQC. Aussi, le programme
d'attribution automatique repère ces « décalages »
systématiques afin que l'utilisateur puisse modifier en
conséquence les tables de déplacements chimiques de la
protéine en fin d'itération.
b) Le paramètre de seuil
Une fois établie la liste de toutes les
possibilités d'attribution relatives à chaque pic, chacune des
corrélations potentielles entre 2 protons i et j est
associée à une valeur de distance
dx. Le paramètre dx correspond
à la distance moyenne qui sépare les 2 protons i et
j dans les modèles de plus basse énergie de
l'itération précédente (ou dans les structures
initiales aléatoires pour la première itération). Il
constitue la donnée principale utilisée par le programme
pour choisir le nombre d'attributions de chaque pic. La méthode
consiste à convertir chaque moyenne de distance dx,
relative à une possibilité d'attribution, en une
intensité nOe théorique moyenne
Ix selon la relation suivante :
1
Ix = 6
dx
(2.28)
Par cette définition, la contribution
Ix traduit véritablement le poids
d'une attribution par
rapport à une autre : plus la distance moyenne
dx entre 2 protons est courte, et plus la contribution
moyenne associée Ix sera importante. Ainsi, à
l'issue de cette seconde étape, chaque possibilité
d'attribution d'un pic donné est associée à un poids
différent.
A ce stade du traitement des données, le programme utilise
un paramètre de seuil p, compris entre 10-4 et 0.3,
pour déterminer le nombre d'attributions retenues pour chaque pic :
les contributions Ix sont
classées par ordre décroissant, puis chacune d'entre elles
est
comparée à la précédente en
commençant par la contribution la plus forte (systématiquement
sélectionnée). Si pour une possibilité donnée,
la valeur de son poids Ix-1 est supérieure au produit
pIx, alors l'attribution est retenue et le poids Ix-2 est
ensuite comparé au produit pIx-1
de la même manière. Dans le cas contraire, si
Ix-1 < pIx, alors
l'attribution est rejetée ainsi que
toutes les autres dont la valeur de contribution
Ix-n est inférieure à Ix-1. Le processus
de sélection du nombre d'attributions est alors terminé. Au
final, ce nombre ne peut excéder 20 attributions par pic.
Prenons l'exemple d'un pic pour lequel 5 attributions
sont possibles. Dans le cas le plus simple, considérons les
corrélations potentielles d'un proton A avec respectivement, un proton
B, C, D, E, ou F. A chacune de ces possibilités d'attribution
correspond, une distance moyenne séparant chacun des 2 protons
respectifs dans les modèles précédents (dAB,
dAC, dAD, dAE, et dAF), et une contribution
moyenne associée (IAB, IAC, IAD,
IAE, et IAF). La figure 3.13 représente l'histogramme
des contributions classées par ordre décroissant.

Ix
pIAB
pIAC
pIAD
AB AC AD AE AF
Figure 3.13 : Principe de la
sélection du nombre d'attributions d'un pic nOe en fonction du
paramètre de seuil p. Les contributions sont dans un premier
temps triées par ordre décroissant puis chacune d'elles est
comparée à la précédente. Une possibilité
d'attribution
est sélectionnée si et seulement si Ix-1 >
pIx.
Dans notre exemple, le poids IAB constitue la
contribution la plus importante et sert donc de point de départ. La
valeur de contribution IAC étant supérieure au produit
pIAB, la possibilité d'attribution AC est par
conséquent sélectionnée. La valeur de IAC
est ensuite utilisée pour définir le seuil
d'attribution suivant et conduit à la sélection de la
possibilité AD car
IAD > pIAC. Chaque contribution est ainsi
comparée à la précédente tant que la valeur de
Ix-1 est
supérieure au produit pIx. Dans notre exemple, ce
n'est plus le cas pour la possibilité AE car
IAE <
pIAD, ce qui amène à rejeter les
attributions AE et AF. Finalement, seules les attributions
AB, AC, et AD sont maintenues après application du
paramètre de seuil.
Lors des premières itérations, l'utilisation d'une
valeur faible de paramètre de seuil p
favorise les attributions multiples et par conséquent, la
constitution de contraintes ambiguës.
Au fil des calculs, l'augmentation de la valeur de
p permet de restreindre le nombre d'attributions possibles pour
chaque pic, et donc de diminuer la quantité de contraintes
ambiguës au profit des contraintes non ambiguës.
La sélection du nombre d'attributions par un
paramètre de seuil présente plusieurs atouts. Le premier
réside dans la capacité du programme à
intégrer un maximum de possibilités d'attribution lors
des premières itérations, et donc à explorer
« l'espace conformationnel des nOe ». En effet, l'ajout
d'attributions ambiguës à une contrainte de distance ne
conduit pas à une violation énergétique tant que la
véritable attribution figure dans
les différentes possibilités. La multiplication
des contraintes ambiguës ne pénalise donc pas le système
lors des premières itérations, mais elle contribue à une
dilution de l'information, et limite la convergence des modèles.
C'est pourquoi, la valeur de paramètre de seuil est
incrémentée au fil des calculs afin de réduire
progressivement le nombre d'attributions de chaque pic. Par ailleurs,
notons ici le rôle important du fichier de topologie ss.cons qui
permet d'orienter le repliement de la protéine dès les premiers
calculs et par conséquent, de guider indirectement le programme vers les
attributions les plus probables d'un point de vue de la structure 3D.
Finalement, l'augmentation progressive du paramètre de seuil
conduit à la convergence des structures
générées vers un repliement unique dont les
distances intra-
moléculaires 1H correspondent le mieux à
un jeu de données expérimentales nOe.
c) Nombre de structures contribuant à une
attribution
Comme nous venons de l'évoquer, la sélection
du nombre d'attributions par pic est obtenue à partir de l'analyse
des 10 structures de plus basse énergie de l'itération
précédente
via le paramètre dx. Aussi, la distance rij
relative à une possibilité d'attribution peut être
très variable d'un modèle à l'autre, et notamment lors des
premières itérations où la convergence des structures est
faible. C'est pourquoi, le programme utilise une valeur seuil cut-off
de 20 Å pour considérer une possibilité d'attribution
dans un modèle. Dès lors, seules les possibilités
qui répondent à la condition rij
< 20 Å dans un nombre suffisant de structures sont
sélectionnées. On parle ainsi de nombre de
structures contribuant à une attribution. La valeur
de ce paramètre est progressivement augmentée au
fil des calculs (de 4 à 6), ce qui contribue à
la convergence des attributions, et donc des structures au fil
des itérations.
d) Gestion des pics intra-résiduels
Deux modes sont proposés par le programme d'attribution
automatique pour traiter les nOe relatifs aux corrélations
intra-résiduelles. En mode intra, chaque possibilité de
type intra- résiduel est associée à un
paramètre dx de 1.0 Å, ce qui favorise
considérablement ce type d'attribution par rapport aux autres
possibilités via une valeur forte de Ix. En mode
non-intra,
le paramètre dx est
utilisé comme défini dans le paragraphe précédent,
et quel que soit le type
de corrélation. Le mode intra,
utilisé pour la plupart des itérations, permet de limiter
le nombre de contraintes ambiguës au profit d'attributions plus
évidentes, ce qui conduit à une convergence plus rapide des
structures. Ce mode favorise toutefois à outrance les attributions
intra-résiduelles. Aussi, le mode non-intra est utilisé
lors des 3 dernières itérations, lorsque la protéine est
repliée, afin de tenir compte du repliement de la protéine de
manière plus réaliste pour réaliser l'attribution
automatique des pics.
e) Gestion des pics symétriques
Sur les spectres NOESY-HSQC éditée
13C, la présence d'un pic de corrélation entre 2
protons aliphatiques i et j sur un plan
13Ci peut être accompagnée d'un pic
réciproque ou symétrique sur le plan 13Cj
lorsque la résolution spectrale le permet. Dans le cas d'une
corrélation de type inter-résiduelle, l'identification de 2
pics réciproques est un argument supplémentaire qui
conforte leur attribution. Aussi, lorsque 2 pics symétriques sont
repérés
par le programme, l'attribution de type «
réciproque » est favorisée par rapport aux autres
possibilités en recevant une valeur de paramètre dx de
1.5 Å. Après application du paramètre
de seuil, si ces 2 pics sont dotés de la même
attribution, le programme supprime alors une des
2 informations et convertit la distance moyenne en contrainte.
3.3.4) Les filtres d'attribution
Une fois établie la liste des attributions
sélectionnées, le programme met à disposition
de l'utilisateur un certain nombre de filtres
d'attribution dont l'objectif est d'améliorer la qualité
des attributions, et donc des structures générées,
en favorisant les conformations
vraisemblables. L'application de ces différents
filtres conduit à une diminution du nombre
d'attributions et constitue la dernière étape avant
la conversion en contraintes de distance au format CNS.
a) Le filtre dRMN trop grande
Malgré la notion d'intervalle de distances
permises introduite pour tenir compte de l'incertitude de la
quantification du nOe, il est possible que les distances
associées aux volumes de certains pics soient démesurées
par rapports aux distances correspondantes dans
les structures calculées. Ceci est notamment
observé, pour des effets à longue distance, lors des
premières itérations où la structure n'est encore
que partiellement repliée. Aussi, l'intégration de ce type de
contraintes peut conduire à des violations et pénalités
énergétiques importantes. Le filtre dRMN trop grande
repère et supprime ces pics trop intenses avant leur conversion en
contraintes. Lors des itérations finales, l'application de ce
filtre ne doit conduire qu'à rejeter un très faible nombre de
pics pour lesquels la distance déduite du nOe apparaît trop courte
en raison de problèmes d'homogénéité de la
calibration ou de dynamique intra-moléculaire inhérents à
la technique RMN.
b) Le filtre DIAG
Le filtre DIAG (pour diagonal plot) est
utilisé pour éviter le calcul de conformations
qui correspondent à un repliement inexact ou
incohérent d'une partie de la protéine. Il recherche et
supprime les attributions longue distance dites « isolées ».
En pratique, lorsque l'attribution d'un nOe est associée à
la corrélation entre 2 protons appartenant à 2
résidus éloignés dans la séquence primaire, si
aucun autre pic ne correspond à un contact longue distance
entre protons appartenant aux mêmes résidus ou à
des résidus voisins, alors l'attribution est considérée
comme suspecte. Le pic est alors filtré mais l'attribution n'est pas
définitivement supprimée. Au fil des itérations, le
pic peut être à nouveau considéré si
l'évolution du repliement fait apparaître d'autres nOe
longue distance qui impliquent les mêmes régions de la
protéine.
c) Le filtre SUPRES
L'option SUPRES permet d'utiliser les informations
de flexibilité des résidus non structurés des
extrémités N- et C-terminales pour diminuer le nombre
d'attributions. En effet,
ces résidus en échange conformationnel très
rapide ne peuvent donner naissance à des nOe
longue distance sur les spectres de type NOESY
où l'information est moyennée sur l'ensemble de l'espace
conformationnel. Par conséquent, à partir d'une liste de
ces résidus fournie par l'analyse du nOe
hétéronucléaire 1H-15N (cf.
§ 2.2.4), l'option SUPRES rejette systématiquement
chaque attribution de type longue distance qui corrèle un proton de
résidu quelconque avec un autre appartenant à un résidu de
cette liste.
d) Le filtre SUPRES_LAT
Selon le même principe, cette option supprime
l'ensemble des attributions inter- résiduelles qui associent un
proton de résidu quelconque avec un proton de chaîne
latérale d'un résidu de la liste SUPRES_LAT. Ainsi, les
filtres SUPRES et SUPRES_LAT permettent
de tenir compte de données dynamiques obtenues par
RMN, et par conséquent, d'éviter le calcul de structures
incohérentes avec ces paramètres.
e) Le filtre SUP2_ini
Le fichier SUP2_ini est une liste arbitraire
qui définit l'ensemble des attributions interdites pour chaque pic
donné. Cette option offre à l'utilisateur la possibilité
de contrôler partiellement l'attribution de certains pics nOe. Aussi, les
filtres d'attribution ne sont utilisés qu'après application du
paramètre de seuil p. Par conséquent, le filtre
SUP2_ini ne permet pas d'influencer de manière directe
l'attribution des nOe, mais conduit au rejet des pics dont une certaine
attribution n'est pas souhaitée.
3.3.5) Gestion des violations
Les violations de distance systématiques
retrouvées à chaque itération sont souvent dues
à des erreurs d'attribution de résonance de la
protéine, ou à l'intégration de pics qui
correspondent en réalité à du bruit spectral. Aussi, les
contraintes qui engendrent ce type de violations supportent une information
erronée, qui peut ralentir la convergence du repliement
au fil des itérations, ou conduire à des
conformations inexactes. Pour pallier ce problème, le programme
d'attribution exploite les fichiers de sortie du logiciel CNS à
la fin de chaque cycle, et établit la liste SUP2 des
violations de distance supérieures à 0.5 Å communes
à au moins 4 des 10 meilleures structures. Chaque attribution
qui figure sur cette liste est alors
supprimée lors de l'itération suivante
après application du paramètre de seuil. Notons par
ailleurs que la composition du fichier SUP2 est
différente à chaque itération : aucune
attribution n'est supprimée définitivement et c'est
à l'utilisateur que revient cette tâche.
3.3.6) Analyse et validation des structures
Les ressources informatiques du Laboratoire de Structure des
Protéines permettent le calcul d'un grand nombre de structures. Une
sélection s'impose donc pour ne conserver que
les meilleures, du point de vue de la
géométrie covalente et du respect des contraintes
expérimentales. Plusieurs critères sont classiquement
utilisés pour évaluer leur qualité :
a) Le logiciel PROCHECK
Les valeurs des angles dièdres et des modèles
générés doivent occuper les zones permises du diagramme
de Ramachandran qui définit les régions favorables de
l'espace conformationnel (Ramachandran et al., 1971). Le logiciel
PROCHECK (Laskowski et al.,
1996) permet d'observer la répartition de ces couples
d'angles, et facilite l'identification des structures secondaires. On
considère généralement que les structures finales
sont de bonne qualité lorsque moins de 1% des valeurs d'angles et
occupent les régions interdites.
b) Les termes énergétiques
La fonction d'énergie est un paramètre
fondamental pour valider les structures calculées. Les valeurs des
différents termes énergétiques reflètent la
qualité de la géométrie covalente, et renseignent sur
le degré de prise en compte des contraintes
expérimentales. Aussi, l'objectif de la répétition
des processus itératifs n'est autre que de générer
des structures dont l'énergie associée est la plus basse
possible. Selon ce principe, les structures finales doivent
présenter une bonne géométrie covalente, des
violations de distance inférieures à 0.5 Å, et des
violations d'angles dièdres inférieures à 10°, pour
être considérées comme étant de bonne
qualité.
3.3.7) Description des cycles
itératifs
La phase préliminaire du processus itératif
consiste à générer 10 structures aléatoires
à partir des fichiers de paramètres CNS qui définissent le
champ de force et la géométrie de la protéine.
L'attribution automatique de la première itération est
réalisée à partir de ces structures aléatoires,
et des différentes listes de déplacements chimiques et de pics
nOe. Le nombre de structures contribuant à une attribution doit
être supérieur à 4, et la valeur de p est
fixée à 0.0001. Les facteurs de calibration sont dans un
premier temps estimés à partir du volume de pics
référents sur les différents spectres NOESY-HSQC.
Après application des filtres d'attribution, le premier jeu de
distances inter-atomiques 1H est constitué. Les
contraintes dièdres et de distance (dont les données de
topologie) sont converties au format CNS, puis intégrées aux
protocoles de dynamique moléculaire. Lors de la première
itération,
un premier jeu de 100 structures est
généré par le logiciel CNS. Le programme d'attribution
prend alors en charge le traitement de ces 100 modèles. Les
20 structures de plus basse énergie sont
sélectionnées, triées par ordre croissant
d'énergie, puis les 10 premières font l'objet d'analyses
systématiques : bilan des énergies moyennes pour chaque terme du
champ
de force, analyse des angles et par le logiciel
PROCHECK, et calcul des écarts quadratiques moyens sur le
squelette. Le programme permet également d'obtenir une
synthèse des violations de distance systématiques sur ces 10
meilleurs modèles à partir des fichiers de sortie du logiciel
CNS.
Après traitement des résultats de la
première itération, le programme entre dans un processus
itératif de 22 cycles où se succèdent l'attribution des
nOe, le calcul des modèles, et l'analyse des 10 meilleures structures
(Figure 3.14). A l'issue de chaque cycle, les 10 modèles
de plus basse énergie, ainsi que la liste de
leurs violations, sont utilisés pour réaliser l'attribution
de l'itération suivante. Ces 10 meilleurs modèles
constituent également les structures de départ des
protocoles de dynamique moléculaire. Au fil des
itérations, les valeurs de paramètre de seuil et de
nombre de structures contribuant à une attribution sont
progressivement augmentées (Figure 3.15). Il en est de même pour
le nombre de structures
calculées qui est fixé à 200 à partir
du treizième cycle, puis à 300 à la vingtième
itération.
Structures 3D aléatoires

Données initiales
· Listes des pics nOe (déplacements chimiques +
volume)
· Tables de déplacements chimiques 1H,
15N, et 13C
· Fichier de topologie ss.cons
· Fichier Talos de prédiction des angles et

début du cycle itératif
Attribution des pics
· Collecte des possibilités d'attribution :
- intervalles de tolérance
- nombre de structures contribuant à une attribution
· Mesure des distances moyennes dx
sur les modèles de l'itération
n-1
· Calcul des contributions associées Ix
· Application du paramètre de seuil p
fin du cycle itératif :
utilisation des structures 3D des
10 meilleurs modèles
Analyse des 10
meilleures structures

Filtres d'attribution
· dRMN trop grande
· DIAG
· SUPRES, SUPRES_LAT
· SUP2_ini
· SUP2 (violations n-1)

création des contraintes
· PROCHECK ( et )
· Bilan des énergies
· RMSD sur le squelette
· Violations systématiques
sélection des 20 structures de plus basse
énergie

Calcul des structures sous CNS
· Dynamique à haute température sous
contraintes RMN (recuit simulé)
· Création des fichiers de sortie (énergies,
violations...)
Figure 3.14 : Description d'un
cycle itératif du programme d'attribution automatique des
nOe développé au Laboratoire de Structure des
Protéines du CEA de Saclay (Savarin et al.,
2001).
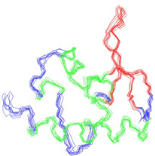
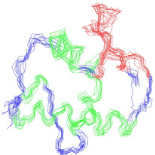
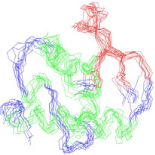
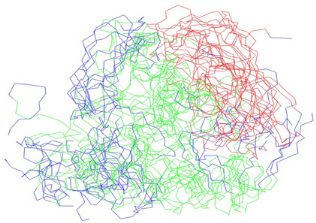
Chapitre 3 : Stratégie d'étude par RMN et
Modélisation Moléculaire du domaine K2
179
p 10-4
Na 4
Ns
10-3
5
3.10-3
100
5.10-3
10-2
0.1
6
0.2
200
0.3
300
It
1 4 7
179
10 13 16 19 22
Figure 3.15 : Evolution de
paramètres du programme d'attribution au fil des 22 cycles (It) du
processus itératif. Les différentes annotations
correspondent au nombre de structures calculées (Ns),
au nombre de structures contribuant à une attribution (Na), et au
paramètre de seuil (p)
Chapitre 3 : Stratégie d'étude par RMN et
Modélisation Moléculaire du domaine K2
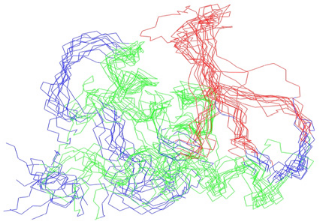
Finalement, les 20 meilleures structures du dernier cycle sont
soumises à une étape de
raffinement dans le champ de force CHARMM22 en solvant
implicite. Les structures convergentes obtenues lors des dernières
itérations sont utilisées pour réajuster les valeurs de
facteur de calibration via le calcul du rapport C. Ces valeurs pourront
être appliquées lors des processus itératifs suivants, puis
à nouveau optimisées en recalculant le rapport C.
Comme nous l'avons évoqué
précédemment, les violations sont dans la plupart des cas dues
à des erreurs d'attribution de résonance de la protéine,
ou à des inexactitudes inhérentes
à la spectroscopie de RMN (recouvrement
spectral, dégénérescence du signal, bruit,
dynamique interne...). L'analyse manuelle des violations en fin de chaque
processus itératif
de 22 cycles est par conséquent nécessaire
afin d'identifier les informations erronées en contradiction avec
le repliement de la protéine. La correction progressive du jeu de
données expérimentales est alors concomitante avec la
baisse des énergies, la convergence des
structures, le respect du diagramme de Ramachandran, et
l'élimination des violations.
Chapitre 4 : Caractérisation structurale du
domaine K2 par RMN et Modélisation Moléculaire
| 


