VII.2.2 - Préparation des cellules
bactériennes par effet thermique
La PCR permettant d'amplifier à partir de petits
fragments le gène ciblé sur le génome, on a
effectué un choc thermique sur les bactéries afin d'avoir
accès à quelques fragments d'ADNr 16S. Pour chaque
bactérie, ce choc thermique a eu lieu sur 1 ml de sa suspension de 3 ml
préalablement cultivée pendant 2 jours dans un
incubateur-agitateur à 30°C. Les différentes
étapes de ce choc thermique sont les suivantes :
- passage sur glace de chaque prélèvement
bactérien (1 ml) contenu dans un tube de 1,5 ml ;
- centrifugation à 12000 t/min pendant 10 min de toutes
les souches prélevées;
- recueil du culot bactérien pour chaque
souche centrifugé;
- reprise du culot dans 100 ul d'eau distillée
stérile et homogénéisation au vortex;
- passage sur glace des différentes souches ;
- chauffage des souches au bain-marie à 100°C
pendant 10 min ;
- enfin, retour sur glace jusqu'à refroidissement total
des souches.
Chaque bactérie ainsi préparée est
prête pour la réalisation de la PCR-RFLP sur l'ADNr 16S.
VII.2.3 - Amplification et digestion de la région
16S de l'ADNr des rhizobia par PCR-RFLP
VII.2.3.1 - Principe de la PCR-RFLP et choix du
gène de l'ADNr 16S
VII.2.3.1.1 - Principe de la
PCR-RFLP
L'approche PCR-RFLP conduit à comparer la longueur des
fragments de restriction d'une région choisie du génome et
préalablement amplifiée par PCR, afin de déterminer le
polymorphisme. Cette région est utilisée comme substrat pour les
enzymes de restriction. Les enzymes de restriction sont des
endonucléases qui reconnaissent spécifiquement une
séquence courte (4 à 8 bases) et coupent la chaîne d'ADN
chaque fois qu'elles reconnaissent cette séquence
élémentaire. L'ADN se retrouve ainsi fragmenté en morceaux
de différentes longueurs séparés en fonction de leur
taille par électrophorèse sur un support physique. Un fragment va
migrer d'autant plus loin qu'il est court. Un polymorphisme de la longueur des
fragments de restriction est ainsi mis en évidence. Les profils
observés permettent l'analyse de la diversité des souches
par :
- la caractérisation de chaque isolat ;
- l'estimation des divergences de séquences entre
isolats et l'établissement de leur proximité
phylogénétique (NDOYE, 1998).
VII.2.3.1.2 - Choix du gène de l'ADNr
16S
Les gènes ribosomiques sont utilisés comme
marqueurs moléculaires dans l'étude phylogénétique
de différents organismes en raison de leur universalité, leur
abondance, leur taille convenable aux analyses comparatives (LUDWIG et
SCHLEIFER, 1999). Les ADNr contiennent par ailleurs des régions de
séquences hautement conservées, très utiles pour la
désignation des amorces (HILLIS et DIXON, 1991 ; STAHL et AMMAN, 1991)
et d'autres régions de séquences suffisamment variables pour
servir comme un excellent moyen taxonomique (GRIMONT et GRIMONT, 1986).
Chez les procaryotes particulièrement, les gènes
codant pour les ARN ribosomaux sont organisés en opérons qui
contiennent également des espaces intra-géniques ainsi que
d'autres gènes codant pour les ARN de transfert (ARNt) (figure
13).
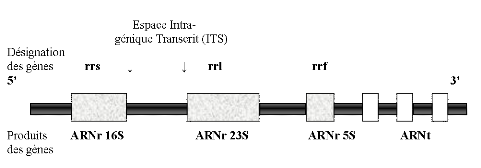
Figure 13 : Organisation de
l'opéron de l'ADNr observé chez les procaryotes
(D'après EL HILALI, 2006)
Comme le montre la figure 13, il existe
trois types d'ADN ribosomique : le 5S, le 16S et le 23S (JENSEN et STRAUS,
1993). L'ADNr 5S est très peu utilisé dans les études de
diversité vu sa petite taille d'environ 120 nucléotides.
Contrairement à ce dernier, l'ADNr 16S codant pour la petite sous
unité ribosomique d'environ 1500 pb (opéron rrs : ribosomal
RNA small subunit) et l'ADNr 23S codant pour la grande sous unité
d'environ 2500 à 3000 pb (opéron rrl : ribosomal RNA large
subunit) (GÜRTLER et STANISICH, 1996) sont très
utilisés. L'espace entre ces deux opérons est transcriptionnel
d'où la désignation ITS (Internal Transcribed Spacer)
(NORMAND et al., 1996). Il est aussi utilisé dans ces
études. Toutefois, depuis que WOESE (1987) a montré que le
gène de l'ADNr 16S est présent chez toutes les bactéries,
qu'il a la même fonction et que sa structure est conservée,
plusieurs chercheurs l'ont préférentiellement utilisé
comme une approche rapide pour évaluer la variabilité
génétique entre les souches de rhizobia (LAGUERRE et
al., 1994 ; NOUR et al., 1994b).
| 


