VII.2.3.2- Réalisation de la PCR-RFLP
VII.2.3.2.1 - Principe de la PCR
La PCR (SAIKI et al., 1985 ;
MULLIS et FALOONA, 1987) se réalise dans un mélange
réactionnel contenant de faibles quantités d'ADN possédant
la séquence à amplifier et utilisées comme matrice, des
paires d'amorces nucléotidiques complémentaires des
séquences qui encadrent la cible à amplifier et un ensemble de
quatre dNTP (dATP, dTTP, dCTP et dGTP) nécessaires à la
synthèse de nouveaux brins d'ADN. A ce mélange, s'ajoute l'enzyme
de polymérisation, la Taq DNA polymérase. C'est une enzyme
thermorésistante qui fut isolée à l'origine de Thermus
aquaticus, une bactérie thermophile.
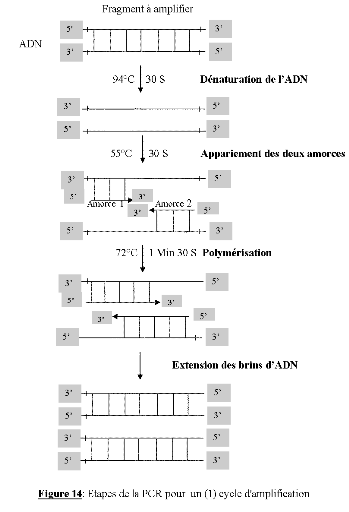
Classiquement, la PCR consiste en la répétition
de trois étapes thermiques réalisées successivement et
connu sous le nom d'étapes d'un cycle d'amplification. Chaque
étape est caractérisée par un temps et une
température de réalisation qui déterminent
l'efficacité de toute la PCR. Dans notre cas, le cycle d'amplification
se présente comme suit :
· Première
étape : le double brin d'ADN est d'abord
chauffé à 94°C pendant 30 secondes ; ce qui permet de
rompre les liaisons entre les deux brins. C'est l'étape de
dénaturation ;
· Deuxième
étape : la température est abaissée
à 55°C pendant 30 secondes. Au cours de cette étape les
amorces s'apparient spécifiquement sur leurs séquences
complémentaires aux bornes de la région à amplifier,
chacune sur son brin. C'est l'étape d'amorçage ou
d'hybridation, annealing. La spécificité de la PCR
repose sur la qualité de cet appariement lié à la taille
des amorces et à leurs séquences ;
· Troisième
étape : la température est
élevée à 72°C pendant 1 minute 30 secondes. A cette
température, la polymérase allonge les nouveaux brins d'ADN
à partir des amorces en assemblant des dNTP. On parle d'étape de
polymérisation, d'extension des amorces ou d'élongation
des brins d'ADN (Figure 14).
À la fin de chaque cycle d'amplification, la
séquence définie par les amorces initiales est dupliquée.
Les brins d'ADN néoformés ainsi que les brins initiaux servent de
matrice dans les cycles successifs d'amplification. C'est la
répétition des cycles de
dénaturation-hybridation-élongation qui conduit finalement
à une accumulation exponentielle de l'ADN, c'est-à-dire à
la réalisation de la PCR. La figure 14
ci-après présente le cycle d'amplification de la PCR que
nous avons réalisée au LCB du CNRA à
Adiopodoumé.
VII.2.3.2.2 - Choix des amorces et milieu
réactionnel de la PCR
Le tableau 4 présente la liste et les
caractéristiques de la paire d'amorces utilisées.
Tableau IV :
Amorces choisies pour l'amplification des fragments ADNr
16S
|
Amorces
|
Séquences 5'3'
|
Sites
|
Références
|
|
FGPS 6
|
GGAGAGTTAAGATCTTGGCTCA
|
ADNr 16S
|
NORMAND et al., 1992
|
|
FGPS1509
|
AAGGAGGGGATCCAGCCGGA
|
ADNr 16S
|
NORMAND et al., 1992
|
Ces deux amorces utilisées sont spécifiques de
l'ADNr 16S des rhizobia. Elles furent mises au point en 1992 (NORMAND
et al., 1992) et utilisées dans plusieurs études de
diversité des rhizobia basée sur cette région (NDOYE,
1998 ; SY et al., 2001 etc.).
Avec ces amorces et les autres réactifs, le milieu
réactionnel de la PCR se présente comme suit (Tableau
5) :
Tableau V : Composition du milieu
réactionnel unitaire commun de la PCR
|
Concentration de travail
|
Volume utilisé pour un échantillon
|
|
Dntp
|
2 ,5 Mm
|
2 ul
|
|
Amorce 1 (FGPS 6)
|
2 uM
|
2,5 ul
|
|
Amorce 2 (FGPS 1509)
|
2 uM
|
2,5 ul
|
|
Tampon X 10
|
1 X
|
2,5 ul
|
|
Taq DNA polymerase
|
-
|
(0,5/15) ul
|
|
Eau PCR
|
-
|
13 ul
|
|
Volume réactionnel partiel
|
22,5 ul
|
Pour réaliser la PCR, chaque tube PCR
a comporté en plus de ce milieu réactionnel commun, 2,5 à
4 ul d'ADN d'une et unique souche bactérienne. Ainsi, le volume
réactionnel total par souche a été de 25 ul, le volume
d'eau PCR ayant été ajusté en conséquence dans
chaque cas. Au total, il y a eu 169 tubes PCR à 25 ul de réactif
pour autant de souches à amplifier. Pour chaque réaction
d'amplification, un microtube contenant uniquement le milieu réactionnel
commun a été utilisé comme témoin
négatif.
VII.2.3.2.3 - Programme de la PCR
Au cours de notre PCR, le cycle
d'amplification à 3 étapes a été
répété 35 fois. Celui-ci définit avec 2 autres
cycles (une phase de préchauffage à 94 °C pendant 5 minutes
et une phase terminale de réparation des bouts des fragments à
72°C pendant 7 min), le programme total de la PCR qui dure 2 heures 7
minutes. Ce programme se présente comme suit :
Etape 1 : 94°C-------- 5 minutes
Etape 2 : 94°C-------- 30
secondes
Etape 3 : 55°C-------- 30 secondes
35 cycles
Etape 4 : 72°C-------- 1 minute 30
seconde
Etape 5 : 72°C-------- 7 minutes
A la fin de la PCR, les amplifiats peuvent
être conservés dans le thermocycleur à une
température de 4°C. Cette phase de conservation correspond à
une étape dite de pause. Dans le cas contraire, vers la fin du temps
nécessaire à la PCR (2 h 7 min), un gel d'agarose à 0,8%
est préparé pour réaliser l'électrophorèse
des gènes une fois l'amplification terminée.
VII.2.3.2.4 - Electrophorèse sur gel d'agarose
et visualisation des gels
· Préparation du gel d'agarose
Le gel d'agarose permet de suivre la
migration des fragments d'ADN en fonction de leur taille et en milieu non
dénaturant. Pour le préparer, 0,8g d'agarose est pesé et
mélangé à 100 ml de tampon TBE (Tris Borate EDTA) 0,5X.
L'ensemble est fondu au four micro-ondes et refroidi partiellement
jusqu'à environ 60°C. Le gel ainsi obtenu est coloré au BET
(Bromure d'Ethydium) et coulé dans un porte gel où des peignes
sont préalablement placés pour créer les puits de
migration. Après solidification complète du gel, les peignes sont
enlevés et ce dernier est ainsi prêt pour
l'électrophorèse.
· Dépôt des amplifiats
Le gel ainsi solidifié est
placé à l'intérieur d'une cuve
d'électrophorèse et recouvert de tampon TBE 0,5X. Dans chaque
puits, est déposé, cinq à huit microlitres (ul) d'ADN
(amplifiats) préalablement mélangés avec deux microlitres
de tampon de charge (bleu de bromophénol). Parallèlement, 3 ul
d'un marqueur de poids moléculaire 1Kb (100 pb à 12000 bp) est
déposé sur le gel, entre 1 et 2 puits, pour vérifier la
taille attendue de l'ADNr 16S amplifié. Enfin, l'allumage de l'appareil
d'électrophorèse de type Mupid-One permet de mettre en migration
pendant 30 minutes et à 100 V, les amplifiats déposés sur
le gel.
VII.2.3.2.5 - Migration de l'ADN et
visualisation
Après le temps de migration
fixé, le gel est retiré de la cuve
d'électrophorèse, puis déposé sur une plaque de
lumière ultraviolette pour la visualisation. Sur cette plaque, la
visualisation des amplifiats est rendue possible grâce au BET. En effet,
celui-ci, en tant que cation, s'intercale entre les brins de l'ADN et rend
ainsi le complexe ion éthidium-ADN fluoresçant sous
l'éclairage UV. La prise d'images est effectuée à l'aide
du transilluminateur à caméra relié à un
ordinateur.
Après cette visualisation et les prises d'images, les
souches dont le gène ADNr 16S a été correctement
amplifié sont sélectionnées en vue de leur
digestion.
VII.2.3.2.6 - Digestion des amplifiats
La digestion enzymatique du produit de la
PCR a été réalisée sur les souches bien
amplifiées. L'enzyme de restriction utilisée est la Tsp
509I (R0576S). Ses caractéristiques sont données dans le
tableau 6 ci après.
Tableau VI : Caractéristiques de
l'enzyme de restriction Tsp 509I
|
Enzyme de restriction
|
Sites à 4 paires de bases
|
|
Tsp 509I (R0576S)
|
5'...A A T T...3'
3'...T T A A...5'
|
Cette enzyme a été déjà
utilisée pour digérer des amplifiats du gène ADNr 16S dans
des études de diversité.
Le milieu réactionnel de la digestion se
présente comme suit (Tableau 7) :
Tableau VII : Composition du milieu
réactionnel unitaire commun de la digestion
|
Concentration de travail
|
Volume utilisé pour un échantillon
|
|
Tampon X 10
|
1 X
|
1,5 ul
|
|
Enzyme de digestion Tsp 509I
|
-
|
(2/10) ul
|
|
Eau PCR
|
-
|
3,5 ul
|
|
Volume réactionnel partiel
|
5 ul
|
Pour réaliser la digestion, chaque tube a
comporté en plus de ce milieu réactionnel commun, 10 à 15
ul d'amplifiat d'une et unique souche bactérienne. Ainsi, le volume
réactionnel total par souche varie entre 15 et 20 ul.
La digestion a eu lieu pendant 3 à 4 heures dans un
bain marie chauffé à 65°C. Un témoin négatif
constitué uniquement de 15 ul d'un bon amplifiat a accompagné
chaque digestion.
À la fin de la digestion, la migration et la
visualisation des fragments de restriction obtenus ont été
effectuées dans les mêmes conditions que celles des produits PCR.
Cependant, la petite taille des fragments de restriction
générés a nécessité la préparation
d'un gel d'agarose plus résolutif (agarose à 3%) et un temps de
migration de 50 min. Aussi, note-on qu'ici, tout le produit de digestion
de chaque souche (10 à 15 ul) a été entièrement mis
en migration contrairement aux amplifiats.
CHAPITRE VIII : RESULTATS ET DISCUSSION
| 


