Annexes
Annexes
Annexe 1 : Les Protéines Phage T4 dans la base
de données (Miller et al., 2003).
|
Protéines
|
Description
|
Banque de données
|
|
AsiA
|
Anti- regulatory protein
|
1JR5, 1KA3
|
|
f3-gt
|
f3 Glycosyltransférase
|
1QKJ
|
|
DenV
|
Pyrimidine-dimer excisionase
|
2END, 1VAS
|
|
gpe
|
Lysozyme
|
1LYD
|
|
I-TevI
|
Intron-homing endonuclease
|
1I3J
|
|
MotA
|
Transcription regulatory factor
|
1BJA, 1I1S
|
|
NrdC
|
Glutaredoxin, thioredoxin
|
1ABA, 1DE1
|
|
NrdD
|
Anaerobic NTP reductase, large chain
|
1H77
|
|
RegA
|
Translation regulatory protein
|
1REG
|
|
Rnh
|
RNase H
|
1TFR
|
|
TS
|
Thymidylate synthase
|
1TIS
|
|
Wac
|
Fibritin deletions E and M
|
1AAO, 1AVY
|
|
gp1
|
dNMP kinase
|
1DEK
|
|
gp5/27
|
Tail-associated lysozyme
|
1K28
|
|
gp9
|
Long-tail fiber connector
|
1QEX
|
|
gp11
|
Baseplate-short-fiber connector
|
1EL6
|
|
gp12
|
Short tail fiber
|
1H6W
|
|
gp31
|
Cochaperone
|
1G31
|
|
gp32
|
ssDNA-binding protein
|
1GPC
|
|
gp42
|
dCMP-hydroxymethylase
|
1B5D
|
|
gp43
|
T4 DNA polymerase fragment, RB69,
DNA polymerase
|
1NOY,
1WAF
|
|
gp45
|
Processivity clamp
|
1CZD
|
|
gp49
|
EndoVII
|
1E7D
|
|
gp59
|
Helicase assembly protein
|
1C1K
|
|
nrdD intron
|
Group IA intron RNA/ribozyme
|
1SUN
|
Annexes
Annexe 2 : Electrophorèse en champ
pulsé (PFGE) de bactériophage digéré par l'enzyme
de restriction XbaI.

Puits 1 : marqueur (PFGE low range DNA
approximately 0.13-194 kb), Puits 2 : p T4b Puits 3 :
p T4c, Puits 4 : p A5a, Puits 5 : p
A5a, Puits 6 : p Ebrios, Puits 7 : p Ebrios,
Puits 8 : marqueur (PFGE low range DNA Marker in agarose
plugs), Puits 9 : p M3, Puits 10 : p M3,
Puits 11 : p M7, Puits 12 : p M7, Puits
13 : p M11, Puits 14 : p M11, Puits
15 : marqueur (PFGE low range DNA Marker in agarose plugs).
Annexe 3: Electrophorèse en champ
pulsé (PFGE) de bactériophage digéré par l'enzyme
de restriction XhoI.

Puits 1 : marqueur (PFGE low range DNA Marker
in agarose plugs), Puits 2 : p T4c, Puits 3 :
p T4c, Puits 4 : p T4b, Puits 5 : p
T4b, Puits 6 : p A5a, Puits 7 : p A5a, Puits
8 : p M3, Puits 9 : p M3, Puits 10 :
marqueur de poids moléculaire GeneRuler 1 kb DNA Ladder, Puits
11 : p M7, Puits 12 : p M7, Puits 13
: p M11, Puits 14 : p M11, Puits 15 :
p Ebrios.
Annexes
Annexe 4 : Profil de digestion des phages par les
enzymes de restrictions XbaI et XmnI
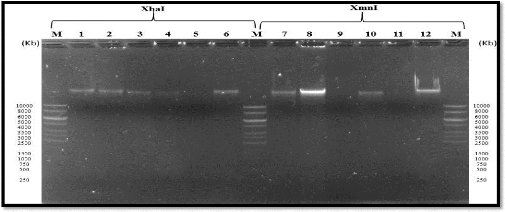
M :Marqueur de poids moléculaire 1 kb;
Puits 1: p M7 ; Puits 2 : p
M3 ; Puits 3 : p M11 ; Puits 4 : p Ebrios ;
Puits 5 : p T4 ; Puits 6 : p A5a ; Puits
7 : p M7 ; Puits 8 : p M3 ; Puits 9
: p M11 ; Puits 10 : p Ebrios ; Puits 11
: p T4 ; Puits 12 : p A5a.
RÉSUMÉ
Les bactériophages ou phages sont les virus des
bactéries et ont la capacité de lyser celles-ci. Cette action
lytique fait des phages une alternative thérapeutique appelée
phagothérapie face à l'antibiothérapie. L'utilisation
abusive de l'antibiothérapie cause aujourd'hui une recrudescence
mondiale de la résistance bactérienne aux anti-infectieux
disponibles. En Côte d'Ivoire, les bactéries
multirésistantes sont rapportées dans les études cliniques
et environnementales. La phagothérapie méconnue du corps
médical professionnel et des populations. Des études
récentes ont rapporté la présence de phages à
potentiel thérapeutique mais aucune application n'est rapportée.
La mise au point de la phagothérapie nécessite une
caractérisation morphologique et génomique complète des
phages à potentiel lytique. L'objectif de déterminer le profil
génomique de bactériophages isolés en Côte d'Ivoire.
Spécifiquement d'analyser leur génome par la technique de
l'électrophorèse en champ pulsé et par la digestion
enzymatique. 5 phages dont 2 phages lagunaires de la Lagune Ebrié (p
A5a, Ebrios) et 3 phages (M3, p M7, p M11) isolés des
micromammifères ont été inclus dans cette étude.
Les enzymes XbaI, XmnI et XhoI ont été utilisés pour la
digestion des génomes. Les résultats obtenus montrent que les
phages sont des phages à ADN double brin. L'analyse en champ
pulsé montrent que les phages p A5a, p M3, p M7, p M11 ont une taille de
génome est supérieure à 164 Kpb. La diversité
génomique des phages est démontrée par les profils de
restriction de l'électrophorèse en champ pulsé et de la
digestion enzymatique. Cette étude montre que
l'électrophorèse en champ pulsé (PFGE) est une
méthode plus sensible que la digestion enzymatique pour la
caractérisation génomique. Par contre la digestion enzymatique
des phages est simple et rapide et pour déterminer le type de
matériel génétique des phages.
Mots clés : Bactériophages,
Diversité génétique, champ pulsé, Digestion
enzymatique, Cote d'Ivoire.
| 


